概要
ものごとの関係性を裏付けたり、予測をしたい場合、**「回帰」**による分析が便利です。
機械学習アルゴリズム一覧

上図のように、「回帰」は機械学習の中でも一大分野を占める重要な手法ですが、
なぜその結果となったかを裏付ける**「解釈」が難しい**分野でもあります。
そこで今回はPythonのグラフ描画ライブラリ「seaborn」をベースにして、
回帰分析の解釈性を飛躍的に向上させる可視化ツールを作成しました!
機能1. 相関分析
機能2. 予測値vs実測値プロット
機能3. 1次元説明変数回帰モデルの可視化
機能4. 2~4次元説明変数回帰モデルのヒートマップ可視化
2021/7 修正:pipでインストールできるよう改良しました
下記コマンドでインストール可能となりました
2021/10 修正:新機能「average_plot」追加
各変数ごとに、他の変数を平均値(または中央値)で固定して予測値をプロットするaverage_plot()メソッドを追加しました。
詳しくはGitHubの説明を参照ください
$ pip install seaborn-analyzer
※関連ツールを1つの記事にまとめました!よければこちらもご覧ください
もしこのツールを良いと思われたら、GitHubにStar頂けるとありがたいです!
ユースケース
業務での回帰分析において、本ツールが有効な場面でのユースケースを作成しました
ユースケース1:2つの値の相関関係
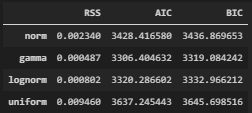
上司「アヤメの花弁(petal_length)とがく(sepal_length)の長さの関係を分析してくれ」
このような2つの値の関係性を見たいとき、よく使用するグラフが散布図です。
import seaborn as sns
iris = sns.load_dataset("iris")
sns.scatterplot(x='petal_length', y='sepal_length', data=iris)
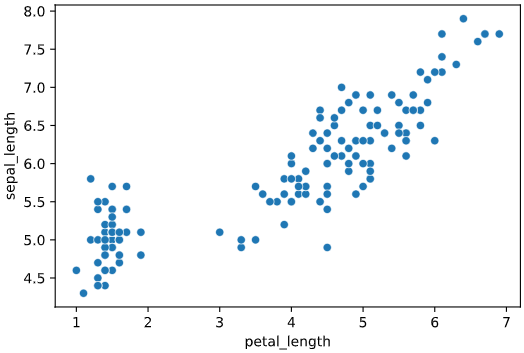
私「散布図で右下から左上にかけてデータが分布しているので、花弁とがくは相関関係がありそうです」
上司「"ありそう"じゃ定性的だなあ!責任ある社会人なら定量的な裏付けが必要だね」
私「ぴえん🥺」
このように、普段何気なく使っている**「相関」ですが、定量的な裏付けを求められると困る**ことも多いかと思います。
ユースケース1の解決策:ピアソン相関係数とP値
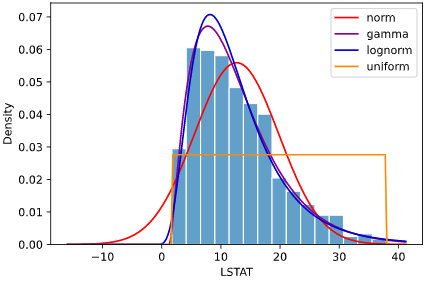
相関の強さを定量的に判断する指標として、「相関係数」という言葉を知っている方も多いかと思います。
相関係数は何種類かありますが、最も一般的なのは**「ピアソン相関係数」**です。
ピアソン相関係数の定量的解釈には幅がありますが、こちらの資料のように、下表の解釈が一般的かと思います。
| 相関係数Rの絶対値 | 解釈 |
|---|---|
| 0.0 < R ≦0.2 | XとYの間にほとんど相関がない |
| 0.2 < R ≦0.4 | XとYの間に弱い相関がある |
| 0.4 < R ≦0.7 | XとYの間にやや強い相関がある |
| 0.7 < R ≦1 | XとYの間に強い相関がある |
また、ピアソン相関係数の弱点として、データ数が少ない時の信頼性が低いことが挙げられます。
例えば、下図のようにデータが2点しかない場合、実際の相関有無に関わらずピアソン相関係数=1となってしまいます。
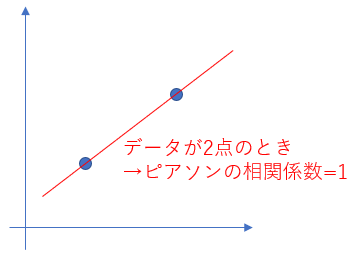
ここで活用できるのが、ピアソン相関係数=0を帰無仮説としたときの**「P値」**です。
P値が有意水準0.05(あるいは0.01)より小さければ、「相関係数が有意に0より大きい(小さき)」と言えるので、ピアソン相関係数と併用することで、
・P値が0.05(あるいは0.01)未満のとき
** →上の表に基づき、ピアソン相関係数から相関を解釈**
・P値が0.05(あるいは0.01)以上のとき
** →データ数不足のため相関分析困難(あるいは相関なし)**
と解釈することができます。
本ツールによる判断方法
本ツールでは、ピアソンの相関係数とP値を散布図とともにプロットする
regplot.linear_plot()
というメソッドを用意しました。
from seaborn_analyzer import regplot
import seaborn as sns
iris = sns.load_dataset("iris")
regplot.linear_plot(x='petal_length', y='sepal_length', data=iris)
・P値が0.05(あるいは0.01)未満のとき
** →上の表に基づき、ピアソン相関係数から相関を解釈**
・P値が0.05(あるいは0.01)以上のとき
** →データ数不足のため相関分析困難(あるいは相関なし)**
のフローでデータの相関に関して判断ができます。
また、ピアソンの相関係数は外れ値の影響を受けやすい等の欠点もありますが、これらも散布図を見てある程度は判断できるかと思います。
ユースケース2:回帰予測精度の可視化
前述の相関関係を利用して、片方の値からもう片方の値を予測する手法が、**「回帰」**です。
前述のアヤメ(iris)データセットの場合、petal_lengthからsepal_lengthを予測する、といった具合です。
このとき、
予測対象の値 (前例でのsepal_length)のことを、「目的変数」
予測に使用する値 (前例でのpetal_length)のことを、「説明変数」
と呼びます。
現実の複雑なデータでは、1種類の説明変数だけでは十分な精度の回帰モデルを作成できず、複数の説明変数が必要となる場合が多いです。
一例として、標高と緯度の2変数から、平均気温を予測する場合を考えます。
気象庁のサイトから下記のようなデータをダウンロードします
※気温は1月の平均を使用
city,altitude,latitude,temperature,pressure
Asahikawa,119.8,43.75666667,-7.5,995.3
Sapporo,17.4,43.06,-3.6,1009.8
Morioka,155.2,39.69833333,-1.9,996.1
Sendai,38.9,38.26166667,1.6,1010.2
Nagano,418.2,36.66166667,-0.6,966.6
Matsumoto,610,36.24666667,-0.4,943.5
Karuizawa,999.1,36.34166667,-3.5,897.6
Kawaguchiko,859.6,35.5,-0.6,913.7
Fujisan,3775.1,35.36,-18.4,626.5
Tokyo,25.2,35.69166667,5.2,1011.4
Nagoya,51.1,35.16666667,4.5,1011.6
Takayama,560,36.155,-1.4,950.9
Osaka,23,34.68166667,6,1009.5
Fukuoka,2.5,33.58166667,6.6,1020.1
Kagoshima,3.9,31.555,8.5,1017.3
Amami,2.8,28.37833333,14.8,1019.6
Naha,28.1,26.20666667,17,1014.5
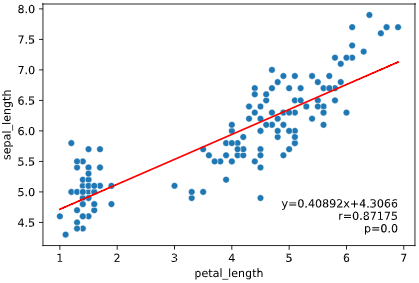
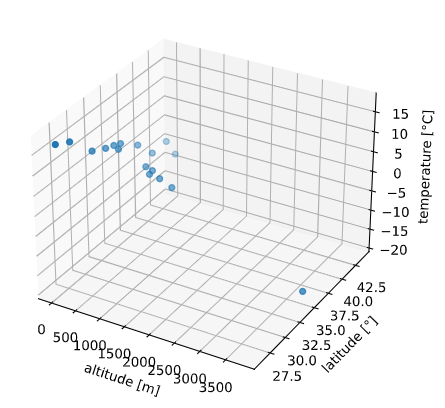
データを可視化すると下記のようになります(標高(altitude)、緯度(latitude)、気温(temperature)で3Dプロット)
import pandas as pd
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
df_temp = pd.read_csv(f'./temp_pressure.csv')
X = df_temp[['altitude', 'latitude']].values # 説明変数(標高+緯度)
y = df_temp[['temperature']].values # 目的変数(気温)
fig = plt.figure()
ax = Axes3D(fig)
ax.scatter3D(X[:, 0], X[:, 1], y)
ax.set_xlabel('altitude [m]')
ax.set_ylabel('latitude [°]')
ax.set_zlabel('temperature [°C]')
このデータに対し、標高(altitude)、緯度(latitude)の2つの説明変数から、目的変数=気温(temperature)を回帰で予測してみます。
回帰アルゴリズムには最もシンプルな「線形回帰」を使用し、ライブラリには機械学習のデファクトスタンダード、scikit-learnを利用します。
from sklearn.linear_model import LinearRegression
from sklearn.metrics import r2_score, mean_absolute_error
lr = LinearRegression() # 線形回帰用クラス
lr.fit(X, y) # 線形回帰学習
y_pred = lr.predict(X) # 学習モデルから回帰して予測値を算出
print(f'R2={r2_score(y, y_pred)}') # R2_Scoreを表示
print(f'MAE={mean_absolute_error(y, y_pred)}') # MAEを表示
> R2=0.9882146530719522
> MAE=0.7357671021553763
上司「平均気温は予測できそうかい?」
私「標高と緯度を利用すれば予測できそうです!」
上司「"できそう"じゃ定性的だなあ!定量的な裏付けはないのかい?」
私「回帰性能を表すR2-Scoreは0.988、誤差を表すMAEは0.736℃と良好です」
上司「数値だけじゃ分かりにくいなあ!グラフで見やすくしてくれ!」
私「説明変数が2次元なので、目的変数と合わせて3次元で散布図が描けません」
上司「本当にグラフ描けないの?なにか手段はあるでしょ?」
私「ぴえん🥺」
このように、説明変数が1次元であれば、目的変数と一緒に散布図プロットして相関や回帰の可否を論ずることもできますが、2次元以上では散布図により可視化できません。
(前述の3Dプロットも回帰精度を見るには不適)
回帰の性能自体はMAE(予測値と実測値の誤差平均値)などの評価指標で評価できますが、直感的な理解が難しく、グラフと比べると解釈性が低下してしまいます。
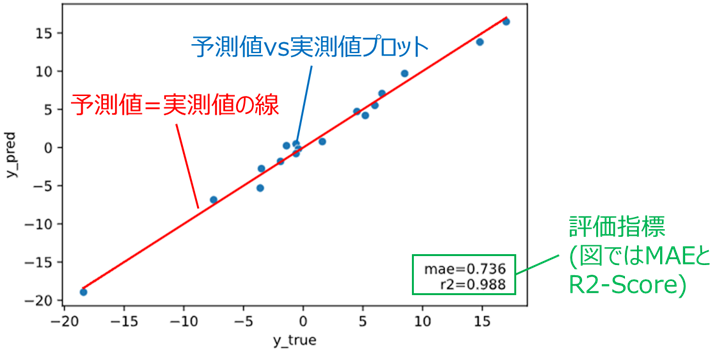
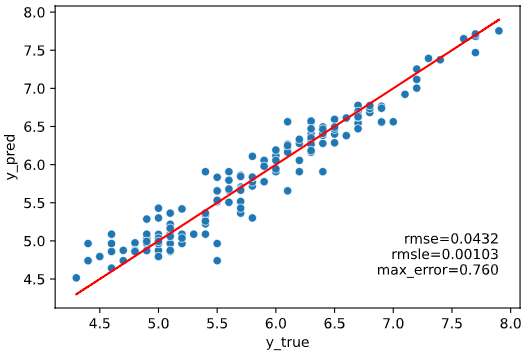
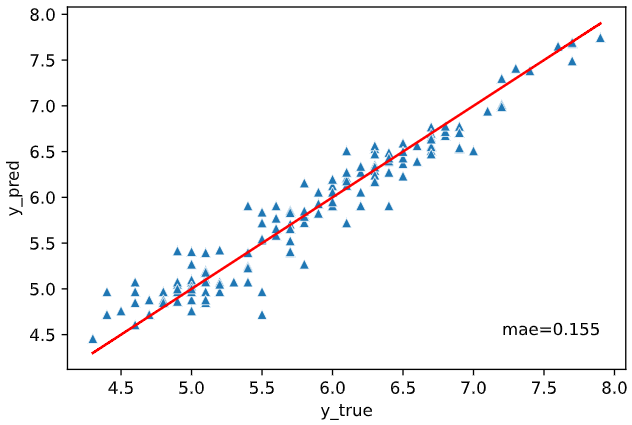
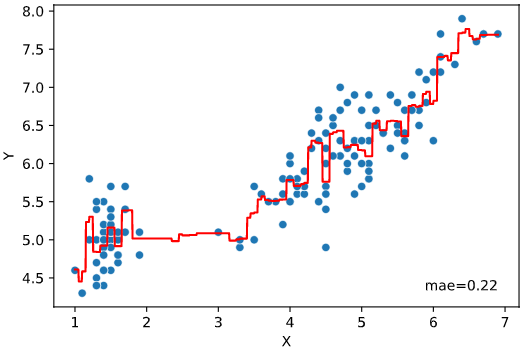
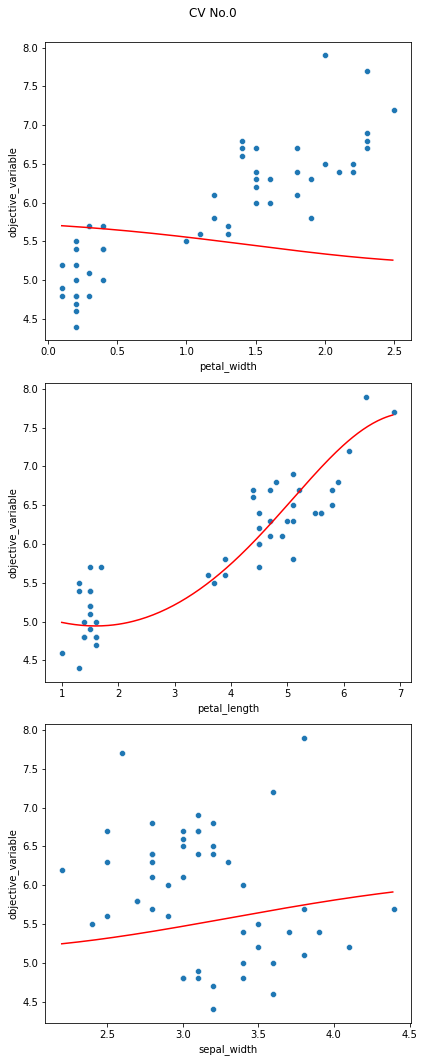
ユースケース2の解決策:予測値vs実測値プロット
回帰性能を可視化する上で有効な方法が、目的変数の実測値を横軸、回帰モデルが予測した予測値を縦軸にとった散布図プロットです。
import seaborn as sns
sns.scatterplot(x=y, y=y_pred)
ax = plt.gca()
ax.set_xlabel('true')
ax.set_ylabel('pred')
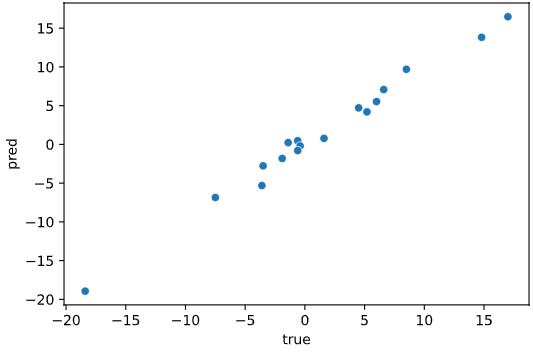
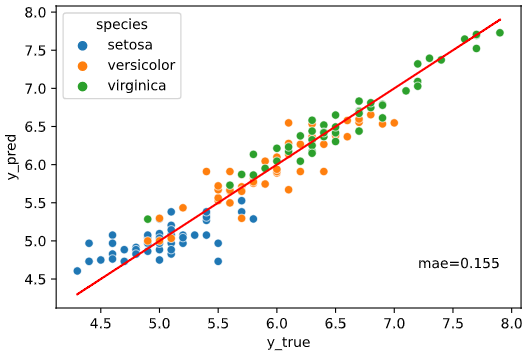
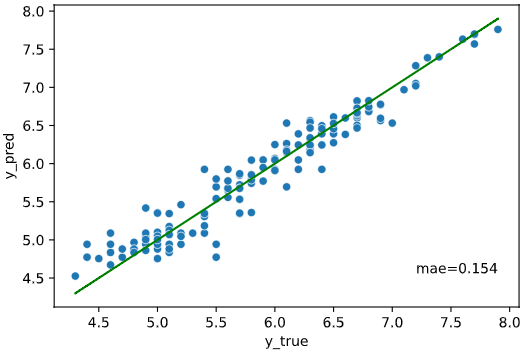
理想的な状態では、下図赤線のように予測値=実測値となるので、ここからのズレを見るのが精度の可視化という意味では最適です。
青い点(データ)が赤線(予測値=実測値の線)の近くに集まっているほど回帰性能が高いとみなすイメージです。
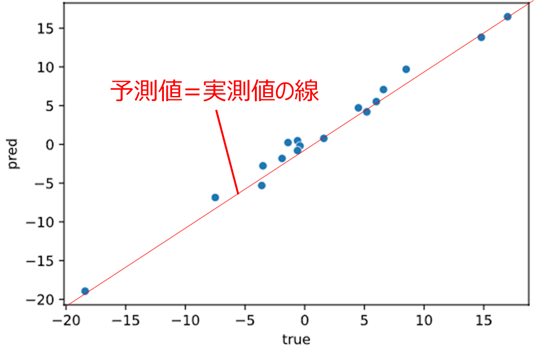
ならば予測値=実測値の線をプロットすれば良いじゃないか!とツッコまれそうですが、
python(matplotlib)では、ゼロから線をプロットするのは意外と手間が掛かります。
これ以外にも
・図だけだと定量指標がない
・評価データと学習データを分けずに評価するのはズルい
・どのデータの誤差が大きいか見たい
等のツッコミポイントが、実際の分析の現場では発生しやすいかと思います
本ツールによる可視化方法
本ツールでは上記のツッコミに対応するため、
予測値vs実測値プロットを、予測値=実測値の線、各種指標ともにプロットする
regplot.regression_pred_true()
というメソッドを用意しました。
from seaborn_analyzer import regplot
import seaborn as sns
from sklearn.linear_model import LinearRegression
iris = sns.load_dataset("iris")
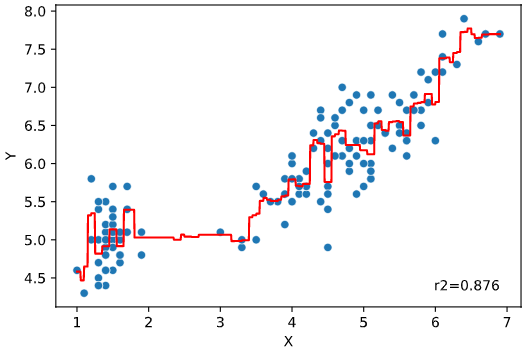
regplot.regression_pred_true(LinearRegression(), x=['altitude', 'latitude'], y='temperature',
data=df_temp, scores=['mae', 'r2'])
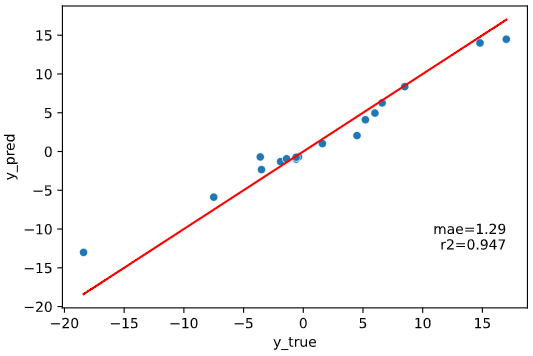
線形回帰(LinearRegression)以外でも、Scikit-learn APIに対応した回帰モデルであれば全て適用可能です(下例はランダムフォレスト回帰)
from sklearn.ensemble import RandomForestRegressor
regplot.regression_pred_true(RandomForestRegressor(), x=['altitude', 'latitude'], y='temperature',
data=df_temp, scores=['mae', 'r2'])
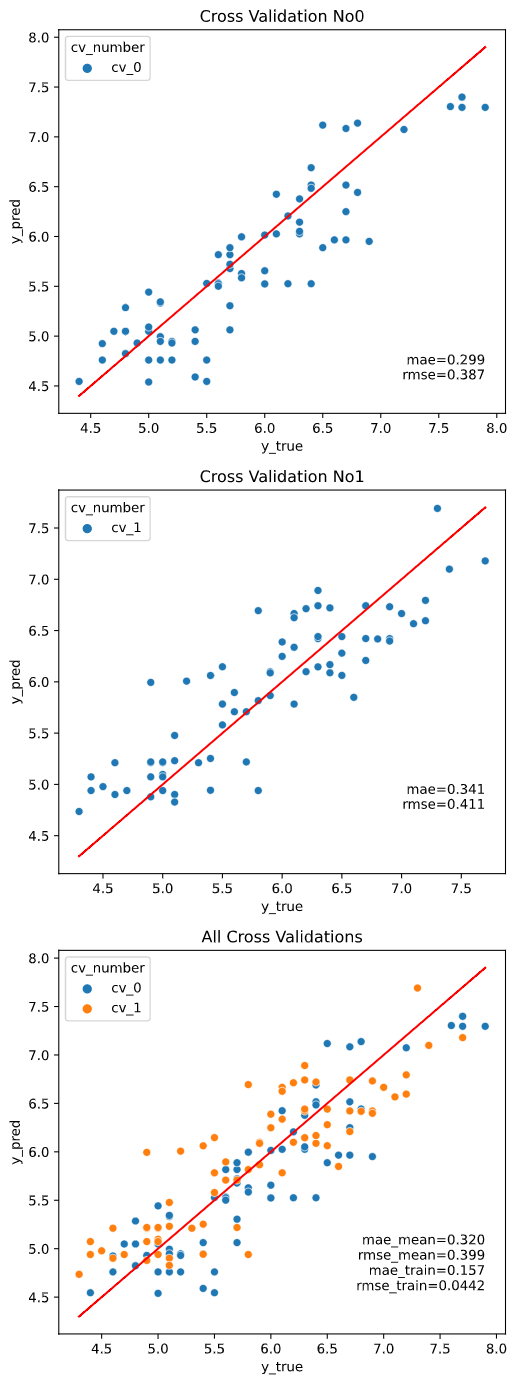
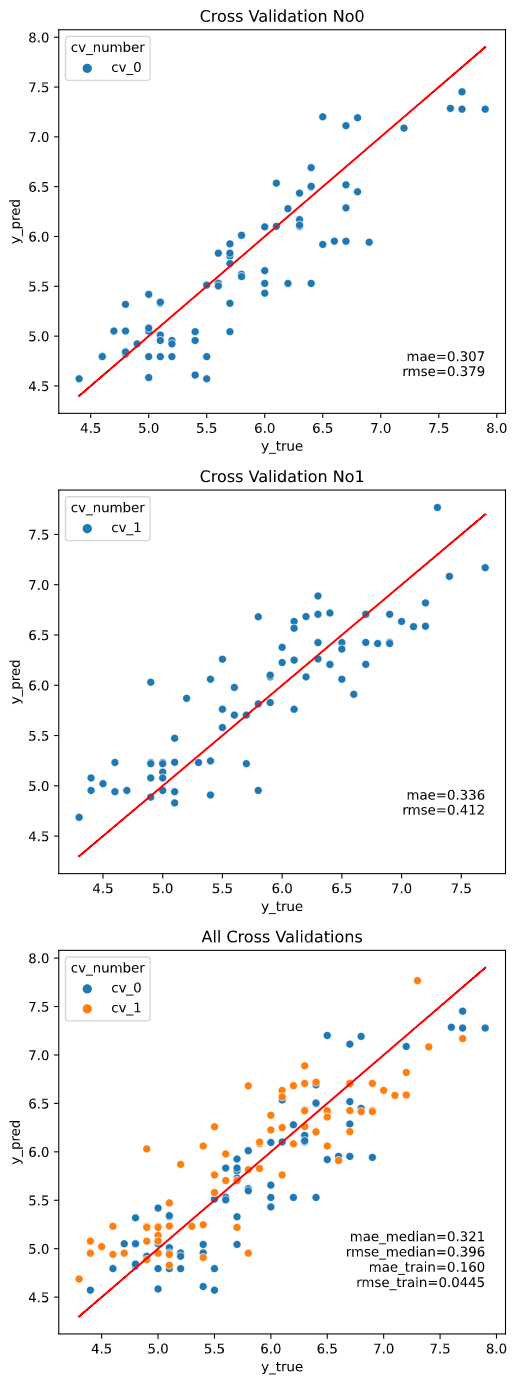
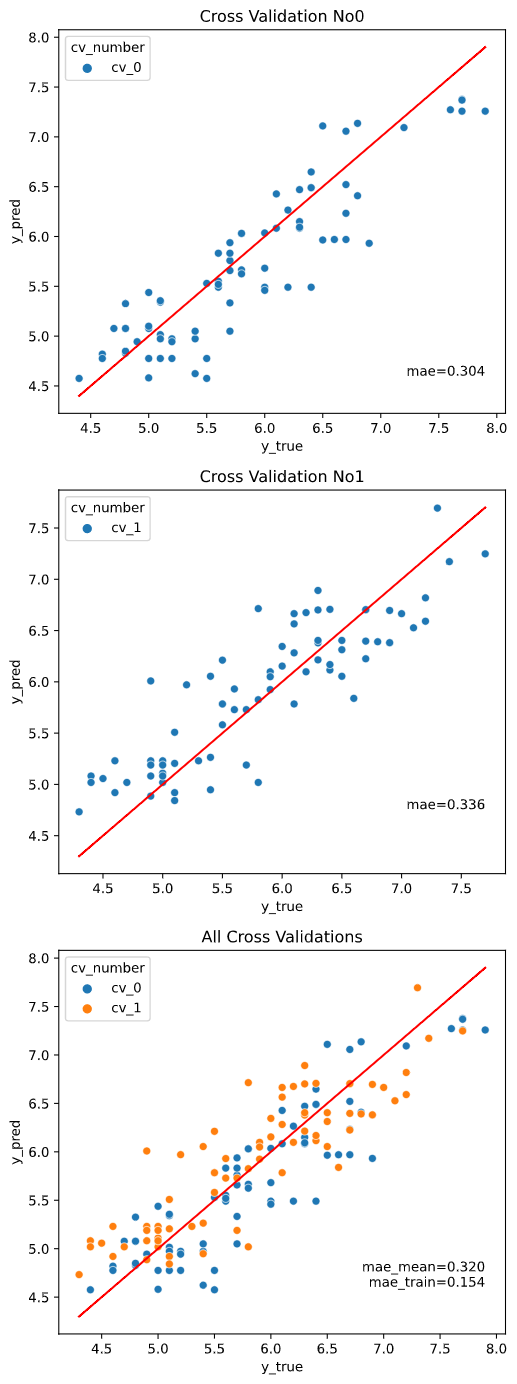
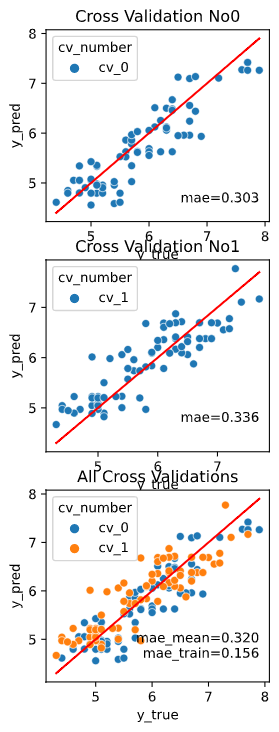
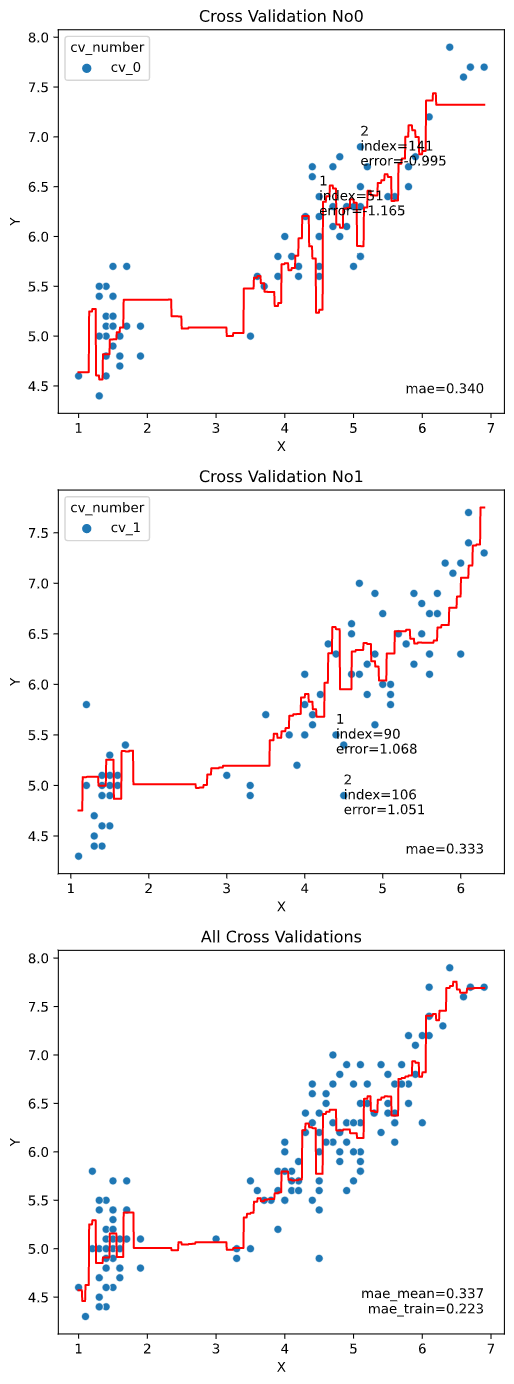
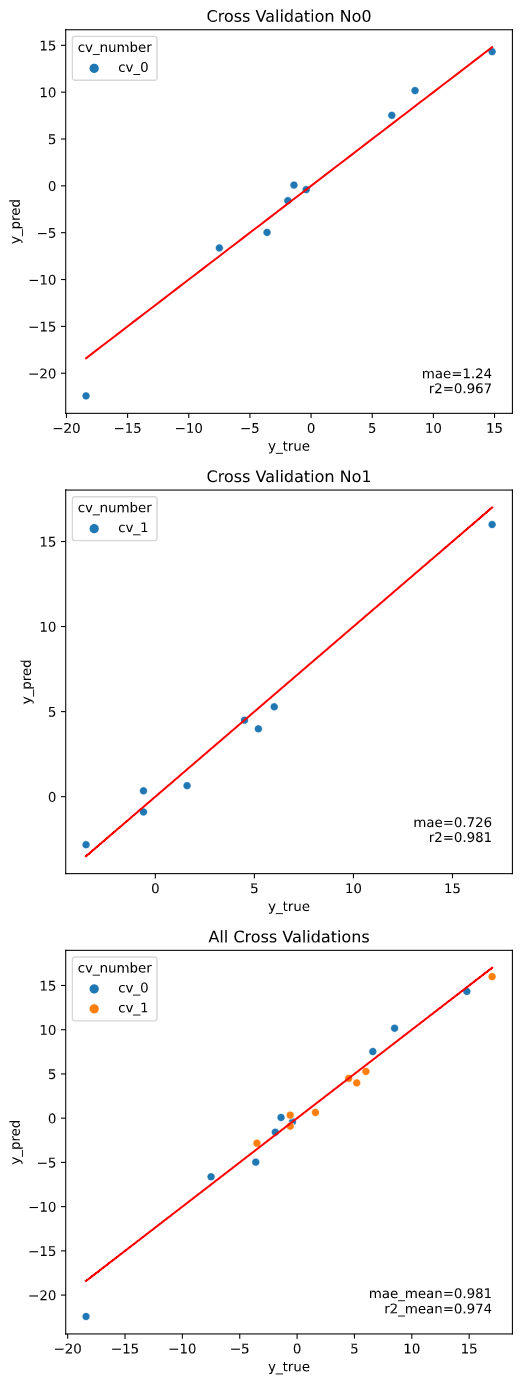
また、評価データと学習データを分けずに評価するのはズルい!
という鋭いツッコミに対応するために、クロスバリデーションでの評価にも対応しています。
※クロスバリデーションの指定法詳細は後述します
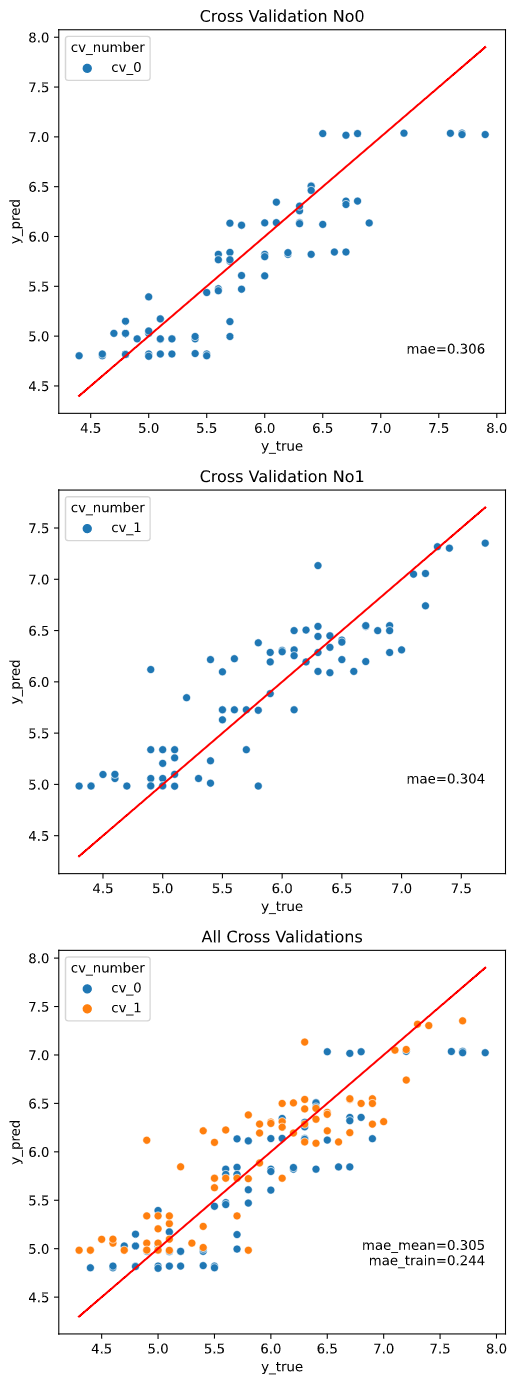
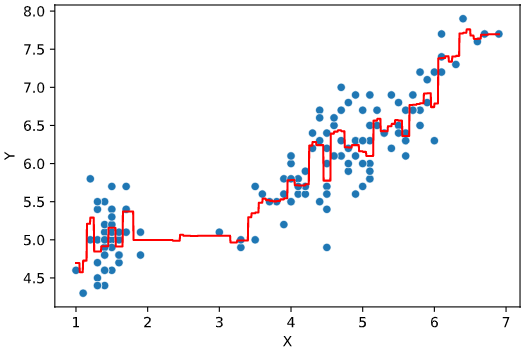
regplot.regression_pred_true(LinearRegression(), cv=2,
x=['altitude', 'latitude'], y='temperature',
data=df_temp, scores=['mae', 'r2'])
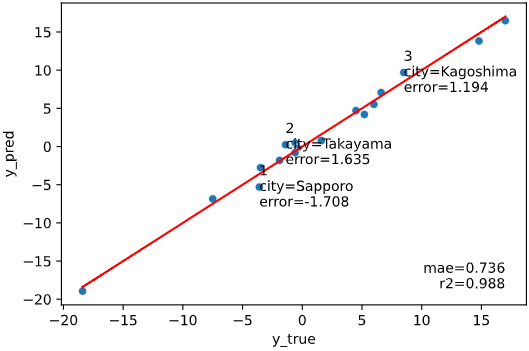
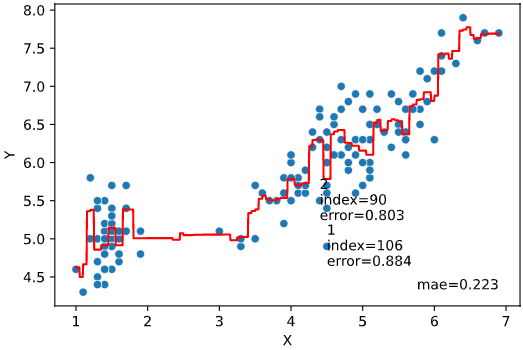
また、どのデータの誤差が大きいか見たいというツッコミが来ても即答できるよう、
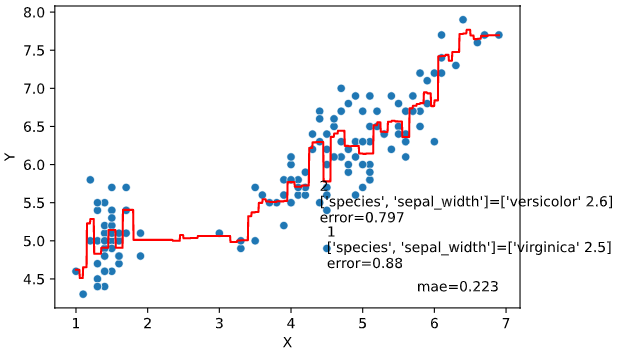
誤差上位を文字表示する機能も付加しています
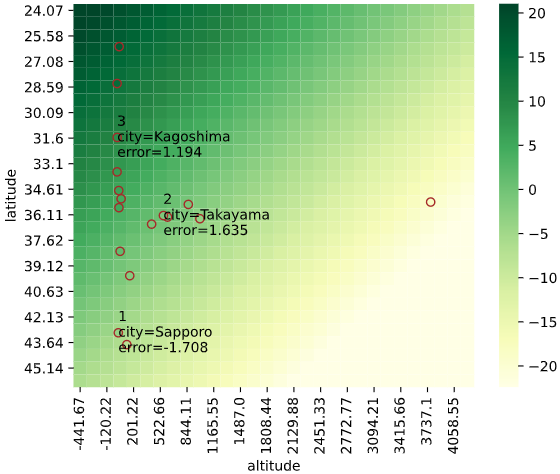
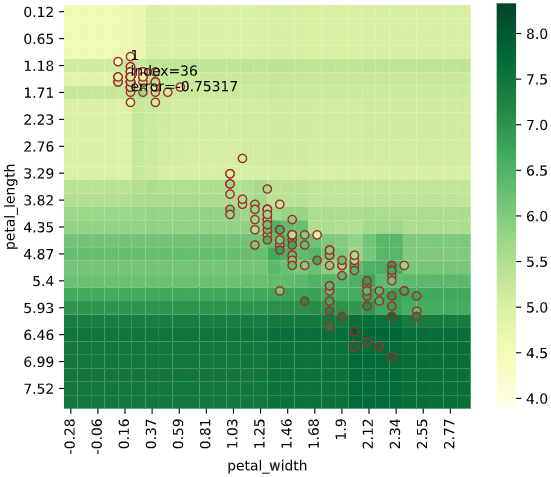
regplot.regression_pred_true(LinearRegression(), rank_number=3, rank_col='city',
x=['altitude', 'latitude'], y='temperature',
data=df_temp, scores=['mae', 'r2'])
ユースケース3:回帰モデル予測値の可視化
ユースケース2の予測値vs実測値プロットは、評価データが存在する部分の性能を可視化することができます。
しかし、以下のようなツッコミが来るかもしれません。
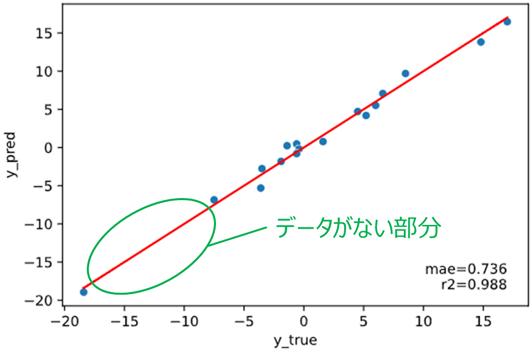
上司「この部分のデータの間隔が空いているけど、データがない部分も正しく予測できるの?」
私「他の部分は精度が高いので、恐らく大丈夫かと思います!」
上司「"恐らく"じゃ根拠が弱いな、データがない部分の予測も可視化できる方法を考えてよ」
私「ぴえん🥺」
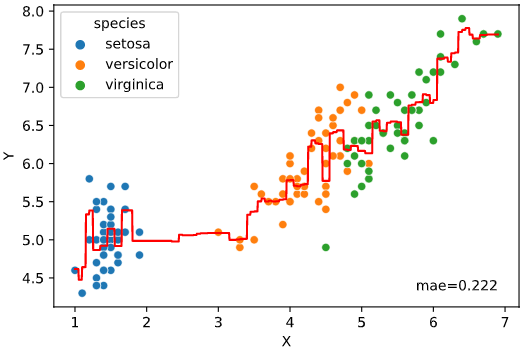
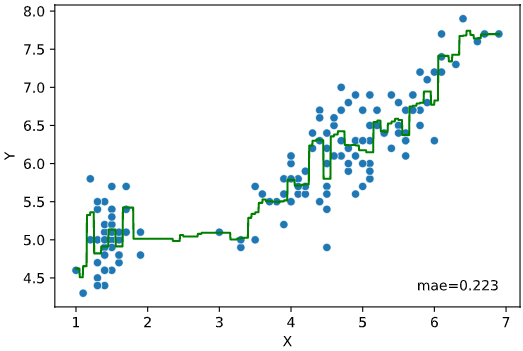
ユースケース3の解決策:回帰線&回帰予測値ヒートマップ
限られたケース(説明変数が3~4次元以下)のみの解決策となりますが、
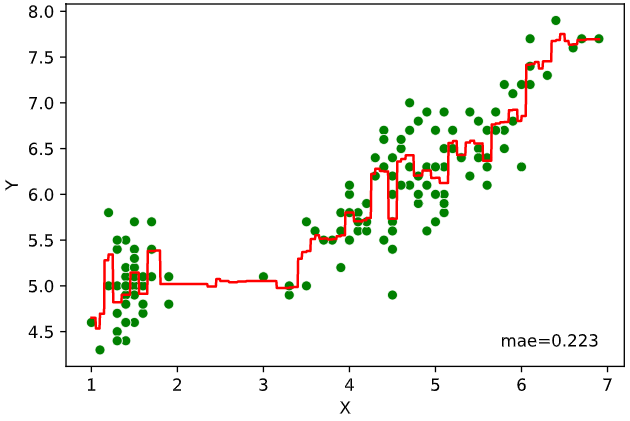
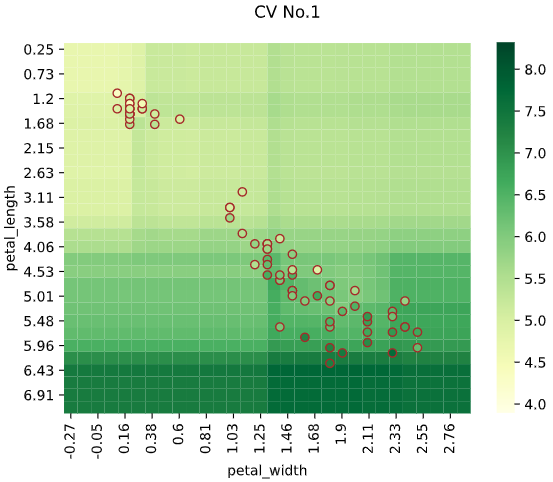
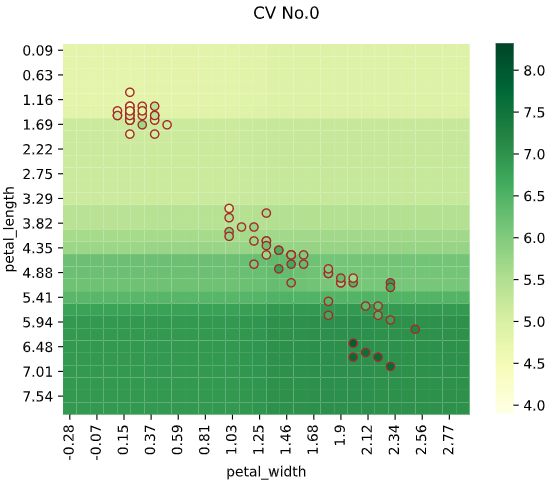
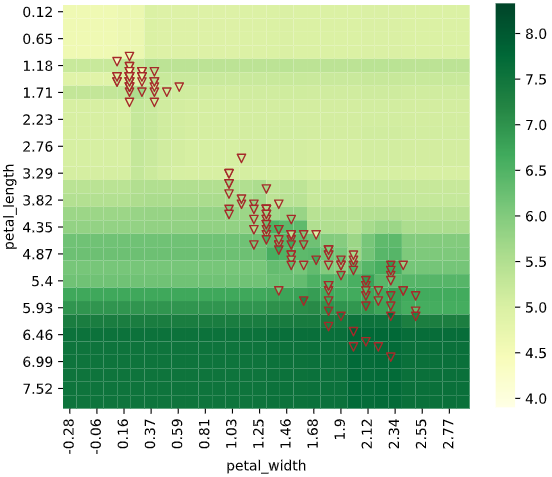
1次元説明変数の場合は回帰線をプロットすることで、データがない部分の予測値が可視化できます。(下の例はサポートベクター回帰の予測値可視化)
import numpy as np
from sklearn.svm import SVR
import seaborn as sns
import matplotlib.pyplot as plt
iris = sns.load_dataset("iris")
# 散布図プロット
sns.scatterplot(x='petal_length', y='sepal_length', data=iris)
# サポートベクター回帰学習
svr = SVR()
X = iris[['petal_length']].values
y = iris['sepal_length'].values
svr.fit(X, y)
# 回帰モデルの線を作成
xmin = np.amin(X)
xmax = np.amax(X)
Xline = np.linspace(xmin, xmax, 100)
Xline = Xline.reshape(len(Xline), 1)
# 回帰線を描画
plt.plot(Xline, svr.predict(Xline), color='red')
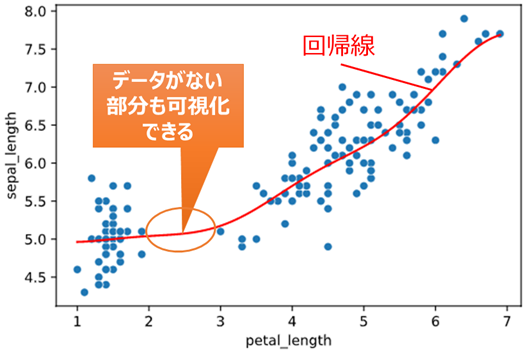
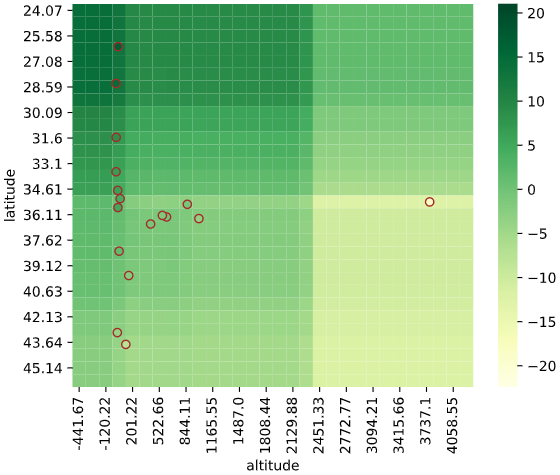
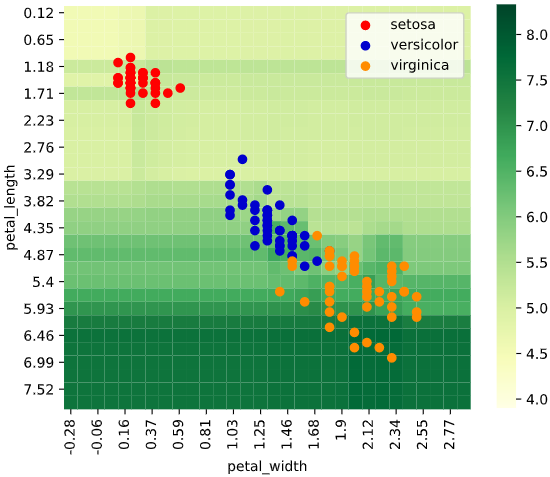
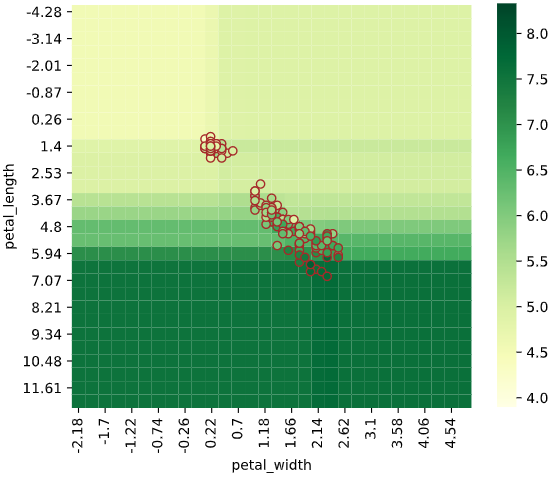
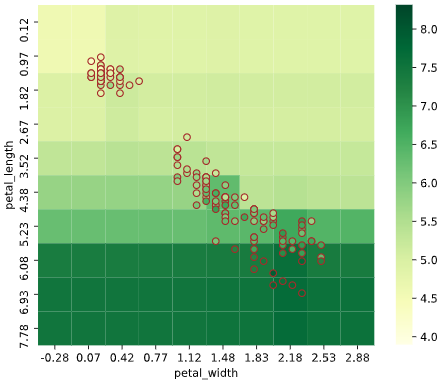
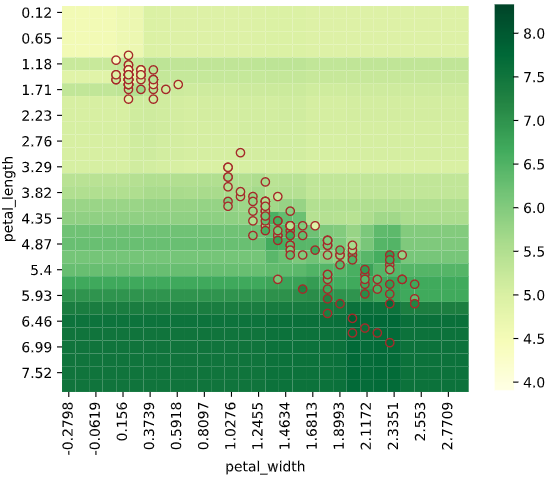
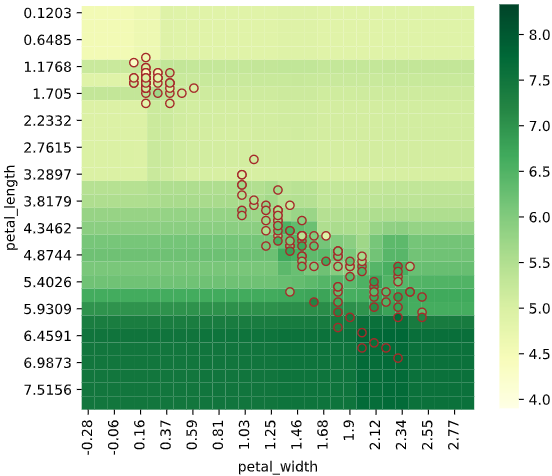
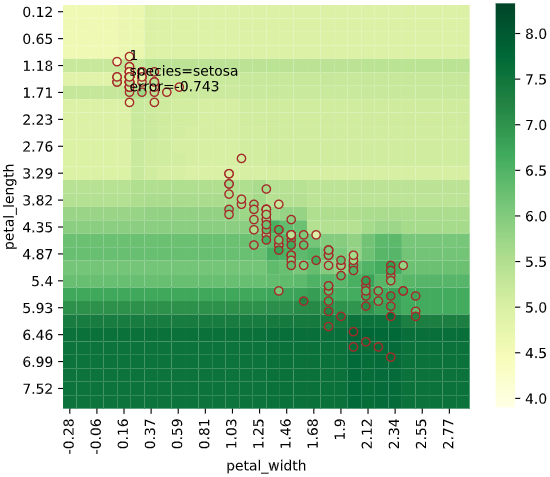
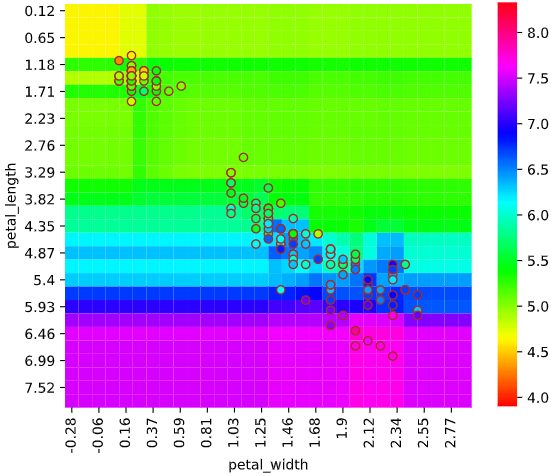
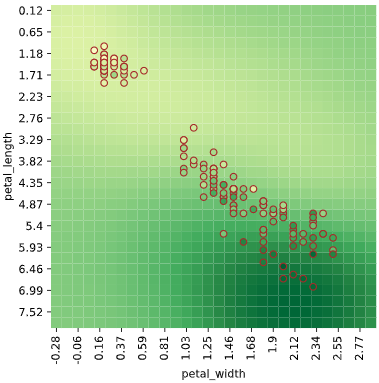
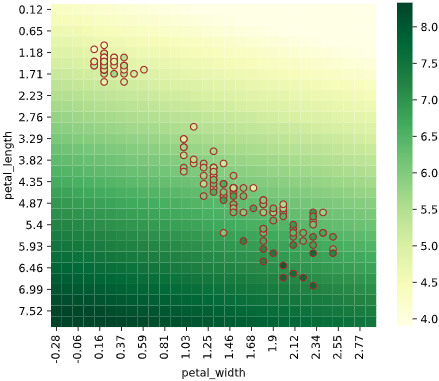
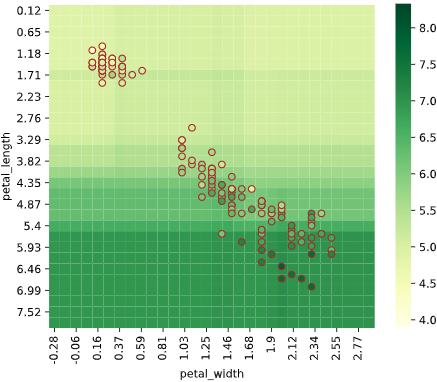
同様に2次元説明変数の場合、ヒートマップでデータがない部分の予測値が可視化できます。
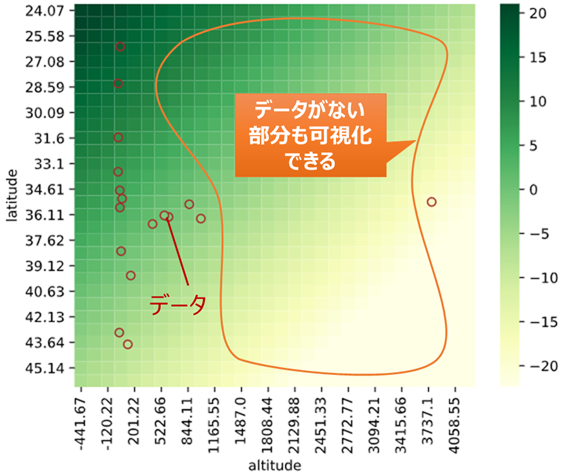
3次元以上の場合も、ヒートマップを複数組み合わせることで、描画できます
本ツールによる可視化方法
本ツールでは回帰の予測値を可視化するため、
1次元説明変数で回帰線表示+実データの散布図をプロットする
regplot.regression_plot_1d()
および、2~4次元説明変数で回帰予測値をヒートマップ表示+実データの散布図をプロットする
regplot.regression_heat_plot()
というメソッドを用意しました。
import seaborn as sns
from seaborn_analyzer import regplot
from sklearn.svm import SVR
iris = sns.load_dataset("iris")
regplot.regression_plot_1d(SVR(), x='petal_length', y='sepal_length', data=iris)
import pandas as pd
from sklearn.linear_model import LinearRegression
from seaborn_analyzer import regplot
df_temp = pd.read_csv(f'./temp_pressure.csv')
regplot.regression_heat_plot(LinearRegression(), x=['altitude', 'latitude'],
y='temperature', data=df_temp)
ユースケース2と同様、Scikit-learn APIに対応した回帰モデルであれば全て適用可能です(下例はランダムフォレスト回帰)
from sklearn.ensemble import RandomForestRegressor
regplot.regression_heat_plot(RandomForestRegressor(), x=['altitude', 'latitude'],
y='temperature', data=df_temp)
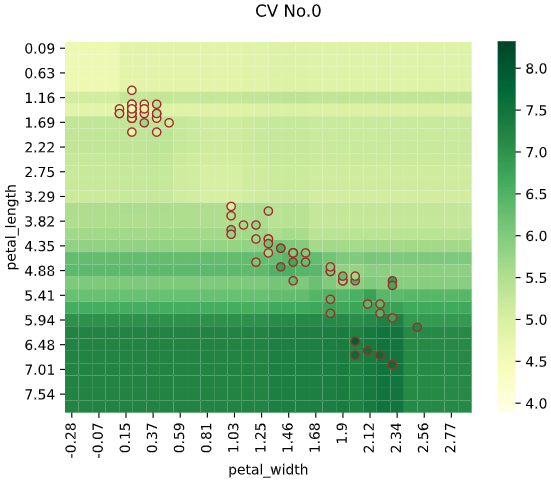
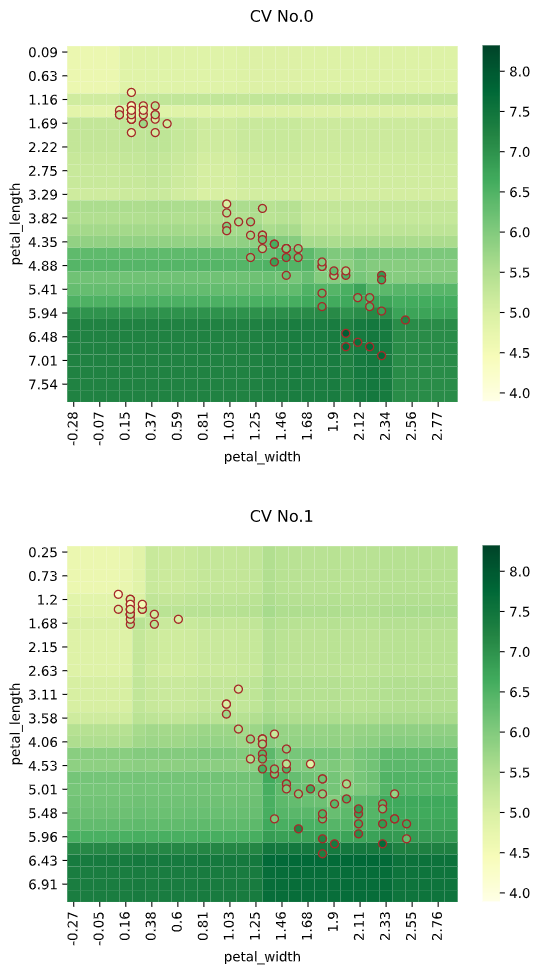
こちらもユースケース2と同様、クロスバリデーションに対応しています
from sklearn.ensemble import RandomForestRegressor
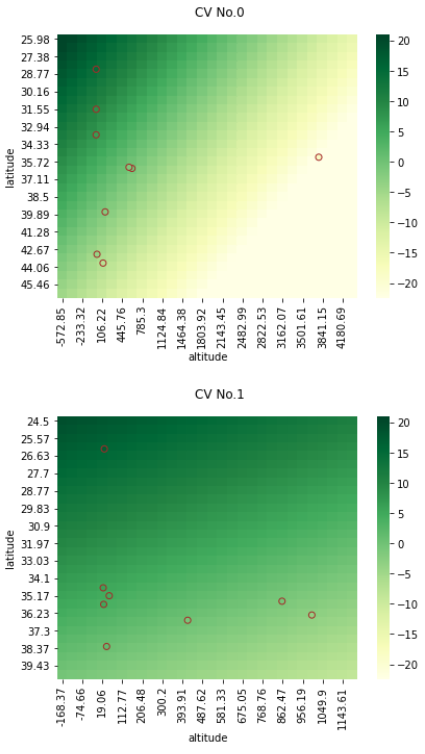
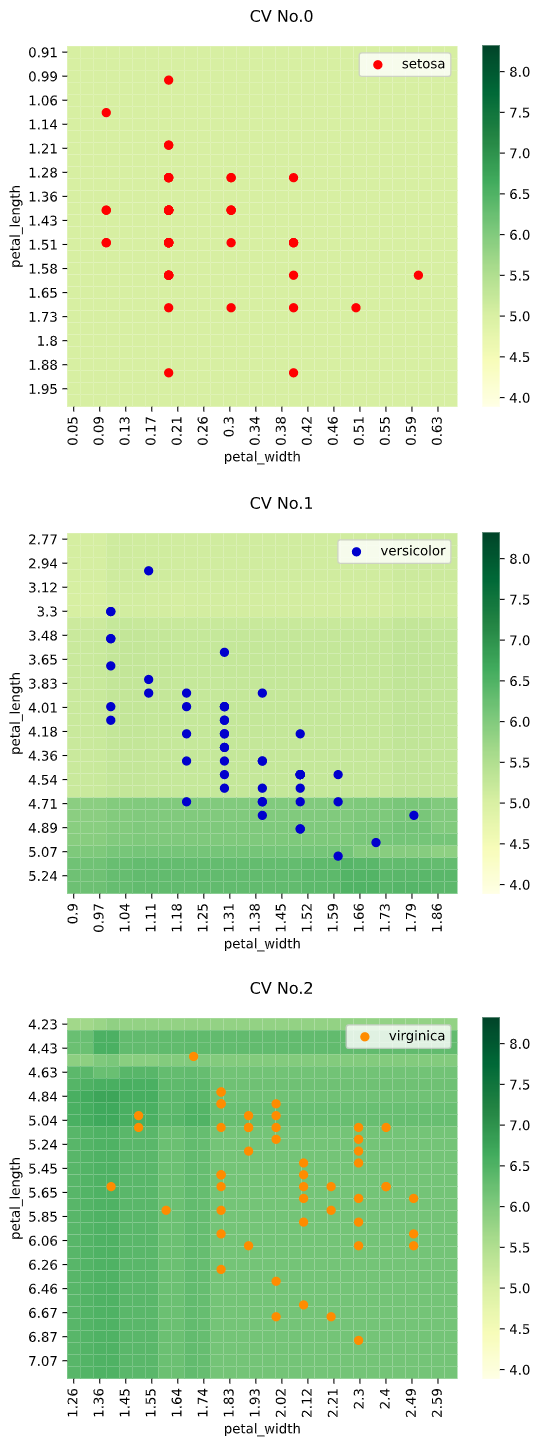
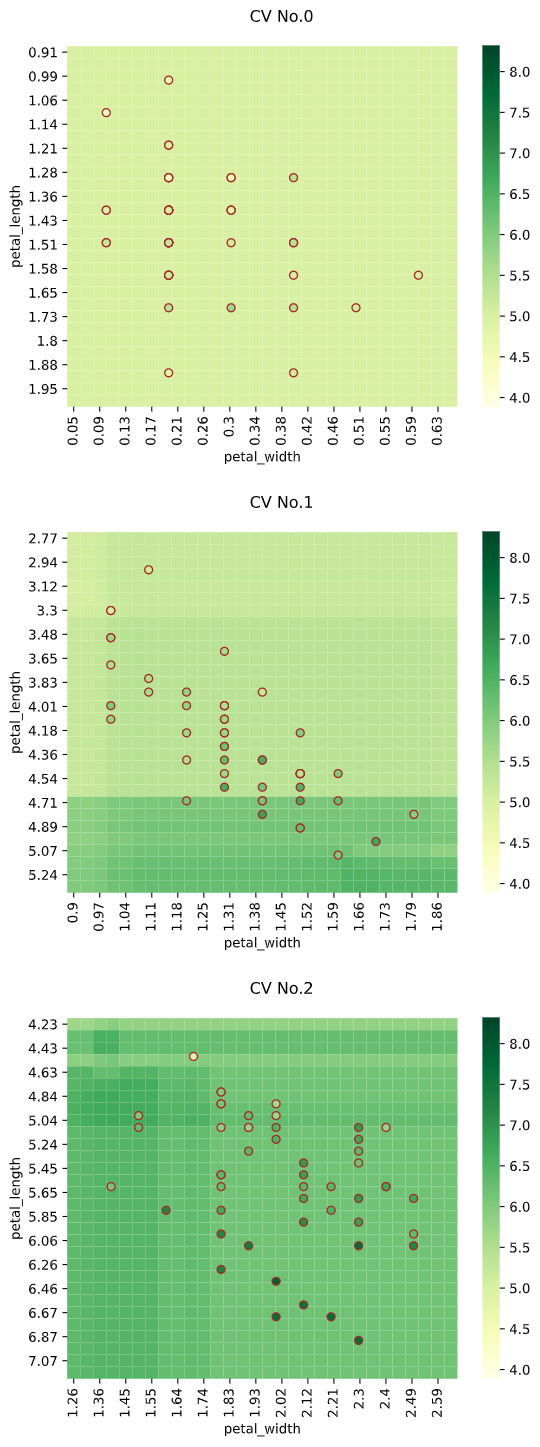
regplot.regression_heat_plot(RandomForestRegressor(), cv=2, display_cv_indices=[0, 1],
x=['altitude', 'latitude'], y='temperature', data=df_temp)
さらにユースケース2と同様、誤差上位を文字表示する機能にも対応しています
from sklearn.ensemble import RandomForestRegressor
regplot.regression_heat_plot(RandomForestRegressor(), rank_number=3, rank_col='city',
x=['altitude', 'latitude'], y='temperature', data=df_temp)
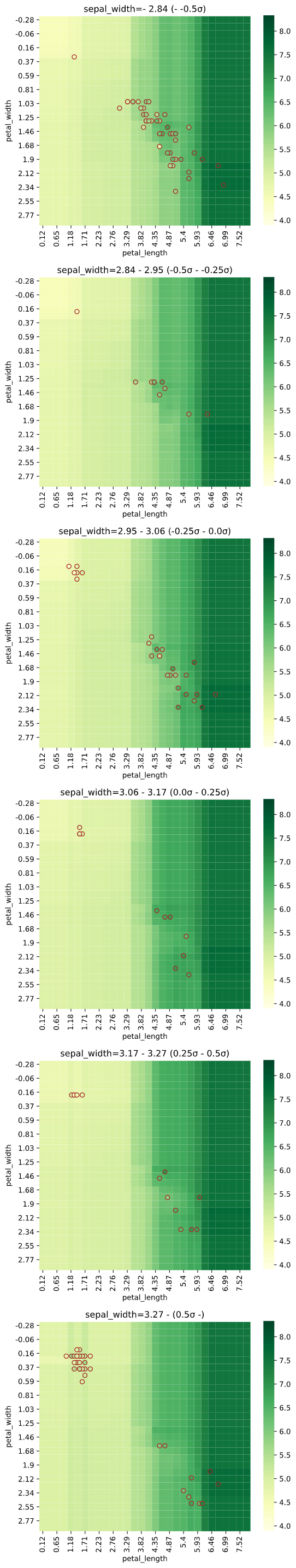
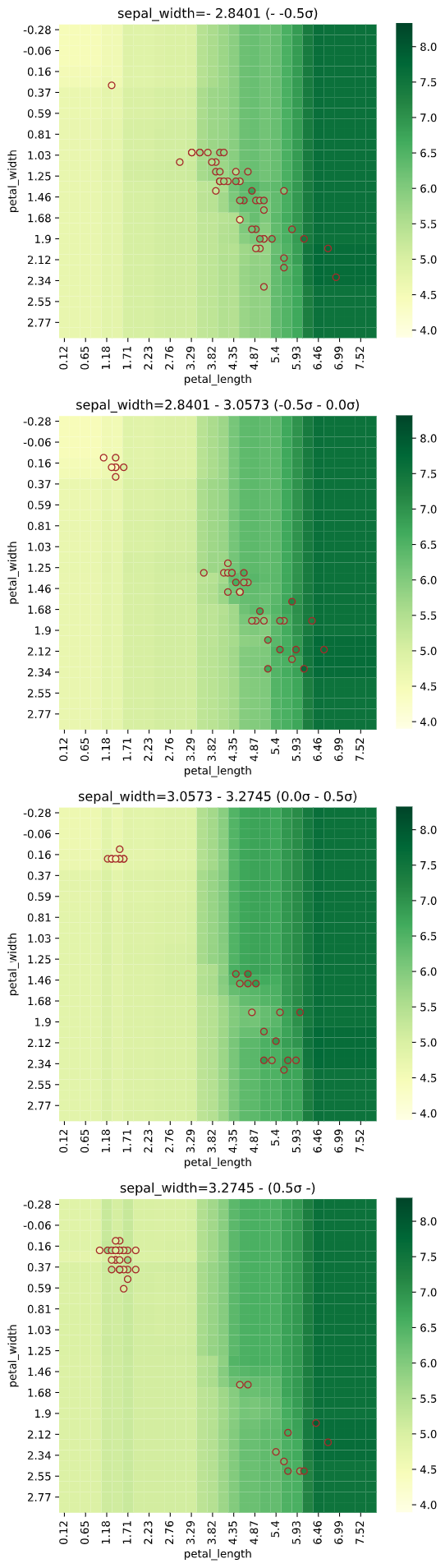
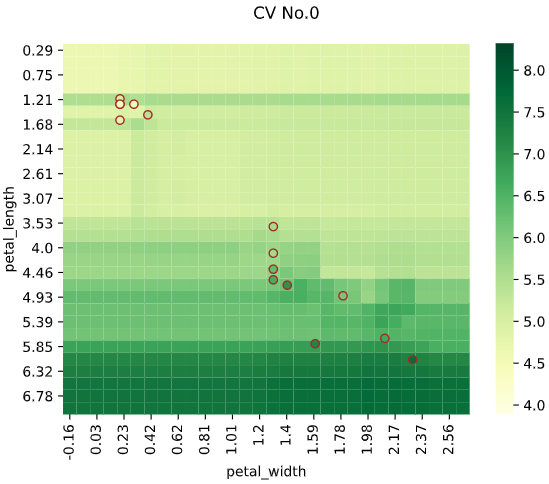
3次元説明変数の場合、3個目の説明変数を一定間隔で分割し、分割された区間の中央値を使用した予測値をヒートマップで、分割された区間内のデータを散布図でプロットします
from seaborn_analyzer import regplot
import seaborn as sns
from sklearn.svm import SVR
iris = sns.load_dataset("iris")
regplot.regression_heat_plot(SVR(), x=['sepal_width', 'petal_width', 'petal_length'],
y='sepal_length', data=iris, x_heat=['petal_length', 'petal_width'],
pair_sigmarange=1.0)
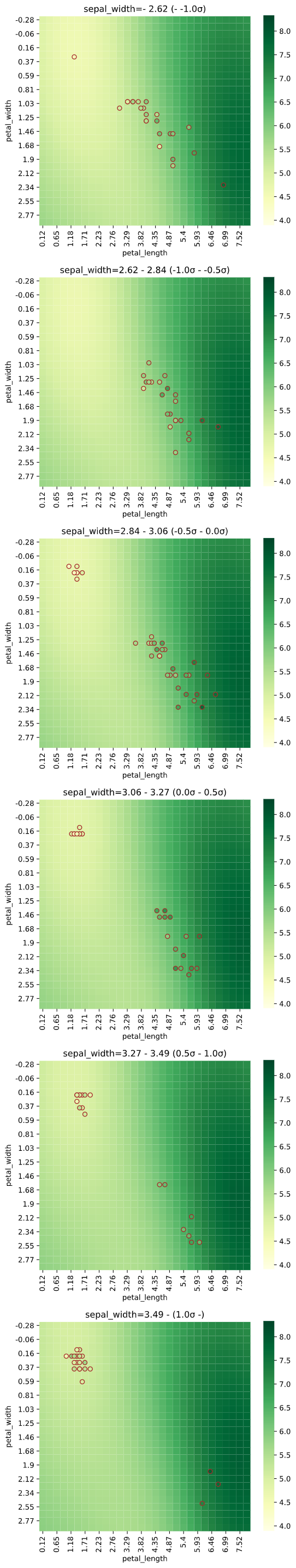
3次元でヒートマップを作成し、一定間隔ごとの断面図を2次元で表示するイメージです
4次元の場合も同様に、3個目・4個目の説明変数を一定間隔に分割し、縦横に図を並べますが、全ての図を見るのが大変なので、あまり実用的ではないかもしれません
使用に必要なもの
・Python本体 (動作確認時は3.9.1を使用)
・Matplotlibで表示可能な環境 (Jupyter等)
・下記ライブラリ (参考として動作確認時のライブラリバージョンも記載)
seaborn (0.11.1)
numpy (1.20.1)
pandas (1.2.2)
matplotlib (3.3.4)
scipy (1.6.0)
scikit-learn (0.24.1)
インストール方法
下記コマンドでインストール可能です
$ pip install seaborn-analyzer
※アンダースコアのpip install seaborn_analyzerでもインストール可能です。
インポート時はアンダースコアのimport seaborn_analyzerやfrom seaborn_analyzer
となるのでご注意ください
コード
モジュールcustom_scatter_plot.py内のクラスregplotに、必要な処理をまとめました。
seabornのregplotぽいAPIで使用できるようにするため、クラスメソッドを利用しています。
・GitHubにもアップロードしています
・該当部分のソースコードはこちら
各メソッドの解説
実行可能なメソッドは下記4個となります
| メソッド名 | 機能 |
|---|---|
| linear_plot | ユースケース1で紹介した、ピアソン相関係数とP値を散布図と共に表示 |
| regression_pred_true | ユースケース2で紹介した、予測値vs実測値プロット |
| regression_plot_1d | ユースケース3で紹介した、1次元説明変数で回帰線表示 |
| regression_heat_plot | ユースケース3で紹介した、2~4次元説明変数で回帰予測値をヒートマップ表示 |
参考までに、クラス内のみで使用されるメソッド一覧も下記します
| メソッド名 | 機能 |
|---|---|
| _round_digits | 小数点以下N桁を丸めるメソッド |
| _round_dict_digits | dictのvalueすべての小数点以下N桁を丸めるメソッド(内部で_round_digitsメソッドを呼び出し) |
| _make_score_dict | 回帰評価指標を算出してdict化するメソッド |
| _rank_display | 誤差上位を文字プロットするメソッド |
| _scatterplot_ndarray | np.ndarrayを入力として散布図表示(scatterplot) |
| _plot_pred_true | 予測値と実測値を、回帰評価指標とともにプロット(regression_pred_trueメソッドの描画処理部分) |
| _estimator_plot_1d | 1次説明変数回帰曲線を、回帰評価指標とともにプロット(regression_plot_1dメソッドの描画処理部分) |
| _reg_heat_plot_2d | 回帰予測値ヒートマップと各種散布図の表示(regression_heat_plotメソッドの描画処理部分) |
| _reg_heat_plot | 回帰予測値ヒートマップ表示の、説明変数の数(2~4)に応じた分岐処理(regression_heat_plotメソッド処理のうち、説明変数の数に応じたデータ分割等を行う) |
使用方法
ユースケースで紹介した4メソッドの使用法を詳説します
linear_plotメソッド実行方法
ユースケース1で紹介した、ピアソン相関係数とP値を散布図と共に表示するlinear_plotメソッドの実行方法です。
実行元スクリプトと同フォルダにcustom_scatter_plot.pyを置き、下記のように実行します
# 参考としてirisデータを読み込んでいます
import seaborn as sns
iris = sns.load_dataset("iris")
# ここから実行部分(引数x, y, dataの指定はMUST)
from seaborn_analyzer import regplot
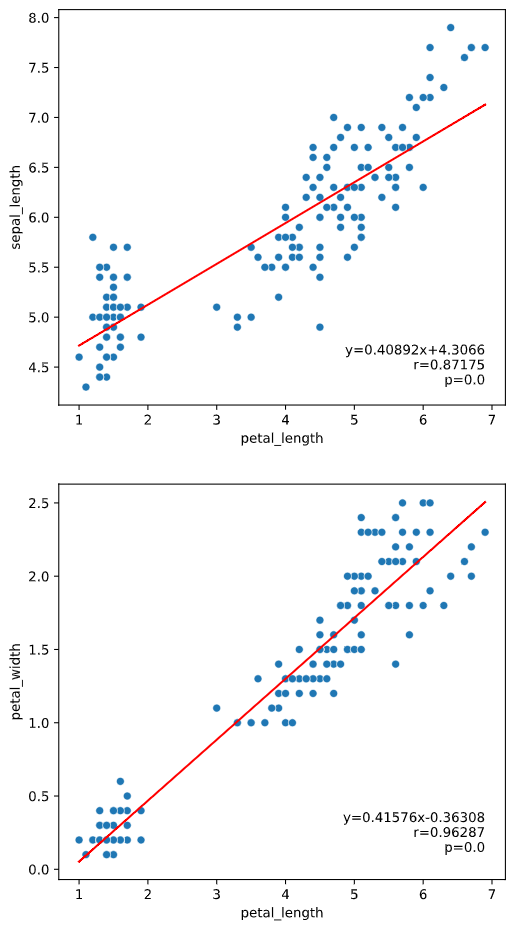
regplot.linear_plot(x='petal_length', y='sepal_length', data=iris)
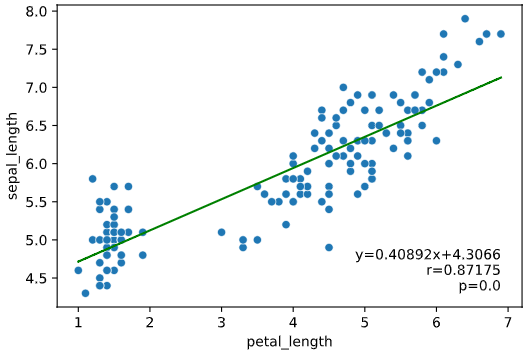
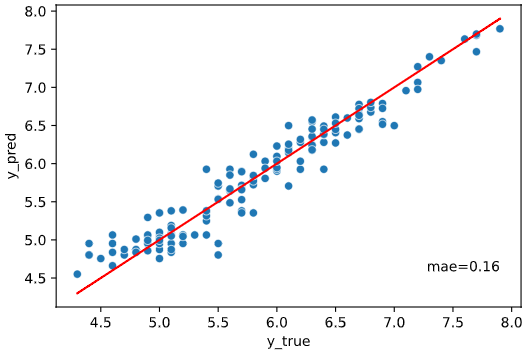
実行すると下図のようなグラフ(ピアソン相関係数とP値を散布図と共にプロット)が表示されます
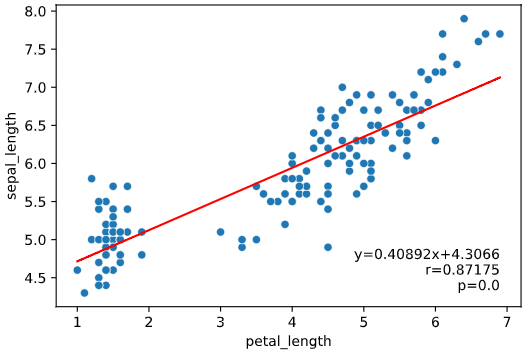
regression_pred_trueメソッド実行方法
ユースケース2で紹介した、予測値vs実測値プロットを表示するregression_pred_trueメソッドの実行方法です。
実行元スクリプトと同フォルダにcustom_scatter_plot.pyを置き、下記のように実行します
# 参考として前述の気象庁データtemp_pressure.csvを読み込んでいます
import pandas as pd
df_temp = pd.read_csv(f'./temp_pressure.csv')
# 回帰モデルとして線形回帰を使用
from sklearn.linear_model import LinearRegression
# ここから実行部分(引数estimator, x, y, dataの指定はMUST)
from seaborn_analyzer import regplot
regplot.regression_pred_true(LinearRegression(), x=['altitude', 'latitude'], y='temperature', data=df_temp)
実行すると下図のようなグラフ(予測値vs実測値プロット+評価指標)が表示されます
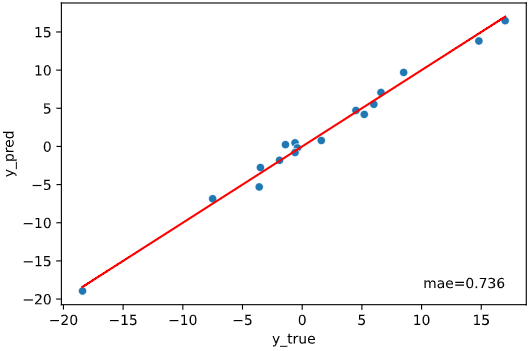
regression_plot_1dメソッド実行方法
ユースケース3で紹介した、1次元説明変数で回帰線表示するregression_plot_1dメソッドの実行方法です。
実行元スクリプトと同フォルダにcustom_scatter_plot.pyを置き、下記のように実行します
# 参考としてirisデータを読み込んでいます
import seaborn as sns
iris = sns.load_dataset("iris")
# 回帰モデルとしてサポートベクター回帰を使用
from sklearn.svm import SVR
# ここから実行部分(引数estimator, x, y, dataの指定はMUST)
from seaborn_analyzer import regplot
regplot.regression_plot_1d(SVR(), x='petal_length', y='sepal_length', data=iris)
実行すると下図のようなグラフ(回帰線プロット+評価指標)が表示されます
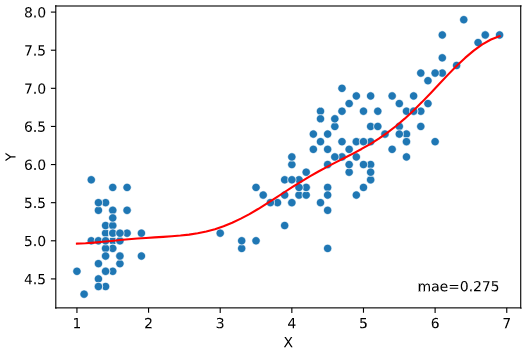
regression_heat_plotメソッド実行方法
ユースケース3で紹介した、2~4次元説明変数で回帰予測値をヒートマップ表示するregression_heat_plotメソッドの実行方法です。
実行元スクリプトと同フォルダにcustom_scatter_plot.pyを置き、下記のように実行します
# 参考として前述の気象庁データtemp_pressure.csvを読み込んでいます
import pandas as pd
df_temp = pd.read_csv(f'./temp_pressure.csv')
# 回帰モデルとして線形回帰を使用
from sklearn.linear_model import LinearRegression
# ここから実行部分(引数estimator, x, y, dataの指定はMUST)
from seaborn_analyzer import regplot
regplot.regression_heat_plot(LinearRegression(), x=['altitude', 'latitude'],
y='temperature', data=df_temp)
実行すると下図のようなグラフ(回帰予測値ヒートマップ)が表示されます
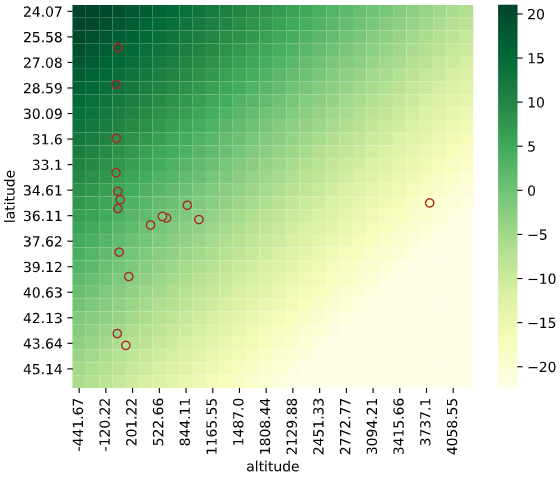
引数の解説
メソッドごとに引数を解説します
linear_plotの引数
| 引数名 | 必須引数orオプション | 型 | デフォルト値 | 内容 |
|---|---|---|---|---|
| x | 必須 | str | - | 横軸に指定するカラム名 |
| y | 必須 | str | - | 縦軸に指定するカラム名 |
| data | 必須 | pd.DataFrame | - | 入力するデータ(Pandasのデータフレーム) |
| ax | オプション | matplotlib.axes.Axes | None | 表示対象のAxes |
| hue | オプション | str | None | 色分けに指定するカラム名 |
| linecolor | オプション | str | 'red' | 回帰直線の色 |
| rounddigit | オプション | int | 5 | 表示指標の小数丸め桁数 |
| plot_scores | オプション | bool | True | 回帰式、ピアソンの相関係数およびp値の表示有無 |
| scatter_kws | オプション | dict | None | seaborn.scatterplot()に渡す引数 |
| オプション引数の詳細は後述します |
オプション引数を指定しないとき
下記の値が自動入力されます(x, y, dataは入力必須)
(x, y, data, ax=None, hue=None, linecolor='red',
rounddigit=5, plot_scores=True)
・表示例
irisデータセットでの実行例
regplot.linear_plot(x='petal_length', y='sepal_length', data=iris)
ax
表示対象のAxes(参考)を指定します
(Noneならmatplotlib.pyplot.gca()で1枚ごとにプロットします)
import matplotlib.pyplot as plt
fig, axes = plt.subplots(2, 1, figsize=(6, 12))
regplot.linear_plot(x='petal_length', y='sepal_length', data=iris, ax=axes[0])
regplot.linear_plot(x='petal_length', y='petal_width', data=iris, ax=axes[1])
hue
散布図の色分け用カラム名(列名)を指定します。
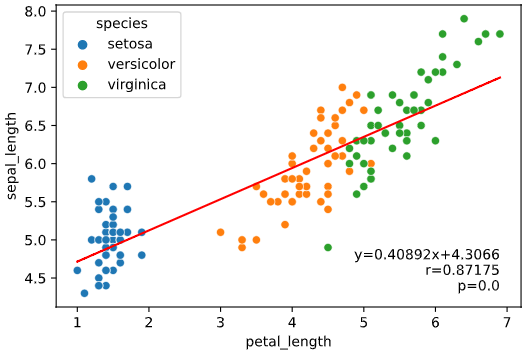
regplot.linear_plot(x='petal_length', y='sepal_length', data=iris, hue='species')
linecolor
回帰直線の色を指定します。色はmatplotlibに準拠します。
regplot.linear_plot(x='petal_length', y='sepal_length', data=iris, linecolor='green')
rounddigit
ピアソンの相関係数やp値など、数値表示の丸め桁数を指定します。
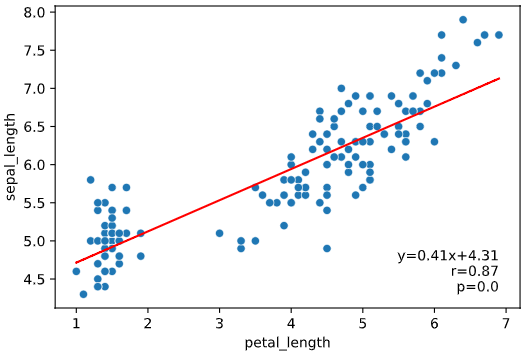
regplot.linear_plot(x='petal_length', y='sepal_length', data=iris, rounddigit=2)
plot_scores
回帰式、ピアソンの相関係数およびp値の文字表示の有無を指定します。
(Trueなら文字表示あり、Falseなら文字表示あり)
regplot.linear_plot(x='petal_length', y='sepal_length', data=iris, plot_scores=False)
scatter_kws
散布図描画用メソッドseaborn.scatterplot()に渡す引数をdict指定できます。
渡せる引数は、こちらをご参照ください
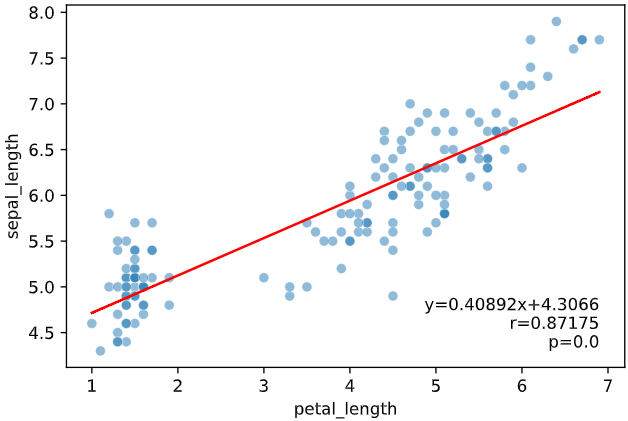
regplot.linear_plot(x='petal_length', y='sepal_length', data=iris, scatter_kws={'alpha': 0.5})
regression_pred_trueの引数
| 引数名 | 必須引数orオプション | 型 | デフォルト値 | 内容 |
|---|---|---|---|---|
| estimator | 必須 | Scikit-learn API | - | 表示対象の回帰モデル |
| x | 必須 | List[str] | - | 説明変数に指定するカラム名のリスト |
| y | 必須 | str | - | 目的変数に指定するカラム名 |
| data | 必須 | pd.DataFrame | - | 入力するデータ(Pandasのデータフレーム) |
| hue | オプション | str | None | 色分けに指定するカラム名 |
| linecolor | オプション | str | 'red' | 予測値=実測値の線の色 |
| rounddigit | オプション | int | 3 | 表示指標の小数丸め桁数 |
| rank_number | オプション | int | None | 誤差上位何番目までを文字表示するか |
| rank_col | オプション | str | None | 誤差上位と一緒に表示するフィールド名 |
| scores | オプション | str | 'mae' | 文字表示する評価指標を指定 |
| cv_stats | オプション | str | 'mean' | クロスバリデーション時に表示する評価指標統計値 |
| cv | オプション | int | int or sklearn.model_selection.* | クロスバリデーション分割法 (Noneのとき学習データから指標算出、int入力時はkFoldで分割) |
| cv_seed | オプション | int | 42 | クロスバリデーションの乱数シード |
| cv_group | オプション | str | None | GroupKFold,LeaveOneGroupOut等のグルーピング対象カラム名 |
| estimator_params | オプション | dict | None | 回帰モデルに渡すパラメータ |
| fit_params | オプション | dict | None | 学習時のパラメータをdict指定 |
| subplot_kws | オプション | dict | None | matplotlib.pyplot.subplotsに渡す引数 |
| scatter_kws | オプション | dict | None | seaborn.scatterplot()に渡す引数 |
| オプション引数の詳細は後述します |
オプション引数を指定しないとき
下記の値が自動入力されます(estimator, x, y, dataは入力必須)
(estimator, x, y, data, hue=None, linecolor='red', rounddigit=3,
rank_number=None, rank_col=None, scores=['mae'], cv_stats='mean', cv=None, cv_seed=42,
estimator_params=None, fit_params=None, subplot_kws=None)
・表示例
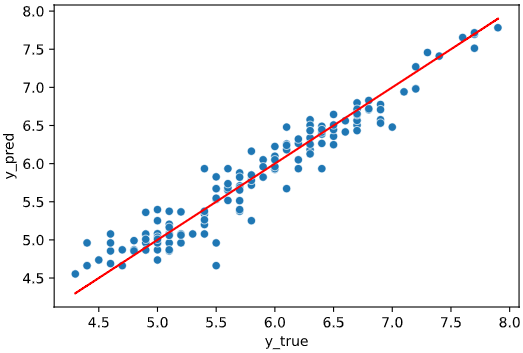
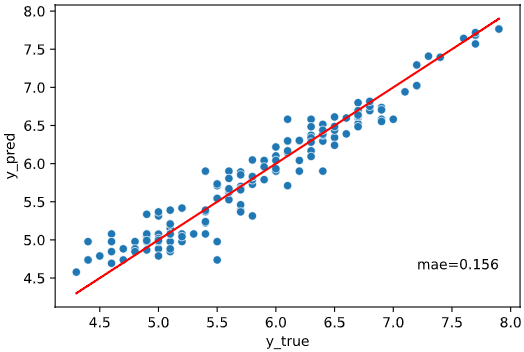
irisデータセット+ランダムフォレスト回帰RandomForestRegressorでの実行例
from sklearn.ensemble import RandomForestRegressor
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris)
実行すると下図のようなグラフ(予測値vs実測値プロット+評価指標)が表示されます
hue
散布図の色分け用カラム名(列名)を指定します。
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
hue='species')
linecolor
予測値=実測値の線の色を指定します。色はmatplotlibに準拠します。
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
linecolor='green')
rounddigit
評価指標の数値表示の丸め桁数を指定します。
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
rounddigit=2)
rank_number
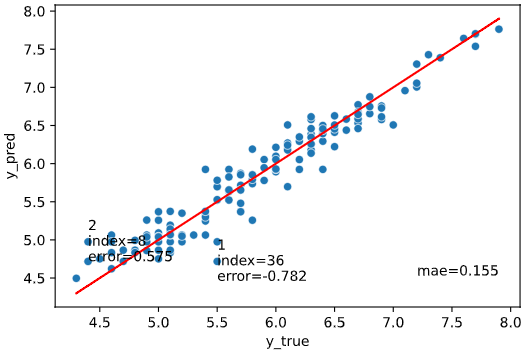
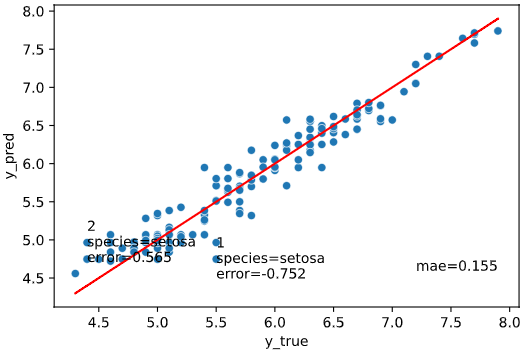
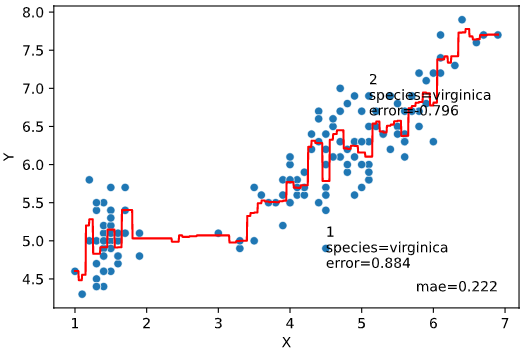
誤差上位何番目までを文字表示するかを指定します。
引数rank_colを指定すれば、一緒に表示する列も指定可能です
(rank_col指定なしのときは、データのインデックスを表示)
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
rank_number=2)
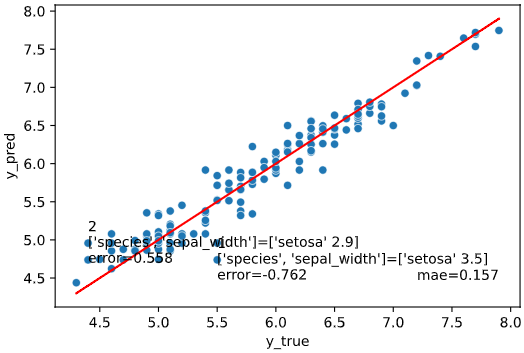
rank_col
引数rank_number指定時に、一緒に表示する列を指定します。
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
rank_number=2, rank_col='species')
リスト指定すれば、複数の列を同時表示することもできます
(表示がごちゃごちゃするのであまりオススメはできませんが‥)
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
rank_number=2, rank_col=['species', 'sepal_width'])
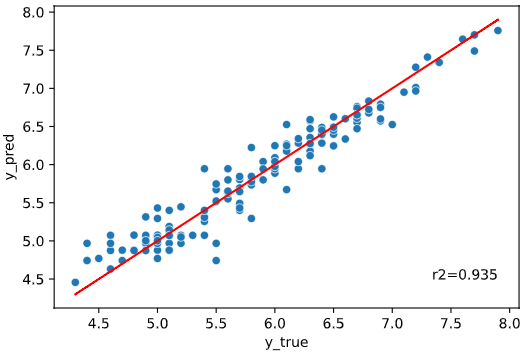
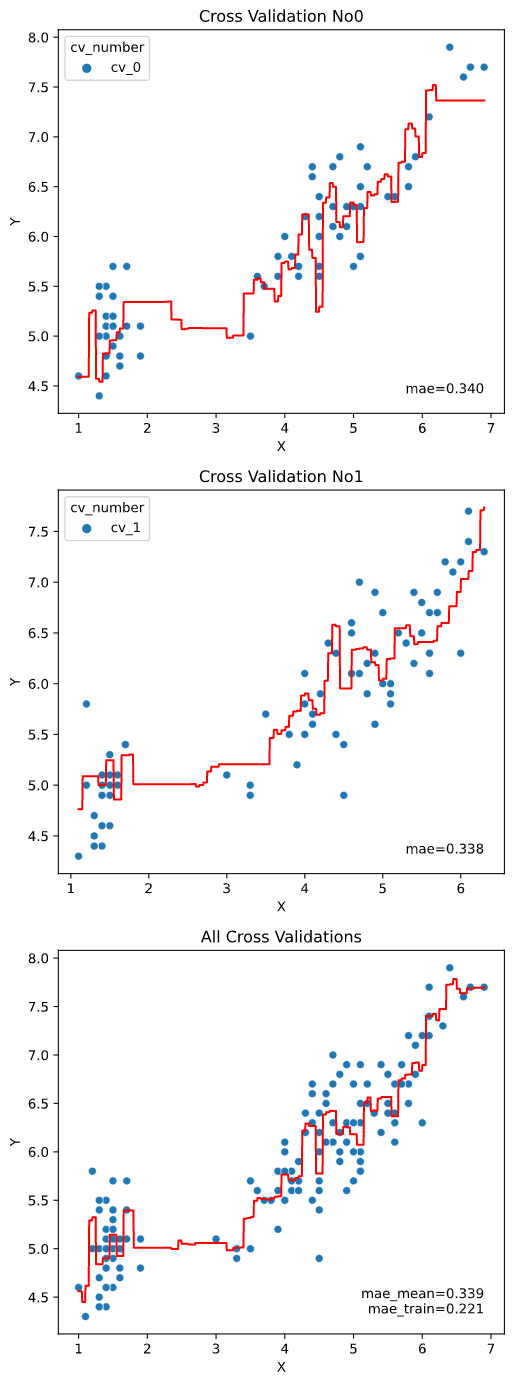
scores
文字表示する評価指標を指定します。
指定できる指標一覧は下記です
| 名称 | 指定時の文字列 |
|---|---|
| R2-Score | 'r2' |
| MAE | 'mae' |
| RMSE | 'rmse' |
| RMSLE | 'rmsle' |
| 誤差最大値 | 'max_error' |
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
scores='r2')
None指定すれば、指標表示を消せます
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
scores=None)
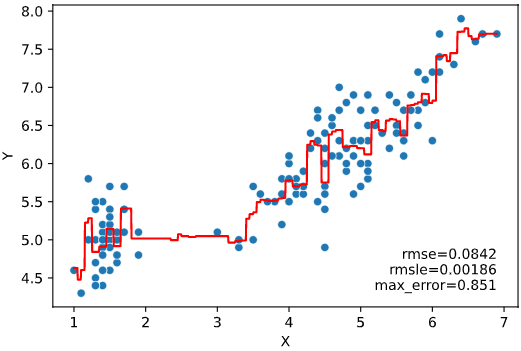
リスト指定すれば、複数の指標を同時表示することもできます
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
scores=['rmse','rmsle','max_error'])
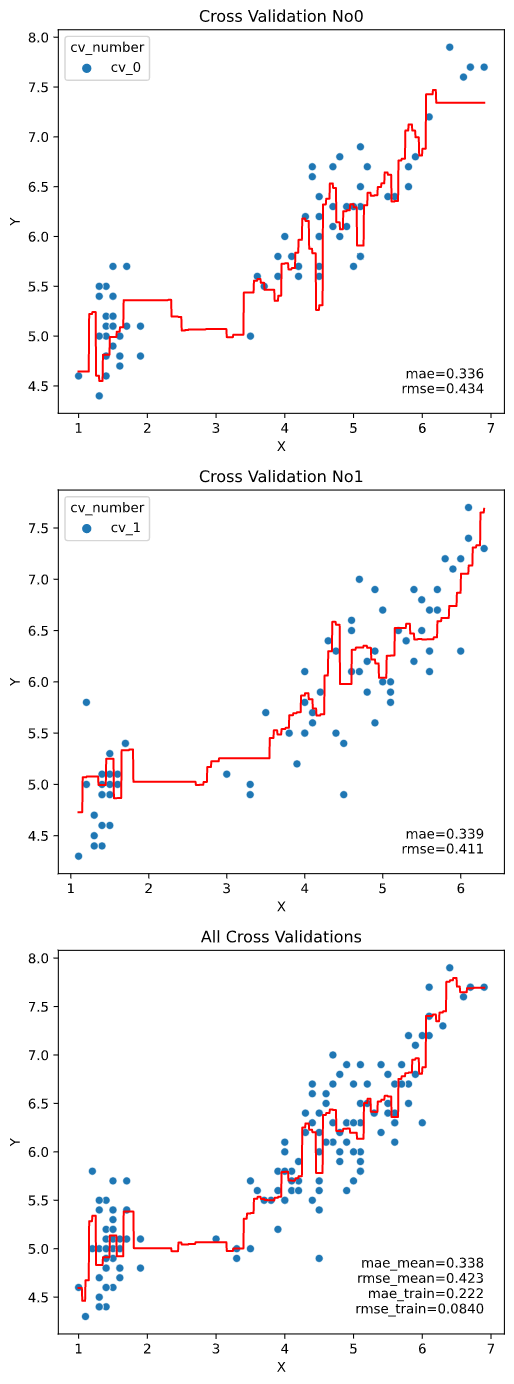
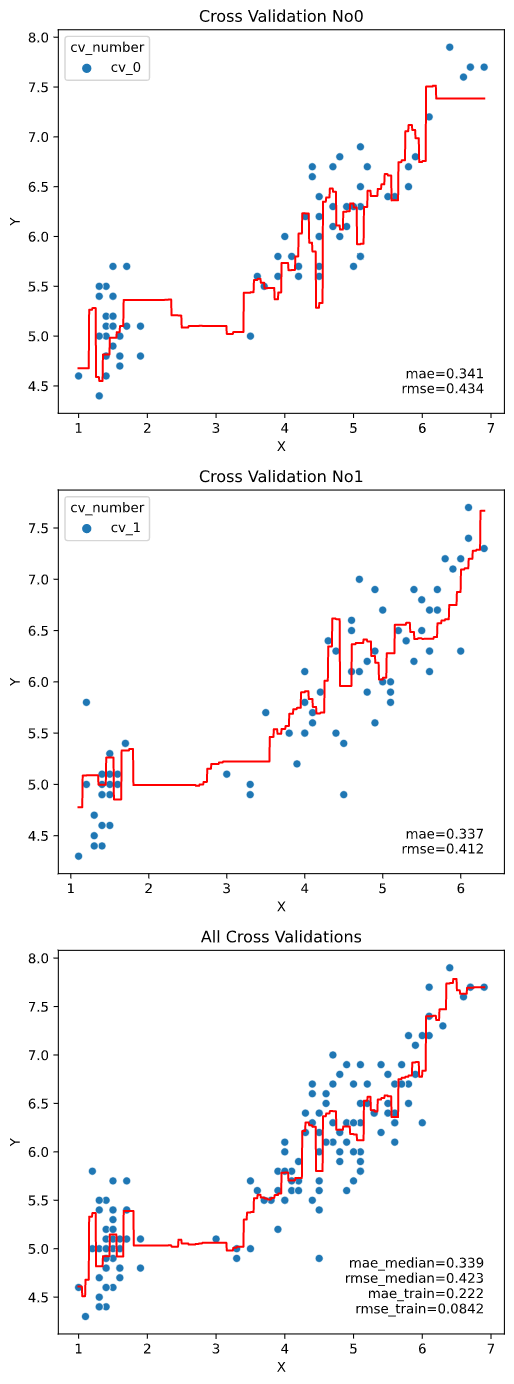
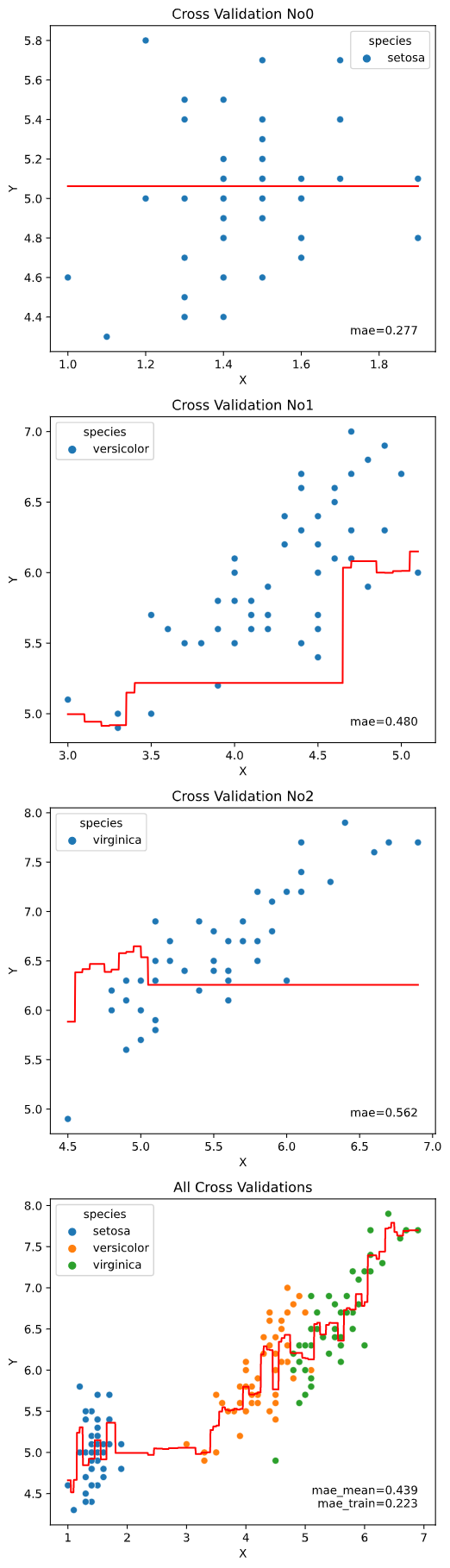
クロスバリデーション指定時は、クロスバリデーションごとの図にはクロスバリデーション内テストデータでの指標を、全体結果の図にはcv_stats引数で指定した指標統計値+全体データで学習&指標算出した場合の値 (_trainとついた指標)をプロットします。
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
scores=['mae', 'rmse'], cv_stats='mean', cv=2)
cv_stats
クロスバリデーション指定時に、全体結果の図に表示する指標の統計値を指定します。
'mean'(平均値)、'median'(中央値)、'max'(最大値)、'min'(最小値)が指定できます。
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
scores=['mae', 'rmse'], cv_stats='median', cv=2)
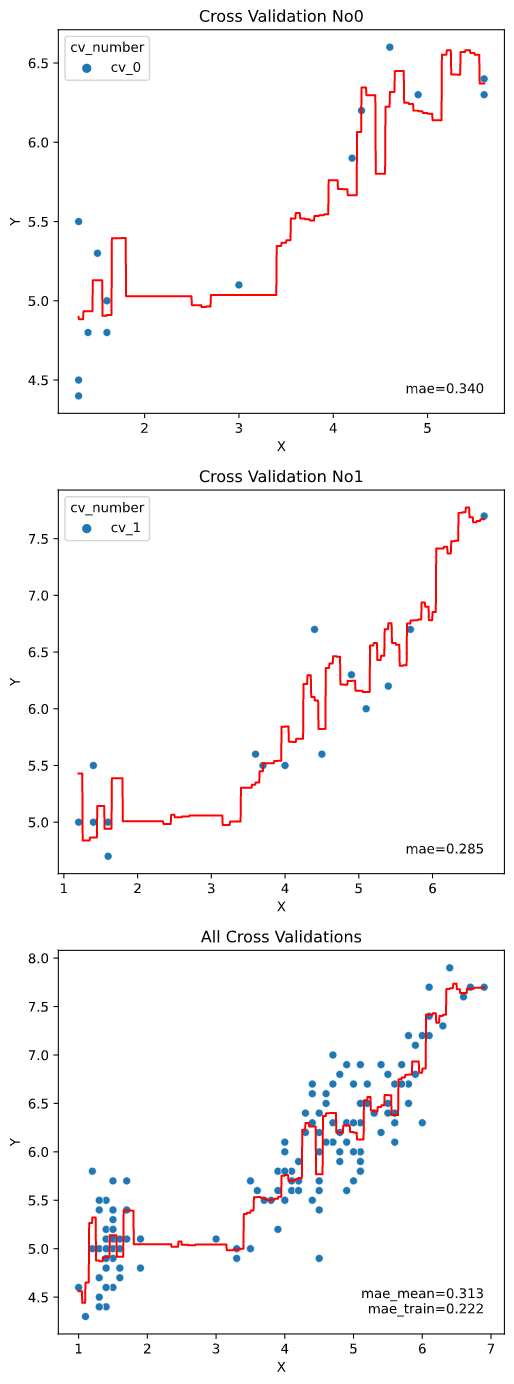
cv
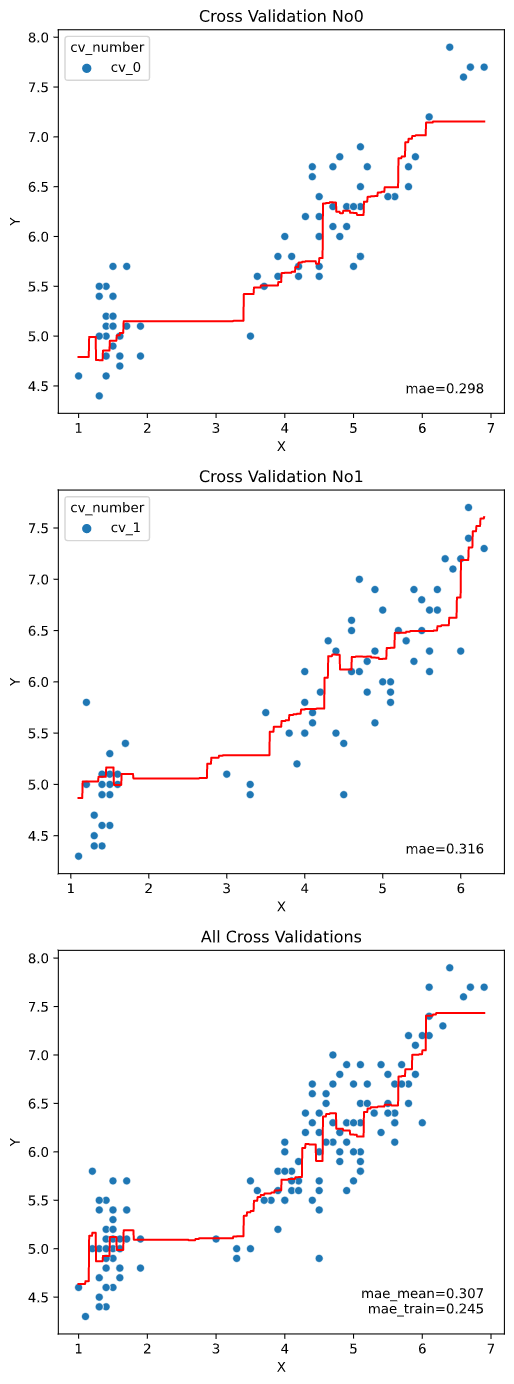
クロスバリデーション分割法を指定します。
数値を指定すれば、指定した数に応じて通常のK-Foldで分割します
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2)
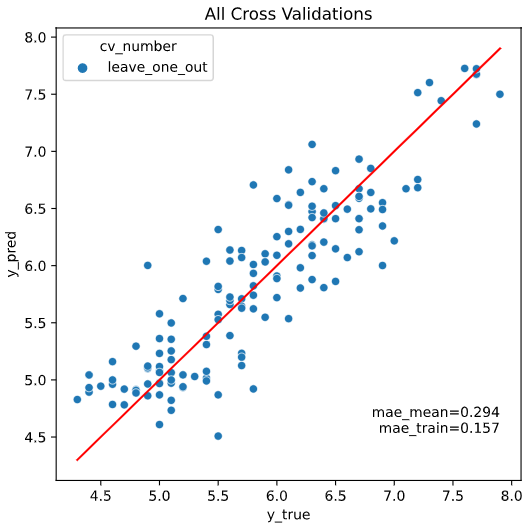
Scikit-LearnのAPIに対応していれば、Leave-One-Out、GroupKFoldなどの特殊な分割法にも対応しています。
※Leave-One-Outのときは、CVごとの図はプロットせず、各CVでの結果を1枚の図に集約プロットします
from sklearn.model_selection import LeaveOneOut
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=LeaveOneOut())
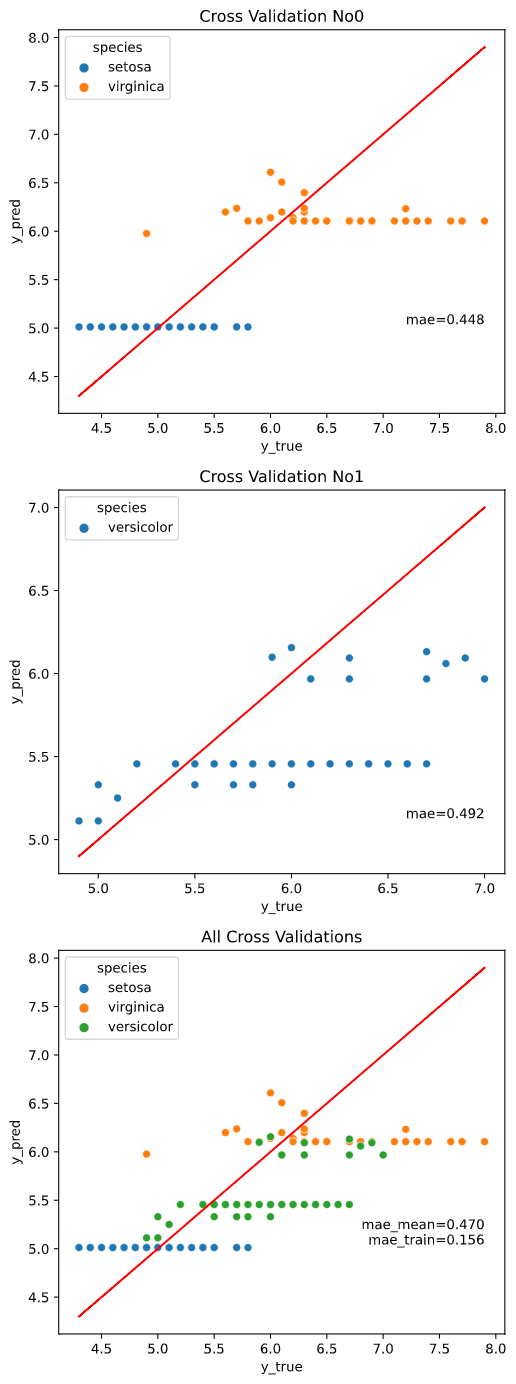
GroupKFold、LeaveOneGroupOutなどのグルーピング系の分割法では、引数cv_groupで指定したグループを分割に使用します。
from sklearn.model_selection import GroupKFold
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=GroupKFold(n_splits=2), hue='species', cv_group='species')
cv_seed
引数"cv"で数値指定した際の、クロスバリデーション指定時の乱数シードを指定します
(デフォルトでは乱数シード42を使用しています)
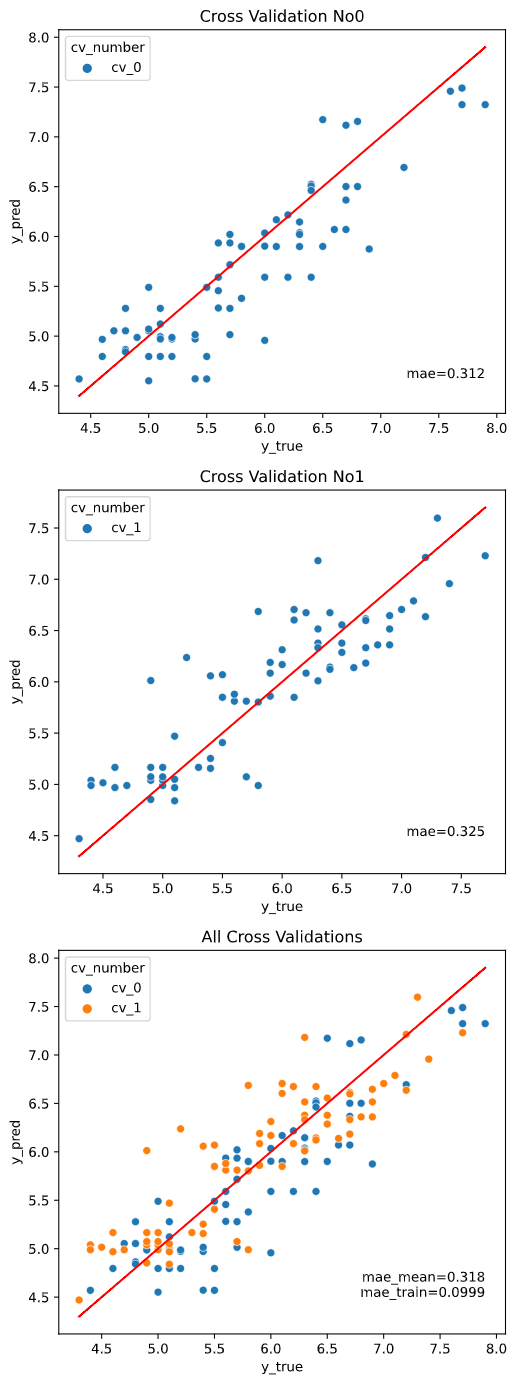
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2, cv_seed=43)
estimator_params
回帰モデルに渡すパラメータをdict指定します。
理想的には、チューニング後のパラメータを渡すのが望ましいです。
(未指定ならば、scikit-learnのデフォルトパラメータを使用します)
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2,
estimator_params={'n_estimators': 108,
'max_features': 2,
'max_depth': 167,
'min_samples_split': 6,
'min_samples_leaf': 5})
fit_params
学習時の"fit"メソッドに渡すパラメータをdict指定します。
XGBoostやLightGBMにおける"early_stoppling_round", "verbose"パラメータなどを想定しています。
from xgboost import XGBRegressor
regplot.regression_pred_true(XGBRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2,
fit_params={'early_stopping_rounds': 20,
'eval_set': [(iris[['petal_width', 'petal_length']].values, iris['sepal_length'].values)],
'verbose': 1})
subplot_kws
画像作成用メソッドmatplotlib.pyplot.subplotsに渡す引数をdict指定できます。
渡せる引数は、こちらやこちらをご参照ください
下の例のように、画像のサイズ('figsize'、デフォルトはCV指定なしなら成り行き、CV指定ありなら画像1枚あたり6×6インチ)を変えたいときなどに便利です
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2, subplot_kws={'figsize': (3, 9)})
scatter_kws
散布図描画用メソッドseaborn.scatterplot()に渡す引数をdict指定できます。
渡せる引数は、こちらをご参照ください
regplot.regression_pred_true(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
scatter_kws={'marker': '^'})
regression_plot_1dの引数
| 引数名 | 必須引数orオプション | 型 | デフォルト値 | 内容 |
|---|---|---|---|---|
| estimator | 必須 | Scikit-learn API | - | 表示対象の回帰モデル |
| x | 必須 | str | - | 説明変数に指定するカラム名 |
| y | 必須 | str | - | 目的変数に指定するカラム名 |
| data | 必須 | pd.DataFrame | - | 入力するデータ(Pandasのデータフレーム) |
| hue | オプション | str | None | 色分けに指定するカラム名 |
| linecolor | オプション | str | 'red' | 予測値=実測値の線の色 |
| rounddigit | オプション | int | 3 | 表示指標の小数丸め桁数 |
| rank_number | オプション | int | None | 誤差上位何番目までを文字表示するか |
| rank_col | オプション | str | None | 誤差上位と一緒に表示するフィールド名 |
| scores | オプション | str | 'mae' | 文字表示する評価指標を指定 |
| cv_stats | オプション | str | 'mean' | クロスバリデーション時に表示する評価指標統計値 |
| cv | オプション | int or sklearn.model_selection.* | None | クロスバリデーション分割法 (Noneのとき学習データから指標算出、int入力時はkFoldで分割) |
| cv_seed | オプション | int | 42 | クロスバリデーションの乱数シード |
| cv_group | オプション | str | None | GroupKFold,LeaveOneGroupOut等のグルーピング対象カラム名 |
| estimator_params | オプション | dict | None | 回帰モデルに渡すパラメータ |
| fit_params | オプション | dict | None | 学習時のパラメータをdict指定 |
| subplot_kws | オプション | dict | None | matplotlib.pyplot.subplotsに渡す引数 |
| scatter_kws | オプション | dict | None | seaborn.scatterplot()に渡す引数 |
| オプション引数の詳細は後述します |
オプション引数を指定しないとき
下記の値が自動入力されます(estimator, x, y, dataは入力必須)
(estimator, x, y, data, hue=None, linecolor='red', rounddigit=3,
scores='mae', cv_stats='mean', cv=None, cv_seed=42,
estimator_params=None, fit_params=None, subplot_kws=None)
・表示例
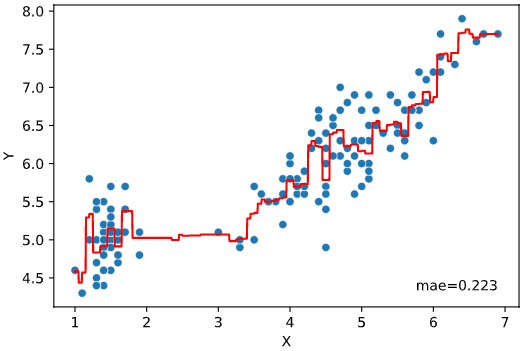
irisデータセット+ランダムフォレスト回帰RandomForestRegressorでの実行例
from sklearn.ensemble import RandomForestRegressor
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris)
実行すると下図のようなグラフ(予測値vs実測値プロット+評価指標)が表示されます
hue
散布図の色分け用カラム名(列名)を指定します。
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
hue='species')
linecolor
予測値=実測値の線の色を指定します。色はmatplotlibに準拠します。
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
linecolor='green')
rounddigit
評価指標の数値表示の丸め桁数を指定します。
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
rounddigit=2)
rank_number
誤差上位何番目までを文字表示するかを指定します。
引数rank_colを指定すれば、一緒に表示する列も指定可能です
(rank_col指定なしのときは、データのインデックスを表示)
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
rank_number=2)
※クロスバリデーション指定時は、全体で順位付けするregression_pred_trueやregression_heat_plotメソッドと異なり、クロスバリデーションごとに順位付けするので注意
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
rank_number=2, cv=2)
rank_col
引数rank_number指定時に、一緒に表示する列を指定します。
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
rank_number=2, rank_col='species')
リスト指定すれば、複数の列を同時表示することもできます
(表示がごちゃごちゃするのであまりオススメはできませんが‥)
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
rank_number=2, rank_col=['species', 'sepal_width'])
scores
文字表示する評価指標を指定します。
指定できる指標一覧は下記です
| 名称 | 指定時の文字列 |
|---|---|
| R2-Score | 'r2' |
| MAE | 'mae' |
| RMSE | 'rmse' |
| RMSLE | 'rmsle' |
| 誤差最大値 | 'max_error' |
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
scores='r2')
None指定すれば、指標表示を消せます
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
scores=None)
リスト指定すれば、複数の指標を同時表示することもできます
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
scores=['rmse','rmsle','max_error'])
クロスバリデーション指定時は、クロスバリデーションごとの図にはクロスバリデーション内テストデータでの指標を、全体結果の図にはcv_stats引数で指定した指標統計値+全体データで学習&指標算出した場合の値 (_trainとついた指標)をプロットします。
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
scores=['mae', 'rmse'], cv_stats='mean', cv=2)
cv_stats
クロスバリデーション指定時に、全体結果の図に表示する指標の統計値を指定します。
'mean'(平均値)、'median'(中央値)、'max'(最大値)、'min'(最小値)が指定できます。
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
scores=['mae', 'rmse'], cv_stats='median', cv=2)
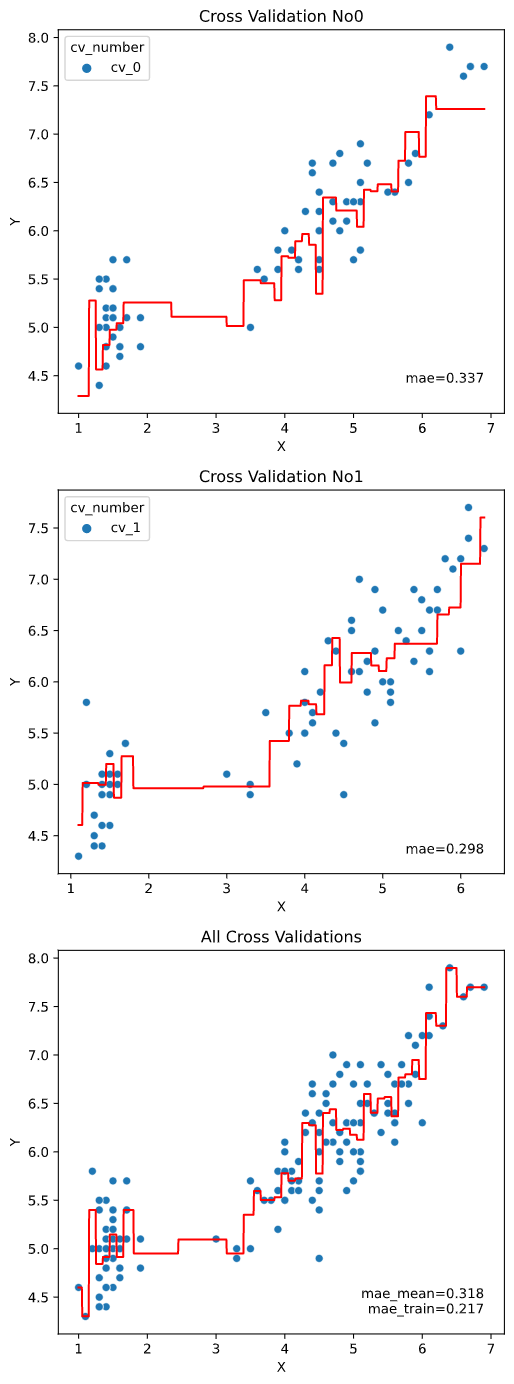
cv
クロスバリデーション分割法を指定します。
数値を指定すれば、指定した数に応じて通常のK-Foldで分割します
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
cv=2)
Scikit-LearnのAPIに対応していれば、ShuffleSplit、GroupKFoldなどの特殊な分割法にも対応しています。
※Leave-One-Outは図の数が激増するのを防ぐため非対応なので、regression_pred_trueメソッドを使用してください
from sklearn.model_selection import ShuffleSplit
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
cv=ShuffleSplit(n_splits=2, test_size=0.1))
GroupKFold、LeaveOneGroupOutなどのグルーピング系の分割法では、引数"hue"で指定したグループを分割に使用します。
from sklearn.model_selection import LeaveOneGroupOut
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
cv=LeaveOneGroupOut(), hue='species')
cv_seed
引数"cv"で数値指定した際の、クロスバリデーション指定時の乱数シードを指定します
(デフォルトでは乱数シード42を使用しています)
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
cv=2, cv_seed=43)
estimator_params
回帰モデルに渡すパラメータをdict指定します。
理想的には、チューニング後のパラメータを渡すのが望ましいです。
(未指定ならば、scikit-learnのデフォルトパラメータを使用します)
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
cv=2,
estimator_params={'n_estimators': 151,
'max_features': 1,
'max_depth': 173,
'min_samples_split': 14,
'min_samples_leaf': 1})
fit_params
学習時の"fit"メソッドに渡すパラメータをdict指定します。
XGBoostやLightGBMにおける"early_stoppling_round", "verbose"パラメータなどを想定しています。
from xgboost import XGBRegressor
regplot.regression_plot_1d(XGBRegressor(), x='petal_length',
y='sepal_length', data=iris,
cv=2,
fit_params={'early_stopping_rounds': 20,
'eval_set': [(iris[['petal_length']].values, iris['sepal_length'].values)],
'verbose': 1})
subplot_kws
画像作成用メソッドmatplotlib.pyplot.subplotsに渡す引数をdict指定できます。
渡せる引数は、こちらやこちらをご参照ください
下の例のように、画像のサイズ('figsize'、デフォルトはCV指定なしなら成り行き、CV指定ありなら画像1枚あたり6×6インチ)を変えたいときなどに便利です
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
cv=2, subplot_kws={'figsize': (3, 9)})
scatter_kws
散布図描画用メソッドseaborn.scatterplot()に渡す引数をdict指定できます。
渡せる引数は、こちらをご参照ください
regplot.regression_plot_1d(RandomForestRegressor(), x='petal_length',
y='sepal_length', data=iris,
scatter_kws={'color': 'green'})
regression_heat_plotの引数
| 引数名 | 必須引数orオプション | 型 | デフォルト値 | 内容 |
|---|---|---|---|---|
| estimator | 必須 | Scikit-learn API | - | 表示対象の回帰モデル |
| x | 必須 | List[str] | - | 説明変数に指定するカラム名のリスト |
| y | 必須 | str | - | 目的変数に指定するカラム名 |
| data | 必須 | pd.DataFrame | - | 入力するデータ(Pandasのデータフレーム) |
| x_heat | オプション | List[str] | None | 説明変数のうちヒートマップ表示対象のカラム名 |
| scatter_hue | オプション | str | None | 散布図色分け指定カラム名(plot_scatter='hue'時のみ有効) |
| pair_sigmarange | オプション | float | 1.5 | ヒートマップ非使用変数の分割範囲 |
| pair_sigmainterval | オプション | float | 0.5 | ヒートマップ非使用変数の1枚あたり表示範囲 |
| heat_extendsigma | オプション | float | 0.5 | ヒートマップ縦軸横軸の表示拡張範囲 |
| heat_division | オプション | int | 30 | ヒートマップ縦軸横軸の解像度 |
| value_extendsigma | オプション | float | 0.5 | ヒートマップの色分け最大最小値拡張範囲 |
| plot_scatter | オプション | str | 'true' | 散布図の描画種類 |
| rounddigit_rank | オプション | int | 3 | 誤差上位表示の小数丸め桁数 |
| rounddigit_x1 | オプション | int | 2 | ヒートマップ横軸の小数丸め桁数 |
| rounddigit_x2 | オプション | int | 2 | ヒートマップ縦軸の小数丸め桁数 |
| rounddigit_x3 | オプション | int | 2 | ヒートマップ非使用軸の小数丸め桁数 |
| rank_number | オプション | int | None | 誤差上位何番目までを文字表示するか |
| rank_col | オプション | str | None | 誤差上位と一緒に表示するフィールド名 |
| cv | オプション | int or sklearn.model_selection.* | None | クロスバリデーション分割法 (Noneのとき学習データから指標算出、int入力時はkFoldで分割) |
| cv_seed | オプション | int | 42 | クロスバリデーションの乱数シード |
| cv_group | オプション | str | None | GroupKFold,LeaveOneGroupOut等のグルーピング対象カラム名 |
| display_cv_indices | オプション | int | 0 | 表示対象のクロスバリデーション番号 |
| estimator_params | オプション | dict | None | 回帰モデルに渡すパラメータ |
| fit_params | オプション | dict | None | 学習時のパラメータをdict指定 |
| subplot_kws | オプション | dict | None | matplotlib.pyplot.subplotsに渡す引数 |
| heat_kws | オプション | dict | None | ヒートマップ用のseaborn.heatmap()に渡す引数 |
| scatter_kws | オプション | dict | None | 散布図用のmatplotlib.pyplot.scatter()に渡す引数 |
| オプション引数の詳細は後述します |
オプション引数を指定しないとき
下記の値が自動入力されます(estimator, x, y, dataは入力必須)
(estimator, x, y, data, x_heat = None, scatter_hue=None,
pair_sigmarange = 1.5, pair_sigmainterval = 0.5,
heat_extendsigma = 0.5, value_extendsigma = 0.5, heat_division = 30,
plot_scatter = 'true', rank_number=None, rank_col=None, rounddigit_rank=3,
rounddigit_x1=2, rounddigit_x2=2, rounddigit_x3=2,
cv=None, cv_seed=42, display_cv_indices = 0,
estimator_params=None, fit_params=None, subplot_kws=None, heat_kws=None, scatter_kws=None)
・表示例
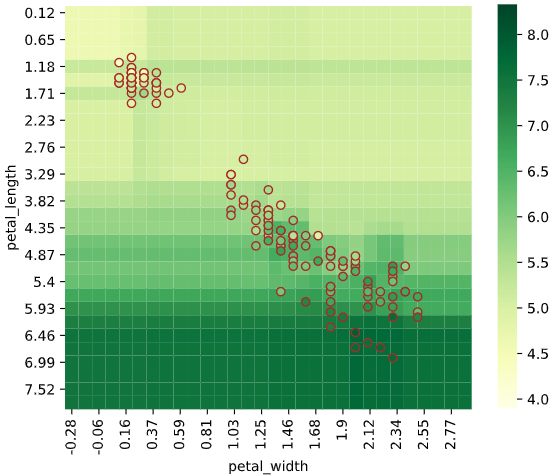
irisデータセット+ランダムフォレスト回帰RandomForestRegressorでの実行例
from sklearn.ensemble import RandomForestRegressor
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris)
実行すると下図のようなグラフ(予測値vs実測値プロット+評価指標)が表示されます
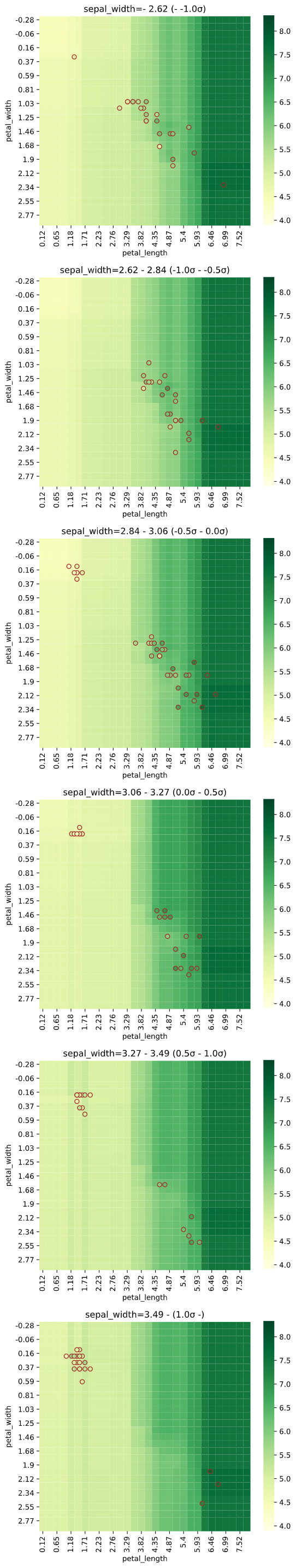
x_heat
3説明変数以上のときのみ有効なのでご注意ください
説明変数のうち、ヒートマップの横軸と縦軸のカラム名を指定します。
指定しなかった説明変数を使い、
「pair_sigmarange引数 × 標準偏差」の範囲内で、
「pair_sigmainterval引数 × 標準偏差」ごとに一定間隔で図を分割します。
※Noneを指定すると、引数"x"で指定した説明変数の前から2個を自動指定します。
regplot.regression_heat_plot(RandomForestRegressor(), x=['sepal_width', 'petal_width', 'petal_length'],
y='sepal_length', data=iris,
x_heat=['petal_length', 'petal_width'],
pair_sigmarange = 1.0, pair_sigmainterval = 0.5)
コツとしては、ヒートマップで目的変数の変化が追いやすいよう、
目的変数に対する影響度が大きい説明変数を指定することです。
(影響度の大きさは、後述の「特徴量重要度」などで算出可能)
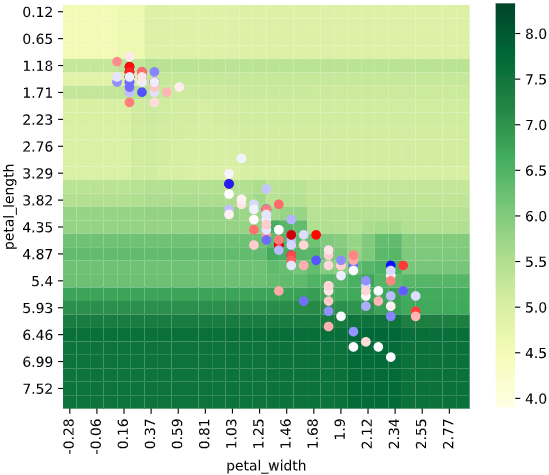
scatter_hue
散布図の色分け用カラム名(列名)を指定します。引数plot_scatter='hue'も同時指定する必要があることにご注意ください
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
scatter_hue='species', plot_scatter='hue')
pair_sigmarange
3説明変数以上のときのみ有効なのでご注意ください
ヒートマップ非使用変数(x_heatで未指定の説明変数)での図の分割範囲を指定します。
「pair_sigmarange引数 × 標準偏差」の範囲内で、
「pair_sigmainterval引数 × 標準偏差」ごとに一定間隔で図を分割します。
regplot.regression_heat_plot(RandomForestRegressor(), x=['sepal_width', 'petal_width', 'petal_length'],
y='sepal_length', data=iris,
x_heat=['petal_length', 'petal_width'],
pair_sigmarange = 0.5, pair_sigmainterval = 0.5)
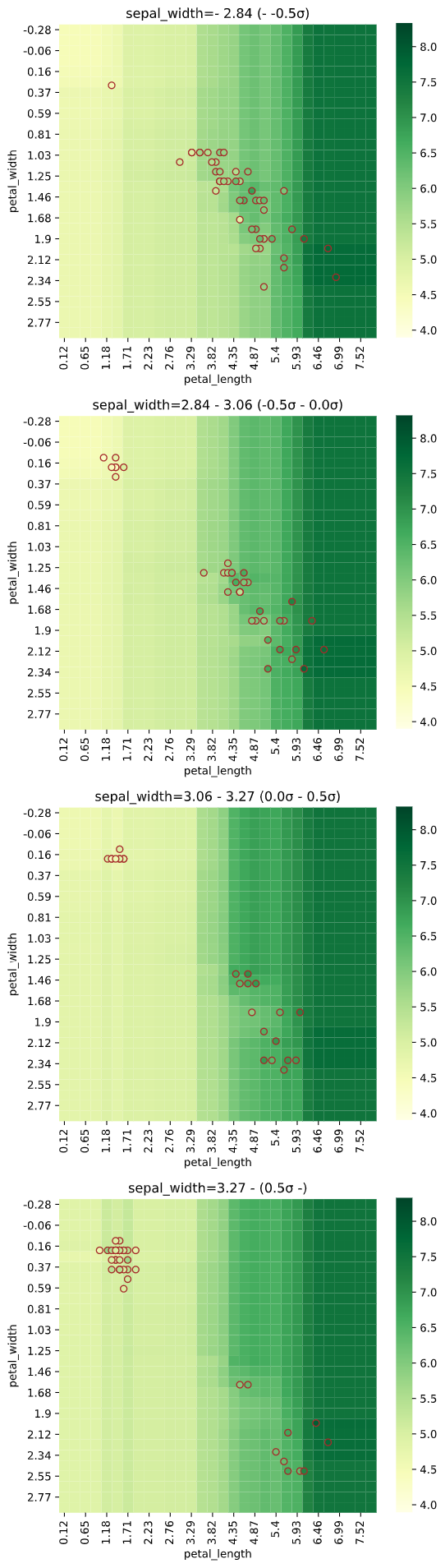
なお、表示されるヒートマップにおけるヒートマップ非使用変数の値は、
・pair_sigmarange範囲内の図:pair_sigmaintervalの中央値
・pair_sigmarange範囲外の図(最初の図):-pair_sigmarange - pair_sigmainterval/2
・pair_sigmarange範囲外の図(最後の図):pair_sigmarange + pair_sigmainterval/2
を使用します
pair_sigmainterval
3説明変数以上のときのみ有効なのでご注意ください
ヒートマップ非使用変数(x_heatで未指定の説明変数)での図の分割範囲を指定します。
「pair_sigmarange引数 × 標準偏差」の範囲内で、
「pair_sigmainterval引数 × 標準偏差」ごとに一定間隔で図を分割します。
regplot.regression_heat_plot(RandomForestRegressor(), x=['sepal_width', 'petal_width', 'petal_length'],
y='sepal_length', data=iris,
x_heat=['petal_length', 'petal_width'],
pair_sigmarange = 0.5, pair_sigmainterval = 0.25)
heat_extendsigma
ヒートマップ縦軸横軸の表示拡張範囲を指定します。
散布図データの最大最小値に対し、「heat_extendsigma × 標準偏差」だけ縦軸横軸範囲を拡張します
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
heat_extendsigma=3.0)
heat_division
ヒートマップ縦軸横軸の解像度を指定します。
ヒートマップ全体を、ここで指定した数のグリッド数で分割します
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
heat_division=10)
plot_scatter
散布図の色分け方法を選択します。下表から選択できます
| plot_scatterの指定値 | 散布図の色分け方法 |
|---|---|
| 'error' | 誤差(予測値-実測値)で色分け |
| 'true' | 実測値で色分け |
| 'hue' | 引数"scatter_hue"指定列で色分け |
| None | 散布図表示なし |
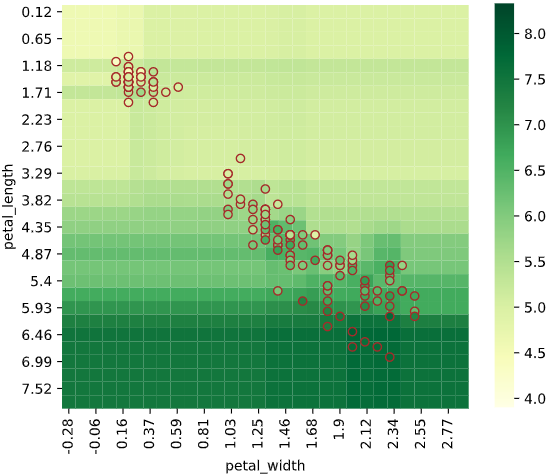
plot_scatter='error'のとき
誤差(予測値-実測値)で散布図を色分けします
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
plot_scatter='error')
plot_scatter='true'のとき
実測値で散布図を色分けします(これがデフォルト設定)
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
plot_scatter='error')
plot_scatter='hue'のとき
引数scatter_hueで指定した列の値で散布図を色分けします。
引数scatter_hueも同時指定する必要があることにご注意ください
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
plot_scatter='hue', scatter_hue='species')
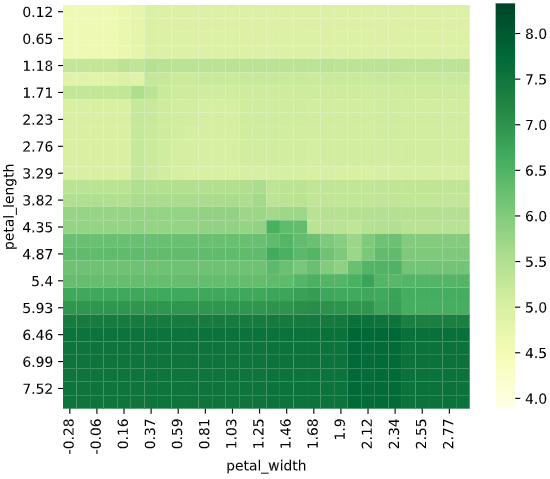
plot_scatter=Noneのとき
散布図をプロットしません
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
plot_scatter=None)
rounddigit_rank
誤差上位表示の小数丸め桁数を指定します。
引数rank_numberも同時指定する必要があることにご注意ください
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
rounddigit_rank=5, rank_number=1)
rounddigit_x1
ヒートマップ横軸の小数丸め桁数を指定します
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
rounddigit_x1=4)
rounddigit_x2
ヒートマップ縦軸の小数丸め桁数を指定します
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
rounddigit_x2=4)
rounddigit_x3
3説明変数以上のときのみ有効なのでご注意ください
ヒートマップ非表示変数の小数丸め桁数の小数丸め桁数を指定します
(各ヒートマップの上に表示される非表示変数の範囲表示の桁数です)
regplot.regression_heat_plot(RandomForestRegressor(), x=['sepal_width', 'petal_width', 'petal_length'],
y='sepal_length', data=iris,
x_heat=['petal_length', 'petal_width'],
pair_sigmarange = 0.5, pair_sigmainterval = 0.5,
rounddigit_x3=4)
rank_number
誤差上位何番目までを文字表示するかを指定します。
引数rank_colを指定すれば、一緒に表示する列も指定可能です
(rank_col指定なしのときは、データのインデックスを表示)
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
rank_number=4)
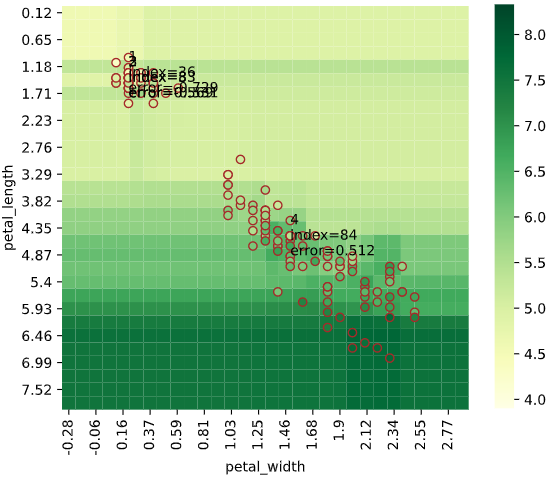
見ての通り表示が重なりやすいので、引数subplot_kwsと併用して図を大きくするのがおすすめです。
rank_col
引数rank_number指定時に、一緒に表示する列を指定します。
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
rank_number=1, rank_col='species')
cv
クロスバリデーション分割法を指定します。
数値を指定すれば、指定した数に応じて通常のK-Foldで分割します。
regression_pred_trueやregression_plot_1dメソッドと異なり、デフォルトではクロスバリデーションの最初のセクション(CV_Number=0)の結果のみがプロットされます。
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2)
全てのセクションの結果をプロットしたい場合、引数"display_cv_indices"でCV_Numberをリスト指定します
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2, display_cv_indices=[0, 1])
Scikit-LearnのAPIに対応していれば、ShuffleSplit、GroupKFoldなどの特殊な分割法にも対応しています。
※Leave-One-Outは図の数が激増するのを防ぐため非対応なので、regression_pred_trueメソッドを使用してください
from sklearn.model_selection import ShuffleSplit
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=ShuffleSplit(n_splits=2, test_size=0.1))
GroupKFold、LeaveOneGroupOutなどのグルーピング系の分割法では、引数"scatter_hue"で指定したグループを分割に使用します。
from sklearn.model_selection import LeaveOneGroupOut
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=LeaveOneGroupOut(), scatter_hue='species',
display_cv_indices=[0, 1, 2], plot_scatter='hue')
引数scatter_hue指定時は本来であればplot_scatter='hue'とする必要がありますが、GroupKFold、LeaveOneGroupOutなどのグルーピング系の分割法指定時のみ、plot_scatter='hue'以外での色分け('true', 'error'など)も可能となっています
from sklearn.model_selection import LeaveOneGroupOut
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=LeaveOneGroupOut(), scatter_hue='species',
display_cv_indices=[0, 1, 2], plot_scatter='true')
cv_seed
引数"cv"で数値指定した際の、クロスバリデーション指定時の乱数シードを指定します
(デフォルトでは乱数シード42を使用しています)
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2, cv_seed=43)
display_cv_indices
クロスバリデーション分割時に、ヒートマップ表示するクロスバリデーション番号を指定します。
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2, display_cv_indices=1)
前述のように、リスト指定で複数のクロスバリデーション番号でのヒートマップを同時表示させることも可能です
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2, display_cv_indices=[0, 1])
estimator_params
回帰モデルに渡すパラメータをdict指定します。
理想的には、チューニング後のパラメータを渡すのが望ましいです。
(未指定ならば、scikit-learnのデフォルトパラメータを使用します)
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2,
estimator_params={'n_estimators': 108,
'max_features': 2,
'max_depth': 167,
'min_samples_split': 6,
'min_samples_leaf': 5})
fit_params
学習時の"fit"メソッドに渡すパラメータをdict指定します。
XGBoostやLightGBMにおける"early_stoppling_round", "verbose"パラメータなどを想定しています。
from xgboost import XGBRegressor
regplot.regression_heat_plot(XGBRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2,
fit_params={'early_stopping_rounds': 20,
'eval_set': [(iris[['petal_width', 'petal_length']].values, iris['sepal_length'].values)],
'verbose': 1})
subplot_kws
画像作成用メソッドmatplotlib.pyplot.subplotsに渡す引数をdict指定できます。
渡せる引数は、こちらやこちらをご参照ください
下の例のように、画像のサイズ('figsize'、デフォルトは画像1枚あたり6×5インチ)を変えたいときなどに便利です
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
cv=2, subplot_kws={'figsize': (3, 3)})
heat_kws
ヒートマップ描画用メソッドseaborn.heatmap()に渡す引数をdict指定できます。
渡せる引数は、こちらやこちらをご参照ください
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
heat_kws={'cmap': 'hsv'})
scatter_kws
散布図描画用メソッドmatplotlib.pyplot.scatter()に渡す引数をdict指定できます。
渡せる引数は、こちらをご参照ください
regplot.regression_heat_plot(RandomForestRegressor(), x=['petal_width', 'petal_length'],
y='sepal_length', data=iris,
scatter_kws={'marker': 'v'})
補足:パイプライン処理への対応
標準化等の前処理と組み合わせた回帰モデルを構築したいとき、Scikit-Learnではパイプライン処理がよく使われますが、本ツールの入力にもパイプライン処理が適用できます
from sklearn.svm import SVR
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
pipe = Pipeline([("scaler", StandardScaler()), ("svr", SVR())]) # 標準化+SVRパイプライン
regplot.regression_heat_plot(pipe, x=['petal_width', 'petal_length'],
y='sepal_length', data=iris)
余談:その他の便利な回帰可視化テクニック
他のライブラリで簡単に実装できるため本ツールでは触れていませんが、回帰の可視化には、以下のようなテクニックもよく使われています。
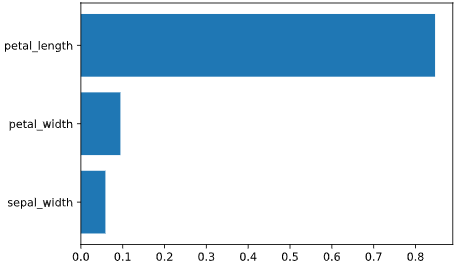
特徴量重要度(Feature Importance)
Random ForestやXGBoost、LightGBMなどの決定木系の回帰アルゴリズムには、
目的変数の予測に対する、各説明変数の寄与度を定量的にランク付けした、
**「特徴量重要度 (feature importance)」**と呼ばれる指標が存在します。
from xgboost import XGBRegressor
import matplotlib.pyplot as plt
# モデルの学習
xgbr = XGBRegressor()
features = ['sepal_width', 'petal_width', 'petal_length']
X = iris[features].values
y = iris['sepal_length'].values
xgbr.fit(X, y)
# 特徴量重要度の取得と可視化
importances = list(xgbr.feature_importances_)
plt.barh(features, importances)
この指標は**「どの変数が予測に寄与したか」**という、回帰結果を解釈する上で重要な知見を与えてくれます
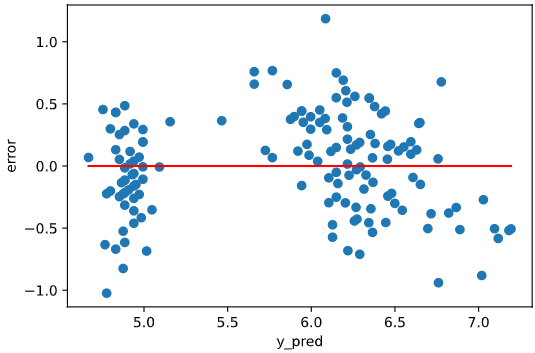
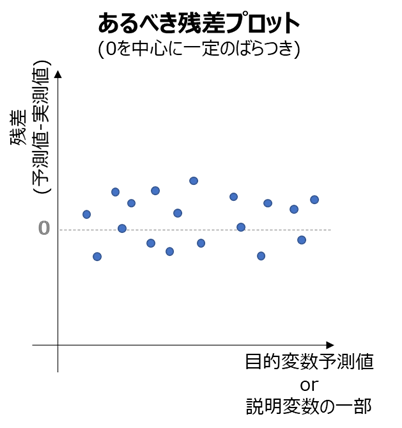
残差プロット
・横軸に目的変数の予測値あるいは説明変数の一部
・縦軸に目的変数の誤差(予測値 - 実測値)
をとった散布図を、残差プロットと呼びます。
残差プロットは誤差が目的変数、または説明変数によらず一定であることを評価します。
from sklearn.linear_model import LinearRegression
import matplotlib.pyplot as plt
import numpy as np
# モデルの学習
lr = LinearRegression()
features = ['petal_width', 'petal_length']
X = iris[features].values
y = iris['sepal_length'].values
lr.fit(X, y)
# 残差プロット(横軸は目的変数予測値)
y_pred = lr.predict(X)
error = y_pred - y
plt.scatter(y_pred, error)
plt.xlabel('y_pred')
plt.ylabel('error')
# 残差=0の補助線を引く
plt.plot([np.amin(y_pred), np.amax(y_pred)], [0, 0], "red")
多くの機械学習による回帰モデルは、誤差の平均および標準偏差が目的変数や説明変数によらず一定であることを前提としているため、
残差プロットが下図のあるべき姿となっていない場合、モデルの適合性不足が疑われ、何らかの改善策を検討する必要が生じます。
代表的な適合性不足の例としては、
・誤差が右肩上がり(右肩下がり)となっているとき(誤差平均の拡大)
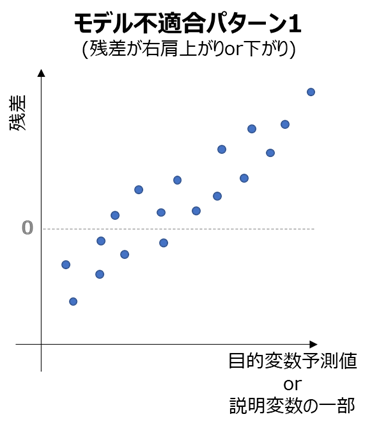
→現状のモデルはデータの傾向にフィットしきれていない(未学習状態)ため、より複雑なモデルを採用
(例:線形回帰 → 多項式回帰、あるいはRBFカーネルSVRなど非線形な回帰手法を採用)
・誤差が末広がりとなっているとき(ばらつきの拡大)
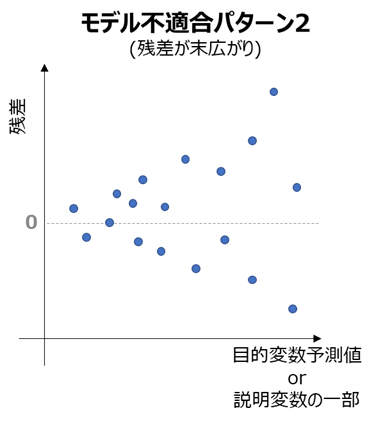
→分散が説明変数に依存して大きくなるため、依存する説明変数を特定したのちモデルに組み込む
(例:線形回帰 → 分散が説明変数に比例するベイズ回帰モデル)
詳細はこちらの資料が分かりやすいです。
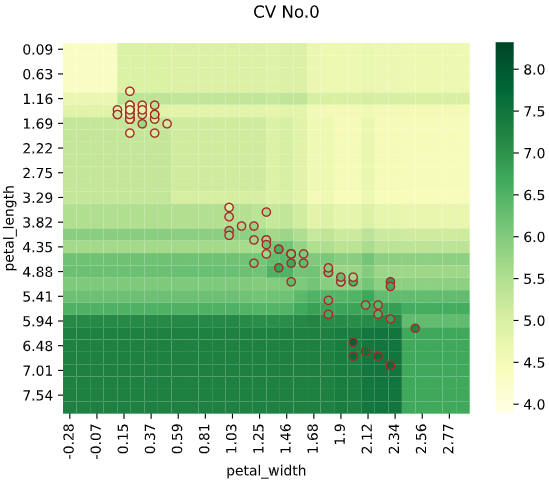
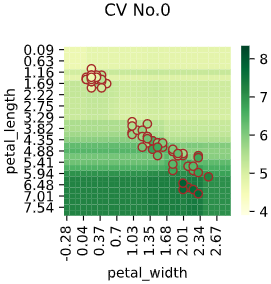
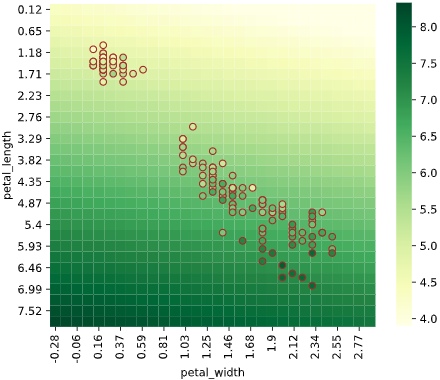
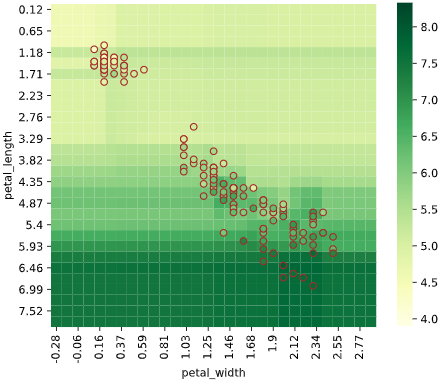
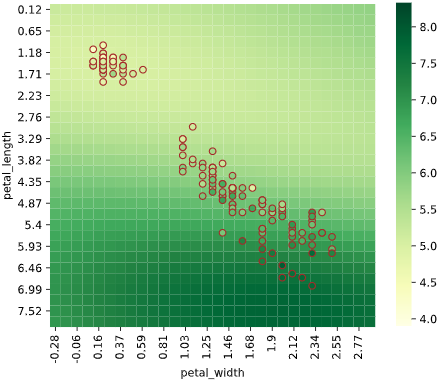
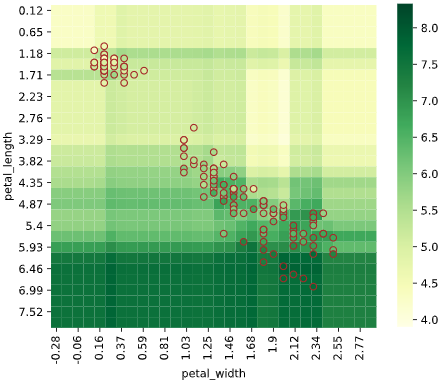
余談:各機械学習アルゴリズムの回帰ヒートマップ可視化
ヒートマップで可視化すると、各アルゴリズムの特性がわかり面白いと感じたので、
Irisデータセット(説明変数['petal_width','petal_length']、目的変数'sepal_length')での可視化例を下記します。
※パラメータチューニングしていないので、各手法の最大性能を出せているわけではありません。あくまで傾向を見るための可視化です
線形回帰
リッジ回帰(α=1)
リッジ回帰(α=100)
ほぼ縦方向(petal_length方向)の傾きのみ
→正則化によりpetal_widthの影響がほぼ消えた
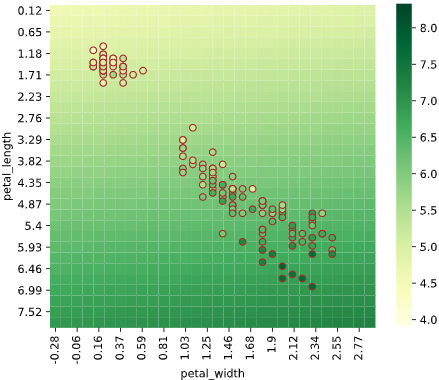
ランダムフォレスト回帰
サポートベクター回帰(RBFカーネル)
XGBoost
LightGBM
関連記事
本記事は、データの可視化をテーマとしたシリーズものです。よければ下記の記事もどうぞ
・分類の可視化
・散布図と相関係数でデータを一括確認

・分布の種類を判断