概要
Kaggleの血液検査データセットを使ってデータ分析をしてみた。
いろいろ試してみた結果、量が多くなったので分割します。
今回はその② (全5回)
他の回はこちらから
①~モデルの性能比較をしてみた~
③~アンサンブル学習をしてみた~
④~外れ値の取り扱い~
⑤~不均衡データの取り扱い~
使用したデータセット:Patient Treatment Classification (Electronic Health Record Dataset)
インドネシアの病院で集められた血液検査の結果から、患者に治療が必要かどうかを判定する
モデル
今回使用したモデルは以下の6種類
- XGBoost
- ニューラルネットワーク
- ランダムフォレスト
- ロジスティック回帰
- 決定木
- k-近傍法
データ確認
データは血液検査の結果。
データの検査方法が分からなかったため、正常値は2016年度国立がん研究センターのデータをお借りしました。(※ 正常値は測定方法などにより若干のばらつきあり)
| 列No | 検査値 | 和訳 | 正常値 | 高いと | 低いと |
|---|---|---|---|---|---|
| 1 | HAEMATOCRIT | ヘマトクリット | 男性:40.7~50.1 %, 女性:35.1~44.4 % |
多血症など | 貧血など |
| 2 | HAEMOGLOBINS | ヘモグロビン | 男性:13.7~16.8 g/dl, 女性:11.6~14.8 g/dl |
多血症など | 貧血など |
| 3 | ERYTHROCYTE | 赤血球 | 男性:4.35~5.55 x 106 /μL, 女性:3.86~4.92 x 106 /μL |
多血症など | 貧血など |
| 4 | LEUCOCYTE | 白血球 | 3.3~8.6 x 103/μL | 感染症・白血病など | 一部感染症・膠原病・貧血など |
| 5 | THROMBOCYTE | 血小板 | 158~348 x 103/μL | 血小板血症・白血病・多血症など | 貧血・紫斑病など |
| 6 | MCH | 平均赤血球ヘモグロビン量 | 27.5~33.2 pg | 巨赤芽球性貧血など | 鉄欠乏性貧血など |
| 7 | MCHC | 平均赤血球ヘモグロビン濃度 | 31.7~35.3 g/dL | 脱水・多血症など | 貧血など |
| 8 | MCV | 平均赤血球容積 | 83.6~98.2 fL | 巨赤芽球性貧血など | 鉄欠乏性貧血など |
| 9 | AGE | 年齢 | ― | ― | ― |
| 10 | SEX | 性別 | ― | ― | ― |
| 11 | SOURCE | 治療が必要か | out: 治療不要, in: 治療が必要 | ― | ― |
前処理は前回と同じなので割愛
特徴選択
モデルの予測精度の改善を目的として、訓練データから予測により強い関連がある特徴を選択すること。
XGBoost
特徴量の重要度
fscore = GBDT_model.get_score(importance_type="total_gain")
# 重要な特徴量を確認
print(sorted(fscore))
fscore = sorted([(k, v) for k, v in fscore.items()], key=lambda tpl: tpl[1], reverse=True)
# 特徴量の重要度を確認
print('---------------')
for k, v in fscore:
print(f'{k}: {v:.3f}')
>>
['f0', 'f1', 'f10', 'f12', 'f13', 'f15', 'f16', 'f17', 'f19', 'f2', 'f22', 'f25', 'f28', 'f3', 'f31', 'f4', 'f5', 'f6', 'f7', 'f8', 'f9']xgboost importance: [('f4', 1295.02294921875), ('f8', 595.5386962890625), ('f3', 580.4332885742188), ('f0', 494.5536193847656), ('f6', 301.5970458984375), ('f7', 256.95867919921875), ('f5', 252.56040954589844), ('f2', 211.041015625), ('f12', 209.5330047607422), ('f1', 188.264404296875), ('f15', 73.5942611694336), ('f9', 61.07915496826172), ('f19', 39.585174560546875), ('f16', 20.44217872619629), ('f17', 18.052425384521484), ('f10', 8.585515975952148), ('f31', 6.885251045227051), ('f13', 4.792502403259277), ('f28', 2.6973867416381836), ('f22', 2.52486515045166), ('f25', 2.124303102493286)]
| 特徴量 | 重要度 |
|---|---|
| f4: | 1295.023 |
| f8: | 595.539 |
| f3: | 580.433 |
| f0: | 494.554 |
| f6: | 301.597 |
| f7: | 256.959 |
| f5: | 252.560 |
| f2: | 211.041 |
| f12: | 209.533 |
| f1: | 188.264 |
| f15: | 73.594 |
| f9: | 61.079 |
| f19: | 39.585 |
| f16: | 20.442 |
| f17: | 18.052 |
| f10: | 8.586 |
| f31: | 6.885 |
| f13: | 4.793 |
| f28: | 2.697 |
| f22: | 2.525 |
| f25: | 2.124 |
ニューラルネットワーク
perm = permutation_importance(my_model, train_X, train_y, n_repeats=5, random_state=42)
perm_imp_df = pd.DataFrame({"importances_mean":perm["importances_mean"], "importances_std":perm["importances_std"]}, index=df_NN.columns)
perm_imp_df_sort = perm_imp_df.sort_values('importances_mean',ascending=False)
print(perm_imp_df_sort)
| 特徴量 | importances_mean | importances_std |
|---|---|---|
| THROMBOCYTE | 0.135567 | 0.006395 |
| AGE | 0.019758 | 0.002807 |
| LEUCOCYTE | 0.012333 | 0.000917 |
| HAEMATOCRIT | 0.011146 | 0.000779 |
| HAEMOGLOBINS | 0.001955 | 0.000061 |
| HAEMOGLOBINS_AB_2 | 0.001517 | 0.000115 |
| HAEMATOCRIT_AB_2 | 0.001465 | 0.000120 |
| HAEMOGLOBINS_AB_0 | 0.001048 | 0.000087 |
| ERYTHROCYTE_AB_2 | 0.000950 | 0.000069 |
| HAEMATOCRIT_AB_0 | 0.000881 | 0.000089 |
| ERYTHROCYTE_AB_1 | 0.000521 | 0.000074 |
| LEUCOCYTE_AB_0 | 0.000521 | 0.000099 |
| LEUCOCYTE_AB_1 | 0.000513 | 0.000167 |
| THROMBOCYTE_AB_0 | 0.000483 | 0.000047 |
| THROMBOCYTE_AB_2 | 0.000431 | 0.000036 |
| MCV_AB_2 | 0.000267 | 0.000116 |
| MCH_AB_2 | 0.000242 | 0.000045 |
| ERYTHROCYTE | 0.000199 | 0.000023 |
| ERYTHROCYTE_AB_0 | 0.000192 | 0.000008 |
| MCV_AB_0 | 0.000110 | 0.000061 |
| MCV | 0.000095 | 0.000215 |
| MCH_AB_0 | 0.000083 | 0.000013 |
| HAEMATOCRIT_AB_1 | 0.000082 | 0.000018 |
| MCHC_AB_2 | 0.000082 | 0.000014 |
| HAEMOGLOBINS_AB_1 | 0.000080 | 0.000012 |
| SEX | 0.000059 | 0.000010 |
| MCH_AB_1 | 0.000049 | 0.000006 |
| MCHC_AB_1 | 0.000039 | 0.000017 |
| MCHC_AB_0 | 0.000036 | 0.000018 |
| THROMBOCYTE_AB_1 | 0.000036 | 0.000031 |
| MCV_AB_1 | 0.000002 | 0.000002 |
| LEUCOCYTE_AB_2 | -0.000008 | 0.000007 |
| MCHC | -0.000074 | 0.000066 |
| MCH | -0.000178 | 0.000066 |
ランダムフォレスト
fi = RF_model.feature_importances_
idx = np.argsort(fi)[::-1]
top_cols, top_importances = df_RF.columns.values[idx], fi[idx]
print("Random Forest importance")
for i in range(len(idx)):
print(i+1, top_cols[i], top_importances[i])
| No | 特徴量 | Random Forest importance |
|---|---|---|
| 1 | THROMBOCYTE | 0.1349386493530065 |
| 2 | LEUCOCYTE | 0.09006417883629521 |
| 3 | ERYTHROCYTE | 0.08848098257028328 |
| 4 | HAEMATOCRIT | 0.08807169432235014 |
| 5 | AGE | 0.0879590158983752 |
| 6 | HAEMOGLOBINS | 0.0762588034928355 |
| 7 | MCV | 0.07482612599756178 |
| 8 | MCH | 0.07138738423084884 |
| 9 | MCHC | 0.0712196204046919 |
| 10 | THROMBOCYTE_AB_2 | 0.04676987232876439 |
| 11 | THROMBOCYTE_AB_0 | 0.01822047906125719 |
| 12 | LEUCOCYTE_AB_0 | 0.013115095527732596 |
| 13 | HAEMOGLOBINS_AB_2 | 0.011861561944217464 |
| 14 | HAEMATOCRIT_AB_0 | 0.01142416947739328 |
| 15 | SEX | 0.011310603125458438 |
| 16 | HAEMATOCRIT_AB_2 | 0.011114960839353765 |
| 17 | ERYTHROCYTE_AB_2 | 0.010879290833220614 |
| 18 | LEUCOCYTE_AB_1 | 0.010015323599044874 |
| 19 | THROMBOCYTE_AB_1 | 0.009901963372281896 |
| 20 | HAEMOGLOBINS_AB_0 | 0.008174127990087232 |
| 21 | MCV_AB_0 | 0.006574645314590906 |
| 22 | ERYTHROCYTE_AB_0 | 0.006542399581846111 |
| 23 | MCV_AB_2 | 0.006032912464580412 |
| 24 | MCH_AB_0 | 0.005133391334790517 |
| 25 | MCHC_AB_0 | 0.004864091535379257 |
| 26 | MCH_AB_2 | 0.004840337089315016 |
| 27 | ERYTHROCYTE_AB_1 | 0.004816498290831876 |
| 28 | LEUCOCYTE_AB_2 | 0.0037256198100022253 |
| 29 | MCHC_AB_2 | 0.003671069260842609 |
| 30 | MCHC_AB_1 | 0.002895116744217623 |
| 31 | HAEMATOCRIT_AB_1 | 0.001525407645089648 |
| 32 | MCV_AB_1 | 0.0012604666189813451 |
| 33 | HAEMOGLOBINS_AB_1 | 0.0011692463589740942 |
| 34 | MCH_AB_1 | 0.0009548947454983618 |
ロジスティック回帰
perm = permutation_importance(LR_model, train_X, train_y, n_repeats=10, random_state=42)
perm_imp_df = pd.DataFrame({"importances_mean":perm["importances_mean"], "importances_std":perm["importances_std"]}, index=df_LR.columns)
perm_imp_df_sort = perm_imp_df.sort_values('importances_mean',ascending=False)
print(perm_imp_df_sort)
| 特徴量 | importances_mean | importances_std |
|---|---|---|
| THROMBOCYTE | 4.316751e-02 | 0.004520 |
| THROMBOCYTE_AB_2 | 2.553526e-02 | 0.003363 |
| HAEMOGLOBINS | 1.571159e-02 | 0.003610 |
| MCH | 1.568010e-02 | 0.003268 |
| MCV | 1.385390e-02 | 0.003027 |
| LEUCOCYTE | 1.344458e-02 | 0.002600 |
| THROMBOCYTE_AB_0 | 1.102015e-02 | 0.002716 |
| MCH_AB_0 | 7.273300e-03 | 0.001513 |
| ERYTHROCYTE_AB_2 | 6.989924e-03 | 0.002107 |
| AGE | 6.045340e-03 | 0.001950 |
| ERYTHROCYTE_AB_1 | 3.715365e-03 | 0.001448 |
| HAEMATOCRIT_AB_2 | 2.550378e-03 | 0.001466 |
| MCH_AB_2 | 2.361461e-03 | 0.001906 |
| HAEMATOCRIT_AB_0 | 2.015113e-03 | 0.001322 |
| MCHC | 1.763224e-03 | 0.001359 |
| ERYTHROCYTE | 1.731738e-03 | 0.001625 |
| MCV_AB_2 | 1.605793e-03 | 0.001231 |
| MCHC_AB_0 | 1.353904e-03 | 0.000631 |
| HAEMATOCRIT | 1.322418e-03 | 0.001134 |
| MCV_AB_0 | 1.227960e-03 | 0.001215 |
| MCHC_AB_2 | 1.196474e-03 | 0.000542 |
| MCHC_AB_1 | 7.241814e-04 | 0.000374 |
| LEUCOCYTE_AB_1 | 6.926952e-04 | 0.002330 |
| HAEMOGLOBINS_AB_0 | 6.612091e-04 | 0.001361 |
| THROMBOCYTE_AB_1 | 2.518892e-04 | 0.000642 |
| HAEMOGLOBINS_AB_1 | 2.204030e-04 | 0.000834 |
| MCH_AB_1 | 1.889169e-04 | 0.000734 |
| HAEMOGLOBINS_AB_2 | 1.574307e-04 | 0.000254 |
| LEUCOCYTE_AB_0 | 1.574307e-04 | 0.002210 |
| MCV_AB_1 | -2.220446e-17 | 0.000199 |
| ERYTHROCYTE_AB_0 | -1.574307e-04 | 0.001805 |
| LEUCOCYTE_AB_2 | -1.889169e-04 | 0.000427 |
| SEX | -2.833753e-04 | 0.000850 |
| HAEMATOCRIT_AB_1 | -4.722922e-04 | 0.000568 |
決定木
fi = DT_model.feature_importances_
idx = np.argsort(fi)[::-1]
top_cols, top_importances = df_DT.columns.values[idx], fi[idx]
print("Decision Tree importance")
for i in range(len(idx)):
print(i+1, top_cols[i], top_importances[i])
| No | 特徴量 | Decision Tree importance |
|---|---|---|
| 1 | THROMBOCYTE | 0.22911978104533764 |
| 2 | LEUCOCYTE | 0.13473908172051005 |
| 3 | AGE | 0.11803281117070838 |
| 4 | HAEMATOCRIT | 0.07404614525647187 |
| 5 | HAEMOGLOBINS | 0.0681531118213668 |
| 6 | MCHC | 0.06788531910725225 |
| 7 | MCV | 0.06745074029423735 |
| 8 | HAEMATOCRIT_AB_2 | 0.06710412920537928 |
| 9 | ERYTHROCYTE | 0.06220313291944446 |
| 10 | MCH | 0.05152663737731115 |
| 11 | SEX | 0.009743815948349414 |
| 12 | LEUCOCYTE_AB_0 | 0.009411776751157556 |
| 13 | MCV_AB_0 | 0.007895357216158095 |
| 14 | MCHC_AB_0 | 0.0036480919311022973 |
| 15 | THROMBOCYTE_AB_0 | 0.003594472724711572 |
| 16 | HAEMOGLOBINS_AB_0 | 0.0031846968801862072 |
| 17 | LEUCOCYTE_AB_2 | 0.002996386885526138 |
| 18 | ERYTHROCYTE_AB_0 | 0.002905366627764105 |
| 19 | ERYTHROCYTE_AB_1 | 0.0026213834235465612 |
| 20 | MCV_AB_2 | 0.002597552665150683 |
| 21 | MCHC_AB_2 | 0.002189450927621275 |
| 22 | THROMBOCYTE_AB_2 | 0.0020095745301139285 |
| 23 | MCH_AB_0 | 0.0015291403304021603 |
| 24 | THROMBOCYTE_AB_1 | 0.0010485533694186242 |
| 25 | MCV_AB_1 | 0.0009830187838299604 |
| 26 | MCH_AB_1 | 0.0009633584081533617 |
| 27 | HAEMOGLOBINS_AB_2 | 0.0009191792939052488 |
| 28 | MCH_AB_2 | 0.0008425875289971085 |
| 29 | LEUCOCYTE_AB_1 | 0.0006553458558866403 |
| 30 | HAEMOGLOBINS_AB_1 | 0.0 |
| 31 | HAEMATOCRIT_AB_1 | 0.0 |
| 32 | HAEMATOCRIT_AB_0 | 0.0 |
| 33 | ERYTHROCYTE_AB_2 | 0.0 |
| 34 | MCHC_AB_1 | 0.0 |
k-近傍法
perm = permutation_importance(KN_model, train_X, train_y, n_repeats=10, random_state=42)
perm_imp_df = pd.DataFrame({"importances_mean":perm["importances_mean"], "importances_std":perm["importances_std"]}, index=df_LR.columns)
perm_imp_df_sort = perm_imp_df.sort_values('importances_mean',ascending=False)
print(perm_imp_df_sort)
| 特徴量 | importances_mean | importances_std |
|---|---|---|
| THROMBOCYTE | 1.755982e-01 | 0.005439 |
| AGE | 9.023929e-02 | 0.006112 |
| HAEMATOCRIT | 3.611461e-02 | 0.003278 |
| LEUCOCYTE | 1.486146e-02 | 0.004174 |
| MCV | 1.486146e-02 | 0.003696 |
| MCH | 1.826196e-03 | 0.002121 |
| LEUCOCYTE_AB_0 | 1.196474e-03 | 0.000560 |
| ERYTHROCYTE_AB_2 | 1.102015e-03 | 0.000323 |
| LEUCOCYTE_AB_1 | 1.007557e-03 | 0.000577 |
| ERYTHROCYTE_AB_0 | 5.667506e-04 | 0.000611 |
| HAEMATOCRIT_AB_2 | 4.408060e-04 | 0.000427 |
| ERYTHROCYTE | 4.408060e-04 | 0.000748 |
| HAEMOGLOBINS_AB_2 | 2.833753e-04 | 0.000535 |
| MCH_AB_2 | 1.574307e-04 | 0.000352 |
| MCH_AB_0 | 1.259446e-04 | 0.000378 |
| THROMBOCYTE_AB_0 | 1.259446e-04 | 0.000154 |
| MCHC | 9.445844e-05 | 0.001082 |
| MCV_AB_0 | 6.297229e-05 | 0.000189 |
| THROMBOCYTE_AB_1 | 6.297229e-05 | 0.000126 |
| MCHC_AB_1 | 3.148615e-05 | 0.000094 |
| MCV_AB_2 | 3.148615e-05 | 0.000220 |
| HAEMATOCRIT_AB_0 | 3.148615e-05 | 0.000571 |
| HAEMOGLOBINS_AB_1 | 0.000000e+00 | 0.000141 |
| THROMBOCYTE_AB_2 | 0.000000e+00 | 0.000000 |
| MCH_AB_1 | 0.000000e+00 | 0.000000 |
| HAEMATOCRIT_AB_1 | 0.000000e+00 | 0.000000 |
| MCV_AB_1 | 0.000000e+00 | 0.000000 |
| HAEMOGLOBINS_AB_0 | -1.110223e-17 | 0.000545 |
| LEUCOCYTE_AB_2 | -3.148615e-05 | 0.000094 |
| SEX | -2.204030e-04 | 0.000509 |
| ERYTHROCYTE_AB_1 | -2.204030e-04 | 0.000283 |
| MCHC_AB_0 | -4.722922e-04 | 0.000211 |
| MCHC_AB_2 | -5.667506e-04 | 0.000189 |
| HAEMOGLOBINS | -1.763224e-03 | 0.001613 |
特徴量の重要度を確認
各モデルの上位10件を表示
| 順位 | GDBT | NN | RF | LR | DT | KN |
|---|---|---|---|---|---|---|
| 1 | THROMBOCYTE | THROMBOCYTE | THROMBOCYTE | THROMBOCYTE | THROMBOCYTE | THROMBOCYTE |
| 2 | AGE | AGE | LEUCOCYTE | THROMBOCYTE_AB_2 | LEUCOCYTE | AGE |
| 3 | LEUCOCYTE | LEUCOCYTE | ERYTHROCYTE | HAEMOGLOBINS | AGE | HAEMATOCRIT |
| 4 | HAEMATOCRIT | HAEMATOCRIT | HAEMATOCRIT | MCH | HAEMATOCRIT | LEUCOCYTE |
| 5 | MCHC | HAEMOGLOBINS | AGE | MCV | HAEMOGLOBINS | MCV |
| 6 | MCV | HAEMOGLOBINS_AB_2 | HAEMOGLOBINS | LEUCOCYTE | MCHC | MCH |
| 7 | MCH | HAEMATOCRIT_AB_2 | MCV | THROMBOCYTE_AB_0 | MCV | LEUCOCYTE_AB_0 |
| 8 | ERYTHROCYTE | HAEMOGLOBINS_AB_0 | MCH | MCH_AB_0 | HAEMATOCRIT_AB_2 | ERYTHROCYTE_AB_2 |
| 9 | HAEMATOCRIT_AB_2 | ERYTHROCYTE_AB_2 | MCHC | ERYTHROCYTE_AB_2 | ERYTHROCYTE | LEUCOCYTE_AB_1 |
| 10 | HAEMOGLOBINS | HAEMATOCRIT_AB_0 | THROMBOCYTE_AB_2 | AGE | MCH | ERYTHROCYTE_AB_0 |
特徴選択して再学習
前回結果が良かったXGBoostとランダムフォレストで特徴選択して学習させてみた
XGBoost
Over_100_importance = [4, 8, 3, 0, 6, 7, 5, 2, 12, 1]
Under_100_importance = [15, 9, 19, 16, 17, 10, 31, 13, 28, 22, 25]
Not_important = [11, 14, 18, 20, 21, 23, 24, 26, 27, 29, 30]
print("--Over_100_importance--")
print(df_GBDT.columns[Over_100_importance])
>>
--Over_100_importance--
Index(['THROMBOCYTE', 'AGE', 'LEUCOCYTE', 'HAEMATOCRIT', 'MCHC', 'MCV', 'MCH',
'ERYTHROCYTE', 'HAEMATOCRIT_AB_2', 'HAEMOGLOBINS'],
dtype='object')
print("--Under_100_importance--")
print(df_GBDT.columns[Under_100_importance])
>>
--Under_100_importance--
Index(['HAEMOGLOBINS_AB_2', 'SEX', 'LEUCOCYTE_AB_0', 'ERYTHROCYTE_AB_0',
'ERYTHROCYTE_AB_1', 'HAEMATOCRIT_AB_0', 'MCV_AB_0', 'HAEMOGLOBINS_AB_0',
'MCHC_AB_0', 'THROMBOCYTE_AB_0', 'MCH_AB_0'],
dtype='object')
print("--Not_important--")
print(df_GBDT.columns[Not_important])
>>
--Not_important--
Index(['HAEMATOCRIT_AB_1', 'HAEMOGLOBINS_AB_1', 'ERYTHROCYTE_AB_2',
'LEUCOCYTE_AB_1', 'LEUCOCYTE_AB_2', 'THROMBOCYTE_AB_1',
'THROMBOCYTE_AB_2', 'MCH_AB_1', 'MCH_AB_2', 'MCHC_AB_1', 'MCHC_AB_2'],
dtype='object')
# 重要度が低い特徴量を削除して再計算
# スコアが表示されないカラムを削除
df_GBDT_drop_zero = df_GBDT.drop(['HAEMATOCRIT_AB_1', 'HAEMOGLOBINS_AB_1', 'ERYTHROCYTE_AB_2', 'LEUCOCYTE_AB_1', 'LEUCOCYTE_AB_2', 'THROMBOCYTE_AB_1', 'THROMBOCYTE_AB_2', 'MCH_AB_1', 'MCH_AB_2', 'MCHC_AB_1', 'MCHC_AB_2'],axis=1)
X_GBDT_drop_zero = df_GBDT_drop_zero.values
# 変数を訓練用とテスト用に分割
train_X, test_X, train_y, test_y = train_test_split(X_GBDT_drop_zero, y_GBDT, test_size=0.2, random_state=42)
train_X, val_X, train_y, val_y = train_test_split(train_X, train_y, test_size=0.1, random_state=42)
# GBDTで学習
dtrain = xgb.DMatrix(train_X, label=train_y)
dval = xgb.DMatrix(val_X, label=val_y)
dtest = xgb.DMatrix(test_X)
params = {'objective': 'binary:logistic', 'silent':1, 'random_state': 71, 'eval_metric': 'auc'}
num_round = 25
watchlist = [(dtrain, 'train'), (dval, 'eval')]
GBDT_drop_zero_model = xgb.train(params, dtrain, num_round, evals=watchlist)
val_pred = GBDT_drop_zero_model.predict(dval)
val_y = val_y.tolist()
score_val = log_loss(val_y, val_pred)
print(f'logloss_val: {score_val: 4f}')
pred_y = GBDT_drop_zero_model.predict(dtest)
test_y = test_y.tolist()
AUC_GBDT_drop_zero = create_ROCcurve(test_y, pred_y)
tn_GBDT_drop_zero, fp_GBDT_drop_zero, fn_GBDT_drop_zero, tp_GBDT_drop_zero, specificity_GBDT_drop_zero, recall_GBDT_drop_zero, f1_GBDT_drop_zero = create_cm(test_y, pred_y, True)
accuracy_GBDT_drop_zero = accuracy_score(test_y, pred_y)
print(f'acc: {accuracy_GBDT_drop_zero}')
AUC_list.append(["AUC_GBDT_NI:", np.round(AUC_GBDT_drop_zero, 3)])
specificity_list.append(["specificity_GBDT_NI:", np.round(specificity_GBDT_drop_zero, 3)])
recall_list.append(["recall_GBDT_NI:", np.round(recall_GBDT_drop_zero, 3)])
f1_score_list.append(["f1_GBDT_NI:", np.round(f1_GBDT_drop_zero, 3)])
ランダムフォレスト
# スコアが0.005以下のカラムを削除
df_RF_drop = df_RF.drop(['MCHC_AB_0', 'MCH_AB_2','ERYTHROCYTE_AB_1', 'LEUCOCYTE_AB_2', 'MCHC_AB_2',
'MCHC_AB_1','HAEMATOCRIT_AB_1', 'MCV_AB_1', 'HAEMOGLOBINS_AB_1', 'MCH_AB_1' ],axis=1)
X_RF_drop = df_RF_drop.values
RF_drop_model = RandomForestClassifier(max_depth=15, n_estimators=82)
RF_drop_model.fit(train_X, train_y)
RF_drop_model.score(test_X, test_y)
pred_y = RF_drop_model.predict(test_X)
AUC_RF = create_ROCcurve(test_y, pred_y)
tn_RF, fp_RF, fn_RF, tp_RF, specificity_RF, recall_RF, f1_RF = create_cm(test_y, pred_y)
AUC_list.append(["AUC_RF_S_NI", np.round(AUC_RF, 3)])
specificity_list.append(["specificity_RF_S_NI", np.round(specificity_RF, 3)])
recall_list.append(["recall_RF_S_NI", np.round(recall_RF, 3)])
f1_score_list.append(["f1_RF_S_NI", np.round(f1_RF, 3)])
結果
_NIは特徴選択した結果
| ROC_AUC | 特異度 | 感度 | f1値 |
|---|---|---|---|
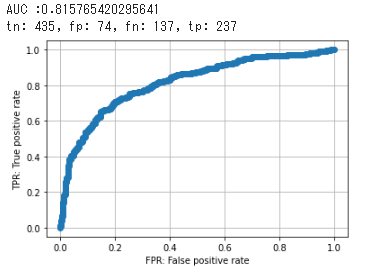
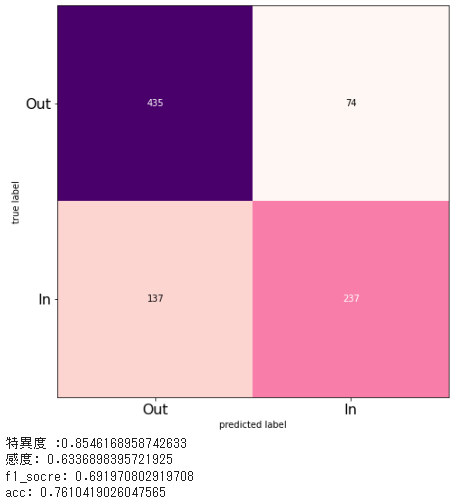
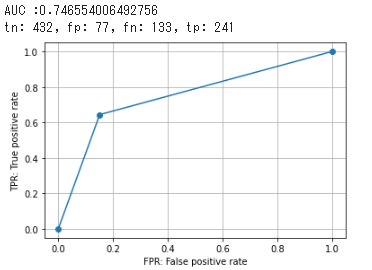
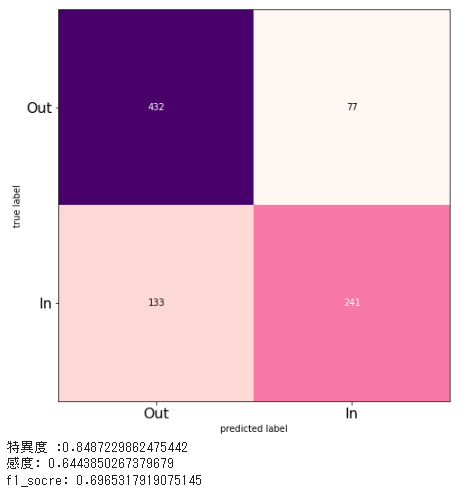
| ['AUC_GBDT:', 0.816] | ['specificity_RF', 0.859] | ['recall_RF_S', 0.658] | ['f1_RF', 0.707] |
| ['AUC_GBDT_NI:', 0.816] | ['specificity_GBDT:', 0.855] | ['recall_RF', 0.652] | ['f1_RF_S', 0.703] |
| ['AUC_RF', 0.755] | ['specificity_GBDT_NI:', 0.855] | ['recall_RF_S_NI', 0.644] | ['f1_RF_S_NI', 0.697] |
| ['AUC_RF_S', 0.75] | ['specificity_RF_S_NI', 0.849] | ['recall_GBDT:', 0.634] | ['f1_GBDT:', 0.692] |
| ['AUC_RF_S_NI', 0.747] | ['specificity_RF_S', 0.843] | ['recall_GBDT_NI:', 0.634] | ['f1_GBDT_NI:', 0.692] |
考察
各モデルの特徴量から
THROMBOCYTE(血小板)が最も重要であり、HEMATCRIT(ヘマトクリット)やAGE(年齢)、LEUCOCYTE(白血球)が重要な要素であることが考えられる。またMCHC(平均赤血球ヘモグロビン濃度), MCV(平均赤血球容積), MCH(平均赤血球ヘモグロビン量),HEMOGLOBINS(ヘモグロビン)などと比較して、ERYTHROCYTE(赤血球)は全体的に影響が小さいことが分かった。
XGBoostとランダムフォレストで特徴選択して学習をしたが、特徴選択により大きな改善はみられなかった。
参考資料
特徴選択とは?機械学習の予測精度を改善させる必殺技「特徴選択」を理解しよう
SageMakerで学習したXGBoostモデルのFeature Importanceを取得するAirflowオペレーター
Permutation Importanceを使って検証データにおける特徴量の有用性を測る
特徴量の重要度評価 ~ "Feature Importance"と"Permutation Importance"の比較 ~
多重共線または相関のある特徴を持つ並べ替えの重要度
【Python 機械学習】特徴量重要度に関してまとめ
ランダムフォレスト系ツールで特徴量の重要度を測る