ChainerやKeras、PandasやDask、Vaex関係などでちらほら見かけるHDF5(.h5とか.hdf5とかの拡張子のやつです)。
知識が無く以前は単なるバイナリフォーマットなのかと思っていましたが、しっかり勉強したら色々機能があって面白かったので、復習も兼ねてまとめておきます。
そもそもHDF5って?
- Hierarchical Data Formatの略(5はバージョン)で、名前の通り階層化された形でデータを保存することができるファイル形式です。
- ある種フォルダやファイルシステムに感覚が近く、1つのファイル内に整理しつつ様々な複数ファイルを保存できます。
HDF5のここが凄い
とりあえず機能に色々触れだすと長くなるので、先にHDF5の、個人的に良さそうに感じた点を書いておきます。
読み書きがCSVなどより大分速い
いくつか記事を見ていた感じ、パフォーマンスはpickleよりも若干遅いか・・・くらいで、CSVよりも大分早くなっています。
| | 読み込み | 書き込み |
|:-:|:-:|:-:|
| CSV | 17.900 | 69.00 |
| Pickle | 0.173 | 1.77 |
| HDF_fixed | 0.196 | 2.03 |
| HDF_tab | 0.230 | 2.60 |
※数値はWhat is the fastest way to upload a big csv file in notebook to work with python pandas?より引用
pickleがPythonオブジェクトをダイレクトに扱う(そのための制約やセキュリティ関係での配慮の必要性などが少しある)のを考えれば、CSVなどと比べればHDF5でも必要十分と言えそうです。
他の言語でも使える
pickleだと基本的に他の言語で扱えません。私の今いる会社だとPython以外にもRだったりを使っている方がいらっしゃいますし、他の会社ではJavaなりGoなりで扱う必要が出てくるケースもあるかもしれません。
また、pickleではPythonバージョンによるプロトコル番号的なところも絡んできます。
(とはいえ、もうPython2系を新しく使い始めるケースはほぼ無いと思いますし、万一必要になったらプロトコル番号を下げて保存すればいい気もしますが・・)
HDF5の場合はpickleよりも各言語跨いで利用したりがしやすいと言えます。
NumPy的なインターフェイスで扱える
スライスやらファンシーインデックスやら、NumPyライクなインターフェイスが色々あり、馴染みのある感じで学習コストを抑えつつ扱うことができます。
読み込み結果もNumPy配列になったりするので、NumPyに慣れていればスムーズに扱うことができます。
dset = f.create_dataset('/sample_data_1', shape=(1000, 10))
dset[:, 0:5] = 1000
dset[:, 5:10] = 2000
dset[0]
array([1000., 1000., 1000., 1000., 1000., 2000., 2000., 2000., 2000.,
2000.], dtype=float32)
圧縮の取り扱いなどがとても楽で、部分的に更新もいける
普通のCSVやpickleだと、圧縮を利用している場合に1つのファイルが大きくなると、更新時に再度圧縮をかける必要が出てきて結構読み書きが速度的に辛くなります。
そのため、基本的に日次単位とかでファイルを分けたりが必要になってきますが、HDF5の場合チャンク(後述)の機能を使うことで、更新領域を一部だけに絞ることができるので、大きなファイルでも細かく更新しても高速に動作させることができます。
さらに、書き込んだ時点でよしなに保存してくれるので、毎回compression引数を設定したりといったことをしなくてもファイルを開く最初だけ設定すれば後は自動でやってくれます。
dset = f.create_dataset(
'/compression_sample', shape=(10000000, 10), dtype=np.int64,
compression='gzip', chunks=(1000, 10))
以下のように、圧縮データでも部分的に更新ができます。配列を更新する感覚で、ファイルの内容を更新できます(圧縮指定なども気にせずに扱えます)。
dset[:100, :] = 1000
dset[100:200, :] = 2000
また、チャンク設定をやっているため、部分的にしか更新が走らないので、圧縮ファイルなのにとても速く保存が終わります(普通に8桁行のデータを圧縮保存すると何秒もかかったりする一方で)。
%%timeit
dset[:100, :] = np.random.randint(1000, 2000)
1000 loops, best of 3: 1.79 ms per loop
データの部分的なアクセスができる
「○○の階層にある、××のインデックス領域のデータだけ読み込みたい」といったケースに、pickleだとバイナリ全体でロードがされるので、pickleサイズが大きいと結構時間がかかります。
CSVであれば行番号の範囲で絞り込んだりもできますが、CSV自体が結構ロードに時間がかかったり、(Pandasなどに乗せる段階では絞れはするものの)列方向でのアクセスがぼちぼち必要になってしまいます。
ただ、CSV は行志向のフォーマットなので不要なカラムであっても必ず読まなければいけないという問題点がある。
Python: Apache Parquet フォーマットを扱ってみる
HDF5であれば、必要なインデックスを指定した際に初めて必要な配列データの必要な箇所のロードがされるので、部分的にデータを扱うのが気軽にできます。
データを階層的に持てるので、1つのファイルなのにデータを整理しつつたくさんのファイルを格納できる
HDF5自体が階層的にデータを持てるので、データの移動などが楽です。
たとえば、S3とかに日別に様々な項目のデータをそれぞれ分けてもっていた際などに、大量のファイルをローカルに落として作業をしたりしないとかだと結構手間です(解凍とかが必要な場合は使い捨てのスクリプトを書いたりも必要になってくるかもしれません)。
(もしくは、クラウドドライブを計算用のインスタンスにマウントして使うケースなどに)
一方で、HDF5だとある程度の件数をまとめることで、ファイル数を減らせるので扱いが楽です。バックアップなども楽かもしれません。
(ただし、あまりに集約しすぎると今度はダウンロードの時間やコストなどが気になってくるのである程度快適なサイズで分割する必要はあると思いますが・・・)
BigQueryやAthenaなどで必要なデータをクエリで絞り込んでPythonに持ってくるという使い方も多いと思いますが、それらは行数が膨大なままPythonに持ってこないといけないケースなどに少し辛い時があるかもしれません(SQLで行数を結構絞れるケースなどはとても快適な一方で。向き不向きがあるので効率的に作業する際に使い分けが必要になったりなど)。
f.create_dataset('/apple/2018/0101', shape=(1000, 20), dtype=np.int64)
f.create_dataset('/apple/2018/0102', shape=(1000, 20), dtype=np.int64)
f.create_dataset('/apple/2018/0103', shape=(1000, 20), dtype=np.int64)
...
f.create_dataset('/orange/2018/0101', shape=(1000, 20), dtype=np.int64)
f.create_dataset('/orange/2018/0102', shape=(1000, 20), dtype=np.int64)
...
デスクトップに色々置くよりも、フォルダをちゃんと挟んでファイルを配置したほうがデータが整理されるのと同様、HDF5では階層を自由に設定できるので整理整頓しつつデータを扱うことができます。
ある種、DBのような使い方ができるわけです。実際に、HDF5を利用したPyTablesというライブラリもあるようです。
PyTables: hierarchical datasets in Python
PyTables is a package for managing hierarchical datasets and designed to efficiently cope with extremely large amounts of data.
ファイルに様々な属性を付与できる
後任の方がデータにアクセスする際に、データの内容がいまいち分からず途方に暮れてしまうケースがあるかもしれません。もしかしたら資料は前任の方が書いているかもしれませんが、その資料がどこにあるのか共有が漏れていたりすると気づけないかもしれません。
HDF5では任意の属性として文字列や数値、テンソルなどをファイルに付与できることができます。
内容はデータの説明や補足、スキーマ、履歴情報、URL、単位など、ケースバイケースで設定されることになりますが、ファイル自身が説明を含んでいるので、ファイルさえあれば後任の方が途方に暮れずに済みます。
MySQLなどでも、テーブルやカラムにコメントなりスキーマ資料などがあった方が引継ぎが楽だと思いますが、それと同様にHDF5でも様々な役立つ情報を付与することができます。
メモリを抑えつつデータの読み書きができる
一度にHDF5のファイル全体を読み込むわけではないのと、条件に応じてスライスなどをしてからNumPy配列などに持ってこれるので、HDF5のファイルサイズがかなり大きくてもメモリが足りない・・・とならずに済みます。
ディープラーニングなどで画像などを扱ったりするケースであれば、数十万画像とかを一つのHDF5ファイルに入れたりもできます(学習時は、メモリが足りるように数千件程度のイテレーションで扱うなどして)。
実際に、論文の実装のgithubのコードなどでは、学習データがHDF5だったりするケースなどもちらほらあると思います。
階数の高いのテンソルも扱いやすい
BigQueryやCSVだと、基本的に行列(一応、列内に配列などを入れたりもできますが、なんだかんだ行列に展開したほうが便利だったり速度が遅くなったり)で扱うことが多いと思いますが、HDF5では基本的にNumPy同様にテンソルを快適に保存・扱うことができます。
HDF5ベースで作られているPyTablesでも、RDBMSと競合するものではなく、多次元のデータが必要な時などに利用するように、役割分担的に使うチームメイトだ、といったことが触れられています(逆にJoinやら内部キー制約やらトランザクションやらが重要なデータであればRDBMSの方が向いていたり等)。
Not a RDBMS replacement
PyTables is not designed to work as a relational database replacement, but rather as a teammate. If you want to work with large datasets of multidimensional data (for example, for multidimensional analysis), or just provide a categorized structure for some portions of your cluttered RDBS, then give PyTables a try.
PyTables: hierarchical datasets in Python
たとえば10万のpng画像(shape例: (100000, 256, 256, 4))などをBigQueryなりに保存する、というのは向いていないですが、HDF5であればそういったデータも快適に保存することができます(参考文献にはエクサバイトのサイズまでスケールできるよ、と書いてあり、流石にそのレベルまでは試していませんがとても大きいサイズでも問題なく扱えるようです)。
ファイルシステムのショートカット的な機能がある
普通のファイルシステムでも、サイズ的にコピーをしたくない(もしくはできない)ケースなどに、ショートカットファイルを用意したりすることが結構あると思います。HDF5でもそういった機能があり、データのコピーをせずに特定の階層に参照のみを設定するリンクという機能があります。
この機能は同一のファイル内だけでなく、他のHDF5のファイル間でリンクさせたりもできますし、リンク先をリネームしても大丈夫なように設定したりもできます。また、リンク先の特定の(スライスなどで得られる)領域のデータのみリンクを張るといったこともできます。
リンク先が少しごちゃっとした際に、データをコピーせず(ディスク領域を節約しつつ)に、特定階層に整理した形で作業で必要なデータ達をリンクで設定したり、といったことをすると快適に作業できるかもしれません。
HDF5の基本と使い方
本記事で使う環境
- Ubuntu
- Python 3.6.1 :: Anaconda 4.4.0 (64-bit)
- Jupyter notebook
- h5py==2.7.0
h5pyはHDF5をPythonで扱うためのオープンソースのライブラリです。Anacondaなどであれば最初から入っていますが、もし入っていない環境の場合にはcondaコマンドやpipなどが必要になります。詳細はInstallationのドキュメントをご確認ください。
ファイルの開き方
扱う際にはまず最初に対象のファイルを開きます。Fileクラスを使います。ほぼPythonのopen関数と似た形で使えます。
import numpy as np
import h5py
f = h5py.File('file_open_sample.hdf5')
# ...必要な処理を記述。
# 終わったらcloseメソッドで閉じます。
f.close()
また、open関数と同様withステートメントが利用して安全に操作することができます。
with h5py.File('file_open_sample.hdf5') as f:
# ...必要な処理を記述。
pass
mode引数があり、必要に応じてrやw、aなどを指定します。
with h5py.File('file_open_sample.hdf5', mode='w') as f:
# ...必要な処理を記述。
pass
グループとデータセットの作り方
※以降では説明のためwithステートメントを挟まないで書く箇所も多いですが、実際に運用する際にはwithステートメントの利用を推奨します。
HDF5での用語として、グループ = フォルダ、データセット = 1つのファイル(CSVなど)といった感覚の扱いになります。
グループを作るにはcreate_groupメソッドを使います。
f = h5py.File('sample_dataset.hdf5', 'w')
group = f.create_group('/apple')
これだけだと空のフォルダを作るようなもので、グループ内には何も入っていません(groupオブジェクトを出力してみると0 membersという表記が出力されます)。
group
<HDF5 group "/apple" (0 members)>
データセットを作るには、create_datasetメソッドを使います。name引数にはそのデータセットの名前を指定し、shapeやdtypeはNumPyと同じような挙動となります。
dataset = group.create_dataset(
name='2019-07-30', shape=(10000, 10), dtype=np.int64)
データセット作成後のgroupオブジェクトにアクセスすると、membersの値が増えていることが分かります。
group
<HDF5 group "/apple" (1 members)>
データセットオブジェクトを出力してみると、名称やサイズなどの情報が確認できます。
dataset
<HDF5 dataset "2019-07-30": shape (10000, 10), type "<i8">
また、直接ファイルオブジェクトでcreate_datasetメソッドを呼ぶこともできます。その際には絶対パスで指定します。未生成のグループ(下記のサンプルのorange部分)がある場合は自動で生成されます。
dataset_orange = f.create_dataset(
name='/orange/2019-07-30', shape=(10000, 10), dtype=np.int64)
最初からテンソルなどをデータセットに設定したい場合には、data引数に配列などを指定します。その場合はshapeやdtypeの引数を省略しても配列を参照して設定してくれます(Pandasと同じような感覚ですね)。
import numpy as np
arr = np.random.random(size=(10, 10))
dataset = f.create_dataset('initial_arr_sample', data=arr)
dataset
<HDF5 dataset "initial_arr_sample": shape (10, 10), type "<f8">
データへのアクセス
生成したデータセットオブジェクトに対して、NumPyのようにインデックスを指定する形でデータにアクセスできます。
インデックスを指定してアクセスした際には、NumPy配列がメモリ上に乗る形で返却されます(そのため、1つのデータセットに対して膨大なサイズのテンソルを指定して、そのほとんどにアクセスするとメモリがきつくなる可能性があるのでご注意ください)。
初期値は、データセットを作る時に指定したdtypeの型に依存します。今回はint64を指定していたので、0の配列で生成されています。
dataset[0]
array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0])
今回はshape=(10000, 10)で作成したので、NumPyのように行番号・列番号といったインデックスの指定もできます。
dataset[0, 0]
0
スライスでのアクセスもNumPyのように動作します。
dataset[:3]
array([[0, 0, 0, 0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0, 0, 0, 0]])
データセット全体の読み込み
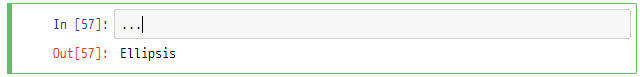
対象のデータセット全体をNumPy配列に読み込む場合は、以下のようにインデックスに...と記載します。
arr = dataset[...]
arr.shape
(10000, 10)
なお、私はHDF5の勉強をして初めて知ったのですが、Pythonで...の表記はEllipsisというオブジェクトとなり、意味を持ちます。Jupyterなどでも...と記載すると確認できます。
データの更新
データセットオブジェクトの(shapeで指定したサイズに収まる範囲での)任意のインデックスに対して代入をするとデータが書き込まれます。NumPyやPandasなどと異なり、to_○○などの保存用の関数はありません。ファイルを閉じた時点でファイル側にデータが流れるようで、閉じるとサイズが増えたりします。(閉じる前でも、別途読み取り専用で開くと、更新内容はちゃんと反映されたりしています。)
dataset[0, :5] = 1
dataset[0]
array([1, 1, 1, 1, 1, 0, 0, 0, 0, 0])
試しに、前述のファイルオブジェクトでcloseメソッドを呼ぶ前に、別途読み取り専用で開いて内容を確認すると、ファイルの内容が更新されていることが分かります。
f2 = h5py.File('./sample_dataset.hdf5', 'r')
f2_dataset = f2['/apple/2019-07-30']
f2_dataset[0]
array([1, 1, 1, 1, 1, 0, 0, 0, 0, 0])
より発展的なデータの選択方法
NumPyやPandasなどでお馴染みのファンシーインデックスやブールインデックスは同様に使えます。
サンプルとして、まずはランダムなデータを設定します。
dataset = group.create_dataset(
name='2019-07-29', shape=(10000, 10), dtype=np.int64)
dataset[...] = np.random.randint(low=0, high=100, size=(10000, 10))
dataset[...]
array([[17, 28, 64, ..., 12, 84, 11],
[16, 96, 58, ..., 96, 56, 53],
[60, 78, 26, ..., 58, 54, 54],
...,
[34, 96, 99, ..., 84, 17, 45],
[28, 89, 99, ..., 33, 12, 80],
[76, 51, 90, ..., 1, 87, 10]])
条件式を指定すると、ブーリアンを格納した同じサイズの配列が返ってきます。
dataset[...] > 50
array([[False, False, True, ..., False, True, False],
[False, True, True, ..., True, True, True],
[ True, True, False, ..., True, True, True],
...,
[False, True, True, ..., True, False, False],
[False, True, True, ..., False, False, True],
[ True, True, True, ..., False, True, False]])
得られたブールインデックスをインデックスに指定することで、取得する対象を絞り込めます。ほとんどNumPyなどと一緒ですね。
dataset[dataset[...] > 50]
array([64, 64, 83, ..., 53, 79, 87])
データとグループの削除
ファイルオブジェクトやグループに対して、delステートメントを指定すると消せます。
del f['/apple/2019-07-29']
消したグループやデータセットにアクセスしようとすると怒られます。
f['/apple/2019-07-29']
KeyError: "Unable to open object (Object '2019-07-29' doesn't exist)"
HDF5上の型
基本的にNumPyと似たような挙動ですが、一部特殊な型やNumPyに無い型、あとはNumPyと挙動が微妙に違う箇所があります。
普通の数値の型
np.int8、np.int64、np.float32、np.float64といったお馴染みの型が指定できます。
バイト数の大きい型を指定すればするほど、許容できる最大値が大きくなる一方で必要なディスクサイズ(もしくは読み込み後の必要メモリ)が大きくなります。
NumPyと少し異なる点として、許容できる最大値を超えた際に、NumPy側はマイナスになったりと値が変わる一方で、HDF5側は上限値で止まります。(UbuntuではなくWindowsで試していたらNumPy側は別の値になったので、OSやNumPyバージョンである程度挙動が変わるかもしれません)
import numpy as np
np.int8(128)
-128
一方で、HDF5は型の上限を超えた場合は上限値で止まります。
dataset = f.create_dataset('dataset_int8', shape=(1,), dtype=np.int8)
dataset[0] = 128
dataset[...]
array([127], dtype=int8)
固定長の文字列
正直使いづらい感があります。Python2系のころの歴史的な理由があるのか・・・結構扱うのが辛い印象です。使うなら後述する可変長の文字列で普通にいいような・・・という印象があります。
特徴として、
- サイズを超えた文字は取り除かれる
- バイトで扱われるので、一旦デコードなどが必要になる
- ASCII以外を指定すると怒られるので日本語とかでの利用に難がある
といった感じなようです(日本語の値の扱いはうまいことする方法があるのだろうか・・・)。
型の指定にはS5やS32といった文字列を指定します。(数値部分は文字列の最大長)
dataset = f.create_dataset('dataset_fixed_str', shape=(1,), dtype='S5')
値を設定する際にはbytes型で指定する必要があるので、文字列の先頭にbと付けておきます。
dataset[0] = b'1234'
dataset[0]
b'1234'
最大長を超える文字列を指定すると末尾の方からデータが欠落します。
dataset[0] = b'123456'
dataset[0]
b'12345'
bytes型なので、decodeしてあげると普通の文字列が取れます。
fixed_str = dataset[0]
fixed_str.decode('utf-8')
'12345'
日本語を指定したり、b表記を省略すると怒られます。
dataset[0] = b'林檎'
SyntaxError: bytes can only contain ASCII literal characters.
dataset[0] = '林檎'
TypeError: No conversion path for dtype: dtype('<U2')
可変長の文字列
可変長(文字列の値に応じた長さ)というだけではなく、ユニコードなどの取り扱いの都合で、基本的にHDF5で文字列を扱う際にはこちらが多くなりそうです。
型の指定用に、special_dtypeクラスを使います。
str_dtype = h5py.special_dtype(vlen=str)
引数のvlenは、初見だと長さの値なのか・・?という感じですが、docstringを読むと、
Base type for HDF5 variable-length datatype
とあるので、値の長さ(variable-length / 可変長)に応じた型、という由来のようです。
strもしくはNumPyの型を設定できます。
これを使って、データセット生成時の型に指定します。
dataset = f.create_dataset(
'str_dataset', shape=(5,), dtype=str_dtype)
生成したデータセットでは、bytes指定無しで値が設定できます。
dataset[0] = 'abcdef'
dataset[0]
'abcdef'
長い文字列でも問題なく設定できます。
dataset[1] = 'Lorem ipsum dolor sit amet, consectetur adipiscing elit, sed do eiusmod tempor incididunt ut labore et dolore magna aliqua. '
dataset[1]
'Lorem ipsum dolor sit amet, consectetur adipiscing elit, sed do eiusmod tempor incididunt ut labore et dolore magna aliqua. '
日本語も問題なく設定できます。
dataset[2] = '林檎オレンジ'
dataset[2]
'林檎オレンジ'
基本的に固定長の文字列よりも便利そうですし、ユニコードを扱う際にはこちらの型を使うこと、参考文献やネットの記事にも書かれているので、こちらでいいような気がしていますが、1点気になる点として、参考文献に「Pythonオブジェクトとして扱われる」といったことが見受けられたのと、special_dtypeという名前などを考えると、他のRなどの言語で利用するときにちゃんと使えるのかな・・?というのが気になりました。HDF5はPythonでしか扱ったことがないので、もし他のRなどの言語でもHDF5を使う環境を用意している方がいらっしゃいましたらコメントなどでご教示ください・・(もしくは、同僚の方にRとPython両方書ける方がいらっしゃるので後でお聞きするかもしれません)
真偽値
普通に型にnp.boolなどを指定すれば使えます。
f = h5py.File('bool_type_sample.hdf5', mode='w')
dataset = f.create_dataset('bool_sample', shape=(5,), dtype=np.bool)
dataset[0]
False
dataset[0] = True
dataset[0]
True
Python上で使う際には特に意識することなく使えますが、内部的にはEnumの機能(0=False, 1=True)を使って実現されているそうです。後述しますが、HDF5ファイルの内部を確認するコマンド(h5ls)を実行すると、Enum情報などが確認できます。(一旦ファイルオブジェクトのcloseを呼んでおかないとコマンドでの結果が確認できないので実行しておきます)
f.close()
$ h5ls -vlr bool_type_sample.hdf5
Opened "bool_type_sample.hdf5" with sec2 driver.
/ Group
Location: 1:96
Links: 1
/bool_sample Dataset {5/5}
Location: 1:800
Links: 1
Storage: 5 logical bytes, 5 allocated bytes, 100.00% utilization
Type: enum native signed char {
FALSE = 0
TRUE = 1
}
Enumerated型
前日の真偽値の型でも少し出てきましたが、いわゆるEnumの型です。HDF5独自の型になります。
special_dtypeのenum引数に番号のための型(np.int8など)とマッピング用の辞書を指定する形で作れます。
f = h5py.File('enum_sample.hdf5', mode='w')
mapping_dict = {'apple': 0, 'orange': 1, 'melon': 2}
dtype = h5py.special_dtype(enum=(np.int8, mapping_dict))
設定されている値は、metadata属性にアクセスしていくことで確認できます。
dtype.metadata['enum']['apple']
0
dtype.metadata['enum']['melon']
2
このEnumの型を使ってデータセットが作れます。
dataset = f.create_dataset('enum_data', shape=(3,), dtype=dtype)
dataset[0] = 0
dataset[1] = 2
注意すべき点として、マッピングの辞書で定義していない番号もエラーなどなく普通に許容されます。
dataset[2] = 100
また、Enumの定義の内容は、真偽値のところと同様に、コマンドで確認することもできます。
f.close()
$ h5ls -vlr enum_sample.hdf5
Opened "enum_sample.hdf5" with sec2 driver.
/ Group
Location: 1:96
Links: 1
/enum_data Dataset {3/3}
Location: 1:800
Links: 1
Storage: 3 logical bytes, 3 allocated bytes, 100.00% utilization
Type: enum native signed char {
apple = 0
melon = 2
orange = 1
}
compound型
HDF5本を読むまで知らなかったのですが、NumPy配列に辞書的な構造を持たせる機能が存在します。
たとえば、id、name、priceといった各キーにテンソルを設定する、といったことができます。
dtype = np.dtype([('id', np.int), ('name', '|U10'), ('price', np.int)])
arr = np.empty(shape=(2, 2), dtype=dtype)
type(arr)
numpy.ndarray
arr['id']
array([[0, 0],
[0, 0]])
arr['name']
array([['', ''],
['', '']], dtype='<U10')
arr['price']
array([[0, 0],
[0, 0]])
こういった型の配列をstructured arraysと呼ばれたり、型名がcompoundなどと呼ばれたりするようです。
HDF5でもそのままcompound型を利用できます。
ただし、NumPyのように|U10といった文字列の型ができないので、前述のspecial_dtypeを利用します
dtype = np.dtype([
('id', np.int),
('name', h5py.special_dtype(vlen=str)),
('price', np.int)])
dataset = f.create_dataset(
'compound_dataset', shape=(2, 2), dtype=dtype)
dataset['id']
array([[0, 0],
[0, 0]])
dataset['name']
array([['', ''],
['', '']], dtype=object)
日付などのデータは?
普通に、整数の型でUnixtimeで保存したり、文字列(%Y-%m-%d)とかで保存する形で良さそうです。
初期値の指定
デフォルトのままだと、整数の型を指定したデータセットでは初期値は0、文字列の型を指定した際には空文字と、型に合わせた初期値が設定されます。
この初期値を変更したい場合は、fillvalue引数を設定します。
f = h5py.File('./sample_dataset_2.hdf5', 'w')
dataset = f.create_dataset(
'fillvalue_sample', shape=(3, 3), dtype=np.int,
fillvalue=12345)
データを確認してみると、確かに指定した値で初期値が設定されています。
dataset[...]
array([[12345, 12345, 12345],
[12345, 12345, 12345],
[12345, 12345, 12345]])
なお、この初期値設定は変更が利かないそうです。
もし変えたい場合は、そのデータセットを削除して再度データセットを作ったりが必要になるかもしれません(もしくは別の名前でコピーしたりなど)。
データセットをリサイズする
デフォルトのままだと、リサイズはできません。
dataset = f.create_dataset(
'resize_sample', shape=(2, 2), dtype=np.int64)
dataset.resize(size=(4,))
TypeError: Only chunked datasets can be resized
変更するには、データセット生成時にmaxshape引数を指定します。maxshapeに指定したサイズ内であれば変更が利きます。((2, 2)から(2, 1)や(1, 1)のサイズにするなど)
dset = f.create_dataset(
'maxshape_sample', shape=(2, 2), dtype=np.int64, maxshape=(3, 3),
fillvalue=100)
dset.shape
(2, 2)
dset.resize(size=(1, 1))
dset.shape
(1, 1)
dset.resize(size=(3, 3))
dset.shape
(3, 3)
また、最初に指定したサイズ以上にデータセットのサイズを大きくした場合に、初期値はfillvalue引数で指定した値になります。
dset[...]
array([[100, 100, 100],
[100, 100, 100],
[100, 100, 100]])
maxshapeで指定したサイズよりも大きいサイズを指定した場合はエラーになります。
dset.resize(size=(4, 4))
ValueError: Unable to set extend dataset (Dimension cannot exceed the existing maximal size (new: 4 max: 3))
最大サイズに制限を設けたくない場合には、maxshapeの対象の軸にNoneを指定します。
dataset = f.create_dataset(
'unlimited_maxshape', shape=(2, 2), dtype=np.int64,
maxshape=(None, 3))
dataset.resize(size=(1000000, 3))
dataset.shape
(1000000, 3)
参考文献には特にmaxshapeにNoneを指定する際のデメリットは書かれていなかったので、基本的にNoneを使う形でいいような気がしますが・・・制限を設けていた方が、誤って極端に大きなサイズを設定してしまって、ディスクサイズが枯渇するようなケースを避ける、といった感じなのでしょうか?
なお、軸を(階数)増やすことはできません((2, 2)から(1, 1, 1)にリサイズするなど)。
dataset = f.create_dataset(
'add_axis_error_sample', shape=(2, 2), dtype=np.int64,
maxshape=(None, None))
dataset.resize(size=(1, 1, 1))
TypeError: New shape length (3) must match dataset rank (2)
属性を設定する
HDF5の大きな特徴として、メタデータとしての属性を自由に設定できるという点があります。
説明やらタイムスタンプやら実験結果やらデータバージョンやら自由に設定でき、説明用のファイルがファイルと一体化するので、データセットの説明資料がどこにあるのか分からない・・・といったことを避けることができます(単純なバイナリ形式のデータセットよりも、長期的な引継ぎ時の保守面などを良くできそうな印象があります)。
設定するには、データセットのattrs属性に辞書的に値を設定していきます。
dataset = f.create_dataset(
'attribute_sample', shape=(100), dtype=np.int)
dataset.attrs['description'] = '2019年7月の東京都での売り上げ'
dataset.attrs['created_at'] = '2019-08-01 10:00:00'
dataset.attrs['location_id'] = 10
アクセスも辞書感覚で使えます。
dataset.attrs['created_at']
'2019-08-01 10:00:00'
存在する属性名のリストの取得なども、辞書のキーと同じインターフェイスで行えます。
list(dataset.attrs.keys())
['description', 'created_at', 'location_id']
keysメソッドの他にも、itemsなどのイテレーション用のインターフェイスも同様です。
for key, value in dataset.attrs.items():
print(key, value)
description 2019年8月の東京都での売り上げ
created_at 2019-08-01 10:00:00
location_id 10
存在しないキーにアクセスするとエラーになります(エラーにしたくない場合は、辞書と同じくgetメソッドなどを使います)。
dataset.attrs['key_error_sample']
KeyError: "Can't open attribute (Can't locate attribute: 'key_error_sample')"
配列なども属性に設定できます。
arr = np.zeros(shape=(2, 5))
dataset.attrs['arr_sample'] = arr
dataset.attrs['arr_sample']
array([[0., 0., 0., 0., 0.],
[0., 0., 0., 0., 0.]])
ただし、1つの属性に設定できるサイズに制限があります。長い文字列や大きな配列を指定する際には注意が必要です(メインのデータはあくまでデータセット内に普通に設定して、属性はデータの説明に使うという立ち位置になります)。
arr = np.zeros(shape=(1000, 1000))
dataset.attrs['big_arr_sample'] = arr
RuntimeError: Unable to create attribute (Object header message is too large)
Pythonの他のオブジェクト(辞書)などは属性には設定できません。どうしても使いたい場合はpickle化する・・・とかで一応できはしますが、基本的に確認しづらくなるため、避ける形が無難かなという印象です(文字列や数値などをメインに使う形が好ましいかなと)。
基本的に属性は型は設定の際の値に応じて設定されます。
そのため、更新時に型を変えたりもできてしまいます。
dataset.attrs['int_attr'] = 100
dataset.attrs['int_attr'] = 'string_value'
dataset.attrs['int_attr']
'string_value'
一方で、「この属性はずっと○○の型で運用したい」というケースには、modifyメソッドを使うと型を引き継いでくれます。
うっかりミスでintがfloatになってしまったりもしないので安心です。
dataset.attrs['int_attr'] = 100
dataset.attrs.modify(name='int_attr', value=103.5)
dataset.attrs['int_attr']
103
圧縮とチャンク設定
データセットの内容を圧縮してディスクスペースを節約したい場合には、create_datasetメソッドでデータセットを作成する際にcompression引数を指定します。
dataset = f.create_dataset(
'compression_sample', shape=(100000, 100), dtype=np.int64,
compression='gzip')
各データセットの圧縮設定を後で確認する際などには、compression属性を参照します。
dataset.compression
'gzip'
その後は圧縮・解凍に関しては特に意識する必要はなく、通常通り値を部分的に更新したりできます。HDF5の内部で圧縮処理を加味しつつよしなに対応してくれます。
dataset[0, 0] = 100
その場合は自動でデータセットが一定のかたまり(chunk)で分割されます。
分割されることによって、大きな圧縮ファイルであっても部分的な更新が高速に処理されます(更新が該当するチャンク領域だけ更新されるため、圧縮データ全体を毎回読み書きするよりは遥かに高速に動作します)。
どのくらいのサイズで分割されているのかはchunks属性で確認できます。
dataset.chunks
(1563, 4)
いくつか圧縮の選択肢がありますが、基本的にはgzipかLZFが選択肢として有力かなという印象があります。
それぞれ、以下のような特徴があります。
- 解凍・圧縮で速度を重視するならLZF
- ただし、LZFはPython以外の環境で使えないケースが多い。gzipは他の環境でも大体使える
- 圧縮率はgzipの方が高い傾向がある(ディスクサイズが小さくて済む)
チャンクサイズは手動でも設定することができます。
パフォーマンス的には、できれば参照するチャンク数は少なくなったほうが速く処理されます。
そのため、(100, 100)といったように更新をよく行うのであれば、前述の(1563, 4)といったような分割よりかは列を一通りカバーできる(200, 100)といったようなチャンクサイズのほうが好ましいかもしれません((1563, 4)といった行方向に長いチャンクだと、更新時に多くのチャンクを参照しないといけなくなり、圧縮処理の範囲が広くなってパフォーマンスが悪くなります)。
設定する際にはchunks引数を指定します。
dataset = f.create_dataset(
'compression_sample_2', shape=(100000, 100), dtype=np.int64,
compression='gzip',
chunks=(200, 100))
dataset.chunks
(200, 100)
Daskなどの他のPythonライブラリのチャンク設定と同様に、チャンクサイズは大きすぎても小さすぎても非効率です。
HDF5では1つのチャンクで1MBを超えるとキャッシュなどの機能的に好ましくなかったり、余分にアクセスしないといけないデータ領域が広まってしまったりで非効率ですし、小さすぎても検索などのオーバーヘッドが大きくなります。程よいサイズで設定するか、自動設定に任せる形がおすすめです。
データアクセスの詳細とイテレーション
サンプルとして以下のようにデータを準備します。
f = h5py.File('./data_access_sample', mode='w')
f.create_group('/group_1')
f.create_dataset('/group_1/dataset_1', shape=(2, 2), dtype=np.int64)
f.create_dataset('/group_1/dataset_2', shape=(2, 2), dtype=np.int64)
f.create_group('/group_1/subgroup_1/')
f.create_dataset('/group_1/subgroup_1/dataset_3', shape=(2, 2), dtype=np.int64)
create_datasetなどで返却されるオブジェクト以外にも、ファイルオブジェクトに辞書のようにアクセスすることで対象のグループやデータセットにアクセスできます。
group_1 = f['/group_1']
group_1
<HDF5 group "/group_1" (3 members)>
dataset_1 = f['/group_1/dataset_1']
dataset_1
<HDF5 dataset "dataset_1": shape (2, 2), type "<i8">
subgroup_1 = f['/group_1/subgroup_1']
subgroup_1
<HDF5 group "/group_1/subgroup_1" (1 members)>
複数の階層を持つグループやデータセットに対しては、以下のようにどちらでもアクセスができますが、前者は少し非効率で後者の方法でのアクセスが無駄が少ないようです。前者の方法だと、一旦group_1のオブジェクトが用意され、次にsubgroup_1のオブジェクトが用意され、最後にデータセットのオブジェクトが用意される一方で、後者だとダイレクトにデータセットのオブジェクトが用意されます。
# アクセスできるけれども少し非効率。
dataset_3 = f['/group_1']['subgroup_1']['dataset_3']
# こちらのアクセスの方が無駄は少ない。
dataset_3 = f['/group_1/subgroup_1/dataset_3']
1つ上の階層のオブジェクトにアクセスしたい時には、parent属性でアクセスできます。
subgroup_1.parent
<HDF5 group "/group_1" (3 members)>
属性と同様に、グループやデータセットに対しても辞書感覚でイテレーションが回せます。
for key, obj in f.items():
print(key, obj)
group_1 <HDF5 group "/group_1" (3 members)>
for key, obj in group_1.items():
print(key, obj)
dataset_1 <HDF5 dataset "dataset_1": shape (2, 2), type "<i8">
dataset_2 <HDF5 dataset "dataset_2": shape (2, 2), type "<i8">
subgroup_1 <HDF5 group "/group_1/subgroup_1" (1 members)>
グループやデータセットの有無のチェックは、辞書のキーの有無の確認と同様inを使ってチェックできます。
'/group_1' in f
True
'/group_2' in f
False
任意のパスから、アクセス面での処理を加えたい場合にはPOSIXスタイルでのパス制御が可能な、ビルトインのposixpathモジュールが便利です(たとえば、../と記載して親の階層を辿りたい場合なと)。
dataset_3.name
'/group_1/subgroup_1/dataset_3'
import posixpath as pp
path = dataset_3.name
path = pp.normpath(pp.join(path, '../../'))
path
'/group_1'
リンク設定
ソフトリンク
デスクトップにショートカットを設置するがごとく、HDF5では特定のグループやデータセットを、コピーせずに任意のパスに設定するソフトリンクという機能が用意されています。
使用するにはパス情報を保持するSoftLinkクラスを使います。
dataset_3.name
'/group_1/subgroup_1/dataset_3'
softlink = h5py.SoftLink('/group_1/subgroup_1/dataset_3')
f['softlink_sample'] = softlink
ソフトリンクオブジェクトを設定したパスにアクセスしてみると、リンク先のデータセットにアクセスできることが分かります。
f['softlink_sample']
<HDF5 dataset "softlink_sample": shape (2, 2), type "<i8">
ソフトリンクはデータのコピーではなく、パス情報のみを保持するため、リンク先が消えたり更新されたりすると影響を受けます。
del f['/group_1/subgroup_1/dataset_3']
f['softlink_sample']
KeyError: 'Unable to open object (Component not found)'
外部ファイルへのリンク
同一のHDF5ファイルだけでなく、他のHDF5ファイル内のデータセットなどをリンクすることもできます。
ただ、リンクが張られているファイルを移動したりするとリンクが切れてしまうので、基本的に動かさないファイルだったり、もしくは集計でやりやすいように一時的に多くのHDF5ファイルで必要なデータのみリンクさせたファイルを用意して、集計終わったらリンク設定をしたHDF5を削除する、とかの用途が無難かもしれません(もしくは、リンクを利用していることが分かるように説明やファイル名などを調整しておくなど)。
設定するには、ExternalLinkクラスを使います。
with h5py.File('./external_link_sample.hdf5', mode='w') as f:
f.create_dataset('/group/external_dataset_sample', shape=(10, 10), dtype=np.int64)
external_link = h5py.ExternalLink(
filename='./external_link_sample.hdf5',
path='/group/external_dataset_sample')
この作成したデータセットを、別のファイルにリンクしてみます。
f = h5py.File('./sample_dataset_4.hdf5', mode='w')
f['link'] = external_link
f['link']
<HDF5 dataset "external_dataset_sample": shape (10, 10), type "<i8">
正常に外部ファイルの(10, 10)のサイズのデータセットにアクセスできているようです。
また、parent属性などはリンク先のものに依存します。そのため、リンクされているオブジェクトだと意識していないと、上の階層を参照したいといった時にうっかりミスしてしまうかもしれませんので注意が必要です。
f['link'].parent
<HDF5 group "/group" (1 members)>
リンク切れしにくいリンクを使う
ソフトリンクなどは便利です。しかし、データセットやグループを移動したりするとリンクが切れてしまい、不便なケースがあります。
そういった場合には、データセットの参照(reference)を属性に保存しておいて、その属性を使ってデータセットにアクセスするようにすると、リンク先のデータセットを移動などしてもエラーになりません。
f.attrs['link_ref'] = dataset_1.ref
f[f.attrs['link_ref']]
<HDF5 dataset "dataset_1": shape (10, 10), type "<i8">
試しにリンク先のデータセットを移動させてみます。moveメソッドでグループやデータセットを移動させることができます。
f.move(source='dataset_1', dest='dataset_2')
ソフトリンクの場合、ソフトリンク側も更新しないとリンク切れでエラーになってしまいますが、この方法の場合は同じコードのまま動作します。
f[f.attrs['link_ref']]
<HDF5 dataset "dataset_2": shape (10, 10), type "<i8">
HDF5関係のコマンドやviewer
ここまで来るまでに、h5lsなどのコマンドを一部使って来ましたが、コマンドだとPythonを経由しなくてもさくっと内容を確認したりできるので便利なケースがあります。
ドキュメントに色々書かれているので、必要な場合は調べてみてもいいかもしれません(インストール自体はAnacondaもしくはh5pyを入れた時点でされるのか、特に作業無く使えるようになっていました)。
HDF5 TUTORIAL: COMMAND-LINE TOOLS FOR VIEWING HDF5 FILES
また、ビューアーのツールなどもあるようです(こちらはちょっとまだ試せていません)。
MySQLなどでもクライアントツールなど使うケースが多いと思いますが、こういったツールを入れておくとデータセットの確認などで便利かもしれません。
並列化に関して
参考文献を読んだ感じ、HDF5の並列(並行)処理に関して以下のような特徴があるようです。
- GILの都合で、マルチスレッドでは基本的に速度は改善しない
- ※GILなどに関しては以前記事にしたのでそちらをご確認ください。Pythonの並列処理・並行処理をしっかり調べてみた
- マルチプロセス(multiprocessモジュール)では改善できるものの、コードが複雑になりがち
- MPI(Message Passing Interface)を使うとコードはシンプルにいける
とのことですが、MPI周りがインストールが若干面倒くさそう・・・という印象を受けました(pipだけでさくっといかなかった・・・)。
そのため、Daskなどでの利用を想定しているので、今回はMPIに関しては割愛し、Daskで色々試してみます(MPIが必要な場合にはドキュメントをご確認ください)。
Daskで使ってみる
PandasやNumPyの並列化や省メモリ化などで利用されるDaskでHDF5を試してみます。
HDF5用のインターフェイスはPandasなどと同様Dask側に用意されており、さくっと使うことができます。
ただ、Daskのドキュメントで「並列化などの面が大変なのでHDF5よりもParquetのほうがお勧め」という記述がある一方で、「データの高速化などで、HDF5などの速いフォーマットの利用がお勧め」といったような記述があって、結局DaskでHDF5ってどうなんだろう・・・ともやもやしていたので、その辺りも含めて試してみます。
Apache Parquet is a columnar binary format that is easy to split into multiple files (easier for parallel loading) and is generally much simpler to deal with than HDF5 (from the library’s perspective).
Best Practices (DataFrame) - Dask documentation
For storage formats you may find that you want self-describing formats that are optimized for random access, metadata storage, and binary encoding like Parquet, ORC, Zarr, HDF5, GeoTIFF and so on
Best Practices - Dask documentation
なお、本記事ではDaskは以下のバージョンを利用しています。
- dask==1.2.2
データの準備
普通の数値の数値のデータセットと、文字列を含んだデータセットの2つ用意してみます。2ヶ月分の時系列データとして考えて、データセットを1日ずつで分ける形で用意します。
数値の方は以下のように1日辺り500万行5列で作成しました。Dask側で入出力用のインターフェイスがあるので、h5py経由ではなくそちら経由で保存します(h5pyで保存してDaskのデータフレームで読み込もうとしたらエラーになった(何か必要な設定が足りていない?)ので、無難にDask側に任せます・・)。
また、Daskを経由するので、文字列はh5pyの可変長のものではなくNumPyやDaskで使われる型で進めてみます。
from datetime import datetime
import h5py
import numpy as np
from pandas.tseries.offsets import Day
import dask.dataframe as dd
target_dt = datetime(2019, 6, 1)
last_dt = datetime(2019, 7, 31)
while target_dt <= last_dt:
print(datetime.now(), target_dt)
date_str = target_dt.strftime('%Y%m%d')
arr = np.random.randint(low=0, high=10000000, size=(5000000, 5), dtype=np.int64)
name = 'int_{date_str}'.format(date_str=date_str)
df = dd.from_array(x=arr, columns=['a', 'b', 'c', 'd', 'e'])
df.to_hdf(
path_or_buf='./Dask_sample.hdf5', key=name)
target_dt += Day()
※データセットの名前に2019-06-01というフォーマットを使っていたところ、警告が出た(軽く調べたら無視してOKな警告らしい?ですが・・)ので、日付はハイフン無しで扱っています。
h5pyで一部データがDaskを経由してどのように保存されているのか確認してみます。
f = h5py.File('./Dask_sample.hdf5')
list(f['int_20190601'].keys())
['_i_table', 'table']
なにやら2つキーが設定されています。
f['int_20190601/table']
DF5 dataset "table": shape (5000000,), type "|V48">
tableの方は、普通にデータセットになっているようです。
f['int_20190601/table'][:5]
array([(0, [ 613201, 8567874, 9117866, 1655147, 1276822]),
(1, [4563397, 7022462, 8275369, 4874325, 1645374]),
(2, [3793055, 6901981, 7093137, 2039387, 1252389]),
(3, [3173753, 2465351, 8636592, 5402520, 7679952]),
(4, [6751103, 2781606, 5385986, 4133449, 156722])],
dtype=[('index', '<i8'), ('values_block_0', '<i8', (5,))])
行列の配列そのままというわけではなく、少し特殊な形になっているようですね・・・
list(f['int_20190601/_i_table'].keys())
['index']
list(f['int_20190601/_i_table/index'].keys())
['abounds',
'bounds',
'indices',
'indicesLR',
'mbounds',
'mranges',
'ranges',
'sorted',
'sortedLR',
'zbounds']
_itableの方はテーブル情報などを格納している、といった感じでしょうか?こうしてみると、普通にh5pyでダイレクトに書き込んだHDF5はDaskでダイレクトに読み込めないのも納得です。
続いて文字列の方も準備します。
以下のように「あ~お」の範囲でランダムな日本語を生成するようにしておきます。
import random
def random_str():
char_num = np.random.randint(low=5, high=100)
return ''.join(random.choices(['あ', 'い', 'う', 'え', 'お'], k=char_num))
random_str()
'えいあいおええいおえおうあういいいいあええいえうおいいうおえお'
文字列の方は1日辺り30万行のデータとして生成しておきます。
target_dt = datetime(2019, 6, 1)
while target_dt <= last_dt:
print(datetime.now(), target_dt)
date_str = target_dt.strftime('%Y%m%d')
name = 'str_{date_str}'.format(date_str=date_str)
str_list = []
for i in range(300000):
str_list.append(random_str())
df = dd.from_array(x=np.array(str_list))
df.to_hdf(
path_or_buf='./Dask_sample.hdf5', key=name)
target_dt += Day()
f['str_20190601/table'][:2]
array([(0, b'\xe3\x81\x88\xe3\x81\x84\xe3\x81\x8a\xe3\x81\x86\xe3\x81\x82\xe3\x81\x88\xe3\x81\x82\xe3\x81\x84\xe3\x81\x88\xe3\x81\x86\xe3\x81\x86\xe3\x81\x8a\xe3\x81\x8a\xe3\x81\x8a\xe3\x81\x84\xe3\x81\x8a\xe3\x81\x86\xe3\x81\x86\xe3\x81\x82\xe3\x81\x84\xe3\x81\x8a\xe3\x81\x88\xe3\x81\x86\xe3\x81\x88\xe3\x81\x82\xe3\x81\x86\xe3\x81\x84\xe3\x81\x86\xe3\x81\x86\xe3\x81\x84\xe3\x81\x8a\xe3\x81\x8a\xe3\x81\x82\xe3\x81\x86\xe3\x81\x84\xe3\x81\x86\xe3\x81\x8a\xe3\x81\x88\xe3\x81\x84\xe3\x81\x88\xe3\x81\x84\xe3\x81\x82\xe3\x81\x86\xe3\x81\x8a\xe3\x81\x86\xe3\x81\x82'),
(1, b'\xe3\x81\x8a\xe3\x81\x84\xe3\x81\x82\xe3\x81\x8a\xe3\x81\x84\xe3\x81\x86\xe3\x81\x88\xe3\x81\x8a\xe3\x81\x88\xe3\x81\x82\xe3\x81\x82\xe3\x81\x88\xe3\x81\x88\xe3\x81\x82\xe3\x81\x86\xe3\x81\x88\xe3\x81\x88\xe3\x81\x8a\xe3\x81\x84\xe3\x81\x8a\xe3\x81\x82\xe3\x81\x84\xe3\x81\x88\xe3\x81\x8a\xe3\x81\x88\xe3\x81\x86\xe3\x81\x82\xe3\x81\x8a\xe3\x81\x8a\xe3\x81\x82\xe3\x81\x86\xe3\x81\x86\xe3\x81\x84\xe3\x81\x84\xe3\x81\x88\xe3\x81\x88\xe3\x81\x8a\xe3\x81\x84\xe3\x81\x88\xe3\x81\x84\xe3\x81\x82\xe3\x81\x82')],
dtype=[('index', '<i8'), ('values', 'S297')])
エンコードされたbytes型で文字列は保存されるようですね。デコードすると普通の文字列が確認できます。
sample_str = f['str_20190601/table'][0][1].decode()
sample_str
'えいおうあえあいえううおおおいおううあいおえうえあういうういおおあういうおえいえいあうおうあ'
とりあえず読み込んでみる
Daskのread_hdfで読み込みができるので、とりあえずデータセット単体を対象として読み込んでみましょう。
df = dd.read_hdf(
'./Dask_sample.hdf5', 'int_20190601')
df.head(3)
| a | b | c | d | e | |
|---|---|---|---|---|---|
| 0 | 7401668 | 898045 | 5748607 | 6887880 | 6220103 |
| 1 | 9096620 | 3875239 | 2957418 | 244275 | 726076 |
| 2 | 8320086 | 5594015 | 3810118 | 8618852 | 3738929 |
ちゃんと読み込めていますね。_itableといった特殊なデータが設定される影響か、カラム名などもちゃんと復元してくれるようです。
続いて文字列のデータセットの方も読み込んでみましょう。1列だけのデータで保存したので、読み込んだ時点だとデータフレームではなくシリーズになっているようです。
sr = dd.read_hdf(
'./Dask_sample.hdf5', 'str_20190601')
type(sr)
dask.dataframe.core.Series
表の表示とかに不便なのでデータフレームに変換しておいて、内容を確認してみましょう。
df = sr.to_frame()
df.head(3)
| 0 | |
|---|---|
| 0 | えいおうあえあいえううおおおいおううあいおえうえあういうういおおあういうおえいえいあうおうあ |
| 1 | おいあおいうえおえああええあうええおいおあいえおえうあおおあうういいええおいえいああ |
| 2 | おあおああえいあううううえあううえああううういい |
ちゃんと表示されています。デコードなどもよしなにやってくれるようですね。
日付のアスタリスク指定のDaskの機能を使って読み込んでみる
DaskにはCSVなどでパスの指定でアスタリスクを指定することで、該当する日付のファイルなどを一括で、並列化しつつ読み込んでくれる機能があります(詳しくは以前書いたPythonのDaskをしっかり調べてみた(大きなデータセットを快適に扱う)で触れています)。
docstringを読む限りHDF5でも対応しているっぽい・・・らしいですが、実際に試してみます。
まずは数値のデータセットで、2019年6月のデータ全体を読み込む想定で指定してみます(日付部分にアスタリスク)。
df = dd.read_hdf(pattern='./Dask_sample.hdf5', key='int_201906*')
len(df)
150000000
500万行ずつの1ヶ月分なので、ちゃんと読み込めていそうですね。
なお、アスタリスク指定はデータセットだけでなくHDF5のファイル名側でも指定できるようです。これなら年ごとにHDF5ファイルを分けたりしてもいいですね。
パーティションは150個で設定されたようです。
df.npartitions
150
タスクグラフを可視化してみます。
df.visualize()
アウトプットが横に長すぎる都合で、縮小されすぎてよく分かりません。
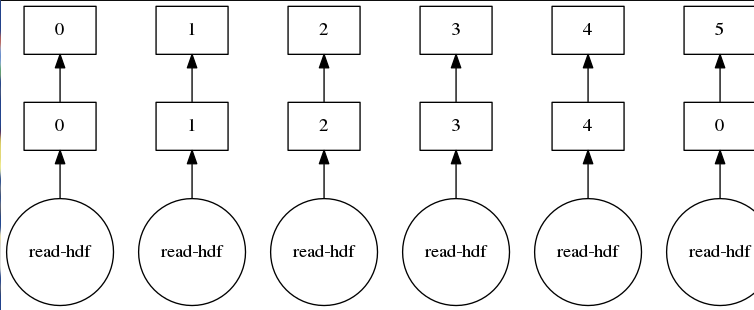
等倍表示してみます。
どうやらパーティションの150個分、タスクが分解されている模様。
これなら普通に並列化してくれていそうですね。
リンクは対応していないっぽい
アスタリスクで複数ファイルを並列化しつつ読み込んでくれることが分かりました。
ただし、これだと年単位や月単位でしか読み込めません。特定の日付範囲(たとえば、7日分だけ読み込みたいとか)だと、処理で無駄が発生してしまいます。
そこで、HDF5でリンク設定をして、その後にアスタリスク指定すればいいのでは?と思ったため試してみました。
が、どうやらDaskではリンクは対応していないっぽいようで・・・エラーになってしまいました。
事前にDaskで保存したHDF5ファイルに対して、別途h5pyでリンク設定をして、Daskで読み込み指定してみた例:
target_date_list = ['20190601', '20190602', '20190603']
with h5py.File('./Dask_sample.hdf5', mode='a') as f:
for date_str in target_date_list:
softlink = h5py.SoftLink('int_%s' % date_str)
f['int_link_%s' % date_str] = softlink
df = dd.read_hdf(pattern='./Dask_sample.hdf5', key='int_link_20190601')
ValueError: No objects to concatenate
Daskの使い方が悪く、もしかしたらちゃんと使う方法がある・・・のかもしれませんが、普通にループを回してconcatでも良さそうなので、ファイルやデータセット個別に対応する際にはconcatしようと思います。
参考 : Load HDF file into list of Python Dask DataFrames
その他気になる点
クラウドストレージをクラウドインスタンスにマウントして使うことがあります(S3をgoofysでマウントしたり、もしくはGoogleDriveなどをColaboratoryにマウントしたりetc)。
そういった場合に、ファイルを扱う際にmode='a'(追記指定)で対応するとgoofysなどで弾かれてしまったような記憶があります。
一方で、HDF5を扱う場合、mode='a'を使うことが多くなる(同一ファイルにデータセットを追加したりする都合)ので、その辺り使えるのだろうか・・・という疑問があります。
その辺りも後日検証できたらと思います。
他にもPythonなどを中心に色々記事を書いています。そちらもどうぞ!
今までに投稿した主な記事たち
参考文献
- Python and HDF5: Unlocking Scientific Data
- What is the fastest way to upload a big csv file in notebook to work with python pandas?
- PyTables: hierarchical datasets in Python
- Installation
- HDF5 TUTORIAL: COMMAND-LINE TOOLS FOR VIEWING HDF5 FILES
- HDF® VIEW
- Parallel HDF5
- Best Practices (DataFrame) - Dask documentation
- Best Practices - Dask documentation
- Pythonを使ってランダムな文字列を生成
- Load HDF file into list of Python Dask DataFrames