PandasやNumPyの並列処理だったり、メモリに乗り切らないデータを扱う際などによく見かけるDaskライブラリ。
ただ、細かいところまで触れている日本語の資料があまり無かったので、公式ドキュメントなどをしっかり読んでみてまとめてみました。
※Daskのドキュメント既に読まれている方はご存知かと思いますが、ドキュメントがかなりのボリュームなのと、細かい所まで把握するのを目的とするため、本記事も長めです。仕事などの都合でさくっと使われたい方には向いておりませんので、そういった場合は別の記事をご参照ください。
どんなライブラリなのか
- Pythonで並列処理・分散処理などを簡単に扱ってくれる。
- Pythonでよく使われるライブラリとかなり近いインターフェイスを提供している(NumPy、Pandas、Scikit-Learnを中心に、他にもTensorFlow・XGBoostなども)。
- 必要な場合には、PySparkなんかでやるような、複数のマシンを用意して分散処理でスケールアップしたり、逆にコストを下げるために気軽にスケールダウンしたりなどができる。
- ※ただし、最初は1つのデスクトップで使い始める人が大半とのこと。
- ※1000台とかくらいであれば、十分効率的に処理してくれるらしい。
- 有名ライブラリだけではなく、Python自体のコードもよしなに、そして簡単に並列化したりしてくれる機能がある。PandasやNumPyのインターフェイスだと対応が難しい際に、独自のコードを書いて例外的な複雑なタスクなどにも柔軟に対応ができる。
- データを全てメモリに乗せず、部分的なかたまり単位でメモリに乗せたりして計算するので、通常はPandasなどでメモリに乗り切らない大きなデータセットでも扱える。
- Daskを経由した後の結果のデータは、NumPy配列やPandasのデータフレームなどになる。そのため、大きなデータの読み込みやスライス、重複削除などの処理を行って行数を減らしてPandasなどで快適に制御できるデータ規模にしてから、さらに細かい計算などはPandasやNumPy配列で継続して行うといったフローが快適に行える。
- NumPyやPandas、Scikit-Learnなどの置き換えとなるライブラリではない。どちらかというとそれらのライブラリをより快適に使えるように拡張するライブラリのようなイメージ。内部でもそれらのライブラリが実際に使われているので、たとえばPandasの改善がそのままDaskにも改善点として反映されたりする。そういった立ち位置であるので、前述のライブラリやJupyterなどのライブラリのコミュニティと密に連携しつつ開発が進められているらしい。
- 2019年現在、Githubのスター数が約5000、contributorの方が250名くらい。現在も頻繁に開発が進められている。
どんなときにDaskが向いているのかの例
Daskも色々便利に使える一方で、銀の弾丸というものでもない。
他の様々なライブラリ同様メリットデメリットあるので、Daskに向いている箇所にDaskを使って、他のもののほうが向いているケースでは他のものを使ったほうがいいと思われる。以下で大雑把に向いている点・向いていない点の特徴をコードに触れていく前に記載(長い記事なので、読むべき記事なのかの判断用として先に向いている点・向いていない点を記載)。
- 普通に扱うとメモリが足りないケース : Pandasなどでは普通は扱えない規模のデータでも、1つのデスクトップでも扱えるようになる。
- 学習コストを抑えたい場合 : NumPy・Pandas・Scikit-Learn・もしくはPython自体のコードで、慣れたインターフェイスで扱えるので、さくっと使い始めることができる。集計などを消化するのが目的で、新しいライブラリを学ぶ手間を減らして他のことに注力したい場合。
- 既にNumPyやPandasなどで書かれたスクリプトなどの資産があり、改善が必要な場合 : それの資産がメモリ的にきつくなってきたり、より高速化が必要になった際に書き直しのコストを低く抑えつつ改善したい場合など。書き直しは、地味で結構しんどい作業なので、そこが軽減されるというのは結構助かるケースがある。
- 1つのPCで最初は進めていたものの、段々分散処理が必要なレベルになってきた場合 : Daskでは1つのマシンでの計算も分散環境での計算も対応しているため、コードの書き直しの手間を少なく抑えつつ途中で切り替えていける。逆に、スケールダウンしてコストを下げたりもやりやすいらしい。
- 大量のデータに対して、データを何度も確認したり、調整したりと、探索的に扱う必要がある場合 : BigQueryやAthenaなどでSQLで扱うよりかは、Pandasなどの感覚でPythonで扱ったほうが長い計算などでは調整がしやすかったり、SQLが複雑になりすぎたりを避けられる。
-
データサイエンティストのノートなどをエンジニアが負荷を下げつつ本番に反映したい場合 :
データサイエンティストからNumPyやPandas、Scikit-Learnなどを使ったJupyterノートをもらって、AIエンジニアもしくはデータエンジニアが本番環境に乗せていく際に、コードをなるべくそのままにしつつ、且つ本番への影響を少なくするためにチューニングやメモリ使用量を低く抑えたい場合。 - データが多くのCSVやHDF5などの様々なフォーマットで保存されている場合 : Pandas同様、読み込みに様々なフォーマットに対応している。また、複数のファイルを個別に読み込んで後でpd.concatなどしなくても、1行書くだけで並列で実行してくれて、concatなどの手間も省いてくれたりなど、ローカルで時系列などで多くのファイルがある場合にも快適に処理ができる。
Daskが向いていないかもしれないケースの例
- 計算が特に複雑にはならず、SQLだけで十分な場合 : 分散環境を用意するコストや、速度や管理の楽さなどを考えると、普通にBigQueryやAthenaなどでいい印象がしている。ただし、どちらか一方ではなく、たとえばS3にCSVとかでパーティション設定しやすいディレクトリ構成で置いておけばDaskでもAthenaでもどちらでも効率的に扱えるので、タスクに応じて使用するものを切り替えたりもしくは併用したりできる形にしておくというのも好ましいかもしれない。
- 既存のPandasなどのコード資産などは関係なく、且つデータがHDF5などで扱えて、速度が重要な場合 : 恐らくvaexなどのほうが向いている。速度自体はvaexの方が大分速そうな印象がある。(vaexはキャッチアップできていないので詳細は割愛・・HDF5のオライリー本は注文したので、もし機会があれば将来まとめる・・かも)
- 1つのマシンで扱う必要があり、しかしDaskでも扱いきれないレベルのデータセットの場合 : Pandasなどに比べればかたまり単位で部分的にメモリに乗せられて処理されるので、大分省メモリで扱える一方で、かたまりを細かく分割しすぎると分割によるオーバーヘッドでDaskでもまともに扱えなくなってくる。そういった場合はBigQueryなどに頼るか、分散処理を検討するか、他のライブラリを検討したりなどが必要になってくる。合計100GBのCSVなどであれば余裕で1つのPCで扱える一方で、合計1TBを大きく超えるようなCSVなどであればどうしてもメモリをたくさん積んだPCじゃないと効率面で厳しくなってくる。
- そもそも小規模なデータセット : 余裕でメモリに乗り切る、小さいデータなどであればDaskを使わずにそのままPandasやNumPyなどを利用するのが好ましいケースも多い。Pandasなどと似たようなインターフェイスといっても、ある程度はDaskを使うことで複雑になったり無駄にオーバーヘッドが絡んできたりするので、闇雲にDaskに書き換えるのではなく、特にデータ量的に問題なく、且つ今後も日次のデータなどが増加していかないデータであればシンプルにPandasなどを使う。
- 単純に高速化のみが必要な場合 : メモリなどは特に問題にはならないものの、計算などの高速化のみが必要な場合には、もしかしたらCythonやNumba、PyPyなどの方が向いているかもしれない。もしくは適したデータ構造を選択すれば、Daskを導入しなくても十分要件を満たせるかもしれない。(参考 : Pythonを速くするTIPS集(計測・ビルドインの各機能・Cython・Numba・etc))
Daskの制限
試してはいないものの、1つのマシンで50コアを超えるレベルになってくると、スケールがそれ以上はできないかも?という印象の記述がドキュメントにあった。
They scale their algorithm between 1 and 50 cores on single workstations
公式ドキュメント Use Casesより。
そこまでのマシンを使う機会はほぼなさそう(最近(2019年)発表されたMac ProでもMax28コアくらい?)ではあるものの、クラウドインスタンスなどを利用する場合はもしかしたら引っかかるかもしれない。
それ以上に高速化などが必要であれば、1つのマシンをとにかくスペックアップするのではなく、複数のマシンでのDaskの分散処理などを検討する必要がある。
本記事で扱うDaskの内容
- Pandasのインターフェイス(Dask DataFrame)
- NumPyのインターフェイス(Dask Array)
- Python自体のインターフェイス(Dask Delayed)
- DaskのパフォーマンスTIPS、プロファイリング
- デバッグTIPS
本記事で扱わないDaskの内容
- Pythonのインターフェイスの中で、Bagsの詳細だったり、即時での計算が走るタイプのもの(Futures)など。
- Scikit-Learnなどの機械学習関係
- 機械学習関係を除いてもドキュメントは結構なボリュームになっており、機械学習まで手を出すと相当長くなりそうな気配がしたため今回は割愛。
- 分散処理関係(Dask Distributed)
- ※今のところは1つのマシンで使う予定なのと、ほとんどの方が1つのマシンで動かすと思うため。必要になってきたら調べて別の記事に対応。
サンプルコードなどで使う環境
- Python 3.6.1 :: Anaconda 4.4.0 (64-bit)
- Ubuntu
- Jupyter notebook
- Dask==1.2.2
※Windowsでは一部動かないケースがあるかもしれない。
インストール
Anacondaを使っている場合は最初から入っている(そもそもAnaconda自体がDaskのメインのスポンサーだったり、コピーライトを見るとAnacondaの表記がある)。
ただし、0.x.x系と1.x.x系だと結構インターフェイスが違う(0.x.x系に無いインターフェイスなども多い)。
確認してみて、もし古ければアップデートしたほうが無難な印象がある。
バージョン確認方法 :
import dask
dask.__version__
'0.14.3'
もしMinicondaなどを使っているケースなどで、Daskが入っていないケースであれば以下のようにインストールすることができる。
$ conda install dask
pipであれば以下のように指定する。
$ pip install "dask[complete]"
※バージョンによってはNumPyやPandasなどにも更新がかかるので、変更したくない場合は注意が必要。事前に別途Anaconda環境を用意するか、影響が出ないDaskの古いバージョンを選択するなどが必要になってくる。
[complete]といったように、特殊な指定になっている。
これは、依存しているライブラリなどを一通りインストールする指定となっている。pip install daskとすると、dask自体の最低限のものしかインストールされない(動かないモジュールが結構出てくる)ので注意。依存するものが色々あるので、基本的には手動で依存関係を解決せずにDaskに任せたほうが無難な印象がある。
[complete]のほかにも、以下のように一部だけインストールする選択肢が用意されている。ただし、基本的には本番環境のディスク容量を少しでも節約したいとかでなければ基本的に[complete]でいいような気がしている。
-
NumPyのインターフェイスのみをインストールする :
$ pip install "dask[array]" -
Pandasのインターフェイスのみをインストールする :
$ pip install "dask[dataframe]" - etc...
インストールバージョンを指定したい場合には、通常のpipと同様に==で指定することで設定できる。
$ pip install "dask[complete]"==1.2.2
※今回は1.2.2を使っていくので、上記のコマンドで対応している。
importの慣習
NumPyのモジュールを、短くas npといったようにするのと同様、Daskでもドキュメントを見ていた感じでは、以下のように統一されてimportのasの記述がされていた。
-
Dask 配列 : Dask Arrayということで、daとなっている。
import dask.array as da -
Dask データフレーム : Dask DataFrameということで、ddとなっている。
import dask.dataframe as dd -
Bag : Dask Bagということで、dbとなっている。
import dask.bag as db
Daskの計算の特長
通常のPandasやNumPyなどでの計算と異なり、計算のコードで即時で計算が走らない。
たとえば、NumPyのインターフェイスと同じように、以下のように書いてもこの時点ではデータが用意されたり、計算が走ったりはしない。
import dask.array as da
x = da.random.uniform(low=0, high=1.0, size=(300000000, 100))
y = x.max(axis=0).max()
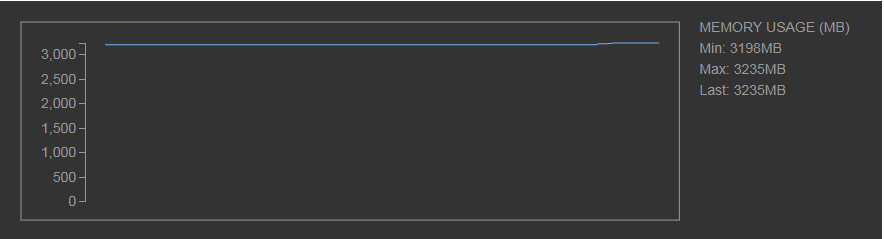
3億行100列という、普通にPandasやNumPyで扱うとPCのスペックによってはひやっとする規模の行列を指定してもこの時点ではメモリが急増したりはしない。
必要な計算を書き終わって、computeメソッドを呼び出した時点でデータが確保され、計算がまとめて走り始める。
y.compute()
上記のように、computeメソッドが呼ばれた時点でメモリが変動し始める。
また、データをある程度のかたまりで分割して、そのかたまり単位でメモリに乗って計算がされていく(NumPyインターフェイスではチャンク(chunk)、Pandasインターフェイスではパーティション(partition)という単語でよく出てくる)ので、一度に全てをメモリに乗せるNumPyやPandasと比べると瞬間的なメモリはぐっと低く抑えられる。
前述のサンプルでは、3億行100列のデータでも、タイミングである程度ばらつくものの、大体1GB弱程度のメモリで処理ができている。
このcomputeのインターフェイスはDaskの各機能のインターフェイスで共通で存在する。
ただし、NumPyインターフェイスを使った場合はcompute後の返却値はNumPyの配列となり、Pandasインターフェイスを使った場合はPandasのデータフレーム、Pythonのインターフェイスを使った場合はintなどのPythonオブジェクトになるという違いがある。
過程で使われたDaskのデータは計算が終わってNumPyやPandasのデータになった時点で、そのデータのみのメモリが確保される。
つまり、Daskの計算の過程でデータの読み込みからスライスなり重複削除などをすることで、最終的に確保されるメモリを小さくすることができる。
このようにPandasのデータフレームやNumPyの配列を小さくするまでの箇所だけDaskで置き換えれば、その後のコードはそのままPandasやNumPyのままでも快適に計算などを行なうことができる。(既存のコードの置き換えがやりやすい)
※逆にいえば、最終的にメモリに展開されるPandasのデータフレームや、NumPy配列がメモリに収まらないサイズのままだと、Daskを経由しても結局メモリが足りなくなる。Dask上で計算やスライスなどを行なって、最終的なデータを小さくするのが大事。
なぜDask即時で計算が走らないようになっているのか、という点は、以下のような理由からそうなっている。
- 必要な計算を書いていった時点で、computeメソッドの前でどんな感じのタスクグラフ(Dask上の計算グラフのようなもの。Daskでタスク単位でグラフに表示される)が必要なのかが確定する。
- その確定したタスクグラフを参照して、Dask側でどのように並列化すると効率がいいのか、どんな感じにデータのかたまりを分けたほうがいいのかが判定できる。結果として、計算が効率的に実行される。
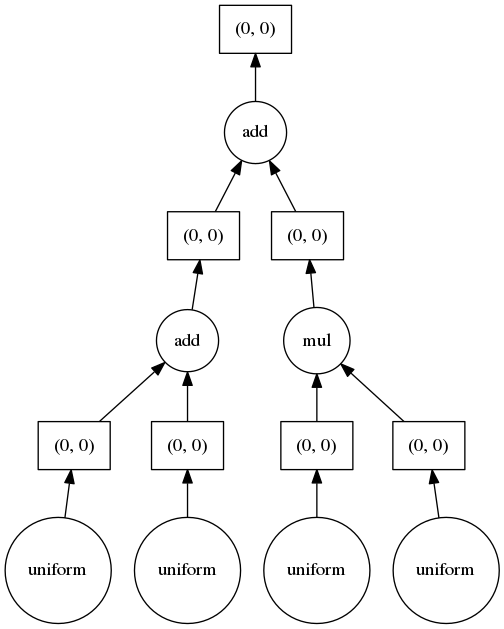
これだけだと分かりづらいので、実際にDaskのタスクグラフの可視化の機能(後述)を使って見てみると、並列化などがどのようにされるのかがイメージがつきやすい。
x1 = da.random.uniform(low=0, high=1.0, size=(100, 100))
x2 = da.random.uniform(low=0, high=1.0, size=(100, 100))
x3 = da.random.uniform(low=0, high=1.0, size=(100, 100))
x4 = da.random.uniform(low=0, high=1.0, size=(100, 100))
y = x1 + x2 + x3 * x4
result_arr = y.compute()
上記のコードは以下のようなタスクグラフになる。
タスクグラフを見ると、computeの時点で必要な計算では、最初は4つで並列処理をして、その後2つ、最後に1つのノードが必要なことが分かる。
1行ずつ、Pandasのように即時で計算してしまうと、どのように並列化すべきなのかが分からない。
ただし、即時で計算がされたほうが便利なときもあるので、そういった場合のための機能が用意されていたり、もしくはcomputeを複数挟む(中間データを挟む)という使い方もできる(その分のPCリソースを必要としたりはする)。
Dask配列(NumPyインターフェイス)
以降では、いくつかの節に渡ってNumPyインターフェイスのDask配列(Dask Array)に関して記載。
Dask配列は以下のような特長がある。
- 行列よりも次元が深いテンソルなどで、サイズがメモリに収まりきらないデータに対して計算が行なえる。
- 構成としては、以下のようにいくつかのNumPy配列をグリッドとして配置された状態で構成される。このグリッドの単位はかたまりという意味のチャンク(chunk)という単語で引数などでよく出てくる(パフォーマンスを考える上で重要になってくるので、詳細は後述)。
Dask配列でサポートしているものの例
-
基本的な演算処理 :
+や%のオペレーターなどでの基本的な計算。
import dask.array as da
arr_1 = da.from_array(x=[1, 2, 3])
arr_2 = da.from_array(x=[4, 5, 6])
arr_3 = arr_1 + arr_2
arr_3.compute()
array([5, 7, 9])
-
要約統計量関係 :
sumやmeanやstdなどの関数。
arr_1 = da.from_array(x=[1, 2, 3])
y = arr_1.sum()
y.compute()
6
- 内積や行列の乗算など :
arr_1 = da.from_array(x=[1, 2, 3])
arr_2 = da.from_array(x=[4, 5, 6])
y = da.dot(a=arr_1, b=arr_2)
y.compute()
32
- 転置 :
arr_1 = da.from_array(
x=[[1, 2, 3],
[4, 5, 6]])
y = arr_1.T
y.compute()
array([[1, 4],
[2, 5],
[3, 6]])
- スライス関係 :
arr_1 = da.from_array(x=[1, 2, 3, 4])
y = arr_1[arr_1 >= 3]
y.compute()
array([3, 4])
- その他 : APIのドキュメントページに、サポートされている関数などが膨大に記載されている。NumPy自体がAPIが色々あるので、Dask側でもAPIが膨大になっている。
前述の通り、一部computeやNumPy配列などからも生成できるfrom_arrayなどのDask特有のものがある(da.from_arrayはnp.arrayとほぼ似たような感覚ではある)ものの、大体のインターフェイスはNumPyそっくりで、返却値の挙動も馴染みがあるので、NumPyに慣れている方ならすぐに使いこなせる印象がある。
Dask配列で対応していないものの例
-
一部を除いた線形代数関係 :
da.linalg関係は、NumPyと比べると多くのものがサポートされていないらしい。NumPy内部で使われているBLASなどのライブラリが絡んで、あまりサポートされていないとのこと。 - sortなどの処理 : 膨大なデータでのフルソートなどは制限がされつつ、ソート後の上位の値を部分的に取ったりなどはサポートされている。
- tolistなどの関数 : そもそも膨大なデータで過程でPythonの配列に変換するとメモリが死にそうなので使わない気もする。compute関数の後がNumPy配列などになるので、必要であればそちらから変換すれば大丈夫そうではある。
- その他のあまり使われないマイナーな関数など
Dask配列とチャンクサイズの話
前述の通り、Dask配列の内部ではチャンクサイズ単位での多くのNumPy配列で構成されている。
このチャンクサイズをどのように設定するかによって、パフォーマンスが変わってきたりするので、頭の片隅に入れておくと「なんだかやけに遅い」みたいなことを避けることができる。
基本的に、Dask配列の生成処理などの関数では、チャンクサイズを指定するchunksという引数で、チャンクサイズを指定することができる。チャンク数ではないので注意(chunk shape的な意味でのchunksという引数名であり、chunk_num的な意味ではない)。
チャンク数を1つだけにしたいと考えて、chunks=1などと指定すると、スカラー値ごとにチャンクが作られて、オーバーヘッドで相当遅くなるので注意。
なお、デフォルト値ではchunks='auto'となっている。
Dask側でよしなに決めてくれるので、細かいチューニングが面倒であればautoが事故りにくくて無難そうな印象はしている。
手動で指定する場合には、以下のような点を参考にすると良い。
- チャンクサイズは大きすぎても小さすぎても良くない : データ量に対して大きすぎるとメモリが多く必要になる。小さすぎると大量の分割による都合で、オーバーヘッドで速度が犠牲になる。
-
チャンクサイズは基本的に100MB以上のメモリサイズを目安にする : それ以下になると、扱うデータサイズによっては分割が多すぎてオーバーヘッドが大きい。float64などの値であれば必要メモリが8バイトなので、行列であれば
(4000, 4000)くらいのサイズで、4000 * 4000 * 8 / 1000 / 1000 = 128MBといったように100MBを超えてくる。 -
チャンク単体のメモリ × 10 ×コア数で計算したときに、PCのメモリを超えないようにする : 基本的に瞬間的な計算ではチャンク単体で扱われる。コアの数だけ並列でメモリに確保されたりするので、チャンク単体のメモリ量 × コア数のメモリが最低限必要になる。また、効率化的に読み込み待ちなどを避けるため、Dask側で近いうちに必要になるチャンクを計算中に読み込んだりするらしく、チャンク10個分くらいは最大で確保される可能性があるらしい(そこまではほぼいくことは無いとは思われる)。そのため、たとえばチャンク単体で100MBのメモリサイズのデータで、コアを4つ積んでいるマシンで計算する場合には、
100 * 10 * 4 = 4000MBとなり、4GBくらいはマシンにメモリが必要になってくる。メモリ32GBのマシンであれば、チャンク単体のメモリサイズを800MBくらいまでは無難に扱えることになる。 - チャンク分割によるオーバーヘッドに配慮する : マシンスペックに依存するものの、一つのチャンクを計算などで扱う際のオーバーヘッドが1ミリ秒程度かかったりするらしい。細かくチャンクを分割しすぎるとこのオーバーヘッドが結構響いてくる。まあでも、たとえばfloat64の型で500万行10列のサイズの行列でチャンクサイズを設定した(チャンク単位のメモリ400MBくらい)としても、チャンク500個で25億行とかの規模なので、1つ1ミリ秒程度なら少し待ったりしていれば十分対応できそうな気もしている。それよりもはるかにデータサイズが大きくなってきたら流石に分散環境などを使ったほうが快適そうな印象はある。
-
行列などが正方行列ではない場合はその形に合わせたチャンクサイズにする : たとえば、
(10000000, 10000)といったようなサイズの行列を扱う場合には、チャンクサイズの方もそちらに合わせて長方形で指定するのが好ましい((1000000, 1000)といった具合に)。
チャンクサイズとファイル単位
もし可能であればチャンクサイズ単位でファイルが分割されていると、読み込みが並列でできて且つチャンクの設定がスムーズで好ましいらしい。
チャンクのauto時の挙動と手動でのサイズ指定時の挙動
どのようにチャンクサイズが指定されているかは、chunksize属性で確認ができる。
arr = da.random.uniform(low=0.0, high=1.0, size=(10000000, 1000))
arr.chunksize
(16000, 1000)
chunksの引数を省略したので、デフォルトのchunks='auto'の
16000 * 1000 * 8 / 1000 / 1000 = 128MBといったように、100MBを超えるチャンクサイズで自動で設定されていることが分かる。(且つ、行方向の方が長い行列を指定したので、チャンクサイズも行方向が長いサイズになっているのが分かる)
以下では、上記のデフォルトのchunks='auto'ではなく手動で指定した際の分割の挙動に関して記載していく。
サンプルとして、10 × 10の行列を利用する。
arr = da.arange(0, 100)
arr = arr.reshape((10, 10))
arr.compute()
array([[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9],
[10, 11, 12, 13, 14, 15, 16, 17, 18, 19],
[20, 21, 22, 23, 24, 25, 26, 27, 28, 29],
[30, 31, 32, 33, 34, 35, 36, 37, 38, 39],
[40, 41, 42, 43, 44, 45, 46, 47, 48, 49],
[50, 51, 52, 53, 54, 55, 56, 57, 58, 59],
[60, 61, 62, 63, 64, 65, 66, 67, 68, 69],
[70, 71, 72, 73, 74, 75, 76, 77, 78, 79],
[80, 81, 82, 83, 84, 85, 86, 87, 88, 89],
[90, 91, 92, 93, 94, 95, 96, 97, 98, 99]])
- スカラーの値を指定した場合(
chunks=5など)、各方向に対してその数で分割される。行列であれば5 × 5のブロックで分割、階数3のテンソルであれば5 × 5 × 5といった感じで分割される。
設定後のサイズはchunks属性で確認ができる。((行の各チャンクのサイズ), (列の各チャンクのサイズ))といったように表示される。
また、チャンクサイズを再調整する場合にはrechunk関数を使う。
y = arr.rechunk(chunks=5)
y.chunks
((5, 5), (5, 5))
可視化すると、以下のように分割されている。
[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9],
[10, 11, 12, 13, 14, 15, 16, 17, 18, 19],
[20, 21, 22, 23, 24, 25, 26, 27, 28, 29],
[30, 31, 32, 33, 34, 35, 36, 37, 38, 39],
[40, 41, 42, 43, 44, 45, 46, 47, 48, 49],
[50, 51, 52, 53, 54, 55, 56, 57, 58, 59],
[60, 61, 62, 63, 64, 65, 66, 67, 68, 69],
[70, 71, 72, 73, 74, 75, 76, 77, 78, 79],
[80, 81, 82, 83, 84, 85, 86, 87, 88, 89],
[90, 91, 92, 93, 94, 95, 96, 97, 98, 99]]
- タプルなどで、例えば
(5, 2)といったように指定した場合、行方向・列方向...といった感じで、その個数分で分割される。
y = arr.rechunk(chunks=(5, 2))
y.chunks
((5, 5), (2, 2, 2, 2, 2))
列方向のサイズを小さくしたので、列方向が5つに分割されていることが分かる。
以下のように分割されている。
[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9],
[10, 11, 12, 13, 14, 15, 16, 17, 18, 19],
[20, 21, 22, 23, 24, 25, 26, 27, 28, 29],
[30, 31, 32, 33, 34, 35, 36, 37, 38, 39],
[40, 41, 42, 43, 44, 45, 46, 47, 48, 49],
[50, 51, 52, 53, 54, 55, 56, 57, 58, 59],
[60, 61, 62, 63, 64, 65, 66, 67, 68, 69],
[70, 71, 72, 73, 74, 75, 76, 77, 78, 79],
[80, 81, 82, 83, 84, 85, 86, 87, 88, 89],
[90, 91, 92, 93, 94, 95, 96, 97, 98, 99]]
- その他にも、chunksに指定するタプルの次元を増やして、個別に指定するような方法も使える。ただし、ほとんど使われないとのこと(確かに、あまりメリットが思いつかない・・)。
y = arr.rechunk(chunks=((2, 2, 3, 3), (3, 7)))
y.chunks
((2, 2, 3, 3), (3, 7))
[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9],
[10, 11, 12, 13, 14, 15, 16, 17, 18, 19],
[20, 21, 22, 23, 24, 25, 26, 27, 28, 29],
[30, 31, 32, 33, 34, 35, 36, 37, 38, 39],
[40, 41, 42, 43, 44, 45, 46, 47, 48, 49],
[50, 51, 52, 53, 54, 55, 56, 57, 58, 59],
[60, 61, 62, 63, 64, 65, 66, 67, 68, 69],
[70, 71, 72, 73, 74, 75, 76, 77, 78, 79],
[80, 81, 82, 83, 84, 85, 86, 87, 88, 89],
[90, 91, 92, 93, 94, 95, 96, 97, 98, 99]]
基本的にはスカラーでchunks=4000といった指定か、シンプルなタプルでchunks=(8000, 2000)といったような指定が多いのではという印象。
チャンクサイズの再設定(rechunk関数)は、比較的効率的に処理されるらしい。あまり頻繁に実施するのも微妙かもしれないとは思う一方で、スライスなどでデータの件数が結構変動し、チャンクが細かくなってしまった際などには再調整したほうが効率的に計算が行なえるケースが出てくる。
自動のチャンクサイズ設定の補足
-
chunks='auto'といった指定の他に、タプルで一部の次元だけautoを指定することができる。
arr = da.random.uniform(size=(1000000, 1000), chunks=(20000, 'auto'))
たとえば、上記のように行方向だけ固定のチャンクサイズを指定して、列方向はautoと指定すると、Dask側が列方向だけ良い感じにサイズを選択してくれる。(NumPyでいうところのshape属性的にDaskのチャンクサイズを見たい場合にはchunksize属性で確認ができる。)
arr = da.random.uniform(size=(1000000, 1000), chunks=(20000, 'auto'))
arr.chunksize
(20000, 500)
- チャンクサイズに
-1を指定すると、その次元はチャンクの制御を行なわない(全体のデータを利用する)ようにする、という指定になる。NumPyのreshapeなどだと、-1の指定がDaskの'auto'に近い挙動になるので注意。
以下のように、-1を指定した次元は、そのDask配列のサイズと同じ値(データ全体)のチャンクとなる。
arr = da.random.uniform(size=(1000000, 1000), chunks=(20000, -1))
arr.chunksize
(20000, 1000)
- rechunkなどでチャンクサイズを調整する際に、一部の次元にNoneを指定すると、Noneが指定されていない次元のみ変更される。行方向はそのまま、列方向側だけ変更したいといったときに便利。
arr = da.random.uniform(size=(1000000, 1000), chunks=(50000, 500))
arr.chunksize
(50000, 500)
arr = arr.rechunk((None, 1000))
arr.chunksize
(50000, 1000)
-
'auto'を指定した際のサイズ上限は、dask.config.get('array.chunk-size')で確認ができる。
import dask
dask.config.get('array.chunk-size')
'128MiB'
この記事を書いているDaskのバージョンでは、デフォルトでは128MBになっている模様。ただし、前述の計算で必要メモリ量とPCのスペックを考えると、128MBだと少し小さい気もする。ノートPCでメモリが少ないケースや、CPUの数の割りにメモリが少ないケース、もしくは他にも色々な処理でメモリを確保しなければいけないなどのケースを除いて、メモリ設定を大きくしたほうが速度は速くなるので、必要に応じて調整を行なう。
以下のように、configの値の変更前後で、'auto'指定時のチャンクサイズが変わっている。
import dask
dask.config.get('array.chunk-size')
'128MiB'
arr = da.random.uniform(size=(1000000, 1000), chunks=(20000, 'auto'))
arr.chunksize
(20000, 500)
dask.config.set({'array.chunk-size': '256MiB'})
arr = da.random.uniform(size=(1000000, 1000), chunks=(20000, 'auto'))
arr.chunksize
(20000, 1000)
DaskのデータフレームからDaskの配列に変換する際の注意点
- 基本的にDaskのデータフレーム(後述)から配列へ変換する処理は、Pandasのような感覚で扱える。
to_dask_arrayメソッドやPandasのようにvalues属性でもアクセスができる。
import pandas as pd
import dask.dataframe as dd
df = pd.DataFrame(data=[{
'a': 1,
'b': 2,
}, {
'a': 3,
'b': 4,
}])
ddf = dd.from_pandas(data=df, npartitions=1)
arr = ddf.to_dask_array()
arr.compute()
array([[1, 2],
[3, 4]])
ただし、DaskのデータフレームはPandasのデータフレームと異なり、データを全てメモリ上には展開しない都合、そのままだとチャンクサイズの行数が欠落する(チャンク関係の処理がされない)。このままだと、メモリ量が多く必要になったり並列化などの面で配列の計算が好ましくない。
arr.chunksize
(nan, 2)
これを避けるには、to_dask_arrayなどのメソッドの引数でlengths=Trueと指定すると、行数などをちゃんと取得して、配列のチャンクサイズに設定してくれるようになる。ただし、行数算出で変換自体は計算が走るので負荷は高いらしい。もし配列が大きく、処理も多いのであれば恐らくlengths=Trueを、そうではなくて配列操作などをほとんどしないのであればデフォルトのままlengthsの引数を指定しない形がいいのだろうか・・という印象。
arr = ddf.to_dask_array(lengths=True)
arr.chunksize
(2, 2)
一部、Daskにscipyのstatsモジュールのインターフェイスがある
Dask配列の中に、dask.array.statsモジュールとして、scipyのインターフェイスのものが用意されている。scipyの全てはないものの、Daskでそのまま利用できるので、もしかしたら便利なときがあるかもしれない。
Daskデータフレーム
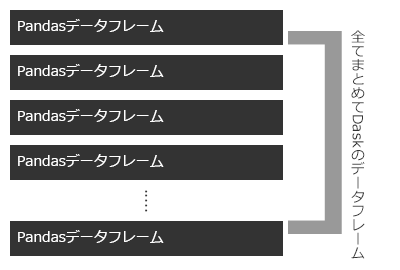
Daskのデータフレームは、Dask配列が多くのNumPy配列で構成されるのと同様、多くのPandasのデータフレームで構成される。
Dask配列の時は分割の単位がチャンクと呼ばれていたが、こちらはパーティション(partition)という名称で分割単位が呼ばれる(引数名なども含め)。
※Dask配列のように列方向での分割などはできない。
他のDaskのインターフェイスと同様に、必要な計算を書くとタスクグラフが生成され、最後にcomputeを読んで計算を実行する。実行後はPandasのデータフレームなどで返却される。
Daskデータフレームでのファイルの読み込み
Pandasで良く使うread_csvなどの各読み込みの関数はDaskでも用意されている。
サンプルコード用として、時系列のデータを想定して、以下のように3つのファイルを事前に用意した。
import pandas as pd
import numpy as np
df_20190601 = pd.DataFrame(columns=['date', 'price'], index=np.arange(0, 1000000))
df_20190601.date = '2019-06-01'
df_20190601.price = np.random.randint(low=80, high=150, size=(1000000,))
df_20190602 = df_20190601.copy()
df_20190602.date = '2019-06-02'
df_20190602.price += 10
df_20190603 = df_20190602.copy()
df_20190603.date = '2019-06-03'
df_20190603.price += 10
df_20190601.to_csv('./2019-06-01.csv', index=False)
df_20190602.to_csv('./2019-06-02.csv', index=False)
df_20190603.to_csv('./2019-06-03.csv', index=False)
df_20190601.head(3)
| date | price | |
|---|---|---|
| 0 | 2019-06-01 | 139 |
| 1 | 2019-06-01 | 98 |
| 2 | 2019-06-01 | 127 |
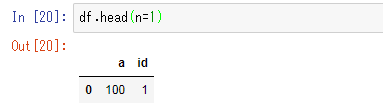
- 単一のファイルの読み込みは何も考えなくても、Pandas感覚で行なえる。
df = dd.read_csv('./2019-06-01.csv')
df.head(n=1)
| date | price | |
|---|---|---|
| 0 | 2019-06-01 | 139 |
- 特記すべき点として、read_csvなどの読み込み系の関数がアスタリスクの表記を使うことで、複数ファイルに対応しているという点が上げられる。最初にDaskのデータフレームを触り始めた際に大分感動した。特殊な書き方をしなくても、並列化のコードを書いたりしなくても、concatなどの関数を挟まなくても、read_csvの1行書くのみで並列化などまでしつつ複数ファイルの読み込みがされる。これだけでもなんだかDaskを学んでも良かったと思える(私だけ?)。
たとえば、時系列のCSVがあるとして、ファイル名に%Y-%m-%dのフォーマットで保存されているとする。
もし対象の月のデータを一括で読み込みたければ、以下のように日の部分をアスタリスクにすると、該当するファイルを全て読み込んでくれる。concatなども不要。
df = dd.read_csv('./2019-06-*.csv')
df = df.compute()
len(df)
3000000
df.date.unique()
array(['2019-06-01', '2019-06-02', '2019-06-03'], dtype=object)
用意したCSV3つ分が全て読み込まれていることが分かる。
対象の年を指定したい場合には、月の部分もアスタリスクを指定すると、その年のファイルを全て読み込むことができる。
たとえば、2019年のファイルを全て読み込みたいのであれば、dd.read_csv('./2019-*-*.csv')というように指定する。
このような複数ファイルのアスタリスクの指定は、ディレクトリ名などでも動作した。そのため、たとえばS3にHiveフォーマットでCSVなどを配置しておいて、同一のDWHにしつつ必要なタスクに応じてAthenaやDaskなどを使い分ける、といったことも選択肢にできそうではある。(AthenaをBigQuery的に使いつつ、複雑な計算などをgoofysなんかでAWSインスタンスにマウントして、Daskで扱ったりなど。)
参考 :
Daskのデータフレームでサポートしていて、且つ速いインターフェイスの例
-
要素ごと(Element-wise)の操作 : データフレーム同士の
+や*などの計算など。
df_1 = dd.from_pandas(pd.DataFrame(data=[{
'a': 100,
'b': 200,
}]), npartitions=1)
df_2 = dd.from_pandas(pd.DataFrame(data=[{
'a': 300,
'b': 400,
}]), npartitions=1)
df_3 = df_1 + df_2
df_3.compute()
| a | b | |
|---|---|---|
| 0 | 400 | 600 |
- スライス関係 :
df = dd.from_pandas(pd.DataFrame(data=[{
'a': 100,
'b': 200,
}, {
'a': 300,
'b': 400,
}]), npartitions=1)
df = df[df.a >= 200]
df.compute()
| a | b | |
|---|---|---|
| 1 | 300 | 400 |
- locでのインデックスを用いた要素(もしくは要素の範囲)の選択 :
df = dd.from_pandas(pd.DataFrame(data=[{
'a': 100,
'b': 200,
}, {
'a': 300,
'b': 400,
}, {
'a': 500,
'b': 600,
}]), npartitions=1)
df = df.loc[1:2]
df.compute()
| a | b | |
|---|---|---|
| 1 | 300 | 400 |
| 2 | 500 | 600 |
- 要約統計量などの算出 : データフレーム全体でも、行単体や列単体(Dask Series Structureという表示がされたので、Daskでもシリーズと呼んで問題なさそう)でも動作する。
df = dd.from_pandas(pd.DataFrame(data=[{
'a': 100,
'b': 200,
}, {
'a': 300,
'b': 400,
}]), npartitions=1)
sr = df.max()
sr.compute()
a 300
b 400
dtype: int64
- isinなどでのデータフレームスライス処理 :
df = dd.from_pandas(pd.DataFrame(data=[{
'a': 100,
'b': 200,
}, {
'a': 300,
'b': 400,
}, {
'a': 500,
'b': 600,
}]), npartitions=1)
df = df[df.a.isin([300, 500])]
df.compute()
| a | b | |
|---|---|---|
| 1 | 300 | 400 |
| 2 | 500 | 600 |
- groupbyと、その結果の要約統計量の算出 :
df = dd.from_pandas(pd.DataFrame(data=[{
'a': 100,
'date': '2019-06-10',
}, {
'a': 300,
'date': '2019-06-10',
}, {
'a': 400,
'date': '2019-06-10',
}, {
'a': 500,
'date': '2019-06-11',
}]), npartitions=1)
grouped_df = df.groupby(by='date')
df = grouped_df.max()
df.compute()
| a | |
|---|---|
| date | |
| 2019-06-10 | 400 |
| 2019-06-11 | 500 |
※groupbyした結果に対して、applyなどを実行する場合はインデックスを事前に設定しておいて、groupbyなどの対象にインデックスを含めないと遅いという記述がドキュメントにある。インデックス関係は、Pandasのときと比べてパフォーマンスに大きく絡んでくる模様。このあたりの感覚はPandasとぼちぼち異なるので注意。MySQLなんかのインデックス云々の話に近い感覚を受けた。
- 値の件数のカウント :
df = dd.from_pandas(pd.DataFrame(data=[{
'a': 100,
}, {
'a': 300,
}, {
'a': 300,
}, {
'a': 500,
}]), npartitions=1)
sr = df.a.value_counts()
sr.compute()
300 2
500 1
100 1
Name: a, dtype: int64
- 重複削除 :
df = dd.from_pandas(pd.DataFrame(data=[{
'a': 100,
}, {
'a': 300,
}, {
'a': 300,
}, {
'a': 500,
}]), npartitions=1)
df = df.drop_duplicates(subset=['a'], keep='first')
df.compute()
| a | |
|---|---|
| 0 | 100 |
| 1 | 300 |
| 3 | 500 |
-
インデックス同士の連結 : ※インデックスを利用しない連結はかなり遅くなりやすいので、Pandasの感覚でmergeなどを行なうと一気に遅くなるケースがあるので注意。
left_indexとright_indexにTrueを指定したりといった引数設定をすると、連結時にインデックスを利用した連結となる。ただし、インデックスの設定自体は遅いので、最初に1回だけインデックスを設定するなどして対応するのが好ましい。なお、片側のデータフレームのみインデックスを参照できる場合には、インデックスのパフォーマンス改善の恩恵を半分だけ享受することができる。
df_1 = dd.from_pandas(pd.DataFrame(data=[{
'id': 1,
'a': 100,
}, {
'id': 2,
'a': 300,
}]), npartitions=1)
df_2 = dd.from_pandas(pd.DataFrame(data=[{
'id': 1,
'b': 200,
}, {
'id': 2,
'b': 400,
}]), npartitions=1)
df_1 = df_1.set_index(other='id')
df_2 = df_2.set_index(other='id')
merged_df = dd.merge(
left=df_1, right=df_2,
left_index=True, right_index=True)
merged_df.compute()
| a | b | |
|---|---|---|
| id | ||
| 1 | 100 | 200 |
| 2 | 300 | 400 |
Daskのデータフレームでサポートしているものの、遅いもの
-
インデックスの設定 :
set_indexなどで行なう。頻繁にインデックスを再設定したりせずに、なるべく少ない回数(最初の方に1回だけ設定して、その後はそのインデックスを使って計算していくなど)で済ませるのが好ましい。
df = dd.from_pandas(pd.DataFrame(data=[{
'id': 1,
'a': 100,
}, {
'id': 2,
'a': 300,
}]), npartitions=1)
df = df.set_index(other='id')
- 1行1行更新を行なう制御 : この辺りはPandasでも遅かったりするが、Daskでも同様に遅い。
- インデックスを使わない連結や、groupby後のオブジェクトの値を更新するような処理
※一部のPandasのAPIはサポートがされていない(Pandas側がインターフェイスが膨大にあるため)。対応しているものはドキュメントのAPIのページで確認できる(Dask側も膨大にある・・・)。
Daskのデータフレームを計算過程で一部確認したい場合
Daskのデータフレームは、Pandasのデータフレームと異なりメモリに過程でデータを載せない作りになっている都合、直接データの確認ができない。以下のように値の部分が...という表記になる。
df = dd.from_pandas(pd.DataFrame(data=[{
'id': 1,
'a': 100,
}, {
'id': 2,
'a': 300,
}]), npartitions=1)
df
部分的に過程のデータを確認したい場合には、headやtailなどのメソッドであれば、内容の確認が効くのでそちらを使う。
df.head(n=1)
df.tail(n=1)
データフレームのインデックス
前述したように、mergeやgroupbyなどの制御の際に、DaskではPandasの時よりもインデックスが重要になってくる。(一方で、インデックスの設定自体が遅いというジレンマはある・・・)
1回だけしかインデックスを使う制御をしないようなあれば、set_indexなどでインデックスを設定するのはあまり効率的にならないかもしれない。一方で、何度もインデックスが使われるようなケースであれば、事前にset_indexでインデックスを設定しておいたほうがトータルでは恐らく速くなる。
また、たとえばデータが日付などで分割されていてそれらを統合した際に、Dask側で自動で各データがパーティションが設定されるが、インデックスを貼ることで各データの日付の境界などがDask側で分かるようになる。そのような状態では、たとえばインデックスを参照した日付のスライスなども非常に高速に動作する。そういった処理が高速に動作する状態になっているかどうかは、データフレームのknown_divisions属性で確認ができる。
df_1 = pd.DataFrame(columns=['date', 'a'], index=np.arange(0, 3000000))
df_1.date = '2019-06-10'
df_1.a = 100
df_2 = pd.DataFrame(columns=['date', 'a'], index=np.arange(0, 3000000))
df_2.date = '2019-06-11'
df_2.a = 200
df_3 = pd.DataFrame(columns=['date', 'a'], index=np.arange(0, 3000000))
df_3.date = '2019-06-12'
df_3.a = 300
df = dd.concat([df_1, df_2, df_3])
この時点ではまだ統合のみで、インデックスを貼っていないのでknown_divisionsはFalseになっている。
df.known_divisions
False
インデックスを貼るとknown_divisionsがTrueになる。
df = df.set_index(other='date')
df.known_divisions
True
データフレームを確認すると、以下のような表示になり、パーティションの境界がソートされていることが分かる(2019-06-12が2個ある?とも思ったが、ドキュメントを読んでいた限り大丈夫そうな印象があるのでスルーする)。
試しに、2019-06-10, 2019-06-12, 2019-06-11という順番でデータフレームをconcatしてみたところ、前述のスクショの12 tasksとなっている箇所のタスク数が増えた。ドキュメントに、インデックスはソートされているとインデックスの設定が早く終わると書かれていたので、恐らくソートの制御が増える分計算が遅くなるのかな、という印象。
データの指定順など(場合によってはCSVなどで保存時などにも、ソートしておくなど)には気を配ったほうが良いケースがあるかもしれない。
さらに言うと、事前にソートが確実にされている前提で、set_indexメソッドでsorted=Trueを引数に指定するとインデックスの設定が高速にされるらしい。試しに、日付がソートされていない状態のset_indexとソートされている状態でconcatされ、sorted=Trueを指定してインデックスを設定した際の速度の差を確認してみる(300万行のデータフレーム × 3)。
まずはソートされていない条件から。
%%timeit
df = dd.concat([df_1, df_3, df_2])
df = df.set_index(other='date')
1 loop, best of 3: 5.49 s per loop
大分遅い。
続いてソートされている条件。
%%timeit
df = dd.concat([df_1, df_2, df_3])
df = df.set_index(other='date', sorted=True)
5倍弱程度速くなった。
パーティションの話
Daskのデータフレームでは、指定を省略した場合などは読み込みで指定されたファイル数や、concatなどでデータの連結をした場合はその個数に応じたパーティションが自動で設定される。
ただし、Dask配列のチャンクサイズと同様、このパーティションのサイズも、小さすぎても大きすぎてもパフォーマンス面で好ましくない。(小さすぎると分割によるオーバーヘッドが大きくなり、大きすぎると瞬間的にメモリが多く必要になってしまう)
パーティションが数万とかになっていたらオーバーヘッド的に大分きついかもしれない。
好ましいサイズ的には、大体チャンクサイズのときと同じような感覚で問題はない。使うマシンのメモリやコア数に合わせて、1つのパーティションで大体100MB~数百MB程度の範囲になるようにしておくと好ましい。
よくあるケースでは、計算の途中でスライスなどをして、データ量は少なくなっているにも関わらず、大量のパーティションのまま計算を色々やってしまうケース。そういった、過程でデータが大分少なくなる条件があり、且つその後も計算が結構続く場合には一旦repartitionなどのメソッドでパーティション数を減らしておくと、その後の計算が効率的に実行される。
スライスで減った件数に比率に応じて、パーティション数も減った比率を加味して減らす設定をする例 :
row_num = 10000000
df = pd.DataFrame(
columns=['a', 'b', 'c'],
index=np.arange(0, row_num))
df.a = np.random.randint(0, 100, size=(row_num,))
df.b = np.random.randint(0, 100, size=(row_num,))
df.c = np.random.randint(0, 100, size=(row_num,))
df = dd.from_pandas(data=df, npartitions=10)
before_row_num = len(df)
df = df[df.a >= 75]
after_row_num = len(df)
print('before_row_num:', before_row_num, ', after_row_num:', after_row_num)
before_row_num: 10000000 , after_row_num: 2500901
npartitions = int(df.npartitions / (before_row_num / after_row_num))
npartitions
2
df = df.repartition(npartitions=npartitions)
連結の話
既にインデックスなどの節である程度触れている、mergeなどの連結関係の操作に関して、インデックス同士の連結以外でも以下のようなケースでは速くなる。
- 複数のパーティションを持つデータフレームと、1つのパーティションしか持たないデータフレームを連結する場合 : 複数のパーティションを持つ大きな既存のデータフレームに対して、1つのパーティションしか持たない小さなデータフレーム(追加で必要になって読み込まれたデータフレームなど)を連結するケースは速く動作する。
- DaskのデータフレームとPandasのデータフレームを連結する場合 : このケースも、実質Pandas側のデータフレームがパーティションが1つのデータフレームとして扱えるので速く動作する。
上記のケース以外で、且つインデックスも加味しない連結の場合、パーティション間のデータの移動や検索などが色々必要になるため、処理が遅くなる。(Pandasと異なり、Daskのデータフレームは複数のパーティションでデータが分けられており、一部しかメモリ上に展開されていないので、Pandasの感覚で扱うとぐっと遅くなる)
locとiloc
Pandasでは特定の位置の要素を参照する際に、locやilocが必要になるケースがぼちぼちある。
Daskでは、locの方は基本的にPandasの感覚で扱える。
たとえば、以下のような書き方はサポートされている。
df.loc[0]
df.loc[1]
df.loc[:, ['id']]
一方で、ilocの方は、Daskが行数を常には保持していないため、特定行の選択には使えなかったりする。列番号の指定には対応しているので、基本的に行の方はコロンで全行指定をする形でしか利用できない。
2番目の列のみのデータフレームを取得する例 :
df.iloc[:, [1]]
なお、Pandasの場合はlocと異なりilocでの[行, 列]といった選択は確かできない気がする(iloc[行インデックス][列名]などと指定する必要がある)ものの、Daskだと前述のような書き方ができる。
ilocで特定行の選択をしようとすると、特定行の選択には対応していないのでコロンで行全体を選択してくれと怒られる。
df.iloc[0, [1]]
NotImplementedError: 'DataFrame.iloc' only supports selecting columns. It must be used like 'df.iloc[:, column_indexer]'.
タスクグラフの可視化
前述したタスクグラフを可視化する機能がDaskには用意されている。
利用するには、graphviz関係のライブラリが必要になる。pipなどだと、graphviz自体のインストールとPythonのgraphvizライブラリの2つが必要になる。
Anacondaのコマンドだと1回のコマンドでよしなにインストールしてくれる。
依存関係とかが若干面倒な印象があるので、Anacondaのコマンドの方が楽な印象はある。
$ conda install python-graphviz -y
The following NEW packages will be INSTALLED:
graphviz: 2.38.0-5
python-graphviz: 0.5.2-py36_0
可視化自体はvisualizeメソッドで実行できる。これはcomputeメソッド同様、Daskの各インターフェイス共通で用意されている。
import dask.array as da
x1 = da.random.uniform(low=0, high=1.0, size=(100, 100))
x2 = da.random.uniform(low=0, high=1.0, size=(100, 100))
x3 = da.random.uniform(low=0, high=1.0, size=(100, 100))
x4 = da.random.uniform(low=0, high=1.0, size=(100, 100))
y = x1 + x2 + x3 * x4
y.visualize()
ただし、あまりにノードが多いものなどは可視化にかなり時間がかかったり、失敗したりするので注意。
可視化に関しては、内容が確認できるだけでなく、ケースによっては遅くなりそうな箇所の特定にも役に立つ場合がある。
たとえば、ノード数が統合されて減っている箇所があれば、並列で処理される件数も減ったり、それぞれのノードが終わるまで統合ができないので処理を待ったりする。
そのため、たとえば片側だけ極端に遅く、そのノードが統合予定であれば、統合時にそのタスク消化待ち(ボトルネック)が発生したりする。
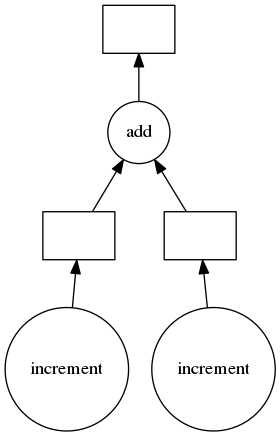
Dask Delayed
NumPyインターフェイスのDaskの配列や、PandasインターフェイスのDaskのデータフレームのほかに、Python自体のコードのインターフェイスとして使えるDelayedというインターフェイスが用意されている。
通常のPythonコードで即時で処理が走るケースとは違い、処理が遅延評価(他のDaskインターフェイス同様、即時では計算が走らず、compute呼び出し時点で計算などが開始)されるため、恐らくこういった名前が付いている。
Pythonビルトインの、普通のマルチスレッドなどの記述よりも記述がシンプルに書けて並列化とタスクグラフの利用などが実現できる。
使い方は、遅延評価を設定したい関数をdask.delayed関数で囲むか、デコレーターを利用することでシンプルに書くことができる。
-
delayed関数を挟む例 :
dask.delayed(対象の関数)(関数に設定する引数)といった書き方になる。
import dask
def increment(x):
return x + 1
def add(a, b):
return a + b
x = dask.delayed(increment)(5)
y = dask.delayed(increment)(10)
z = dask.delayed(add)(x, y)
dask.delayed(increment(5))といった書き方と間違えないように注意。引数部分は外に出さないと遅延評価されずに即時で計算が走ってしまう。
この時点ではまだ計算などは走っていない。zの内容を見ても数値の内容などは確認できない。
compute関数を呼び出した時点ではじめて各計算が走り始め、数値が取得できる。
z.compute()
17
また、Dask配列などのインターフェイスと同様、こちらもタスクグラフの可視化ができる。
自分で計算を書く形になるので、グラフ内容も分かりやすい。
z.visualize()
-
デコレーターを付けて対応する場合 :
@dask.delayedとタスクグラフで扱いたい関数に設定する。こちらはキーワード引数なども受け付けてくれる(ただし、デコレーターを付けると引数が*args、**kwargsという表記になって入力補間などが利かなくなる)のと、記述も自然で違和感がない。
@dask.delayed
def increment(x):
return x + 1
@dask.delayed
def add(a, b):
return a + b
x = increment(x=5)
y = increment(x=10)
z = add(a=x, b=y)
z.compute()
17
これだけで並列化されたコードになる。とてもシンプル。
Pythonビルトインの並列処理モジュールの完全な代替にはならないけれども、Delayedで必要十分な際にさくっと並列化したいときなど便利な印象は受けた。
Daskの配列やDaskのデータフレームが苦手とする、Python自体のループを回したりなどにも向いている。
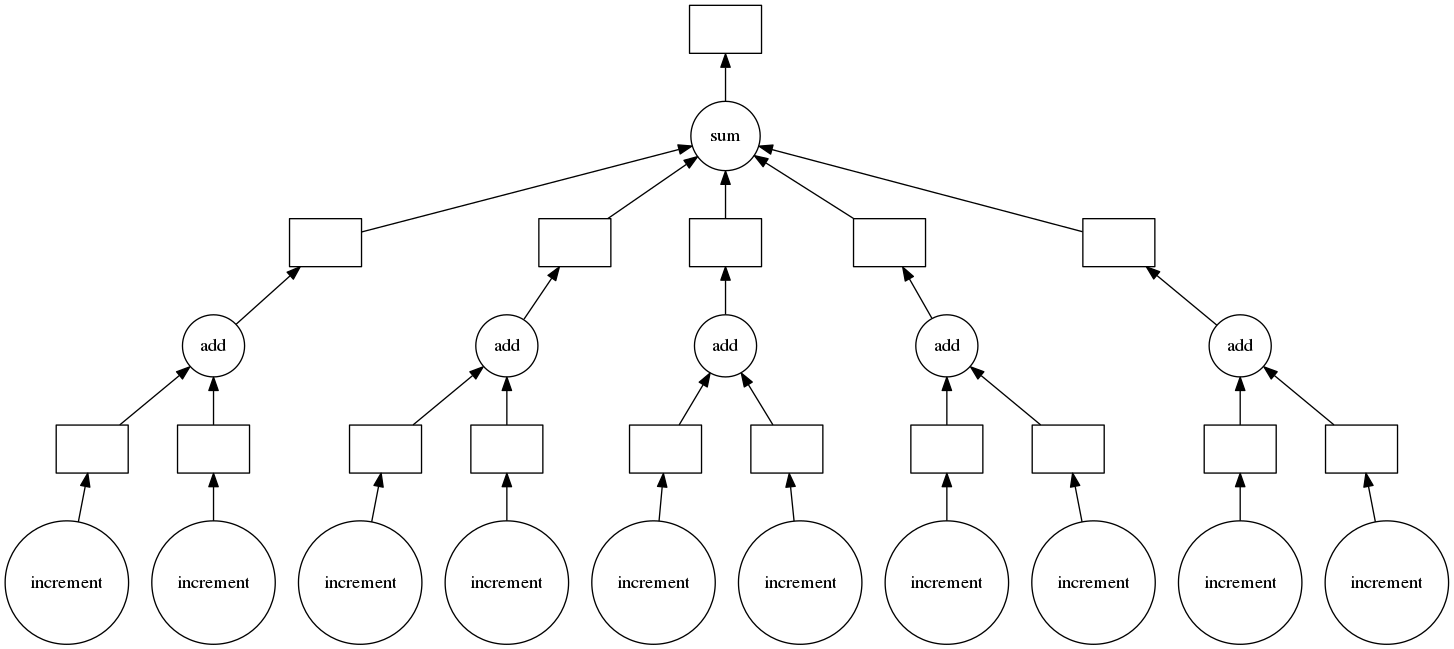
また、Pythonビルトインの関数などに対してもdelayed関数を挟んで遅延評価にしたりもできる(下記コードのsum部分など。ほとんどのPython関数をサポートしているらしい)。
data_tuple = ((1, 2), (3, 4), (5, 6), (7, 8), (9, 10))
output_list = []
for x, y in data_tuple:
x = increment(x=x)
y = increment(x=y)
z = add(a=x, b=y)
output_list.append(z)
total = dask.delayed(sum)(output_list)
total.compute()
65
タスクグラフを表示してみると、ループ分ノードが増えていっていることが分かる。
どんなときにDaskのDelayedを使うかの例
- Daskの配列やデータフレームで用意されているAPIだけでは足りないケースが発生し、ループを回したりが必要になった場合。過程でDelayedを経由する場合でも、配列やデータフレームのインスターフェイスで
from_delayedやto_delayedなどのインターフェイスが用意されており、相互に変換ができる。 - Pythonのビルトインの並列処理を使って細かい調整をしたり・・が不要な場合。前述の通り、シンプルな記述で並列処理などが実現できる。
良くないDelayedの書き方
- computeはなるべく少ない数を呼び出す : できれば1回にまとまるようにする。回数が多くなると、それぞれのcomputeが終わらないと次の計算に移らなくなり、並列化された計算だとそこがボトルネックになったりする。multiprocessingモジュールなどのjoinをループ内で基本書かないのと同様、ループ内でcomputeを呼び出したりしないように注意。
- 入力値を直接更新しない : 引数に渡される値をそのままインクリメントしたりなどは好ましくないらしい。動作はすることは確認した。並列化時のメモリのロックなどの都合なんだろうか・・?以下良くないコード例。
@dask.delayed
def test_func(x):
x += 1
return x
x = test_func(x=1)
x.compute()
2
前述のようなコードが必要な場合には直接インクリメントではなくreturn x + 1といった別の変数で扱われる形で書くか、copyなどを挟むといいらしい。
from copy import copy
@dask.delayed
def test_func(x):
x = copy(x)
x += 1
return x
x = test_func(x=1)
x.compute()
2
- グローバル変数などにはアクセスしない : Daskはマルチスレッドやマルチプロセス、もしくは分散環境などで扱えるが、マルチスレッド以外ではグローバル変数などにアクセスしていると動かないケースが出てくる。マルチスレッドから別のものに切り替えた際に躓く要因になるので、基本的にグローバル変数などにアクセスしている書き方は好ましくない。以下良くない書き方。
global_list = []
@dask.delayed
def test_func(x):
global_list.append(x + 1)
x = test_func(x=1)
-
タスクが膨大になるケースは避ける : 細かい関数で膨大にDelayedを反映したものを呼び出したりすると、1つ1つのDelayedのオーバーヘッドは些細でも、地味に馬鹿にならないオーバーヘッドになる。そういった場合はある程度まとめられるところは複数の関数の処理をまとめて、オーバーヘッドが減るように調整するか、ビルトインの
multiprocessing.Poolなどの方が向いているかもしれない。 -
Delayedの指定が入れ子になるような書き方は避ける : たとえば、デコレーターがつけられた関数内でさらにdelayedを挟んで他の関数を呼び出したりなど。基本的にそういった入れ子になる使い方はNGとのことで、どちらかのみに設定する形に調整する必要がある。
-
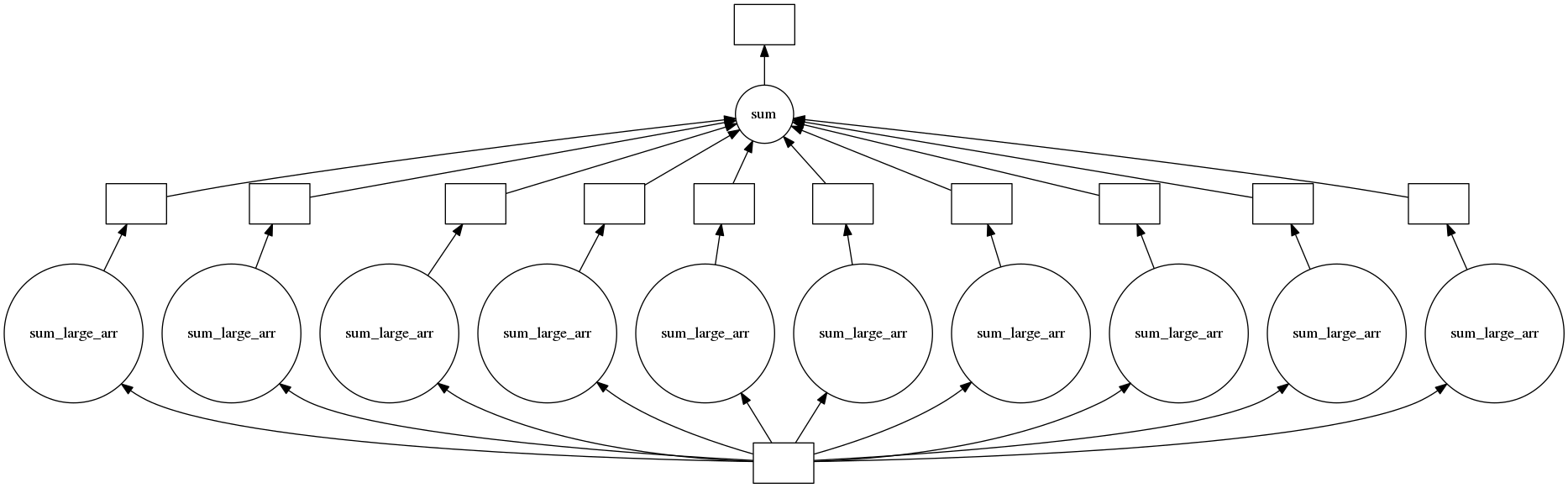
Delayed指定された関数に、直接大きなデータの引数を何度も指定する : そういった場合は、先にそのデータ自体にDelayedを挟んでおくと好ましい(関数以外もDelayedを挟むことができる)。そうするとやりとり面でオーバーヘッドが少なくなるらしい(恐らく、)。以下少し大きめの配列に対して、
delayedを挟んでみた例。
large_arr = np.ones(shape=(100000,))
large_arr = dask.delayed(large_arr)
@dask.delayed
def sum_large_arr(large_arr):
return sum(large_arr)
output_list = []
for i in range(10):
x = sum_large_arr(large_arr=large_arr)
output_list.append(x)
total = dask.delayed(sum)(output_list)
total.compute()
1000000.0
タスクグラフを可視化してみる。
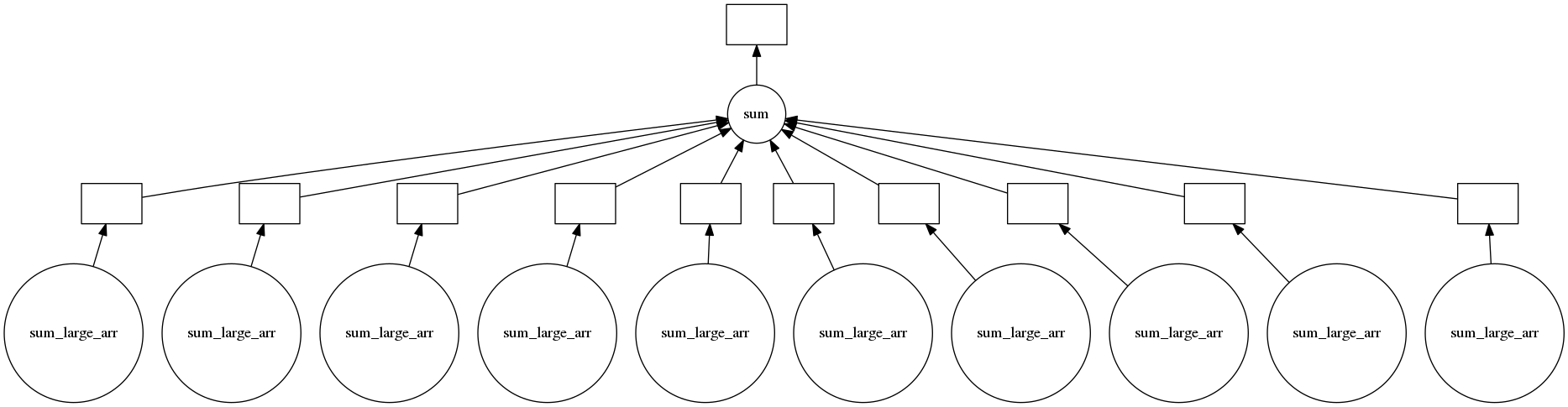
スタートが1つになっている。配列にdelayedを挟んでいるので、各ノードに渡されるものが同一のものが参照されていることが分かる。恐らく並列化時に渡すデータのコピーなどはされず、同じものが使いまわされているので効率的に処理される・・・ということなのだろうか。ちなみに、配列にdelayedを挟まないもののタスクグラフを確認すると、以下のように開始ノードが1つにまとまったりはしていない。
パフォーマンスチューニング
そのままだと、Daskは並列処理や分散処理が絡む都合で、関数単位での処理時間のプロファイリング用の%%prunのマジックコマンドや、そのほかのプロファイリング用の機能が動かなかったりする。
さらに言うと、そういった計測が必要なところ以外にも、並列処理や分散処理によるデータの変換のやりとりやデータ転送時間、タスクグラフ化によるオーバーヘッドなど、通常のPythonでは計測が難しい箇所が色々とある。
そのため、計測などに関してはDask自体にある程度用意されており、遅くなっている箇所の特定などに役立てることができる。
並列環境の計測・診断
分散環境だとまた話が変わってくるところもあるが、並列環境であれば以下のような機能が用意されている(キャッシュ周りなど一部割愛)。
(本記事では割愛するものの、分散環境であればTensorBoardみたくwebベースで確認できる計測機能などもある)
- プログレスバー : tqdm的な表示を、Daskの並列処理環境で利用できる。
- タスク単位のプロファイラー : タスク単位での処理時間が確認できる。
- リソースプロファイラー : メモリ使用量や、CPU使用率などを確認できる。
プログレスバー
並列環境で各処理が全部でどのくらい進んでいるのかをtqdm的に確認ができる。
dask.diagnostics.ProgressBarクラスを使う。
使う場合には、withステートメントで計測したいcomputeの箇所のみ囲うか、もしくはregisterメソッドを利用する。(後者はコード全体に反映される)
- withステートメントで囲う場合 :
import dask.array as da
from dask.diagnostics import ProgressBar
arr = da.random.randint(low=0, high=100, size=(500000000,))
arr += 1
arr += 2
arr += 3
mean_val = arr.mean()
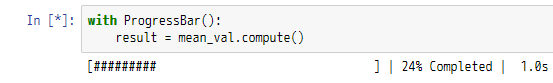
with ProgressBar():
result = mean_val.compute()
- registerで登録する場合 :
こちらはグローバルに反映される。withステートメントなどは不要になるので、複数個所で何度もcomputeを呼ぶ場合などでは便利。
部分的に計測したい場合などには注意。表示自体はwithステートメントを使った際と変わらない。
progress_bar = ProgressBar()
progress_bar.register()
result = mean_val.compute()
タスク単位のプロファイラー
こちらは各タスクの開始時間・終了時間などの計測として使う。dask.diagnostics.Profilerクラスを使う。
プログレスバーと同様に、withステートメントやregisterなどで利用できる。
Profilerクラスのオブジェクトは後で利用するので、一旦変数に設定している。
from dask.diagnostics import Profiler
profiler = Profiler()
with profiler:
result = mean_val.compute()
計測結果はプロファイラーオブジェクトのresults属性に格納される。
ただし、そのまま確認してもなんだかタスク情報がたくさん情報がある・・・というくらいしか分からない。
profiler.results
[TaskData(key=('randint-mean_chunk-812e8f0cb4ac9321a67b998f47ae5f64', 0), task=(subgraph_callable, 3, 2, (<function _apply_random at 0x7fcf6a384598>, None, 'randint', array([1483484144, 3925864007, 3606941276, 3622924718, 2457601011,
2992342959, 3580848644, 3214909470, 229022156, 2784167203,
1520372, 1072208554, 870698918, 2640623133, 1663924999,
71001927, 3028677620, 1358626999, 3744700271, 2796033730,
2908454865, 4008216102, 728249598, 4040528145, 3680559835,
...
そのため、基本的には可視化して使う。プロファイラーにvisualizeメソッドが用意されているので、そちらを使う。
内部でbokehが使われているので、Jupyter上で使う場合には以下のようなbokehの記述をしないと表示してくれない(%matplotlib inline的な記述と思っていただければ)。
from bokeh.plotting import output_notebook
output_notebook()
BokehJS 0.12.5 successfully loaded.
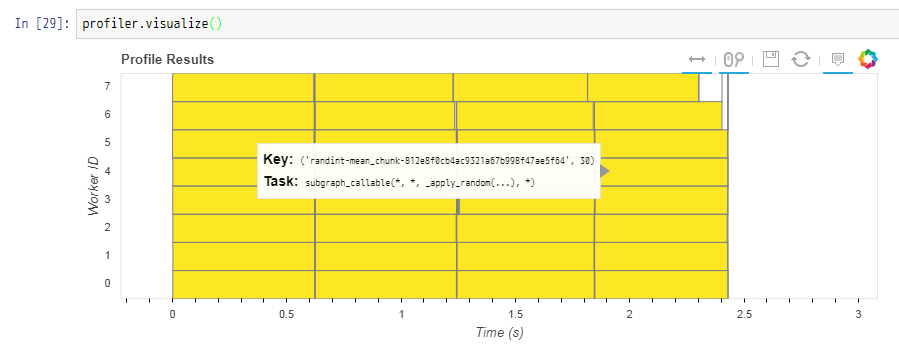
縦軸がワーカーごと(コアが8個のマシンで動かしているため、8個分分割されている)、横軸が経過時間で、タスクごとに四角のブロックで表示されている。
各ブロックにマウスオーバーすると、そのタスクのキーや番号、簡単な内容などが見れる。どのタスクなのか・・ということは正直分かりづらい感もある。(タスクグラフを作るときに名前とか付けられるのだろうか・・?)
今回のケースでは、黄色の色が付いているブロックが各ワーカーで処理を実行中、白い領域がリソースが空いている状態(他のワーカーの処理待ちなど)かと思われる(スクショの右上を見ると、若干の白いブロック領域があることが分かる)。
今回のサンプルコードでは、正直大きくボトルネックになっているものも無さそう(ほとんど全面が黄色くなっている)で、調整も不要そうなプロット結果となった。
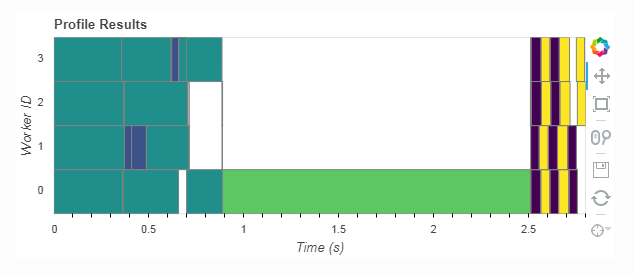
公式のドキュメントを見ていると、もっとボトルネックが顕著なプロットになっているので、恐らくアンバランスな複雑なタスクになってきたらもっと有益なプロットになるのだろう・・という印象。
※上記の図は公式のドキュメント Diagnostics (local)から引用。
なお、registerでグローバルに登録していると、複数のcomputeでの計算の内容がプロットに入ってくる。あまり見やすいとは言えない印象を受けたので、場合によってはwithステートメントで単体のcomputeのみを対象にしたほうが見やすくて好ましいかもしれない(これは、後述のリソースプロファイラーでも同様)。
リソースプロファイラー
リソースプロファイラーでは、並列環境でのCPU使用率やメモリ使用量(MB単位)の確認ができる。
基本的な使い方は、使うクラスがResourceProfilerクラスになるだけで、他のプログレスバーやプロファイラーとほぼ変わらず、withステートメントやregisterなどのメソッドで登録して利用する。
from dask.diagnostics import ResourceProfiler
resource_profiler = ResourceProfiler()
with resource_profiler:
result = mean_val.compute()
results属性を見てみると、タスクごとのプロファイラーとは異なり、こちらはプロットしなくても十分分かる内容となっている。
resource_profiler.results
[ResourceData(time=903533.34111646, mem=632.418304, cpu=0.0),
ResourceData(time=903534.38281667, mem=3682.156544, cpu=786.1999999999999),
ResourceData(time=903535.38432508, mem=2337.964032, cpu=779.7)]
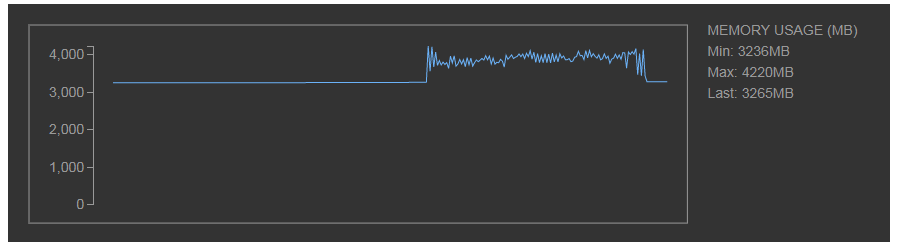
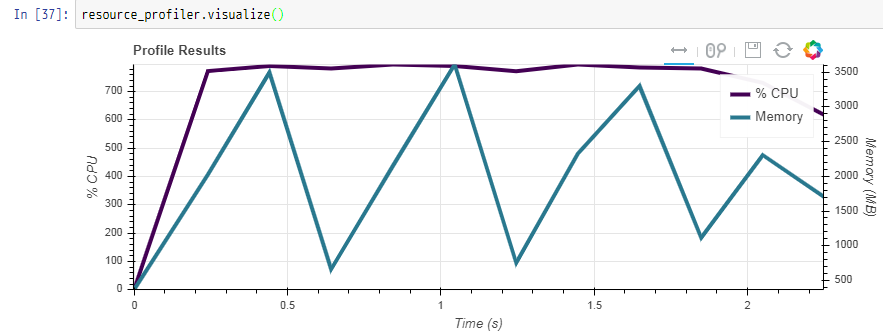
デフォルトでは1秒ごとに計測される。今回の計算は2.5秒かかったので、3件分計測されている。
時間の情報と、そのタイミングのメモリ使用量・CPU使用量が確認できる。CPUは、Kaggleカーネルみたいに、コア数×100%がMAXとして表示されるので、今回は8個のコアのマシンで動かしているので、完全にCPUリソースが使われていれば800%となる(780程度の数字になっているので、ほとんど無駄なくCPUを使っていることが分かる)。
1秒後との計測を、0.5秒ごとにしてもっと細かく計測したい(もしくはもっと長いスパンで計測したい)場合には、ResourceProfilerクラスのdt引数に秒数を指定すると調整ができる(dtはdatetimeなどではなく、delta timeだと思われる)。たとえば、0.2を指定すれば0.2秒間隔で計測され、先ほどまでよりも細かく計測される。(結果のデータの件数が増える)
resource_profiler = ResourceProfiler(dt=0.2)
with resource_profiler:
result = mean_val.compute()
resource_profiler.results
[ResourceData(time=904075.205010556, mem=366.73536, cpu=0.0),
ResourceData(time=904075.445630621, mem=2024.218624, cpu=772.9),
ResourceData(time=904075.646472633, mem=3496.87808, cpu=790.6),
ResourceData(time=904075.847534518, mem=662.540288, cpu=781.8),
ResourceData(time=904076.048325532, mem=2146.500608, cpu=795.9),
ResourceData(time=904076.249606525, mem=3610.038272, cpu=790.3),
ResourceData(time=904076.450497805, mem=754.847744, cpu=772.3),
ResourceData(time=904076.651280945, mem=2326.319104, cpu=796.0),
ResourceData(time=904076.852340879, mem=3300.59776, cpu=785.7),
ResourceData(time=904077.05340738, mem=1114.50112, cpu=781.8),
ResourceData(time=904077.254200403, mem=2304.077824, cpu=731.4),
ResourceData(time=904077.455156886, mem=1699.852288, cpu=617.8)]
先ほどよりも結果の件数が多いことが分かる。また、タスク単位のプロファイラーで、最後のほうが微妙に空いているワーカーがあったという点から分かるように、こちらでも最後の方の計測が微妙にリソースが空いていることが分かる。
こちらも同様に、visualizeメソッドでプロットを表示することができる。
resource_profiler.visualize()
紫がCPU使用率、青緑がメモリ使用量。
CPUはほとんどフルに使われており、メモリのほうはタスクごとに必要なところだけ確保して、次のタスクで不要になったところを破棄して・・とやっている都合、山あり谷ありな結果になっている。
通常のデバッグや計測のツールを使いたい場合
通常は、Daskで並列化されていても、エラーなどがちゃんと検知できたり、計測用の機能が用意されているのでDaskだけでもなんとかなったりする。
しかし、Dask以外の%pdbだったり、%debugのデバッグのマジックコマンドだったり、%prunなどを利用したりがしたくなるケースがある(Dask側で並列化や分散処理などされる都合、通常はこれらは使えない)。
そういった場合には、Daskのスケジューラー(並行処理・並列処理・分散処理などの計算方式的なものの選択)を、1つのスレッドのものに一時的に設定することで、並列化などが一通り無効化され、前述のマジックコマンドも利用できるようになる。
並列化がされなくなるので、もちろん遅くなるものの、デバッグなどの面で必要になった場合に利用することができる。(デバッグなどが終わったら指定を消して元に戻したりなどを行う)
computeメソッドのschedulerの引数に'single-threaded'と指定すると、単一のスレッドで実行されるようになる。
resource_profiler = ResourceProfiler()
with resource_profiler:
result = mean_val.compute(scheduler='single-threaded')
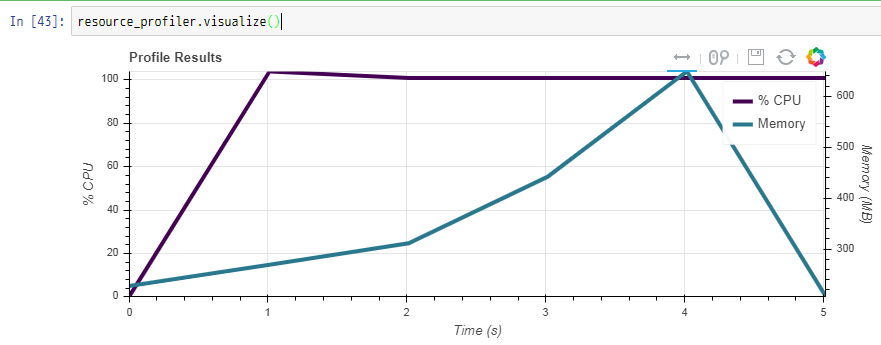
resource_profiler.visualize()
[########################################] | 100% Completed | 5.2s
先ほどは2秒台で終わっていたので、並列化がされなくなったので遅くなったことが分かる。また、CPU使用率の最大が100%になっていることも分かる。
あとは、必要なマジックコマンドを挟めば、通常のPythonコードのように調査ができる。
他にもPythonなどを中心に色々記事を書いています。そちらもどうぞ!
今までに投稿した主な記事たち
参考文献まとめ
- Dask公式 トップページ
- Dask
- Install Dask
- Use Cases
- Why Dask?
- User Interfaces
- Array
- Best Practices (Array)
- Chunks
- Internal Design (Array)
- Stats
- Slicing
- Stack, Concatenate, and Block
- Bag
- DataFrame
- Best Practices (DataFrame)
- Joins
- Internal Design (DataFrame)
- Shuffling for GroupBy and Join
- Indexing into Dask DataFrames
- Delayed
- Best Practices (Delayed)
- Working with Collections
- Best Practices (Dask全体)
- Scheduling
- Visualize task graphs
- Diagnostics (local)
- Debugging
- Amazon Athenaのパーティションを理解する
- s3fsよりも高速に使えるgoofysを試してみた