はじめに
思い立って、PDB形式のタンパク質の構造データをもとに、SketchUpで3次元立体構造を描いてみることにしました。Rubyは素人ですが、SketchUpのAPIがrubyなので、勉強しながら作っていくつもりです。
ご意見、コメントなど頂けますと大変参考になります。
解析対象のPDBデータについては前回の記事をみてください。
タンパク質の立体構造をSketchUpでモデル化 その1
タンパク質の立体構造をSketchUpでモデル化 その2【アミノ酸の側鎖を描画】
タンパク質の立体構造をSketchUpでモデル化 その3【アミノ酸の側鎖も含めて描画】
タンパク質の立体構造をSketchUpでモデル化 その4【主鎖を立体に】
タンパク質の立体構造をSketchUpでモデル化 その5【玉の描画】
タンパク質の立体構造をSketchUpでモデル化 その6【ついでにDNAをモデル化】
タンパク質の立体構造をSketchUpでモデル化 その7【最終回】
今回のテーマ
今回のテーマはアミノ酸の側鎖の描画です。アミノ酸20種類それぞについて処理が必要です。
例えばフェニルアラニンのデータは以下の通りです。3つめのフィールドがそれぞれの原子を意味しています。最初の一文字目が元素記号で、C(炭素)に続くアルファベットは、炭素原子を区別する記号です。A、B、G、D、E、Zは、α、β、γ、δ、ε、ζを表すようです。問題は、どの炭素原子が隣り合っているのかよくわからない、ということです。どの原子とどの原子をedgeで連結すべきでしょうか?
また20種類あるアミノ酸それぞれについて、どう描画すべきかを簡潔に整理したいと思います。
ATOM 184 N PHE A 250 -27.579 31.712 -9.871 1.00 19.79 N
ATOM 185 CA PHE A 250 -27.914 30.830 -8.719 1.00 19.26 C
ATOM 186 C PHE A 250 -28.791 31.632 -7.752 1.00 22.63 C
ATOM 187 O PHE A 250 -29.974 31.534 -7.841 1.00 32.67 O
ATOM 188 CB PHE A 250 -26.662 30.210 -8.066 1.00 18.50 C
ATOM 189 CG PHE A 250 -25.814 29.377 -9.043 1.00 16.76 C
ATOM 190 CD1 PHE A 250 -26.430 28.357 -9.799 1.00 15.96 C
ATOM 191 CD2 PHE A 250 -24.481 29.634 -9.242 1.00 15.84 C
ATOM 192 CE1 PHE A 250 -25.705 27.632 -10.747 1.00 15.68 C
ATOM 193 CE2 PHE A 250 -23.749 28.829 -10.161 1.00 16.68 C
ATOM 194 CZ PHE A 250 -24.405 27.813 -10.836 1.00 16.39 C
計画
- アミノ酸ごとに、どの原子とどの原子を連結するかのデータをまとめる。
- 連結データは、配列の配列とする。
まずはフェニルアラニン
フェニルアラニンのedgeを3つに分けて書くことにします。
主鎖はN->CA->C->0、CAから芳香環までは、CA->CB->CG、芳香環はCG->CD1->CD2->CE1->CE2->CZだとして描いてみます。それぞれのつながり方を配列にし、それをさらに配列に入れます。
まずは連想配列の使い方を調べてみました
# 連想配列(Hash)の初期化
atomsAndCoordinates = Hash.new
# 連想配列への、keyとvalueをセット
atomsAndCoordinates["key"] = "value"
データファイルの行、228行目から238行目にフェニルアラニンのデータがあります。
# モデルを取得
model = Sketchup.active_model
entities = model.active_entities
# データファイルの読み込み。chomp: true を引数にすると機能しません....parse error, unexpected ':', expecting ')'と出ます。
lines = IO.readlines("/Users/yoho/Downloads/mmdb_5EPW.pdb")
# フェニルアラニンを描いてみる
PheEdges = [["N","CA","C","O"],["CA","CB","CG"],["CG","CD1","CD2","CE1","CE2","CZ","CG"]]
atomsAndCoordinates = Hash.new
for x in 228..238 do
data = lines[x].split
atomsAndCoordinates[data[2]] = Geom::Point3d.new(data[6].to_f, data[7].to_f, data[8].to_f)
end
# 配列の配列から、配列を取り出して処理
for pheEdge in PheEdges
ps = []
for atom in pheEdge
ps.push(atomsAndCoordinates[atom])
end
entities.add_edges(ps)
end
右下のジグザグな部分が芳香環であると思われます。
ああ、そうか。普段意識してませんでしたが、基準となるカルボキシル基のCの隣からα、β...となるのでした。分岐がある場合は分岐を起点にして、分岐先どちらも同じギリシャ文字+数字がつくのですね。
["CG","CD1","CD2","CE1","CE2","CZ","CG"]ではなくて、["CG","CD1","CE1","CZ","CE2","CD2","CG"]のようです。ここを変更してもう一度描画してみます。
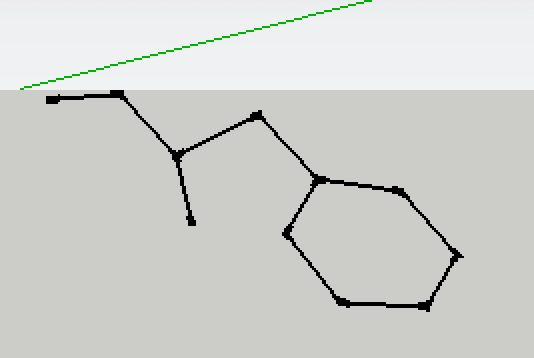
これでOKですね。側鎖だけ考えれば、描画するのは["CA","CB","CG"]と["CG","CD1","CD2","CE1","CE2","CZ","CG"]で良いことになります。
20種類のアミノ酸それぞれについて、側鎖の原子のつながり方情報を準備します。主鎖は主鎖で別に描画しますので、どうしましょうかね。C=Oは残すことにして、主鎖情報をindex 0にして、C=Oを index 1にすることにします。それ以降は側鎖のデータです。側鎖データを、いくつかに区切った方が良いのかわかりませんが、芳香環は、芳香環として、分けておいたほうが良い気がするので分けます。
PheEdges = [["N","CA","C"],["C","O"],["CA","CB","CG"],["CG","CD1","CE1","CZ","CE2","CD2","CG"]]
同じように20種のアミノ酸全部について、データを整備します。以下のようなデータ形式でまとめます。20種類やるのか....めんどくさいなあ。まあやるか。
aminoacidAndBonds = {
"PHE" => [["N","CA","C","O"],["CA","CB","CG"],["CG","CD1","CE1","CZ","CE2","CD2","CG"]],
"GLY" => [["N","CA","C"],["C","O"]]
}
以下のようなコードで、アミノ酸の構造が正しく描画できるかチェック。最初のFor文では、調べたいアミノ酸がファイルの何行目にあるかを記述する(indexが0から始まるので、行番号から1を引いたもの)
atomsAndCoordinates = Hash.new
for x in 719..723 do
data = lines[x].split
atomsAndCoordinates[data[2]] = Geom::Point3d.new(data[6].to_f, data[7].to_f, data[8].to_f)
end
for bonds in aminoacidAndBonds["ALA"]
ps = []
for atom in bonds
ps.push(atomsAndCoordinates[atom])
end
entities.add_edges(ps)
end
20種類のアミノ酸データ
aminoacidAndBonds = {
"ALA" => [["N","CA","C"],["C","O"],["CA","CB"]],#A
"CYS" => [["N","CA","C"],["C","O"],["CA","CB","SG"]],#C
"ASP" => [["N","CA","C"],["C","O"],["CA","CB","CG"],["CG","OD1"],["CG","OD2"]],#D
"GLU" => [["N","CA","C"],["C","O"],["CA","CB","CG","CD"],["CD","OE1"],["CD","OE2"]],#E
"PHE" => [["N","CA","C"],["C","O"],["CA","CB","CG"],["CG","CD1","CE1","CZ","CE2","CD2","CG"]],#F
"GLY" => [["N","CA","C"],["C","O"] ],#G
"HIS" => [["N","CA","C"],["C","O"],["CA","CB","CG","ND1","CE1","NE2","CD2","CG"]],#H
"ILE" => [["N","CA","C"],["C","O"],["CA","CB"],["CB","CG1","CD1"],["CB","CG2"]],#I
"LYS" => [["N","CA","C"],["C","O"],["CA","CB","CG","CD","CE","NZ"]],#K
"LEU" => [["N","CA","C"],["C","O"],["CA","CB","CG"],["CG","CD1"],["CG","CD2"]],#L
"MET" => [["N","CA","C"],["C","O"],["CA","CB","CG","SD","CE"]],#M
"ASN" => [["N","CA","C"],["C","O"],["CA","CB","CG"],["CG","OD1"],["CG","ND2"]],#N
"PRO" => [["N","CA","C"],["C","O"],["CA","CB","CG","CD","N"]],#P
"GLN" => [["N","CA","C"],["C","O"],["CA","CB","CG","CD"],["CD","OE1"],["CD","NE2"]],#Q
"ARG" => [["N","CA","C"],["C","O"],["CA","CB","CG","CD","NE","CZ"],["CZ","NH1"],["CZ","NH2"]],#R
"SER" => [["N","CA","C"],["C","O"],["CA","CB","OG"]],#S
"THR" => [["N","CA","C"],["C","O"],["CA","CB"],["CB","OG1"],["CB","CG2"]],#T
"VAL" => [["N","CA","C"],["C","O"],["CA","CB"],["CB","CG1"],["CB","CG2"]],#V
"TRP" => [["N","CA","C"],["C","O"],["CA","CB","CG"],["CG","CD2","CE2","CZ2","CH2","CZ3","CE3","CD2"],["CG","CD1","NE1","CE2"]],#W
"TYR" => [["N","CA","C"],["C","O"],["CA","CB","CG"],["CG","CD1","CE1","CZ","CE2","CD2","CG"],["CZ","OH"]]#Y
}
となりました。これで側鎖も描画しやすくなりました。
終わりに
次回は、側鎖も含めて立体構造を描画してみたいと思います。
