Rで統計解析を行う際には、しばしばモデルからのデータの抜き出しや結果の描画が面倒です。それはパッケージ間で出力結果の形式が異なったり、出力された結果自体が複雑な構造を持つオブジェクトに格納されるためです。
それを解決するのがeasystatsパッケージ群であり、Qiitaでこの一年その便利さをたびたびご紹介してきました。
せっかくのAdvent Calendarなので、ここではそれらの記事の振り返りもかねて自分なりにeasystatsパッケージ群の便利さをご紹介したいと思います。
他の方による紹介記事であるeasystatsについて①: パッケージ群の紹介もご覧いただくと、より理解が深まるかと思います。
easystatsとは?
easystatsはRのパッケージ群です。このパッケージ群は、おっかないRの統計とそのやっかいなモデルたちを、手懐け、しつけて、うまく利用するための統合的で一貫したフレームワークを提供することを目的としています。
https://github.com/easystats/easystats より
Rで統計解析をしたことがある人なら誰でも、「なんでパッケージが違うとこんなに出方結果が違うんだー!」という絶望を感じたことがあると思います。easystatsは我々にそんなモンスター的なRの統計解析たちを手懐ける道具を提供してくれるパッケージ群です。
現時点では、easystatsには
insight: データ抽出関連のユーティリティ
see: 描画関連のユーティリティ
correlation: 相関関係の確認
parameters: パラメータ推定値の取得
performance: モデルの性能検証
effectsize: 効果量の算出とパラメータの標準化
modelbased: モデルに基づく効果の推定
bayestestR: ベイズ検定
report: 解析結果のレポート形式での出力
の9つのパッケージが含まれています。モデルからの統一的な解析結果の抽出を可能にするinsightを起点として、さまざまな統計解析パッケージに対する対応が行われています。
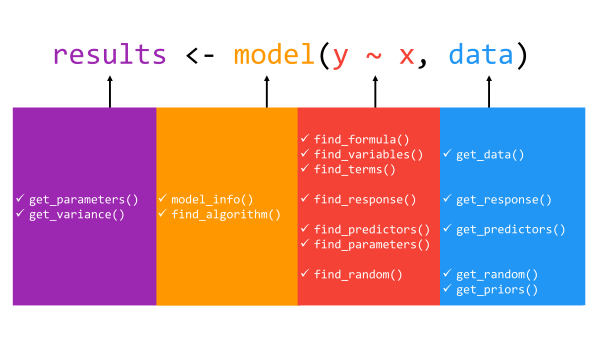
これらのパッケージが各種統計解析関連の関数の出力結果の抽出、要約、描画を強力にサポートします。
要はどんな解析結果でもmodelという名前のオブジェクトに入れてしまえば、ほぼまったく同じコードで、パラメータ推定値と信頼区間の取得、AICやR2等のモデル性能に関する指標の取得、効果量の推定を行うことが出来るようになります。また関連するパッケージであるggeffectsパッケージを使用することでモデルに基づく予想値の描画を簡単に行うことが出来ます。
ここでは各パッケージの簡単な使い方を、実例を交えながらご紹介します。
実例で見る!easystatsパッケージの使い方!
ここではglmとlme4::glmerによる解析を例にとって、easystatsの便利さをご紹介したいと思います。
インストール
easystatsに含まれるパッケージにはCRANに登録されているものもあるのですが、それらを束ねるeasystatsはCRANに登録されていません(各パッケージの更新頻度が高いからかもしれません)。よって、remotesパッケージを用いてGitHubからインストールします。
remotes::install_github("easystats/easystats")
ggeffectsはCRANからでもGitHubからでもインストールできます。
install.packages("ggeffects")
remotes::install_github("strengejacke/ggeffects")
パッケージの読み込みと使用データ
ここでは以下のパッケージを使用します。
library(easystats)
library(ggeffects)
library(tidyverse)
library(MASS)
library(lme4)
またデモ用に以下のデータを使用します。
mtcars
epil0 <- epil %>% mutate(V4 = as.factor(V4), subject = as.factor(subject))
モデルとしては上記のデータに基づく、
model_mtcars <- glm(vs ~ wt + mpg, family = "binomial", data = mtcars)
model_epil0 <- glmer(y ~ lbase*trt + lage + V4 + (1 | subject), family = "poisson", data = epil0)
を使用します。
変数間の相関関係を確認したい! → correlation
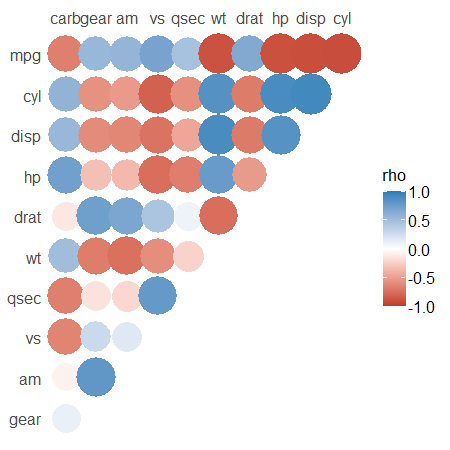
いのちの輝きくんcorrelationは変数間の相関係数の取得を楽々行えるようにするパッケージです。以下のように3行で相関係数の算出、要約、描画を行うことができます。
> cor_mtcars <- correlation(mtcars, method = "spearman")
> head(cor_mtcars)
Parameter1 | Parameter2 | rho | 95% CI | S | p | Method | n_Obs
---------------------------------------------------------------------------------------
mpg | cyl | -0.91 | [-0.96, -0.82] | 10425.33 | < .001 | Spearman | 32
mpg | disp | -0.91 | [-0.95, -0.82] | 10414.86 | < .001 | Spearman | 32
mpg | hp | -0.89 | [-0.95, -0.79] | 10337.29 | < .001 | Spearman | 32
mpg | drat | 0.65 | [ 0.39, 0.82] | 1901.66 | 0.002 | Spearman | 32
mpg | wt | -0.89 | [-0.94, -0.78] | 10292.32 | < .001 | Spearman | 32
mpg | qsec | 0.47 | [ 0.14, 0.70] | 2908.40 | 0.099 | Spearman | 32
> summary(cor_mtcars)
Parameter | carb | gear | am | vs | qsec | wt | drat | hp | disp | cyl
----------------------------------------------------------------------------------------------------------------------
mpg | -0.66** | 0.54* | 0.56* | 0.71*** | 0.47 | -0.89*** | 0.65** | -0.89*** | -0.91*** | -0.91***
cyl | 0.58* | -0.56* | -0.52* | -0.81*** | -0.57* | 0.86*** | -0.68*** | 0.90*** | 0.93*** |
disp | 0.54* | -0.59** | -0.62** | -0.72*** | -0.46 | 0.90*** | -0.68*** | 0.85*** | |
hp | 0.73*** | -0.33 | -0.36 | -0.75*** | -0.67*** | 0.77*** | -0.52* | | |
drat | -0.13 | 0.74*** | 0.69*** | 0.45 | 0.09 | -0.75*** | | | |
wt | 0.50 | -0.68*** | -0.74*** | -0.59** | -0.23 | | | | |
qsec | -0.66** | -0.15 | -0.20 | 0.79*** | | | | | |
vs | -0.63** | 0.28 | 0.17 | | | | | | |
am | -0.06 | 0.81*** | | | | | | | |
gear | 0.11 | | | | | | | | |
> plot(summary(cor_mtcars))
警告メッセージ:
Removed 45 rows containing missing values (geom_point).
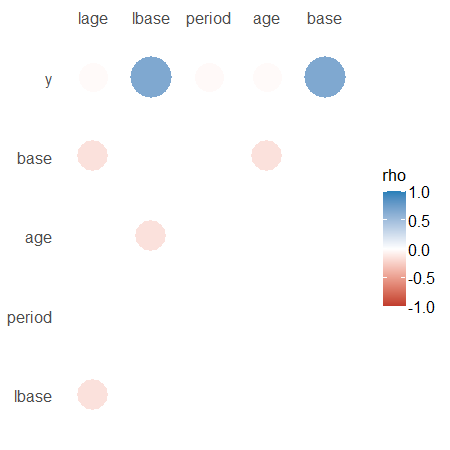
> cor_epil0 <- correlation(epil0, method = "spearman")
> head(cor_epil0)
Parameter1 | Parameter2 | rho | 95% CI | S | p | Method | n_Obs
---------------------------------------------------------------------------------------
y | base | 0.67 | [ 0.59, 0.73] | 7.23e+05 | < .001 | Spearman | 236
y | age | -0.03 | [-0.15, 0.10] | 2.25e+06 | > .999 | Spearman | 236
y | period | -0.03 | [-0.16, 0.10] | 2.25e+06 | > .999 | Spearman | 236
y | lbase | 0.67 | [ 0.59, 0.73] | 7.23e+05 | < .001 | Spearman | 236
y | lage | -0.03 | [-0.15, 0.10] | 2.25e+06 | > .999 | Spearman | 236
base | age | -0.15 | [-0.27, -0.02] | 2.52e+06 | 0.215 | Spearman | 236
> summary(cor_epil0)
Parameter | lage | lbase | period | age | base
--------------------------------------------------------
y | -0.03 | 0.67*** | -0.03 | -0.03 | 0.67***
base | -0.15 | 1.00*** | 0.00 | -0.15 |
age | 1.00*** | -0.15 | 0.00 | |
period | 0.00 | 0.00 | | |
lbase | -0.15 | | | |
> plot(summary(cor_epil0))
警告メッセージ:
Removed 12 rows containing missing values (geom_point).
推定したパラメーター値や信頼区間を取得したい! → parameters
parametersはモデルからのパラメータ推定値の取得を簡単に行えるようにするパッケージです。推定値だけでなく、信頼区間、p値もデータフレーム形式で返してくれます。またplotで簡単に結果を視覚化出来ます。
※詳細記事→glmやglmerによるパラメータ推定値と信頼区間をggplot2で描画する (easystats)
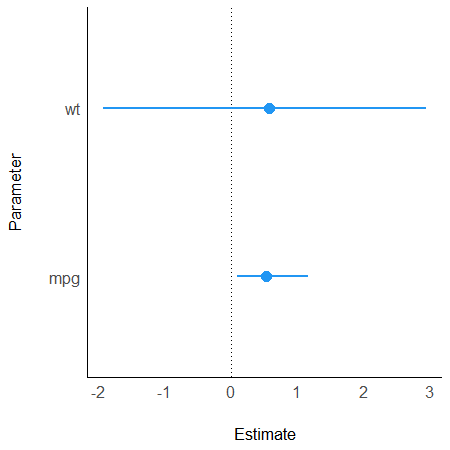
> (param_mtcars <- model_parameters(model_mtcars))
Parameter | Log-Odds | SE | 95% CI | z | p
--------------------------------------------------------------
(Intercept) | -12.54 | 8.47 | [-31.91, 2.92] | -1.48 | 0.139
wt | 0.58 | 1.18 | [ -1.92, 2.94] | 0.49 | 0.623
mpg | 0.52 | 0.26 | [ 0.09, 1.17] | 2.01 | 0.044
> plot(param_mtcars)
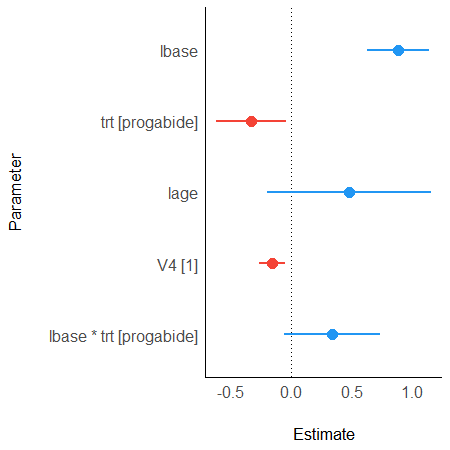
> (param_epil0 <- model_parameters(model_epil0))
Parameter | Log-Mean | SE | 95% CI | z | p
---------------------------------------------------------------------------
(Intercept) | 1.83 | 0.11 | [ 1.63, 2.04] | 17.44 | < .001
lbase | 0.88 | 0.13 | [ 0.63, 1.14] | 6.76 | < .001
trt [progabide] | -0.33 | 0.15 | [-0.62, -0.05] | -2.27 | 0.023
lage | 0.48 | 0.35 | [-0.20, 1.16] | 1.39 | 0.164
V4 [1] | -0.16 | 0.05 | [-0.27, -0.05] | -2.94 | 0.003
lbase * trt [progabide] | 0.34 | 0.20 | [-0.06, 0.74] | 1.67 | 0.094
> plot(param_epil0)
AICやR2とかを確認したい! → performance
performanceパッケージはAICやR2などのモデルの性能評価に関する指標をまとめて返してくれます。また、check_*系の関数を用いることで、多重共線性や過分散などについても検証を行うことが出来ます。
※詳細記事→glmやglmerのR2、多重共線性、正規性、過分散、ゼロ過剰を確認する (easystats)
> # AICやR2の確認
> performance(model_mtcars)
# Indices of model performance
AIC | BIC | R2_Tjur | RMSE | LOGLOSS | SCORE_LOG | SCORE_SPHERICAL | PCP
-----------------------------------------------------------------------------
31.30 | 35.70 | 0.48 | 0.89 | 0.40 | -14.90 | 0.10 | 0.74
> # 多重共線性の確認
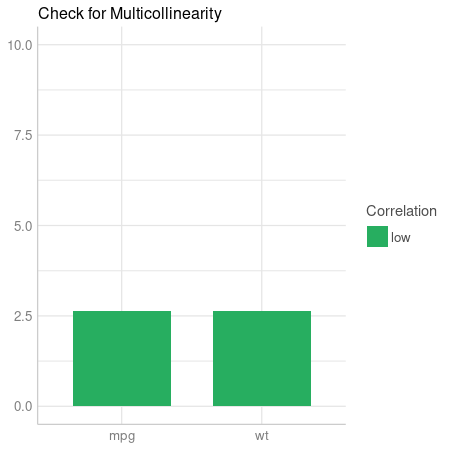
> (vif_mtcars <- check_collinearity(model_mtcars))
# Check for Multicollinearity
Low Correlation
Parameter VIF Increased SE
wt 2.63 1.62
mpg 2.63 1.62
> plot(vif_mtcars)
> # 正規性の確認
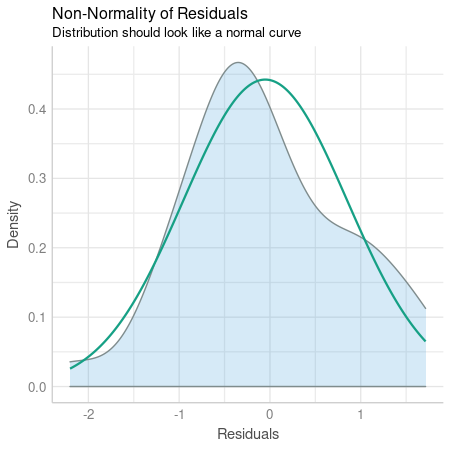
> norm_mtcars <- check_normality(model_mtcars)
OK: Residuals appear as normally distributed (p = 0.601).
> plot(norm_mtcars)
> # AICやR2の確認
> performance(model_epil0)
# Indices of model performance
AIC | BIC | R2_conditional | R2_marginal | ICC | RMSE | SCORE_LOG | SCORE_SPHERICAL
--------------------------------------------------------------------------------------------
1344.95 | 1369.20 | 0.83 | 0.59 | 0.58 | 1.31 | -2.49 | 0.05
> # 多重共線性の確認
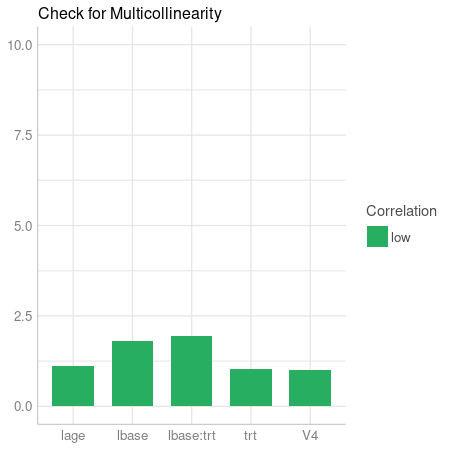
> (vif_epil0 <- check_collinearity(model_epil0))
# Check for Multicollinearity
Low Correlation
Parameter VIF Increased SE
lbase 1.81 1.35
trt 1.03 1.01
lage 1.12 1.06
V4 1.00 1.00
lbase:trt 1.96 1.40
> plot(vif_epil0)
> # 過分散の確認
> (od_epil0 <- check_overdispersion(model_epil0))
# Overdispersion test
dispersion ratio = 1.586
Pearson's Chi-Squared = 363.088
p-value = < 0.001
Overdispersion detected.
パラメータを標準化した際の効果量が知りたい! → effectsize
effectsizeパッケージはパラメータの効果量に関連するさまざまな関数を含んだパッケージです。事後的に標準化したパラメータ推定値を返してくれるだけでなく、一定の基準に基づいて効果量の大小の評価も行ってくれます。
> # パラメータの標準化
> standardize_parameters(model_mtcars)
Waiting for profiling to be done...
Parameter | Coefficient (std.) | 95% CI
------------------------------------------------
(Intercept) | -0.14 | [-1.15, 0.90]
wt | 0.57 | [-1.88, 2.87]
mpg | 3.16 | [ 0.56, 7.02]
> standardize_parameters(model_epil0)
Parameter | Coefficient (std.) | 95% CI
--------------------------------------------------------
(Intercept) | 1.83 | [ 1.63, 2.04]
lbase | 0.66 | [ 0.47, 0.85]
trtprogabide | -0.33 | [-0.62, -0.05]
lage | 0.11 | [-0.04, 0.26]
V41 | -0.16 | [-0.27, -0.05]
lbase:trtprogabide | 0.25 | [-0.04, 0.55]
> # 効果量と評価
> interpret_parameters(model_mtcars)
Waiting for profiling to be done...
Parameter Effect_Size Interpretation
1 (Intercept) NA very large
2 wt 0.5703035 very large
3 mpg 3.1585064 very large
予測値をプロットしたい! → ggeffects
ggeffectsパッケージはモデルに基づいて予測値の算出を行ってくれるパッケージです。特に回帰線を描画する際などに便利で、plotで簡単に描画を行うことも出来ます。またここではご紹介しませんが、出力結果がggplot2と非常に相性がよい形式になっているため、自分で回帰線を書きたいときにもめっちゃ便利です。
※詳細記事→glmやglmerの結果に基づいてggplot2で回帰線を描画する (ggeffects)
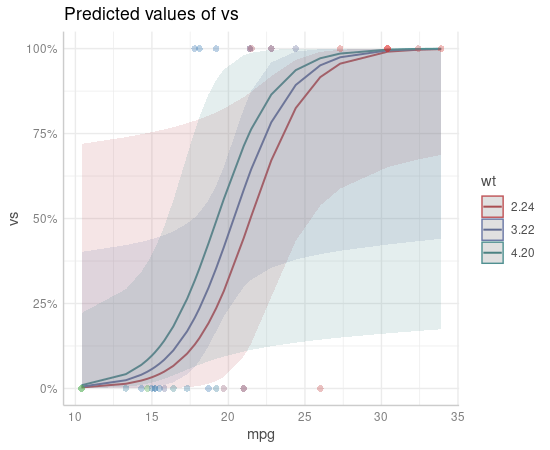
> (predict_mtcars <- ggpredict(model_mtcars, terms = c("mpg [all]", "wt [meansd]")))
# Predicted values of vs
# x = mpg
# wt = 2.24
x | Predicted | SE | 95% CI
---------------------------------------
10.40 | 0.00 | 3.43 | [0.00, 0.72]
15.00 | 0.03 | 2.29 | [0.00, 0.75]
16.40 | 0.07 | 1.96 | [0.00, 0.77]
18.70 | 0.19 | 1.45 | [0.01, 0.80]
21.40 | 0.49 | 0.98 | [0.13, 0.87]
33.90 | 1.00 | 2.93 | [0.69, 1.00]
# wt = 3.22
x | Predicted | SE | 95% CI
---------------------------------------
10.40 | 0.01 | 2.46 | [0.00, 0.40]
15.00 | 0.06 | 1.31 | [0.00, 0.44]
16.40 | 0.11 | 0.98 | [0.02, 0.46]
18.70 | 0.30 | 0.55 | [0.12, 0.55]
21.40 | 0.63 | 0.67 | [0.32, 0.87]
33.90 | 1.00 | 3.75 | [0.44, 1.00]
# wt = 4.2
x | Predicted | SE | 95% CI
---------------------------------------
10.40 | 0.01 | 1.73 | [0.00, 0.22]
15.00 | 0.10 | 0.92 | [0.02, 0.39]
16.40 | 0.18 | 0.87 | [0.04, 0.55]
18.70 | 0.43 | 1.10 | [0.08, 0.87]
21.40 | 0.75 | 1.63 | [0.11, 0.99]
33.90 | 1.00 | 4.71 | [0.17, 1.00]
> plot(predict_mtcars, rawdata = TRUE)
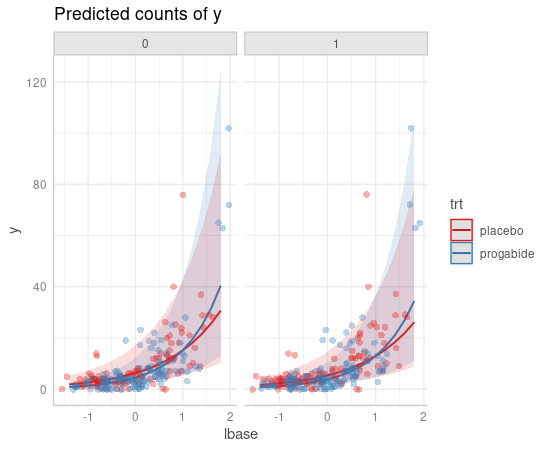
> (predict_epil0 <- ggpredict(model_epil0, type = "re", terms = c("lbase", "trt", "V4"), condition = c(lage = mean(epil0$lage))))
# Predicted counts of y
# x = lbase
# trt = placebo
# V4 = 0
x | Predicted | SE | 95% CI
-----------------------------------------
-1.40 | 1.82 | 0.54 | [ 0.62, 5.28]
-0.80 | 3.08 | 0.52 | [ 1.11, 8.60]
-0.20 | 5.24 | 0.51 | [ 1.92, 14.32]
0.60 | 10.62 | 0.52 | [ 3.85, 29.28]
1.80 | 30.66 | 0.56 | [10.19, 92.24]
# trt = progabide
# V4 = 0
x | Predicted | SE | 95% CI
------------------------------------------
-1.40 | 0.81 | 0.56 | [ 0.27, 2.43]
-0.80 | 1.68 | 0.53 | [ 0.60, 4.76]
-0.20 | 3.51 | 0.51 | [ 1.28, 9.60]
0.60 | 9.32 | 0.52 | [ 3.38, 25.68]
1.80 | 40.40 | 0.57 | [13.13, 124.24]
# trt = placebo
# V4 = 1
x | Predicted | SE | 95% CI
----------------------------------------
-1.40 | 1.55 | 0.55 | [0.53, 4.51]
-0.80 | 2.63 | 0.52 | [0.94, 7.35]
-0.20 | 4.47 | 0.51 | [1.63, 12.24]
0.60 | 9.05 | 0.52 | [3.27, 25.04]
1.80 | 26.13 | 0.56 | [8.66, 78.84]
# trt = progabide
# V4 = 1
x | Predicted | SE | 95% CI
------------------------------------------
-1.40 | 0.69 | 0.56 | [ 0.23, 2.08]
-0.80 | 1.44 | 0.53 | [ 0.51, 4.07]
-0.20 | 2.99 | 0.52 | [ 1.09, 8.21]
0.60 | 7.94 | 0.52 | [ 2.87, 21.95]
1.80 | 34.43 | 0.57 | [11.16, 106.19]
Adjusted for:
* subject = 0 (population-level)
Standard errors are on link-scale (untransformed).
Intervals are prediction intervals.
> plot(predict_epil0, rawdata = TRUE)
parametersやperformanceの結果をまとめて取得したい! → report
reportパッケージはパラメータ推定値やモデルの性能指標をそのままレポートに貼り付けられる形で出力してくれるパッケージです。まとめ的に各数値を確認したい時に便利です。
※詳細記事→glmとかglmerとかの結果を表形式で表示&CSVで出力 (easystats)
> report(model_mtcars) %>% table_long()
Waiting for profiling to be done...
Waiting for profiling to be done...
Waiting for profiling to be done...
Parameter | Coefficient | SE | CI_low.x | CI_high.x | z | df_error | p | Std_Coefficient | CI | CI_low.y | CI_high.y | Fit
----------------------------------------------------------------------------------------------------------------------------------------------
(Intercept) | -12.54 | 8.47 | -31.91 | 2.92 | -1.48 | 29 | 0.14 | -0.14 | 0.95 | -1.15 | 0.90 |
wt | 0.58 | 1.18 | -1.92 | 2.94 | 0.49 | 29 | 0.62 | 0.57 | 0.95 | -1.88 | 2.87 |
mpg | 0.52 | 0.26 | 0.09 | 1.17 | 2.01 | 29 | 0.04 | 3.16 | 0.95 | 0.56 | 7.02 |
| | | | | | | | | | | |
AIC | | | | | | | | | | | | 31.30
BIC | | | | | | | | | | | | 35.70
Tjur's R2 | | | | | | | | | | | | 0.48
RMSE | | | | | | | | | | | | 0.89
LOGLOSS | | | | | | | | | | | | 0.40
SCORE_LOG | | | | | | | | | | | | -14.90
SCORE_SPHERICAL | | | | | | | | | | | | 0.09
PCP | | | | | | | | | | | | 0.74
> report(model_epil0) %>% table_long()
Parameter | Coefficient | SE | CI_low.x | CI_high.x | z | df_error | p | Std_Coefficient | CI | CI_low.y | CI_high.y | Fit
--------------------------------------------------------------------------------------------------------------------------------------------------
(Intercept) | 1.83 | 0.11 | 1.63 | 2.04 | 17.44 | 229 | 0.00 | 1.83 | 0.95 | 1.63 | 2.04 |
lbase | 0.88 | 0.13 | 0.63 | 1.14 | 6.76 | 229 | 0.00 | 0.66 | 0.95 | 0.47 | 0.85 |
trtprogabide | -0.33 | 0.15 | -0.62 | -0.05 | -2.27 | 229 | 0.02 | -0.33 | 0.95 | -0.62 | -0.05 |
lage | 0.48 | 0.35 | -0.20 | 1.16 | 1.39 | 229 | 0.16 | 0.11 | 0.95 | -0.04 | 0.26 |
V41 | -0.16 | 0.05 | -0.27 | -0.05 | -2.94 | 229 | 0.00 | -0.16 | 0.95 | -0.27 | -0.05 |
lbase:trtprogabide | 0.34 | 0.20 | -0.06 | 0.74 | 1.67 | 229 | 0.09 | 0.25 | 0.95 | -0.04 | 0.55 |
| | | | | | | | | | | |
AIC | | | | | | | | | | | | 1344.95
BIC | | | | | | | | | | | | 1369.20
R2 (conditional) | | | | | | | | | | | | 0.83
R2 (marginal) | | | | | | | | | | | | 0.59
ICC | | | | | | | | | | | | 0.58
RMSE | | | | | | | | | | | | 1.31
SCORE_LOG | | | | | | | | | | | | -2.49
SCORE_SPHERICAL | | | | | | | | | | | | 0.05
対応範囲はもっと広い。そして、便利なのはいいこと
このようにeasystatsパッケージを使えばいままで煩雑で面倒だった様々な作業を統一的にそして簡潔に行うことが出来るようになります。
これはみんなのR解析ライフを非常に快適なものにするでしょう。
またeasystatsパッケージが対応しているのはglmやglmerだけではありません。gamをはじめさまざまなモデルに対応しています。ここではまとめきれませんでしたが、ぜひ試してみてください!
insightの対応モデル一覧
もちろん、手作業で情報を抜き出し、処理するのに比べれば、やっていることの中身が確認しにくくなるという点もあると思います。また、統計量の算出などについてはコードやレファレンスを十分確認する必要があると思います。
ですが、こんな便利なパッケージ使わないともったいない!上記のことに注意しながら、うまく使っていければいいのではないかと思います。