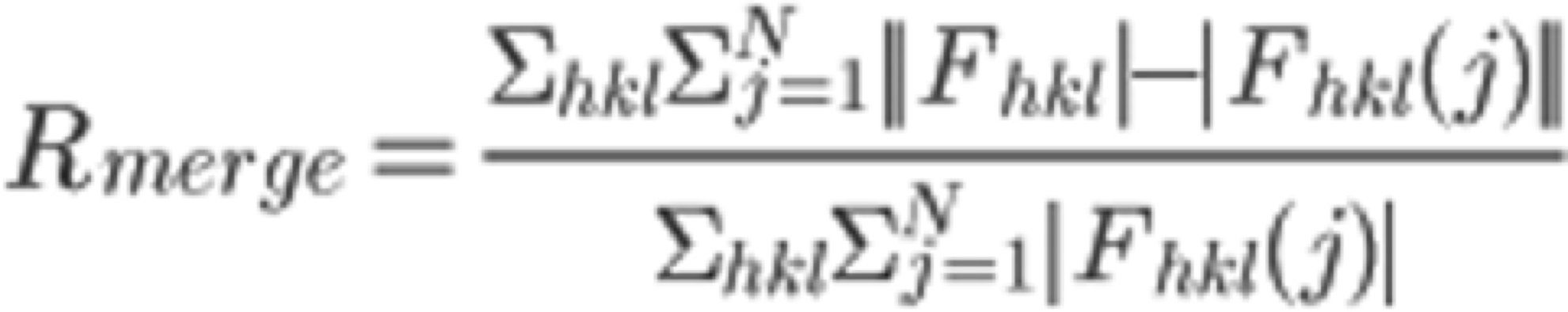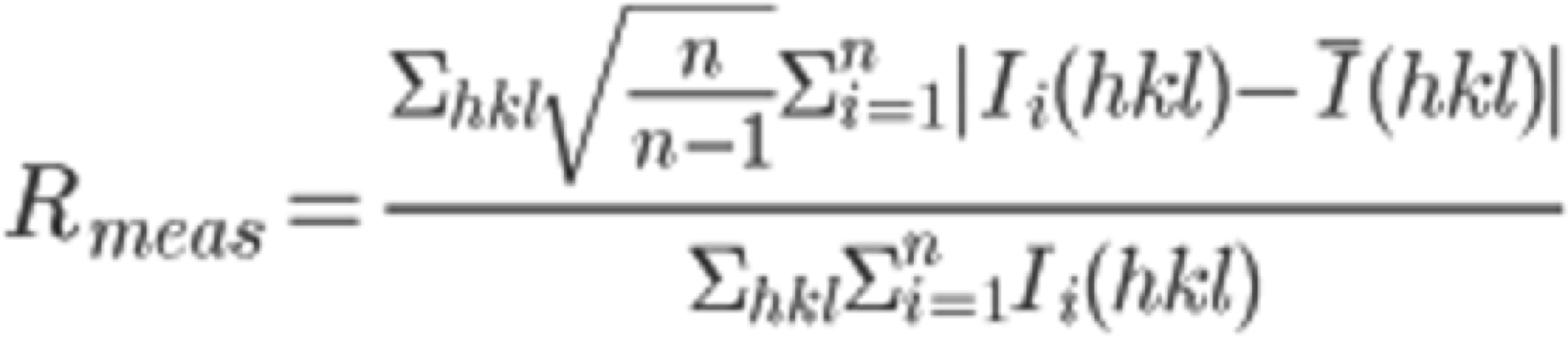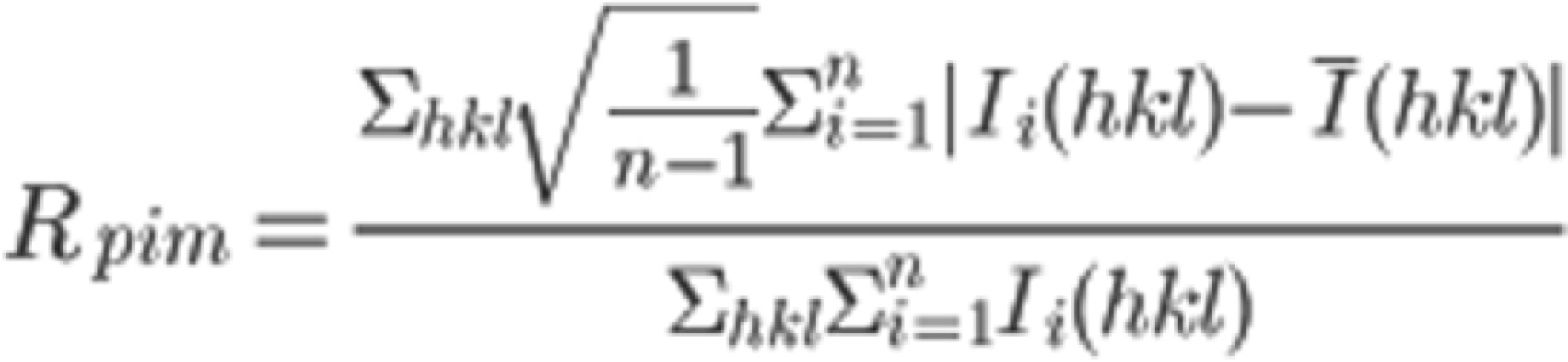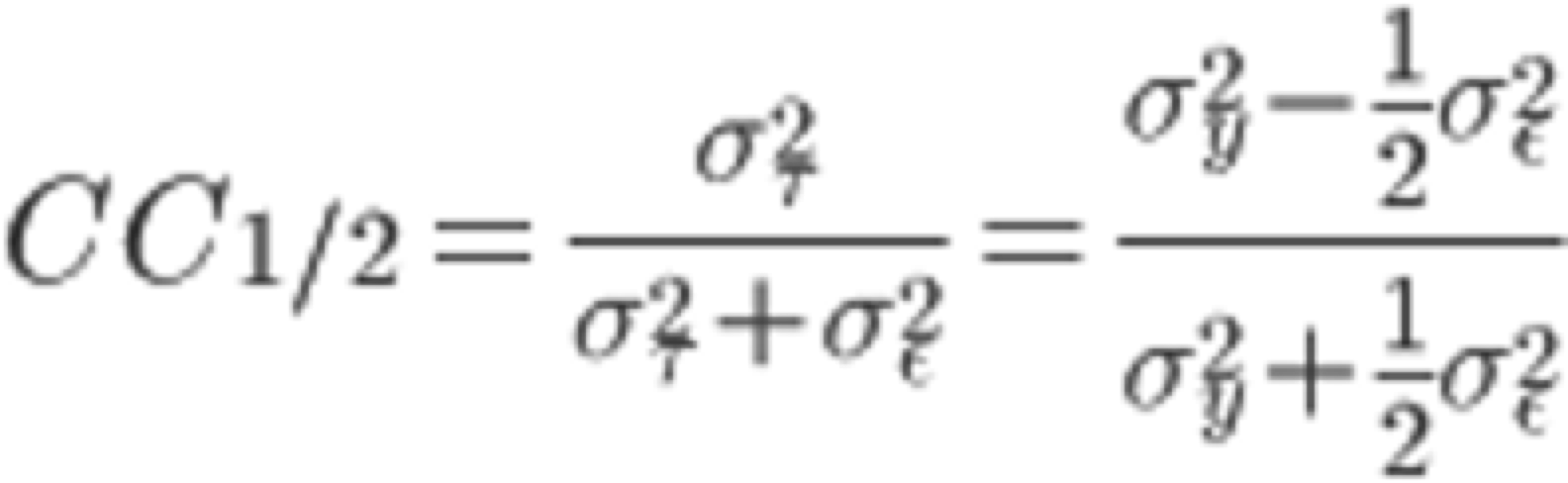はじめに
AlphaFold2時代の構造解析チュートリアルシリーズの連載第2回目(初回はこちら).
今回は回折画像データセットから構造因子$F(hkl)$を求める.
目次
- Step.0 構造決定の流れ
- Step.1 解析ソフトのインストール
- Step.2 回折画像の確認と構造因子の計算 (本記事)
- Step.3 初期位相を求める
- Step.4 構造精密化とバリデーション
この記事の対象と目的
タンパク質X線結晶構造法を専門外とする研究者・大学院生・学部生を対象とし, タンパク質X線結晶構造解析について回折画像から構造決定まで一通りできるようになることを目的とする. 前提として, Linux環境でBashコマンドをある程度打てる方を想定している.
本記事では, タンパク質の構造解析のチュートリアルを全4回の連載でお届けする. 今回は第2回目である.
また, 本記事は完全にオープンである. 学校でも研究機関でも企業でも, ぜひこの記事を広めて, 教育に活用していただければ幸いである. しかし, 著作権は放棄していない.
タンパク質のX先結晶構造解析に必要な計算機環境
筆者の経験から, タンパク質のX線結晶構造解析には以下の計算機環境が適していると考えている. コンピューターパワーが必要であるため, 間違ってもRaspberry Piなどでやってはいけない.
- OS
- Ubuntu, CentOSなどのLinux (強く推奨)
- MacOSX
- Windows 10/11 (ネイティブ環境もしくはWSL2上のLinux環境を使用)
- CPU: > 4 core (推奨:> 16 core)
- Main Memory: > 16 GB (推奨: > 32 GB)
- Storage: > 50 GB (ソフトウェアインストールに約 12 GB.データセット 20 - 30 GB/crystal)
- GPU:必須ではない(推奨: GPU搭載)
Githubレポジトリ
本チュートリアルに関連する代表的なファイルはGithubにアップロードしている. 適宜ご参考いただければ幸いである.
Step.2 回折画像の確認と構造因子の計算
Step.2-1 データの準備
本チュートリアルでは, タンパク質X線結晶構造解析学のHello World構造(?)とも言うべきニワトリの卵白由来の糖質加水分解酵素であるLysozymeのモデル構造を解き明かす. 回折画像とアミノ酸配列を, 以下のリンクからそれぞれ入手する.
| データ | Download URL | 備考 |
|---|---|---|
| 回折画像 | Integrated Resource for Reproducibility in Macromolecular Crystallography, NIH, nsls2_fmx_20161122_lys_266 | 米国にある放射光施設 National Synchrotron Light Sourceのビームライン17-ID-2で, ビームラインの調整目的で取得されたデータ. 約13GB. |
| アミノ酸配列 | UniProt, P00698 LYSC_CHICK | "Sequence"セクションの"Download"ボタンをクリックし, 出てきた配列の19残基目から最後までの配列を, 適当なテキストファイル名(例:lysozyme.fasta)で保存する. 以下に19残基目から最後までのLysozymeの配列を載せるので適宜使用していただきたい. |
KVFGRCELAAAMKRHGLDNYRGYSLGNWVCAAKFESNFNTQATNRNTDGSTDYGILQINSRWWCNDGRTPGSRNLCNIPCSALLSSDITASVNCAKKIVSDGNGMNAWVAWRNRCKGTDVQAWIRGCRL
Step.2-2 回折画像の確認
信頼性のあるタンパク質構造を得るには, 良質な電子雲マップ$\rho (xyz)$が必須である1. 良好な電子雲マップ$\rho (xyz)$のためには, 良質な構造因子$F(hkl)$が必要であり, その決定打となるのは回折画像の質である. つまり, 回折画像の品質確認は良質なタンパク質構造を得るために, 非常に大切なステップである.
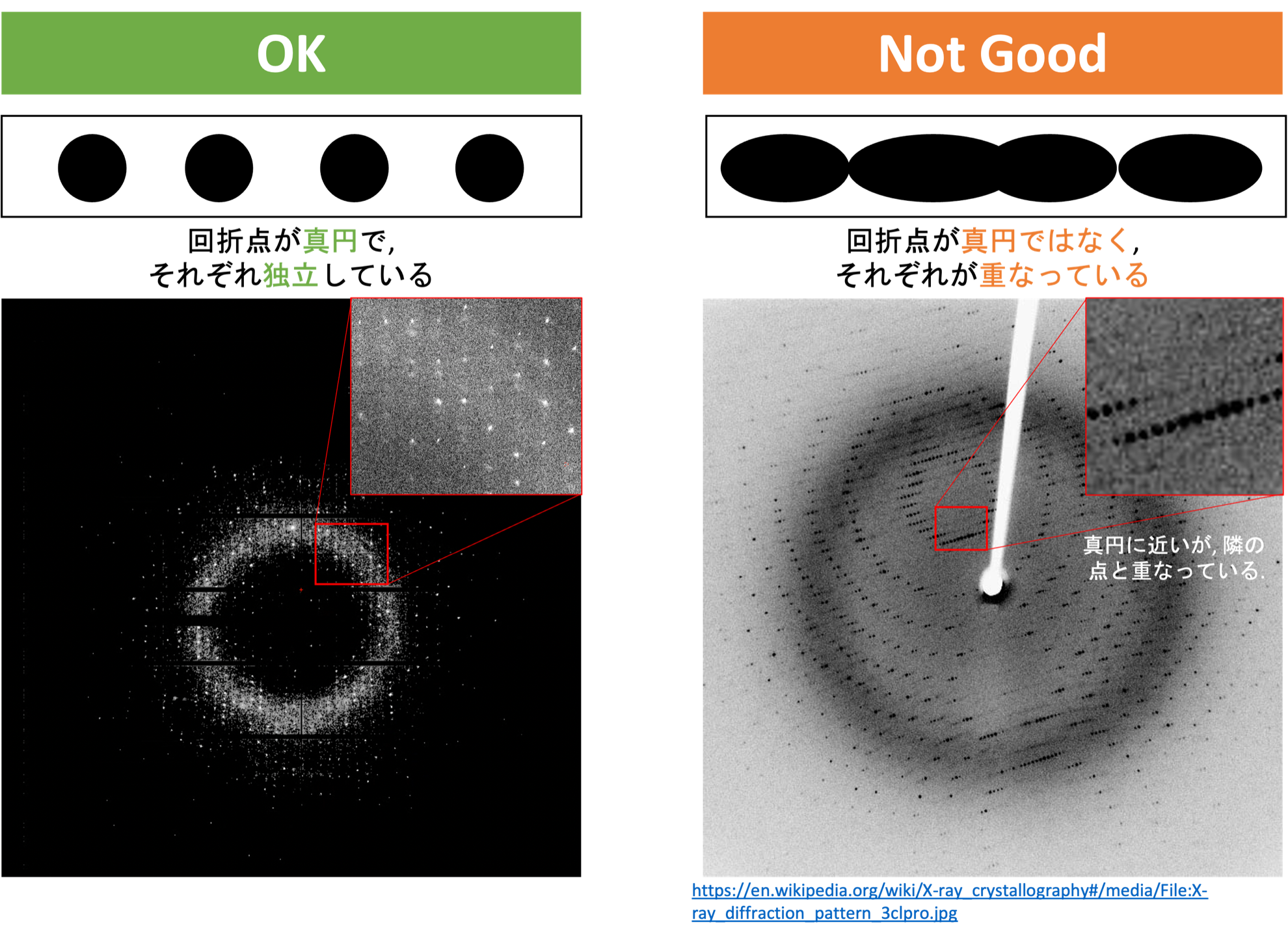
では, 品質が良い回折画像とは一体何なのであろうか?それは「回折点が真円に近く, 回折点同士が重なっていない」画像である. 回折画像の確認ではこの点を主に確認する. 以下に実際の画像を示す.
話が前後するが, そもそも回折画像とは何なのだろうか. タンパク質結晶に限らないが, 結晶にX線を照射すると, 結晶中の分子の並び方によって特定の回折現象が生じ, それが2次元検出器で可視化される. これが回折画像である. なお, 回折画像を得るための実験を"回折実験"と呼称するが、回折実験そのものについてはこのWikipediaの記事で分かりやすく解説されているので参考にされたい.
回折画像には上記図のように規則正しい回折点が観察されるが, 実はこの回折点の位置は以下のパラーメータに依存する事がわかっている.
- 結晶の単位格子の大きさ (3辺の大きさ$a,b,c$と3辺の角度$\alpha, \beta, \gamma$で規定される, これらによって結晶がどのブラベ格子(Bravais Lattice)に属するか決定される.)
- X線の波長(nm)
- 結晶の向き
- 結晶と検出器までの距離
- 検出器の大きさ
構造因子$F(hkl)$を求めるには, 上記全てのパラメーターを明らかにする必要があるが, それは次のステップでソフトウェア(本チュートリアルではXDS)がある程度自動で行ってくれる(Indexingと呼ばれる工程). すべてのパラメータが決定されると, 測定された回折点がどのミラー指数$hkl$に対応したものかが明らかになり, それぞれの回折点強度がミラー指数$hkl$と対応する形でソフトウェアによって算出される(Integrateと呼ばれる工程). それらを統計的にまとめることで構造因子$F(hkl)$が導出される(Scalingと呼ばれる工程). 上記「回折点が真円に近く, 回折点同士が重なっていない」のは, 回折点の位置とその強度を正確に見積もるために必要なのである.
また、最終的にタンパク質のモデル構造を決定するには、結晶の空間群(Space group)の特定が必須である. これはScalingにおいてある程度は消滅則(Extinction rule)によって特定されるが, 最終的には初期位相/タンパク質のモデル構造を解いてみないことには確定できない.
Step.2-3 回折画像フォーマット
さっそく回折画像を見てゆきたいが, その前に二次元検出器から出力される画像フォーマットについて簡単に解説する.
二次元検出器は技術の進化によって, 検出原理がFilm --> CCD --> CMOS-->Hybrid pixelと進化してきており, 検出S/N比・分解能・そして撮像スピードが大幅に向上している. 近年(2014年くらいから)はスイスDECTRIS社の検出器が業界スタンダードになっている.

(現在のフラグシップモデルであるDECTRIS社製EIGER2シリーズ. 出典:https://www.dectris.com/detectors/x-ray-detectors/eiger2/eiger2-for-synchrotrons/eiger2-x/)
回折画像フォーマットは二次元検出器によって異なる. 回折実験はシンクロトロン施設の構造生物学専用ビームラインで行われることが多いが,これまでに様々なタイプの二次元検出器が用いられてきた. CMOS検出器以前に主流であったCCD技術を用いた検出器(~2013年頃)は, 1データセットの測定に20-30分かけて最大360枚程度の回折画像を得ていた. この時代の回折画像は独立した画像ファイルであり, 拡張子は"img"や"osc"であった. しかし, 現在(2023年)のメインはHybrid Pixel技術を用いた検出器群である. これらは1データセットに必要な最大3,600枚程度の回折画像をわずか1-2分で取得する. このように単位時間あたりの撮像枚数の爆発的な増加(そしてS/N比の向上によるX線照射時間の短縮)によって, 研究者は限られたビームタイムにおいて多くの結晶を測定することが可能になった.
一方, 大量の回折画像が短時間で出力されることで, データハンドリング上の課題が生じた2. よって, 近年は深層学習の教師データにも用いられるような階層ファイルコンテナであるHDF5形式で回折画像が保存されることが多い. 本チュートリアルで使用する回折画像もHDF5形式で保存されており, 200枚の画像を1つのHDF5ファイルに統合している.
Step.2-4 回折画像の表示と確認
はじめに回折画像の確認を行い, タンパク質のX線結晶構造解析をスタートする.
- ターミナルで
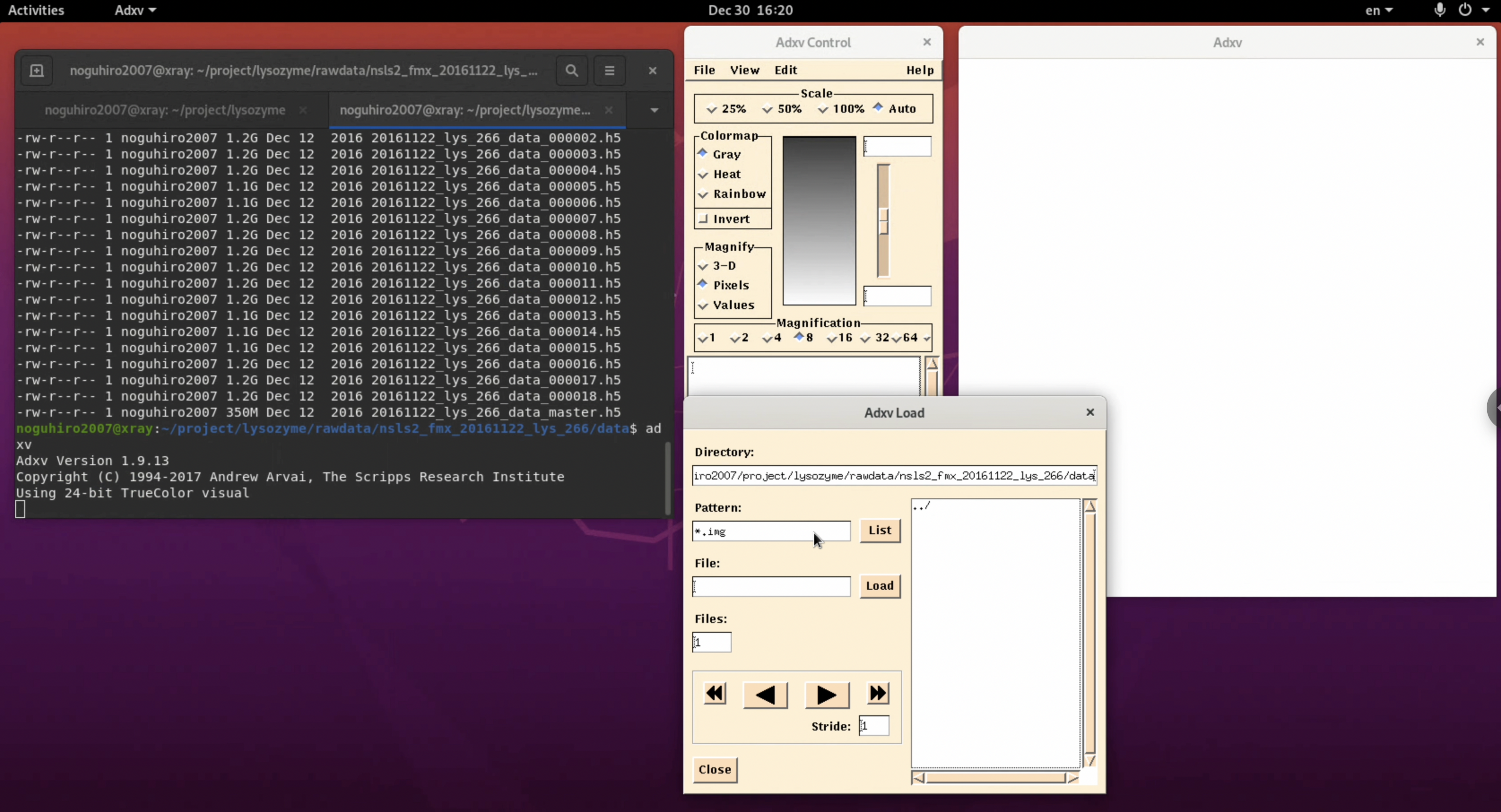
adxvと入力し, adxvを起動する.
$ adxv
2. "Adxv Load" windowのDirectoryで回折画像ファイルが入っているフォルダーを指定し, Patternを" *.img "から" * "に変え, Listをクリックする.
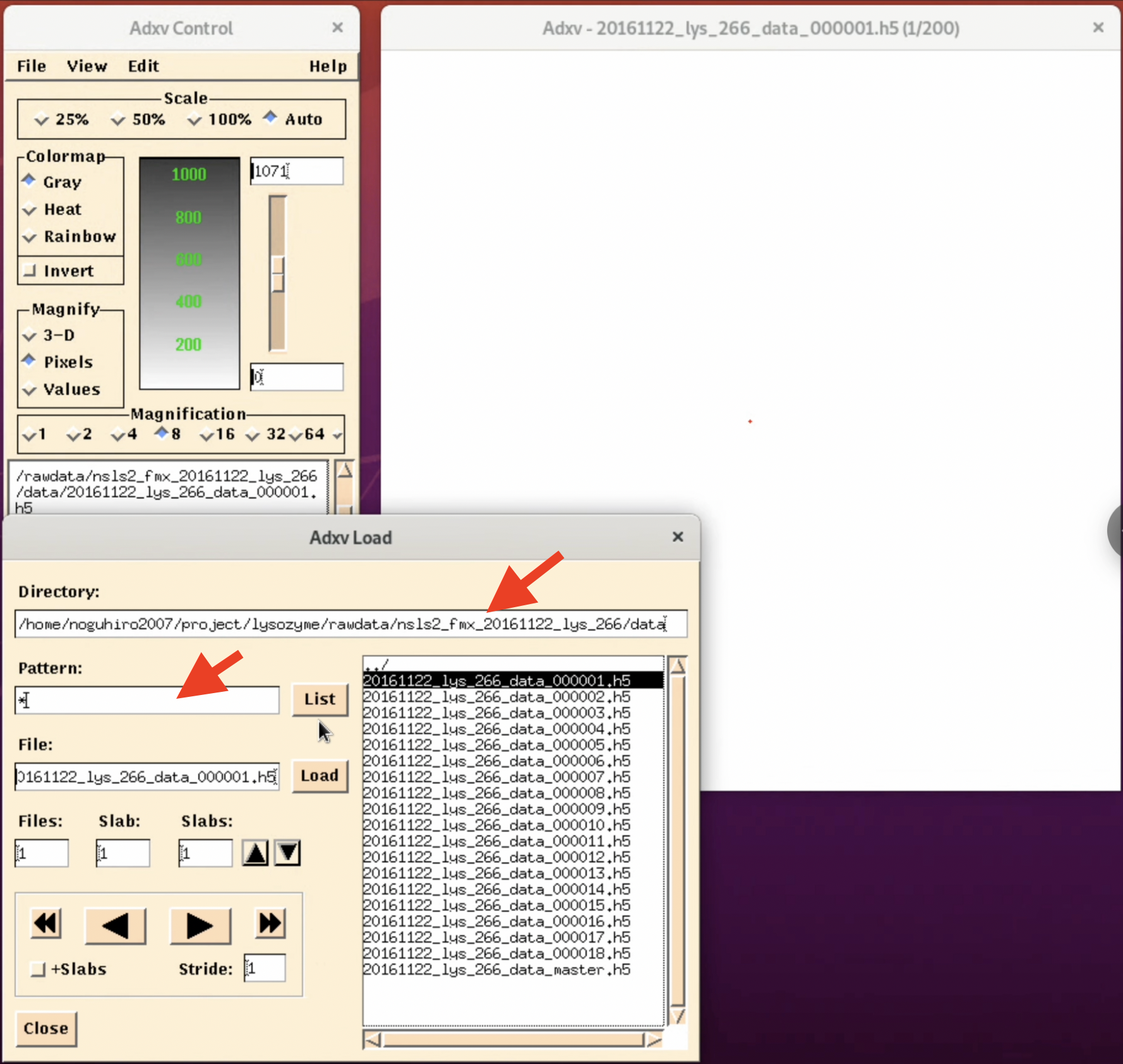
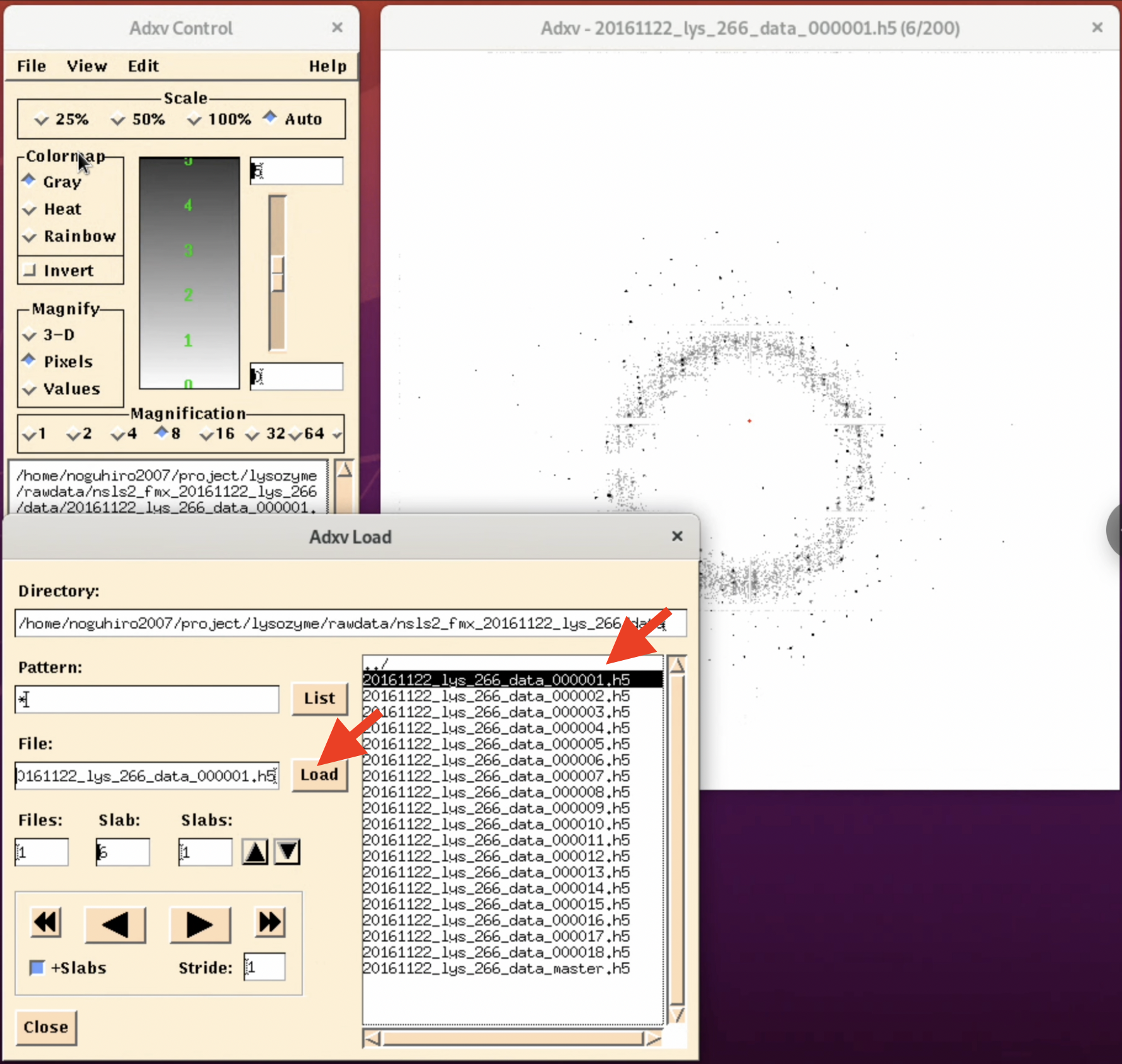
3. "Adxv Load” Windowの右側にファイルリストが一覧で表示される. ここで確認したいimageファイルを選択し, Loadをクリックする.
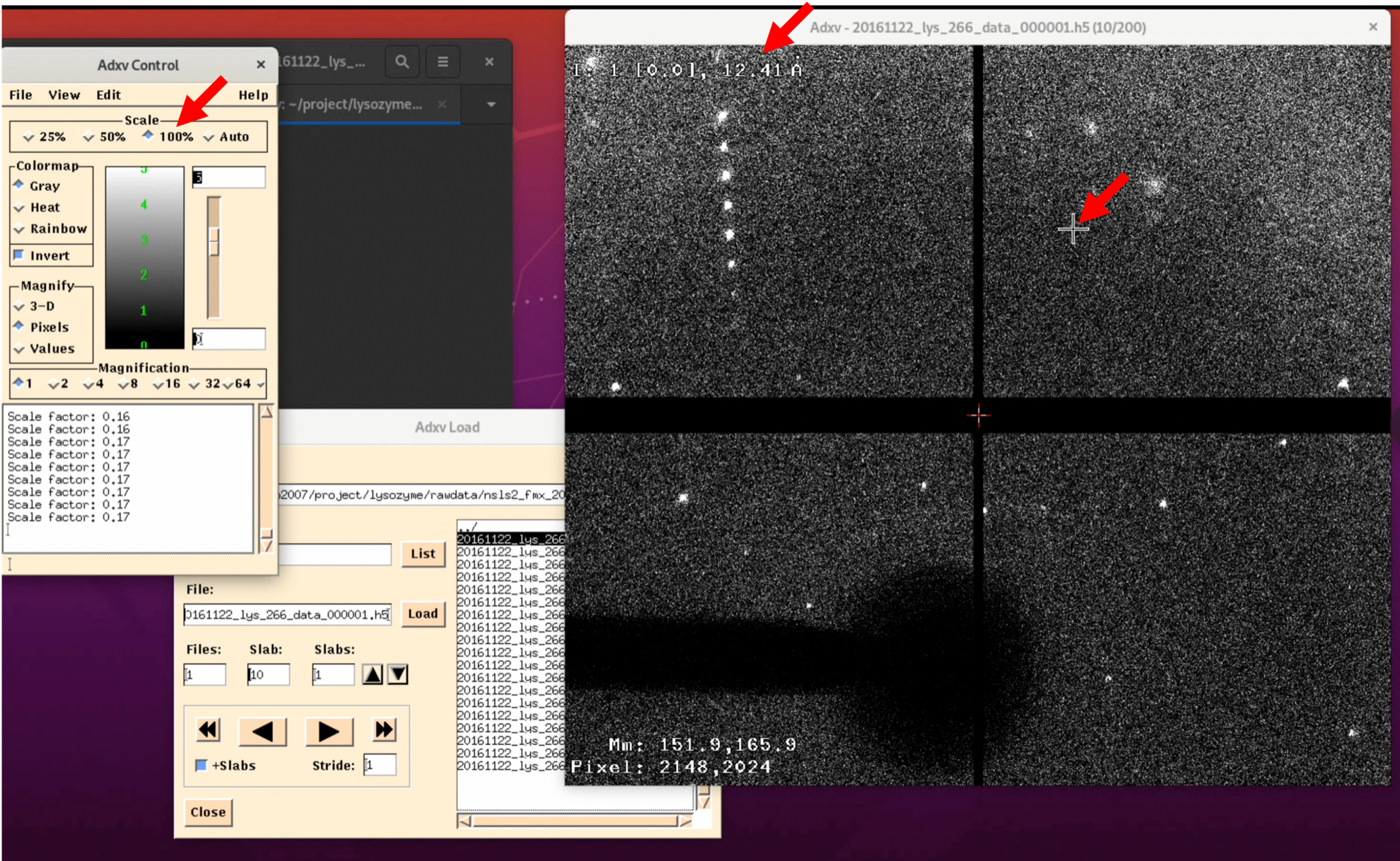
4. より見えやすいように調整をする. "Adxv Control" windowのinvertボタンを選択すると画像が反転し, (人にもよるが)外側の回折点を発見しやすくなる. また, "Adxv Load" windowの矢印ボタンを押すと, 次ファイルの画像を表示する. この際, "+Slabs"をクリックすると, h5の中の回折画像を見れる.
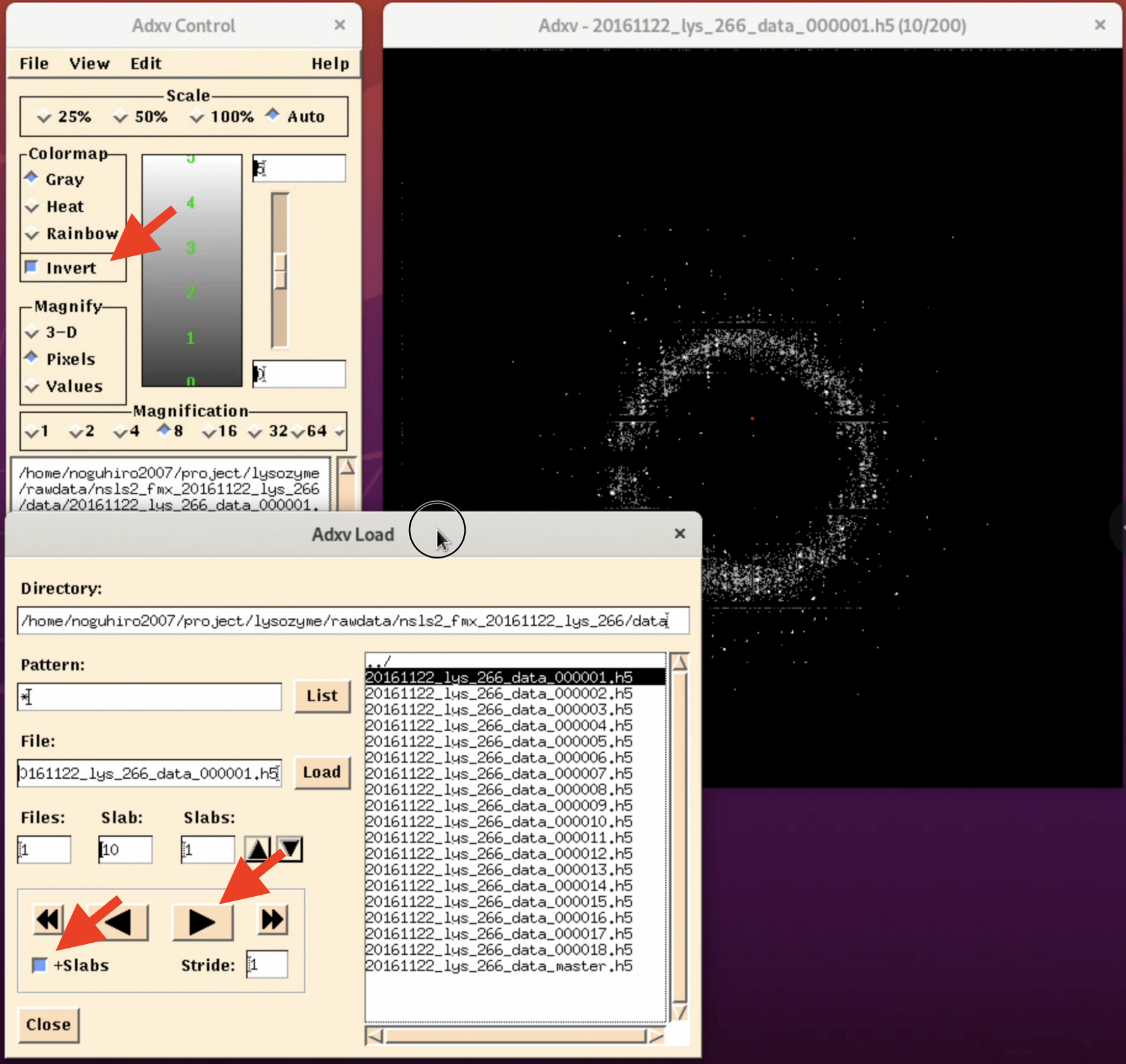
また, "Adxv Control"のScaleは回折画像の拡大率であり, 原寸大だと以下の画像のようになる(実際の拡大率はディスプレイの解像度などに依存する). また, 回折画像の左上にある"12.41 Å"は現在マウスポインタ"+"がある箇所の分解能を示している. 画像の外側に行けば行くほど高分解能であり, そこに回折点が存在すればより信頼性のおける構造を得ることが可能である3. 他の設定もあるので, 色々と探索することをおすすめする.
5. 回折画像のチェック1: 全部を一通り見る
すべての画像をラフに確認し, 途中回折点が抜けていたり, パターンが異なる回折点が出ていないか確認する. 以下の動画のように, "二重再生ボタン"をクリックすると自動で回折画像が再生される.
6. 回折画像のチェック1: 回折点のクオリティをチェックする
0°, +45°, +90°, +135°...と45°ごとの回折画像について, 回折点のクオリティをチェックする. 下図のように回折点が真円で, お互いにかぶさっていなければ問題ない.
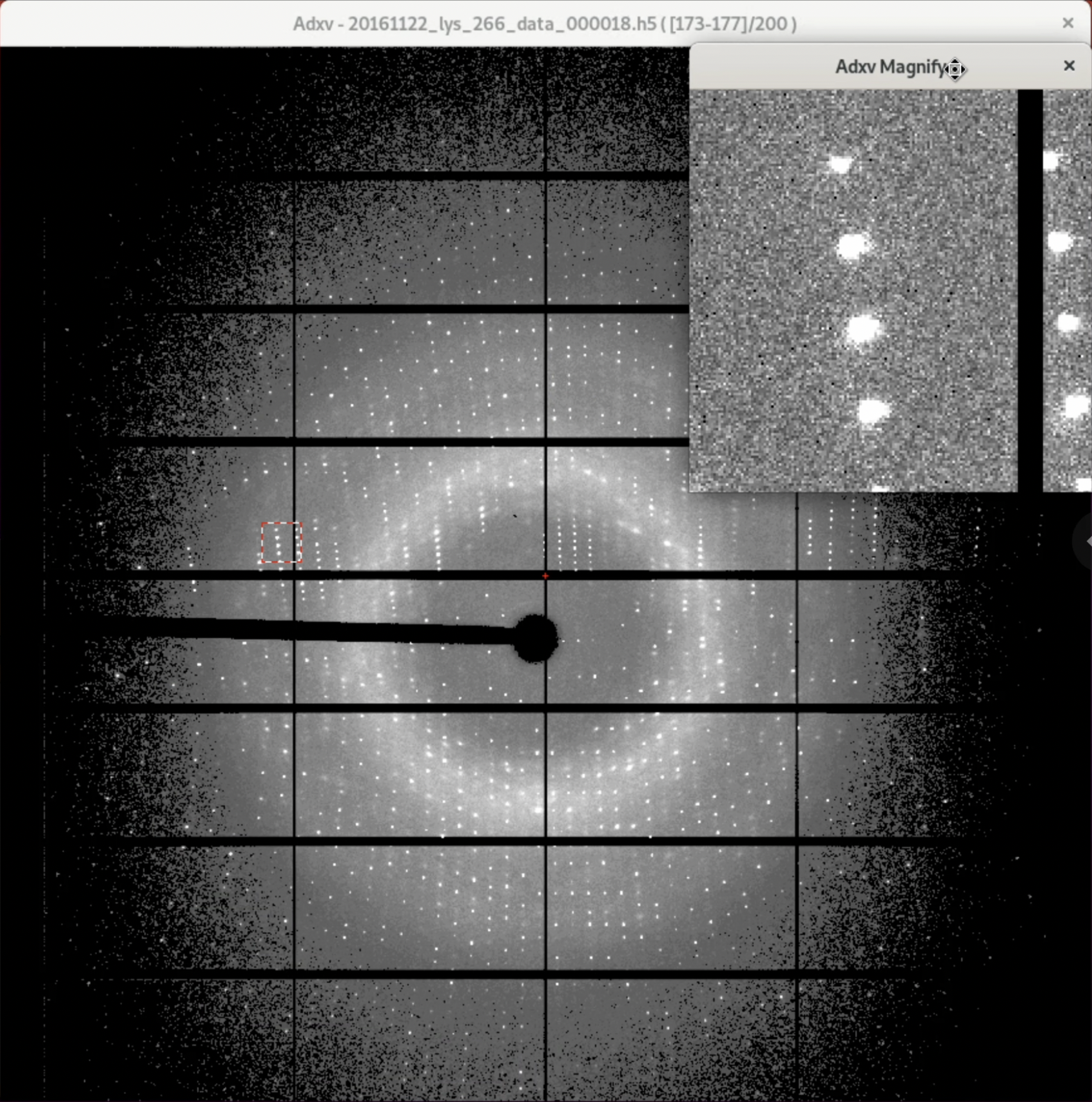
なお, 本チュートリアルで用いるデータセットは非常に品質の良い回折画像である. 他の回折画像をcheckする際のポジティブコントロールとして参考にしていただければ幸いである.
Step.2-5 構造因子の計算
Step.2-5-1 回折画像の処理:XDS
回折画像の品質チェックが終わり, データセットについて大体の感覚がつかめたら次に構造因子$F(hkl)$の計算に取り掛かる.
- ターミナルで
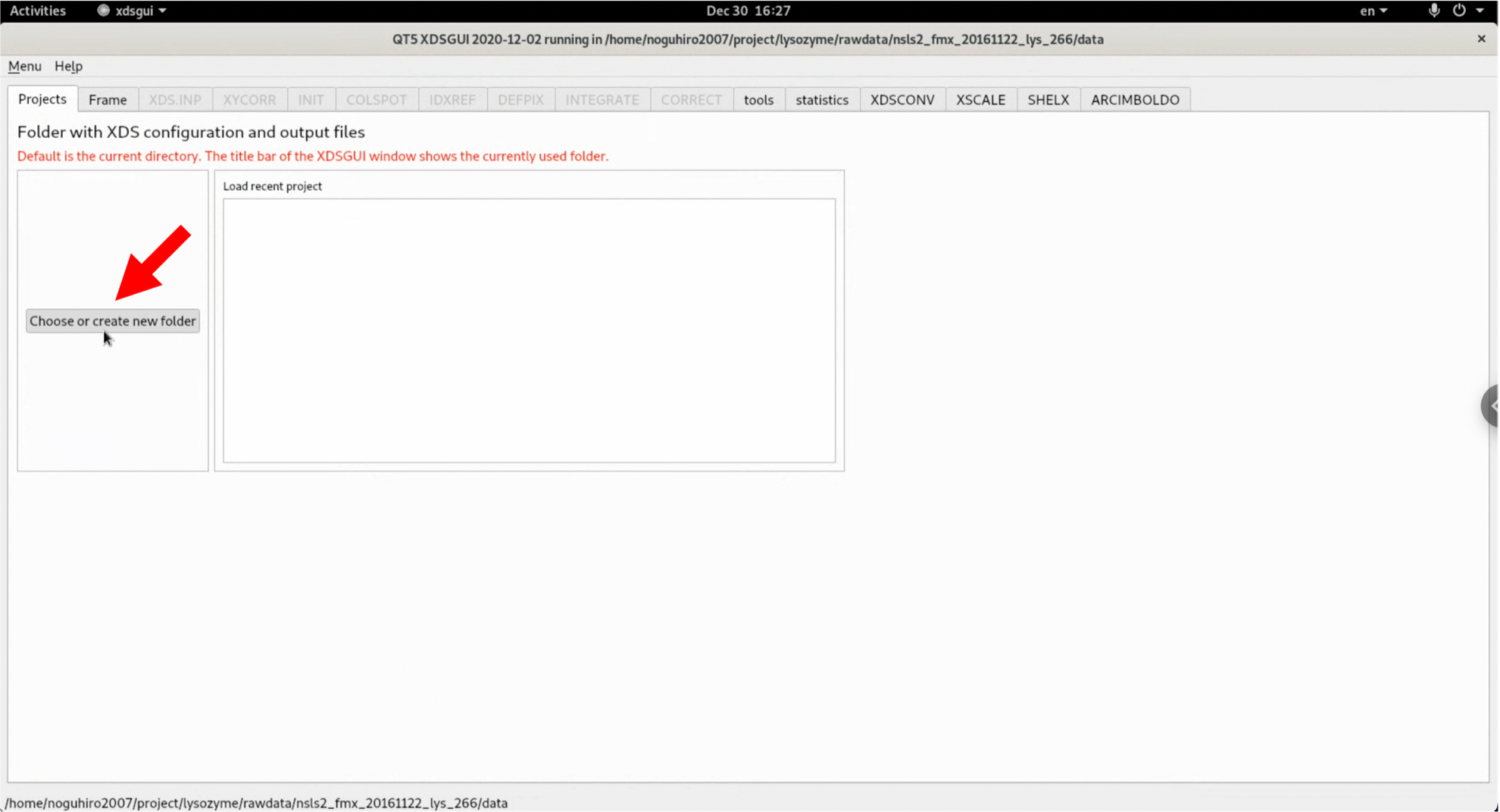
xdsguiと入力し, xdsguiを起動する.
$ xdsgui
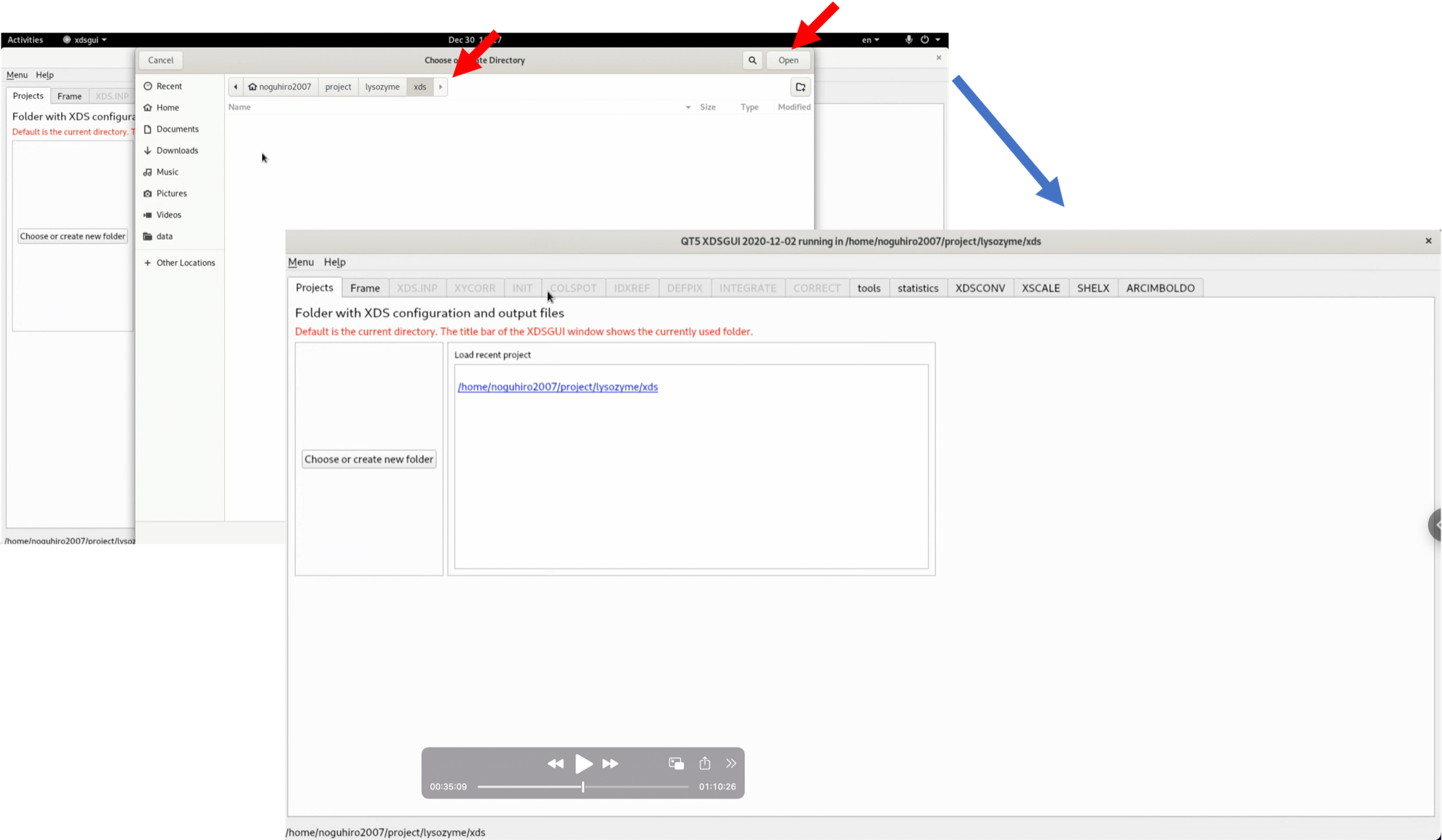
2. xdsguiが起動したら"Choose or create new folder"を選択し, XDSから出力されるファイルの出力フォルダ(project folder)を指定する.
3. "Frame"タブの"Load"をクリックし, ダウンロードして解凍した回折画像ファイルの中から, "20161122_lys_266_data_master.h5"を選択し, Openする. この際, master.h5ファイルを指定する必要があるので注意である.
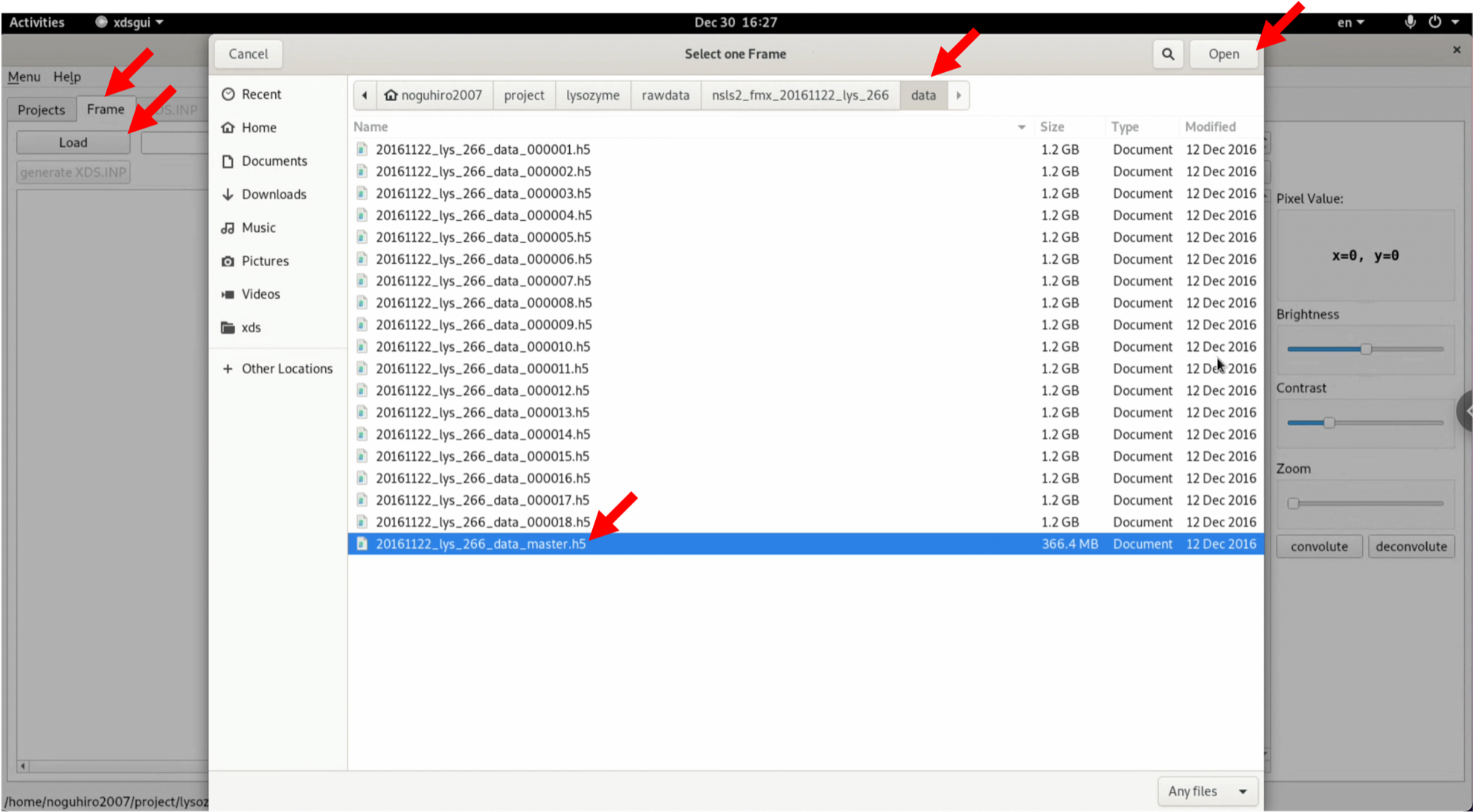
4. 回折画像が表示されるので確認する. 現在, No.0001の画像が見えているが, 何も見えない4. "generate XDS.INP"をクリックし, XDSの処理方法を記述するXDS.INPを自動的に作成する.
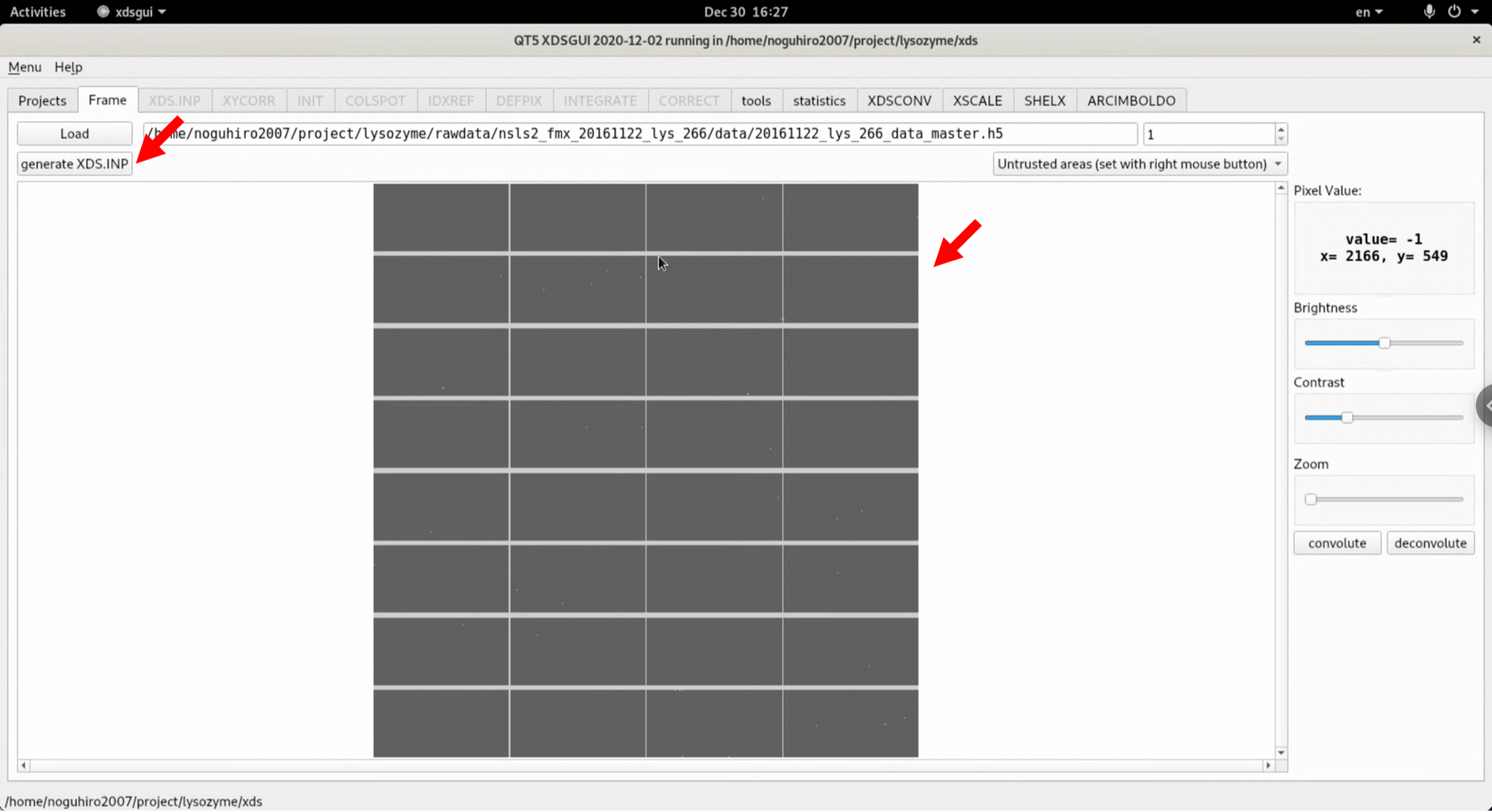
5. "XDS.INP"タブをクリックする. 生成されたXDS.INPが表示され, 編集も可能である. まずは, 生成されたXDS.INPを編集せず, そのまま動かしてみる. 非常にきれいなデータであるなど, いくつかの好条件が揃えば, このようなオート状態でも一発で全体の処理は完了する.
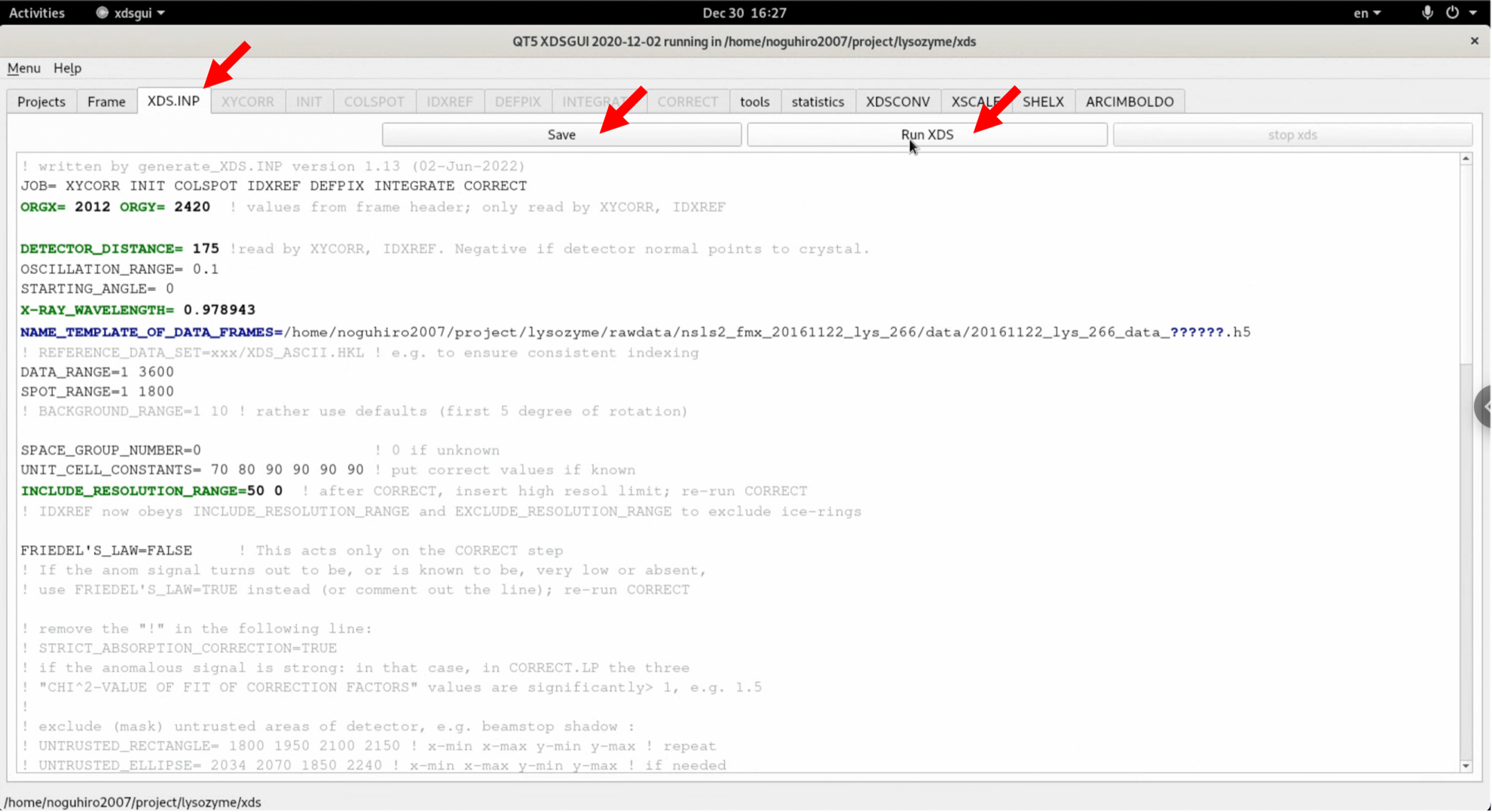
6. 処理が始まる. 処理中はCPUがほぼ100%使われるので, 他の作業をやる際は注意が必要である. 計算機環境にもよるが, 数十分待つ.
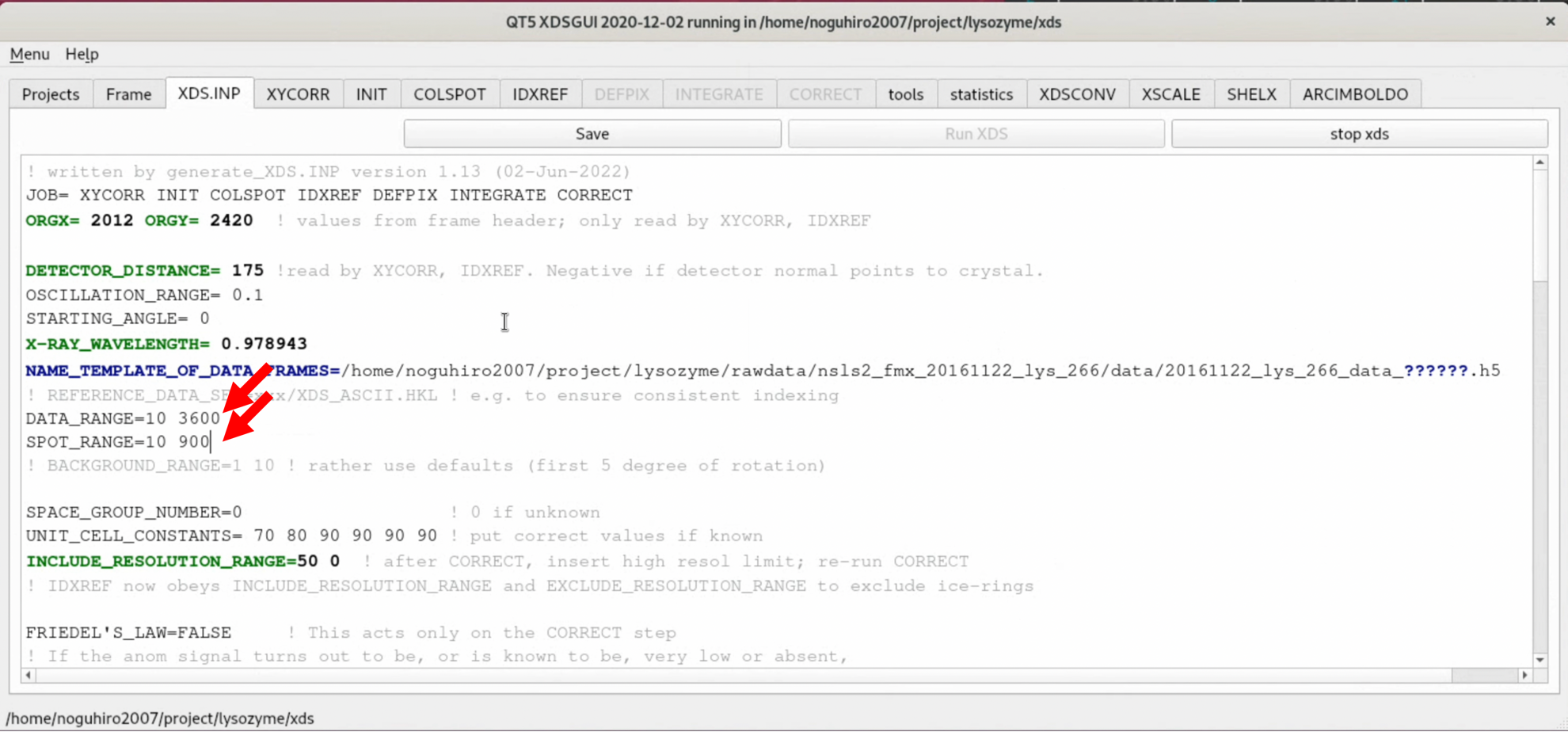
7. 筆者の環境ではIDXREFのステップ(Indexing)でエラーが発生した. どうやらパラメータの精密化工程でエラーが発生したらしい. 回折画像のチェックで明らかなように, 本データセットは最初の数枚に回折点がなく, それが悪さをしている可能性がある. また, 初期設定では1,800枚(180°)のデータを精密化に使用しているが, これは明らかに過剰であるため範囲を狭める.
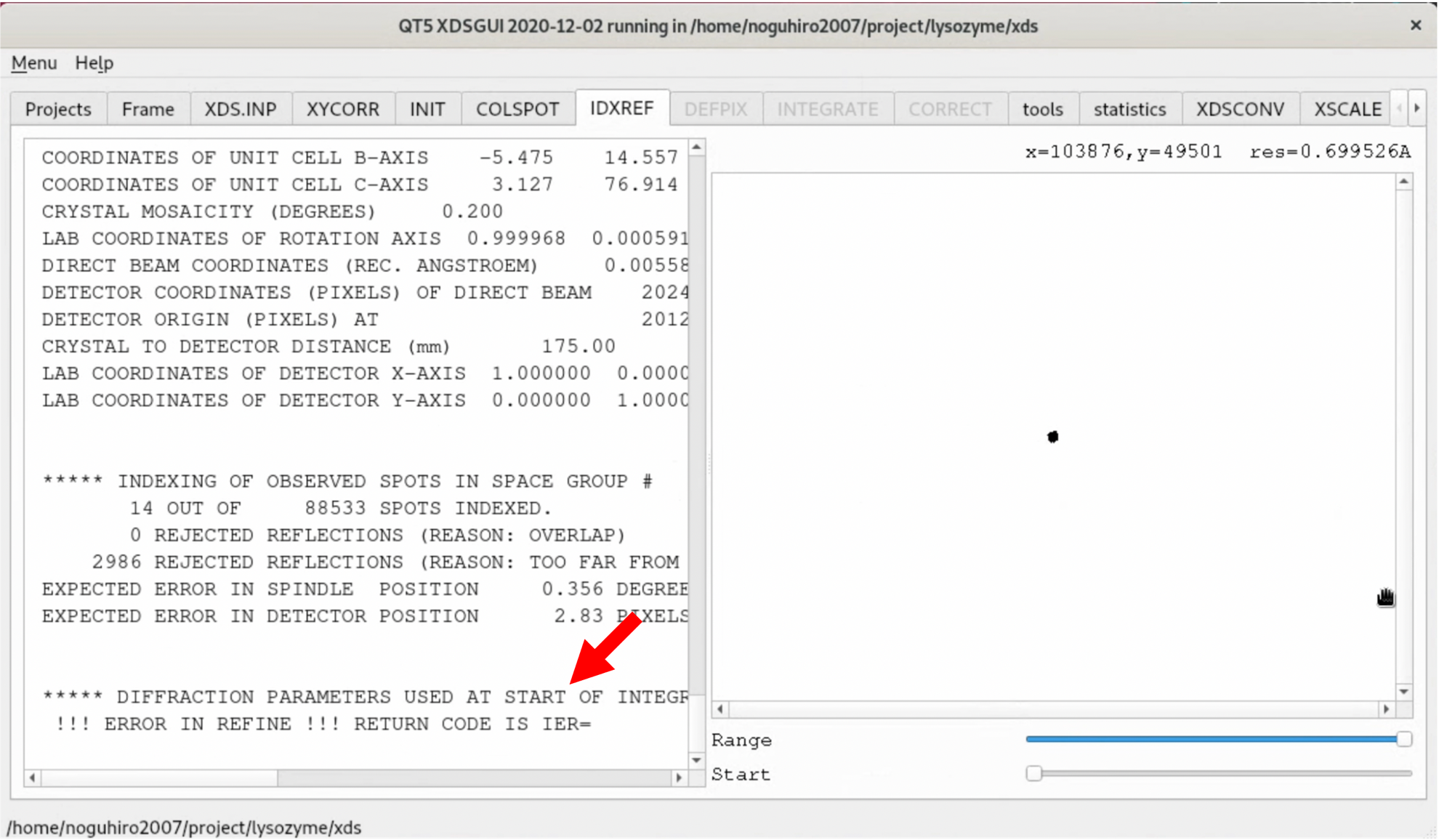
8. "XDS.INP"タブからXDS.INP編集画面を開き, 以下の部分を変更する. これにより, 回折点が存在しない最初の10枚程度を処理から外し, なおかつIndexingの処理に用いる画像を回転角90°までのものに絞る. 変更したら, "Save" --> "Run XDS"して処理を始める.
- DATA_RANGE=0 3600
+ DATA_RANGE=10 3600
- SPOT_RANGE=0 1800
+ SPOT_RANGE=10 900
(RunXDSの様子)
IDXREFでは, COLSPOTでピックアップされた回折点からブラベ格子が予測される(Indexing). ソフトの内部では最も対象性の低いブラベ格子であるaP(三斜格子,$a\neq b \neq c$, $\alpha \neq \beta \neq \gamma$)から徐々に対称性のあるブラベ格子を当てはめてゆき, ピックアップされた回折点に合うギリギリの対称性のそれを見極める. そうやって選ばれた最も対象性の高いブラベ格子が, 本結晶のブラベ格子である可能性が一番高い. 今回の例ではtP(単純正方格子,$a = b \neq c$, $\alpha = \beta =\gamma =90°$)ではないかと予測された(以下のIDEXREF.LPを参照).
...
*********** DETERMINATION OF LATTICE CHARACTER AND BRAVAIS LATTICE ***********
The CHARACTER OF A LATTICE is defined by the metrical parameters of its
reduced cell as described in the INTERNATIONAL TABLES FOR CRYSTALLOGRAPHY
Volume A, p. 746 (KLUWER ACADEMIC PUBLISHERS, DORDRECHT/BOSTON/LONDON, 1989).
Note that more than one lattice character may have the same BRAVAIS LATTICE.
!!! WARNING !!! For unknown crystals an augmented lattice basis may have been
constructed that could obscure the recognition of the correct
unit cell. See: "merged subtrees" in this file (IDXREF.LP).
A lattice character is marked "*" to indicate a lattice consistent with the
observed locations of the diffraction spots. These marked lattices must have
low values for the QUALITY OF FIT and their implicated UNIT CELL CONSTANTS
should not violate the ideal values by more than
MAXIMUM_ALLOWED_CELL_AXIS_RELATIVE_ERROR= 0.03
MAXIMUM_ALLOWED_CELL_ANGLE_ERROR= 2.0 (Degrees)
LATTICE- BRAVAIS- QUALITY UNIT CELL CONSTANTS (ANGSTROEM & DEGREES)
CHARACTER LATTICE OF FIT a b c alpha beta gamma
* 44 aP 0.0 37.2 78.3 78.5 90.1 90.0 90.0
* 31 aP 0.2 37.2 78.3 78.5 89.9 90.0 90.0
* 35 mP 0.4 78.3 37.2 78.5 90.0 90.1 90.0
* 33 mP 2.5 37.2 78.3 78.5 90.1 90.0 90.0
* 34 mP 2.5 37.2 78.5 78.3 90.1 90.0 90.0
* 32 oP 2.7 37.2 78.3 78.5 90.1 90.0 90.0
* 25 mC 7.5 110.8 111.0 37.2 90.0 90.0 89.9
* 23 oC 7.9 110.8 111.0 37.2 90.0 90.0 89.9
* 20 mC 8.1 111.0 110.8 37.2 90.0 90.0 90.1
* 21 tP 10.2 78.3 78.5 37.2 90.0 90.0 90.1
37 mC 249.9 161.3 37.2 78.3 90.0 90.1 76.7
39 mC 250.1 161.1 37.2 78.5 90.0 90.1 76.6
36 oC 252.2 37.2 161.3 78.3 89.9 90.0 103.3
28 mC 252.3 37.2 161.3 78.3 89.9 90.0 76.7
38 oC 252.3 37.2 161.1 78.5 89.9 90.0 103.4
29 mC 252.4 37.2 161.1 78.5 89.9 90.0 76.6
27 mC 500.1 161.1 37.2 110.8 90.0 133.4 76.6
19 oI 507.6 37.2 110.8 117.0 89.9 71.4 90.0
26 oF 622.9 37.2 161.1 161.3 86.9 103.3 103.4
18 tI 630.3 110.8 117.0 37.2 71.4 90.0 90.1
1 cF 999.0 116.9 116.9 117.0 95.9 95.7 142.9
2 hR 999.0 86.8 86.9 117.0 118.0 62.1 100.7
3 cP 999.0 37.2 78.3 78.5 90.1 90.0 90.0
5 cI 999.0 86.9 86.7 110.8 50.4 50.3 79.5
4 hR 999.0 86.8 86.9 116.9 118.0 62.1 100.6
6 tI 999.0 110.8 86.9 86.7 79.5 50.4 50.3
7 tI 999.0 86.9 86.7 110.8 50.4 50.3 79.5
8 oI 999.0 86.7 86.9 110.8 50.3 50.4 79.5
9 hR 999.0 37.2 86.8 250.8 102.6 98.5 115.4
10 mC 999.0 86.7 86.8 78.5 90.0 90.1 129.2
11 tP 999.0 37.2 78.3 78.5 90.1 90.0 90.0
12 hP 999.0 37.2 78.3 78.5 90.1 90.0 90.0
13 oC 999.0 86.7 86.8 78.5 90.0 90.1 50.8
15 tI 999.0 37.2 78.3 179.3 64.1 78.0 90.0
16 oF 999.0 86.7 86.8 179.3 107.7 118.9 50.8
14 mC 999.0 86.7 86.8 78.5 90.0 90.1 50.8
17 mC 999.0 86.8 86.7 86.9 79.5 100.7 50.8
22 hP 999.0 78.3 78.5 37.2 90.0 90.0 90.1
24 hR 999.0 179.1 111.0 37.2 90.0 78.0 108.0
30 mC 999.0 78.3 175.4 37.2 90.0 90.0 63.5
40 oC 999.0 78.3 175.4 37.2 90.0 90.0 116.5
42 oI 999.0 37.2 78.3 179.3 115.9 102.0 90.0
41 mC 999.0 175.4 78.3 37.2 90.0 90.0 63.5
43 mI 999.0 86.7 179.3 78.3 115.9 154.6 61.1
For protein crystals the possible space group numbers corresponding to
each Bravais-type are given below for your convenience. Note, that
reflection integration is based only on orientation and metric of the
lattice. It does not require knowledge of the correct space group!
Thus, if no such information is provided by the user in XDS.INP,
reflections are integrated assuming a triclinic reduced cell lattice;
the space group is assigned automatically or by the user in the last
step (CORRECT) when integrated intensities are available.
****** LATTICE SYMMETRY IMPLICATED BY SPACE GROUP SYMMETRY ******
BRAVAIS- POSSIBLE SPACE-GROUPS FOR PROTEIN CRYSTALS
TYPE [SPACE GROUP NUMBER,SYMBOL]
aP [1,P1]
mP [3,P2] [4,P2(1)]
mC,mI [5,C2]
oP [16,P222] [17,P222(1)] [18,P2(1)2(1)2] [19,P2(1)2(1)2(1)]
oC [21,C222] [20,C222(1)]
oF [22,F222]
oI [23,I222] [24,I2(1)2(1)2(1)]
tP [75,P4] [76,P4(1)] [77,P4(2)] [78,P4(3)] [89,P422] [90,P42(1)2]
[91,P4(1)22] [92,P4(1)2(1)2] [93,P4(2)22] [94,P4(2)2(1)2]
[95,P4(3)22] [96,P4(3)2(1)2]
tI [79,I4] [80,I4(1)] [97,I422] [98,I4(1)22]
hP [143,P3] [144,P3(1)] [145,P3(2)] [149,P312] [150,P321] [151,P3(1)12]
[152,P3(1)21] [153,P3(2)12] [154,P3(2)21] [168,P6] [169,P6(1)]
[170,P6(5)] [171,P6(2)] [172,P6(4)] [173,P6(3)] [177,P622]
[178,P6(1)22] [179,P6(5)22] [180,P6(2)22] [181,P6(4)22] [182,P6(3)22]
hR [146,R3] [155,R32]
cP [195,P23] [198,P2(1)3] [207,P432] [208,P4(2)32] [212,P4(3)32]
[213,P4(1)32]
cF [196,F23] [209,F432] [210,F4(1)32]
cI [197,I23] [199,I2(1)3] [211,I432] [214,I4(1)32]
Maximum oscillation range to prevent angular overlap at high resolution limit
assuming zero (!) mosaicity.
Maximum oscillation range High resolution limit
(degrees) (Angstrom)
2.93 4.00
2.19 3.00
1.46 2.00
0.73 1.00
cpu time used 47.0 sec
elapsed wall-clock time 3.7 sec
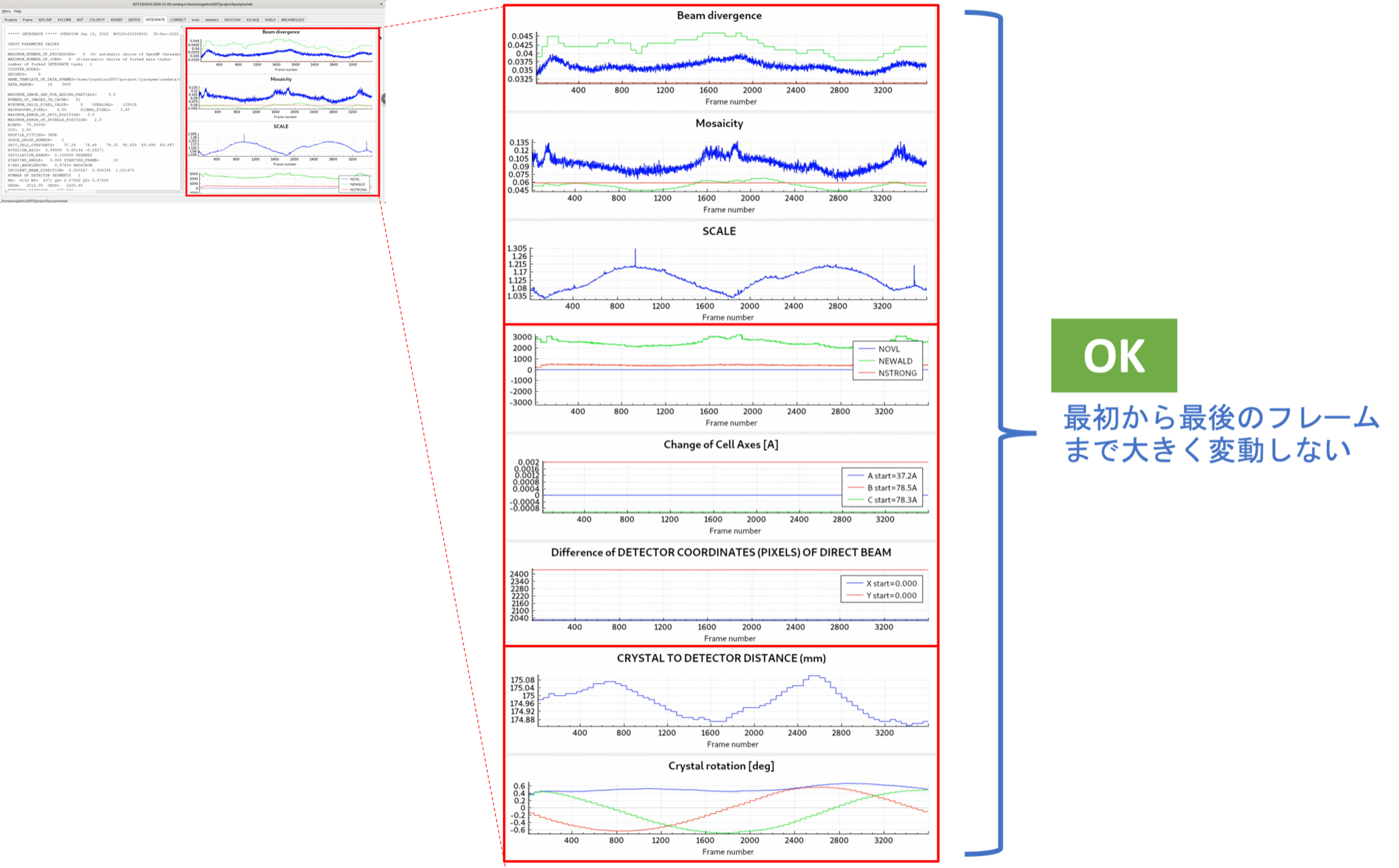
IDXREFで推定されたブラベ格子のパラメーターによって「特定のミラー指数$hkl$の回折点(例えば $h=1, k=1, l=2$)が, 回折画像のココに出るはず!」とソフトが予想できるようになる. INTEGRATEは, 指定した回折画像範囲(DATA_RANGE)の回折点の強度を収集する. INTEGRATEタブの右側には処理中(処理後)の各フレーム別のグラフ化されたパラメータが表示され, 大まかにデータセットの様子を確認できる. 各パラメータのグラフは大きく変動しなければ良い. これが大幅にずれる場合は, IDXREFで推定されたブラベ格子が間違っている場合が多い.
本チュートリアルで使用しているデータセットは非常に品質が良い. 今後の構造解析において, ポジティブコントロールとして参考になる.
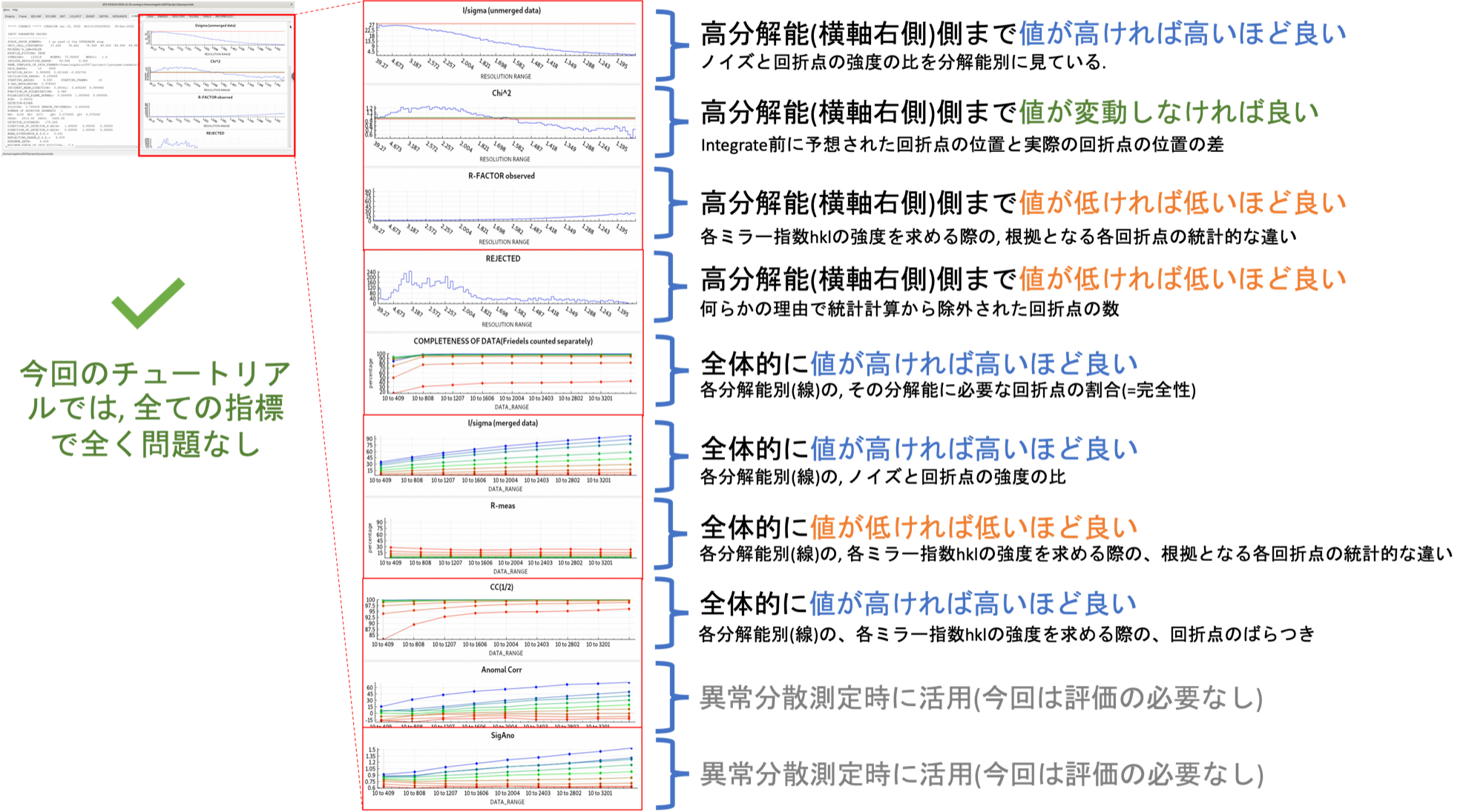
"COLLECT"タブでは, INTEGRATEで収集された各回折点の強度などを統計処理(Scaling)された情報が出力される. xdsguiの右側フレームにはscalingの結果がグラフ化されて表示される. かなり簡単に評価指標などを説明した図5を, 以下に示す. あくまで「そうなんだ」程度でご認識いただきたい. くどいが, 本チュートリアルで使用したデータセットは非常にきれいな統計値であるため, ポジティブコントロールとして今後のご参考にしてほしい.
COLLECTステップが無事に終了すると, 目的のファイルであるXDS_ASCII.HKLを得られる. 本ファイルはファイル名通りTextファイルであり, 各$hkl$の回折強度と標準偏差などに加え, 結晶学的情報なども記載される.
!FORMAT=XDS_ASCII MERGE=FALSE FRIEDEL'S_LAW=FALSE
!OUTPUT_FILE=XDS_ASCII.HKL DATE=30-Dec-2022
!Generated by CORRECT (VERSION Jan 10, 2022 BUILT=20220820)
!PROFILE_FITTING= TRUE
!NAME_TEMPLATE_OF_DATA_FRAMES=/home/noguhiro2007/project/lysozyme/rawdata/nsls2_fmx_20161122_lys_266/data/20161122_lys_266_data_??????.h5 CBF
!DATA_RANGE= 10 3600
!ROTATION_AXIS= 0.999985 0.000941 0.005428
!OSCILLATION_RANGE= 0.100000
!STARTING_ANGLE= 0.000
!STARTING_FRAME= 10
!INCLUDE_RESOLUTION_RANGE= 50.000 1.152
!SPACE_GROUP_NUMBER= 89
!UNIT_CELL_CONSTANTS= 78.538 78.538 37.285 90.000 90.000 90.000
!UNIT_CELL_A-AXIS= 3.607 76.771 16.173
!UNIT_CELL_B-AXIS= 4.469 -16.365 76.684
!UNIT_CELL_C-AXIS= 37.185 -1.235 -2.431
!REFLECTING_RANGE_E.S.D.= 0.059
!BEAM_DIVERGENCE_E.S.D.= 0.031
!X-RAY_WAVELENGTH= 0.978943
!INCIDENT_BEAM_DIRECTION= -0.003116 -0.002702 0.999991
!FRACTION_OF_POLARIZATION= 0.980
!POLARIZATION_PLANE_NORMAL= 0.000000 1.000000 0.000000
!AIR= 0.000316
!SILICON= 3.700630
!SENSOR_THICKNESS= 0.450000
!DETECTOR=EIGER
!OVERLOAD= 125018
!NX= 4150 NY= 4371 QX= 0.075000 QY= 0.075000
!ORGX= 2032.29 ORGY= 2442.09
!DETECTOR_DISTANCE= 175.309
!DIRECTION_OF_DETECTOR_X-AXIS= 1.00000 0.00000 0.00000
!DIRECTION_OF_DETECTOR_Y-AXIS= 0.00000 1.00000 0.00000
!VARIANCE_MODEL= 1.621E+00 7.757E-04
!NUMBER_OF_ITEMS_IN_EACH_DATA_RECORD=12
!ITEM_H=1
!ITEM_K=2
!ITEM_L=3
!ITEM_IOBS=4
!ITEM_SIGMA(IOBS)=5
!ITEM_XD=6
!ITEM_YD=7
!ITEM_ZD=8
!ITEM_RLP=9
!ITEM_PEAK=10
!ITEM_CORR=11
!ITEM_PSI=12
!END_OF_HEADER
0 0 1 -5.993E-03 3.902E-01 2086.1 2430.7 3080.7 0.00203 100 -11 -85.95
0 0 2 5.995E-01 1.671E-01 2147.4 2426.4 3180.0 0.00388 100 18 -75.96
0 0 2 7.318E-01 1.768E-01 2147.6 2444.7 955.6 0.00379 100 21 61.41
0 0 3 1.334E+00 2.725E-01 2209.0 2423.4 3287.9 0.00532 100 20 -65.22
0 0 3 6.267E-01 2.840E-01 2209.1 2448.1 848.0 0.00521 100 11 50.67
...
1 63 4 2.772E+01 1.334E+01 2556.5 9.1 1034.2 0.73636 85 14 -60.96
-1 63 4 2.473E+01 1.385E+01 2551.1 8.1 1015.7 0.73627 77 16 -60.42
1 63 5 3.999E+01 1.502E+01 2648.6 9.4 1036.3 0.73959 87 20 -59.57
-1 63 5 3.925E+01 1.509E+01 2643.2 8.1 1017.9 0.73959 78 18 -59.05
1 63 6 7.619E+00 1.156E+01 2742.3 8.3 1039.1 0.74362 80 10 -58.13
2 63 5 2.280E+01 1.282E+01 2650.6 8.2 1045.7 0.73984 78 13 -59.85
!END_OF_DATA
著者が行ったXDSの処理結果をgithubにアップロードした.
Step.2-5-2 分解能決めとフォーマット変換:CCP4-AIMLESS
XDSの出力データであるXDS_ASCII.HKLをScalingし, 次工程以降で用いられるMTZフォーマットへ変換する. また, この際に構造因子$F(hkl)$の分解能も決定する. なお, AIMLESS内のLogには論文執筆時に非常に扱いやすい形で結果が記述されるため, 筆者はAIMLESSを用いることが多い. しかし, XDSのXDSCONVを用いても同様の変換は可能である.
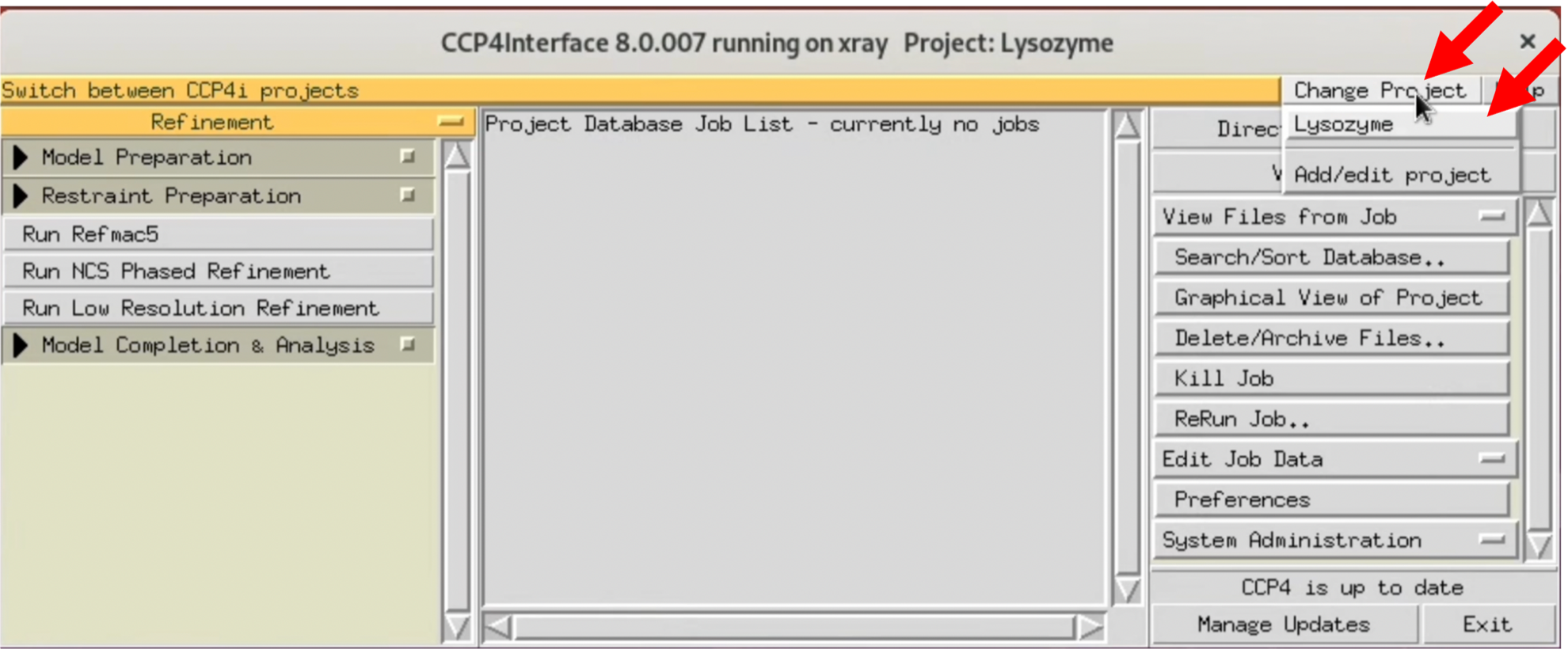
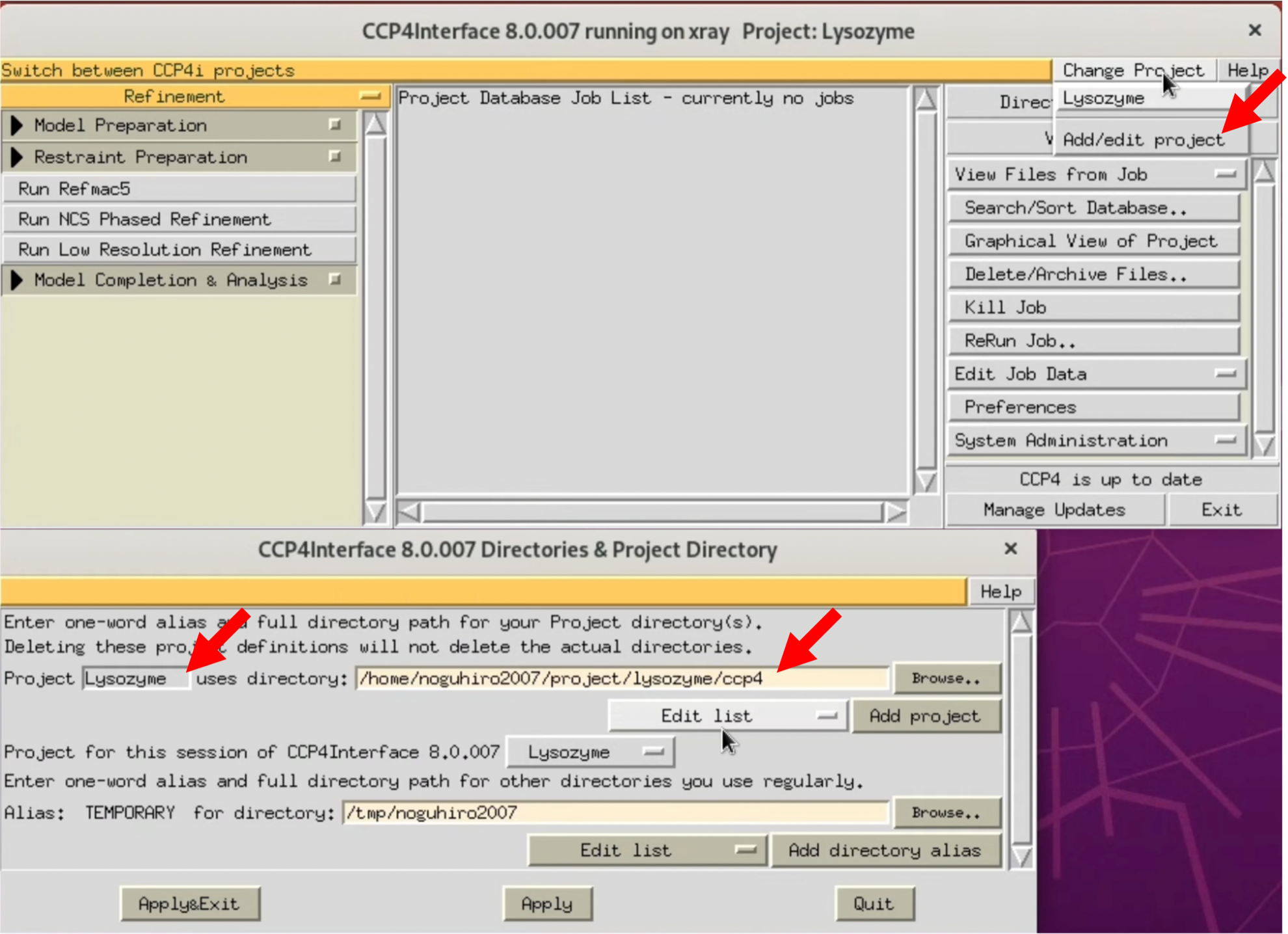
- CCP4iを起動し, Change Projectから本チュートリアルで使用するプロジェクトを選択する.
$ ccp4i
まだProjectを設定しない場合は, Add/Edit projectから新たなプロジェクトを作成する. 基本的にProjectフォルダーにCCP4から出力されるすべてのファイルが保存される. 本チュートリアルでは, Project名をLysozymeに, uses directoryにはCCP4専用のフォルダを作り, それを指定した.
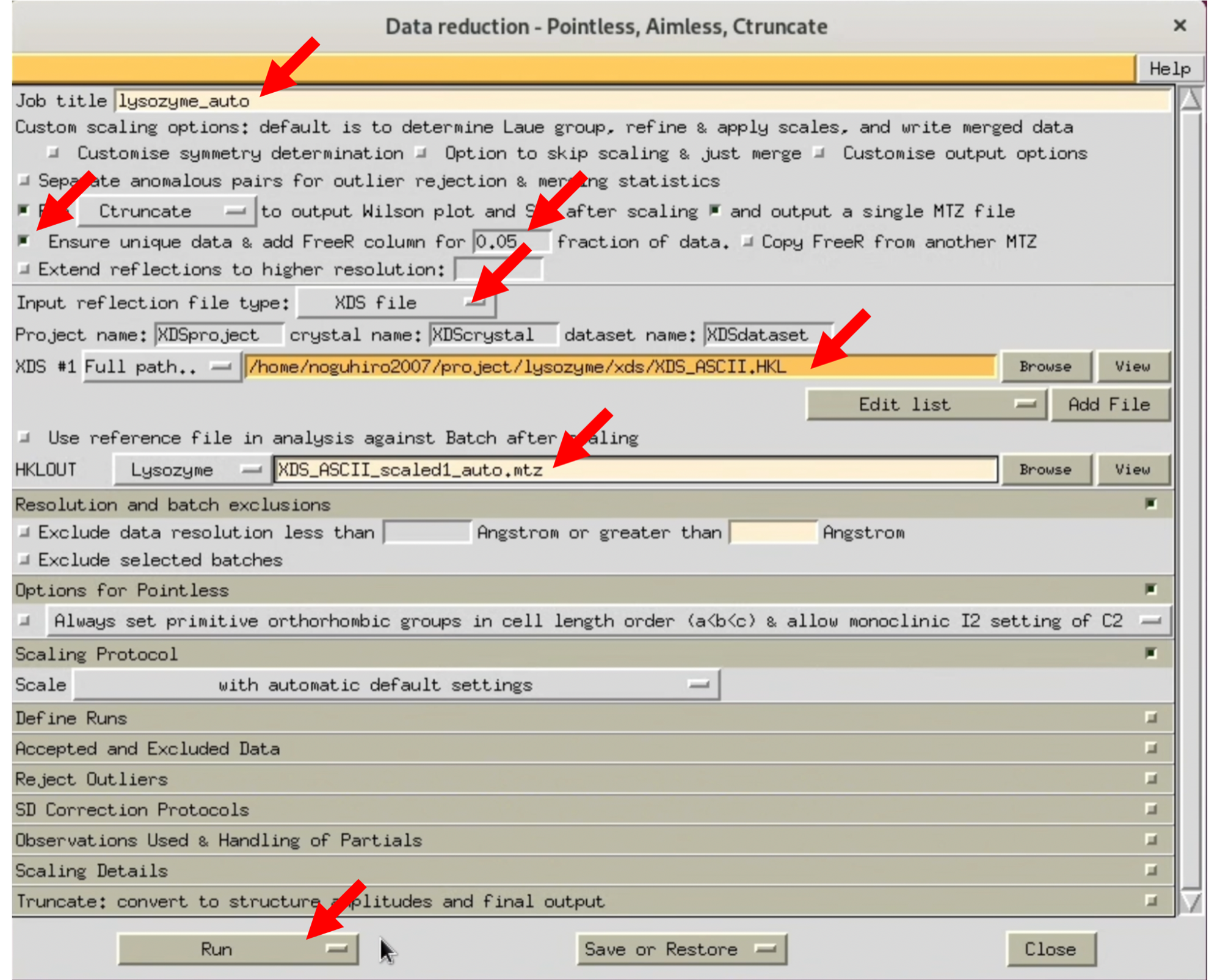
2. CCP4iの左メニューからData Reduction and Analysisを選び, 開いた小メニューからSymmetry, Scale, Merge (Aimless)を選択する.
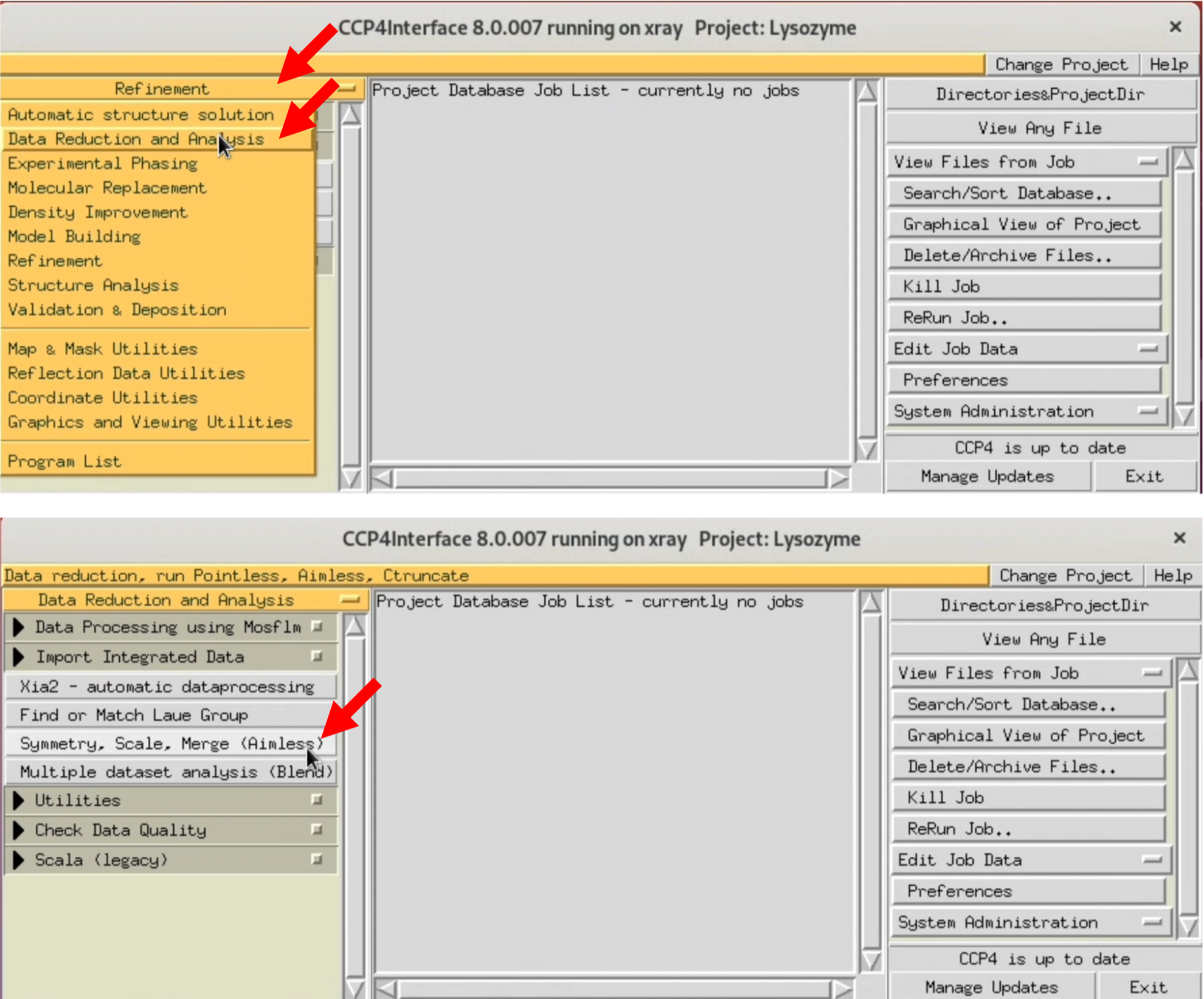
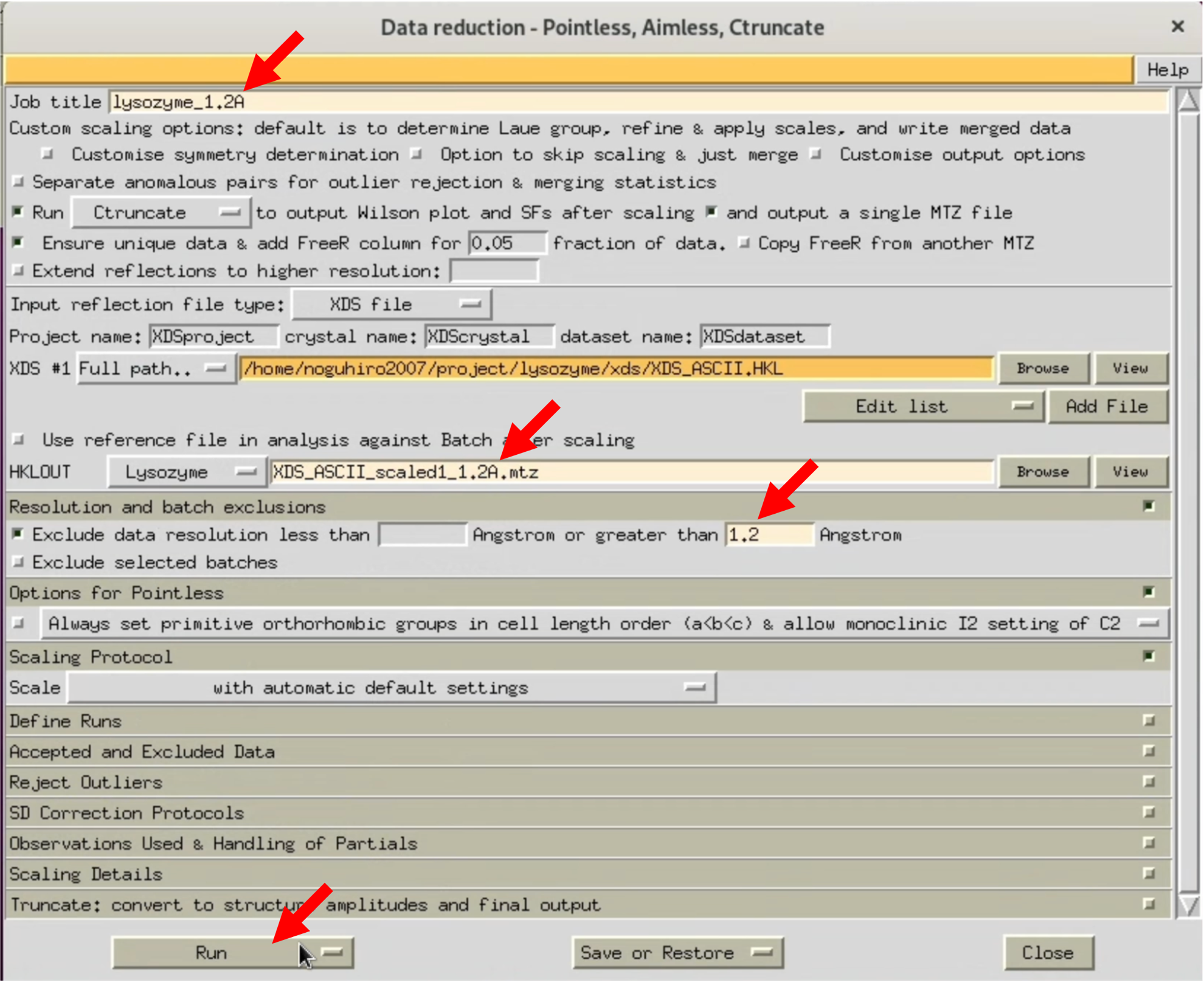
3. AIMESSのWindowが開いたら, 画像の赤矢印を参考に, 各種設定を行う. まずは分解能の指定を行わず, 最高分解能で処理をして, 様子を見る.
著者が行ったXDSの処理結果をgithubにアップロードした. 必要に応じて使用していただきたい.
処理中はStatusがRunningとなり, 処理が終了するとFINISHEDに変化する.
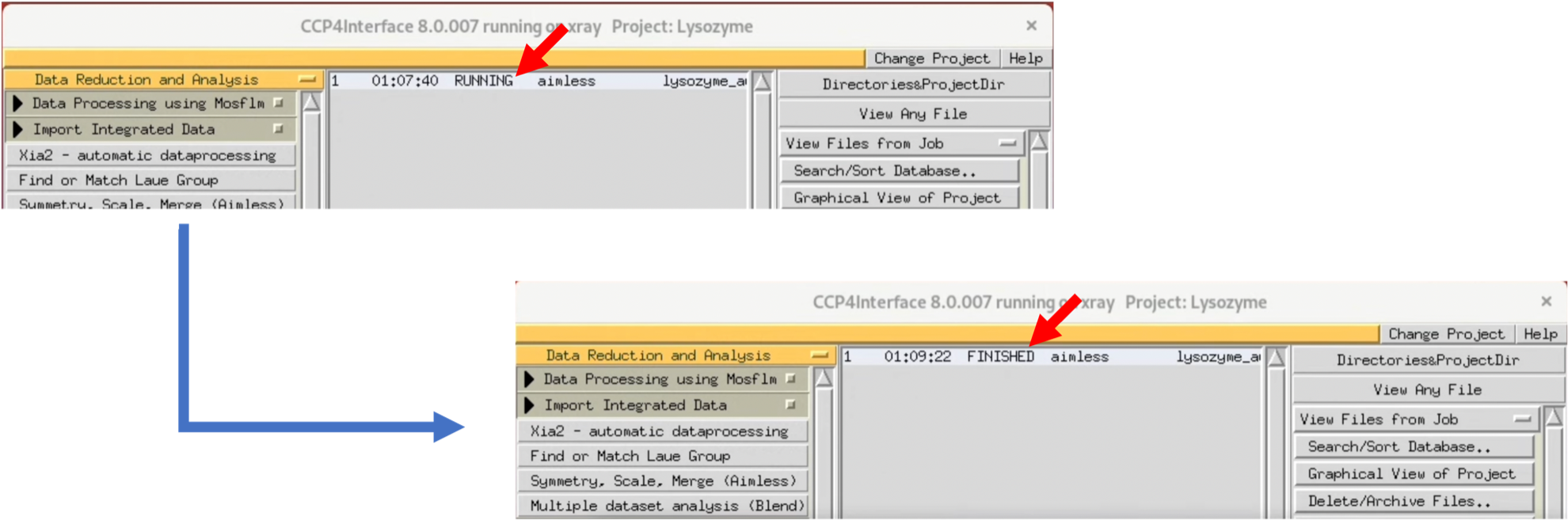
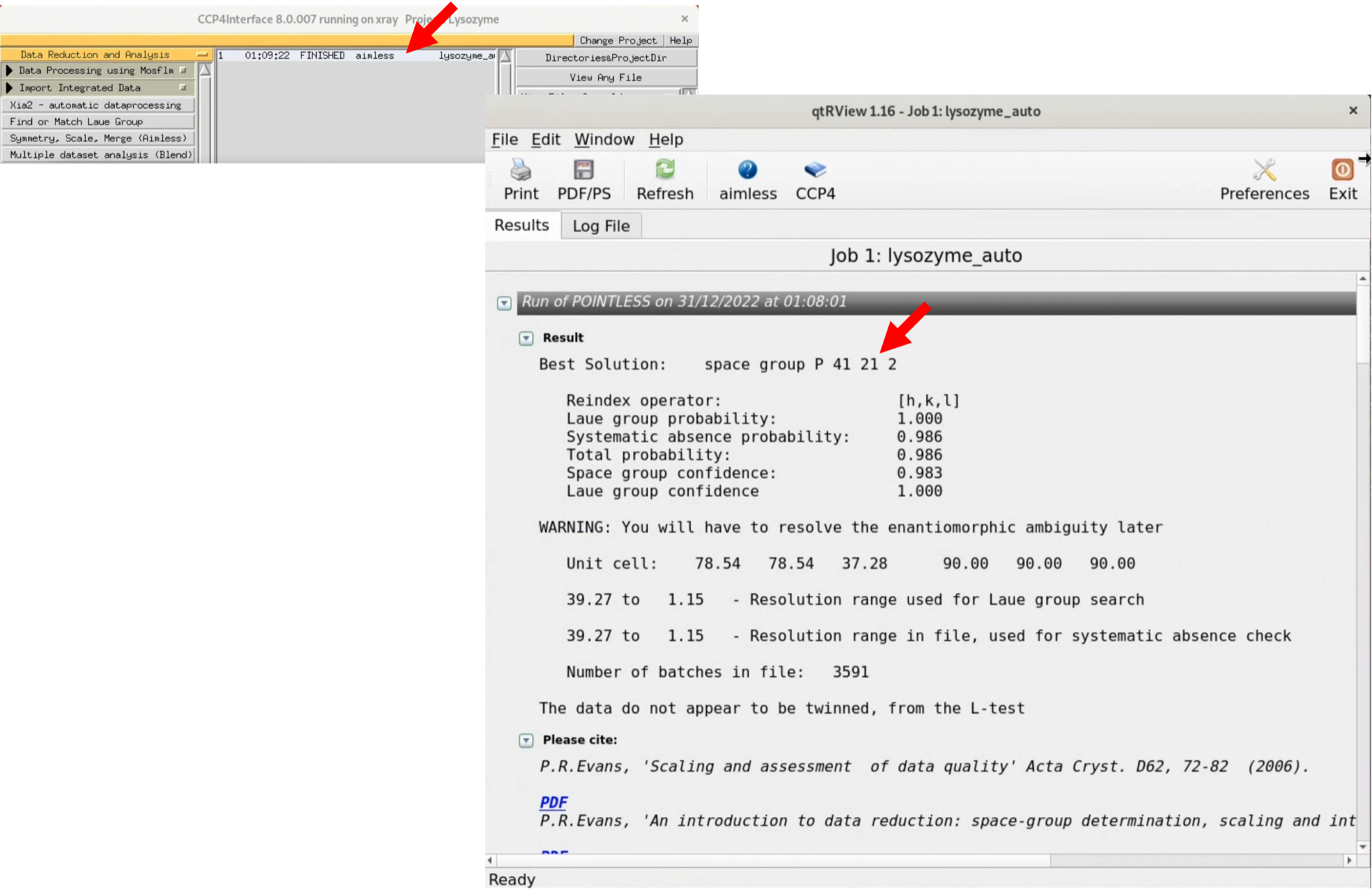
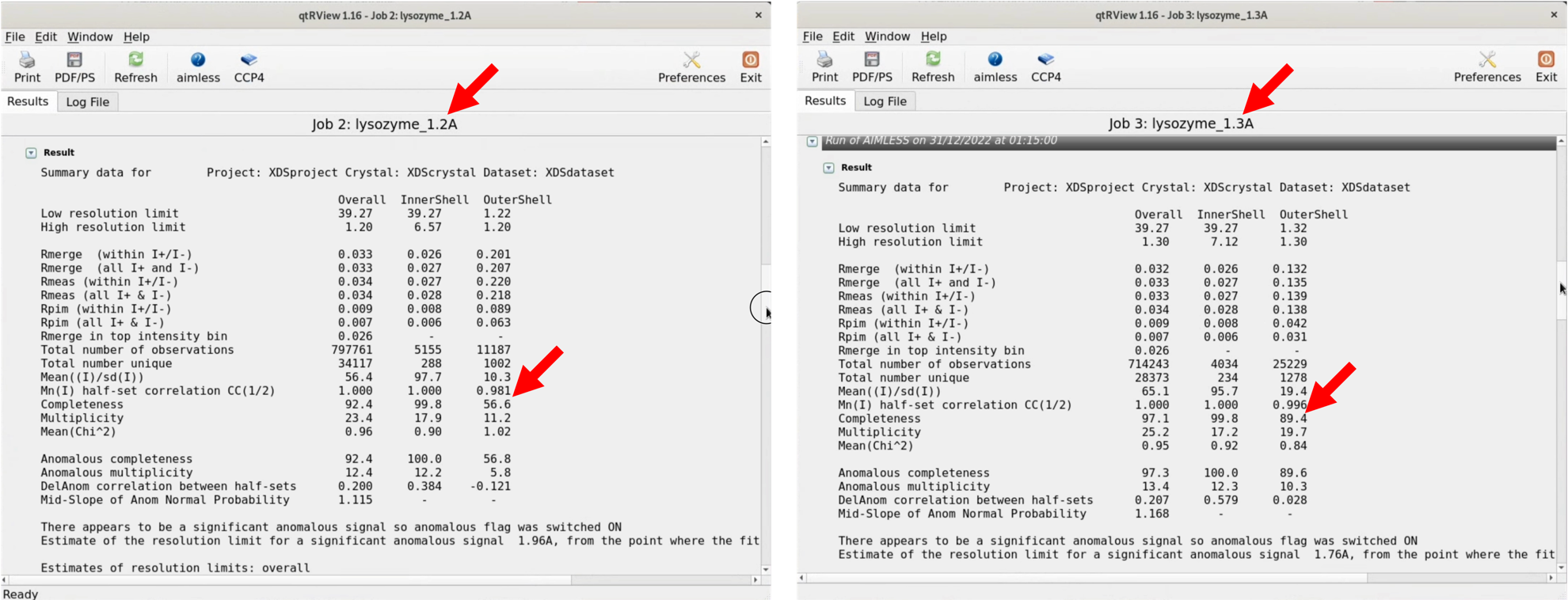
4. StatusがFINISHEDに変化したら, StatusのitemをダブルクリックすることによってグラフィカルなLogを見ることができる(大本はテキストファイルである). AIMLESSは自動的に空間群(Space group)を推定してくれる.
今回は, Best Solutionとしてspace groupがP41212と予測されているが, 実はこの空間群(Space group)は誤りである. しかし, 本チュートリアルではどこでこの間違いに気づくか, ということも重要な学習であると考え, 知らない体でこのまま進める.
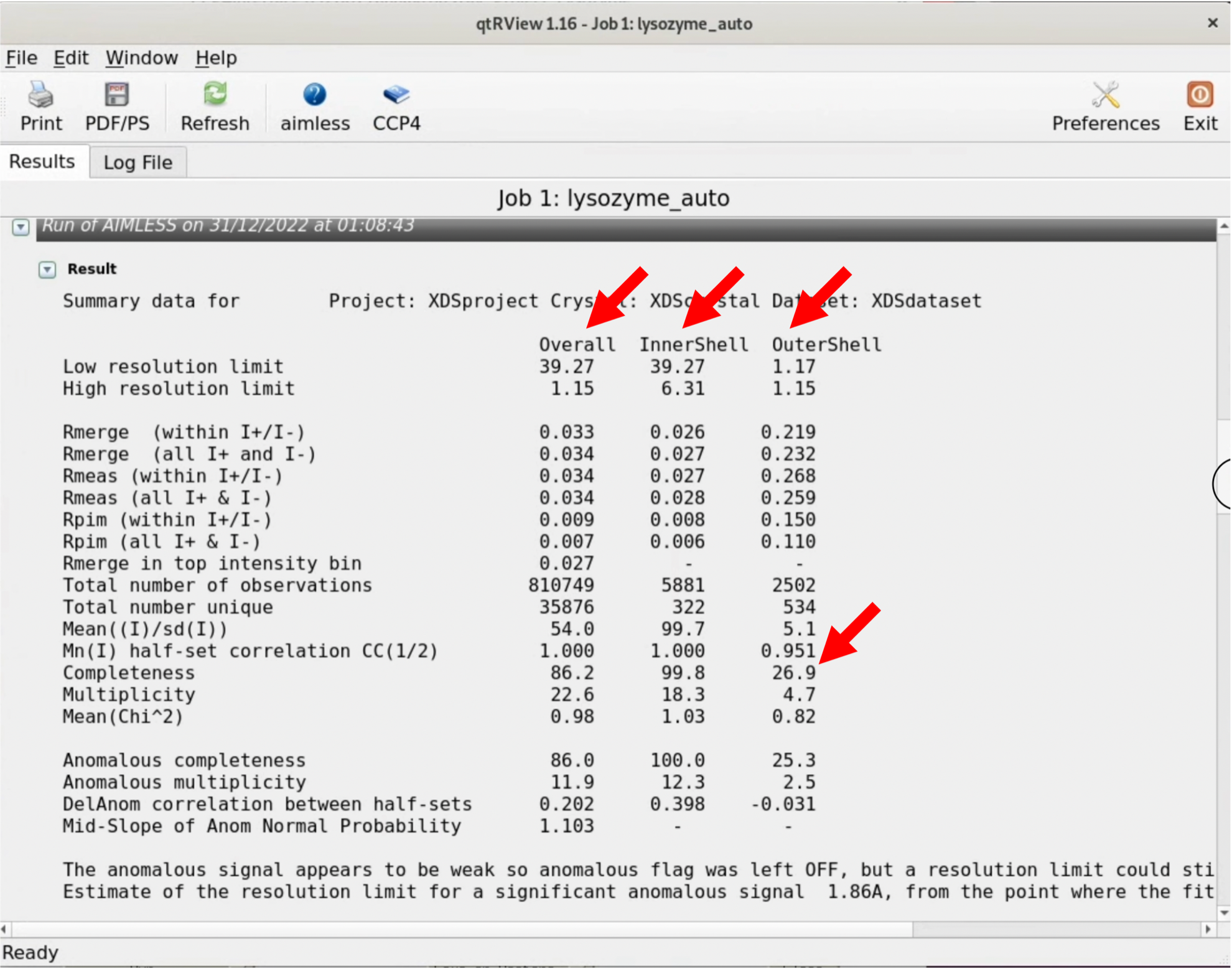
5. グラフィカルなLogをスクロールすると, "Run of AIMLESS on ~~"の項目で, 非常に重要なテーブルを目にする(下図参照). 本テーブルでは, 回折画像データセットから得られた最終的な構造因子$F(hkl)$の品質をチェックすることができる. 特にOuterShellのカラムを用いて最終的なデータセットの分解能を検討することになる. 以下に, 各項目の簡単な説明と, 分解能決定のための指標について記す.
分解能を決定する普遍的な指標は, 現在でも議論があるところであり, 研究者によって流儀が若干異なるのが現状である. 本表の指標はこのようなバックグランドを前提とした上で, あくまで一例としてご参考いただきたい (かなり安全よりの指標と考えている).
<参考資料>
初回処理の最大分解能は1.15Åであるが, OuterShellのCompletenessが26.9%と低すぎるため, 分解能を低く再設定して再びScalingを行う.
6. AIMLESS処理設定Windowの, "Resolution and batch exclusions"の, "Exclude ~ greater than NNN Angstrom"のNNN部分に処理したい最大分解能を入力する. 今回は, 1.20, 1.25, 1.30Åを指定する. 同時に, アウトプットされるファイル名(HKLOUT)やJob titleもそれに合わせて変更を行う. それぞれの分解能設定で, RUNを行う.
7. 結果, 1.3ÅのRUNでOuterShellのCompletenessが89.4%となり, すべての条件を満たす (CC1/2が0.5以上のみを有効な分解能とする判断基準も有り, その場合は1.15Åが分解能となる). 1.3ÅのRUNで出力されたMTZファイル, 筆者の場合はXDS_ASCII_scaled1_1.3A.mtzであるが, はStep.3で使用する.
8. Step.2は以上である. 引き続きStep.3(writting now)で, 初期位相の導出を行う.
著者が行ったCCP4 AIMLESSの処理結果をgithubにアップロードした.
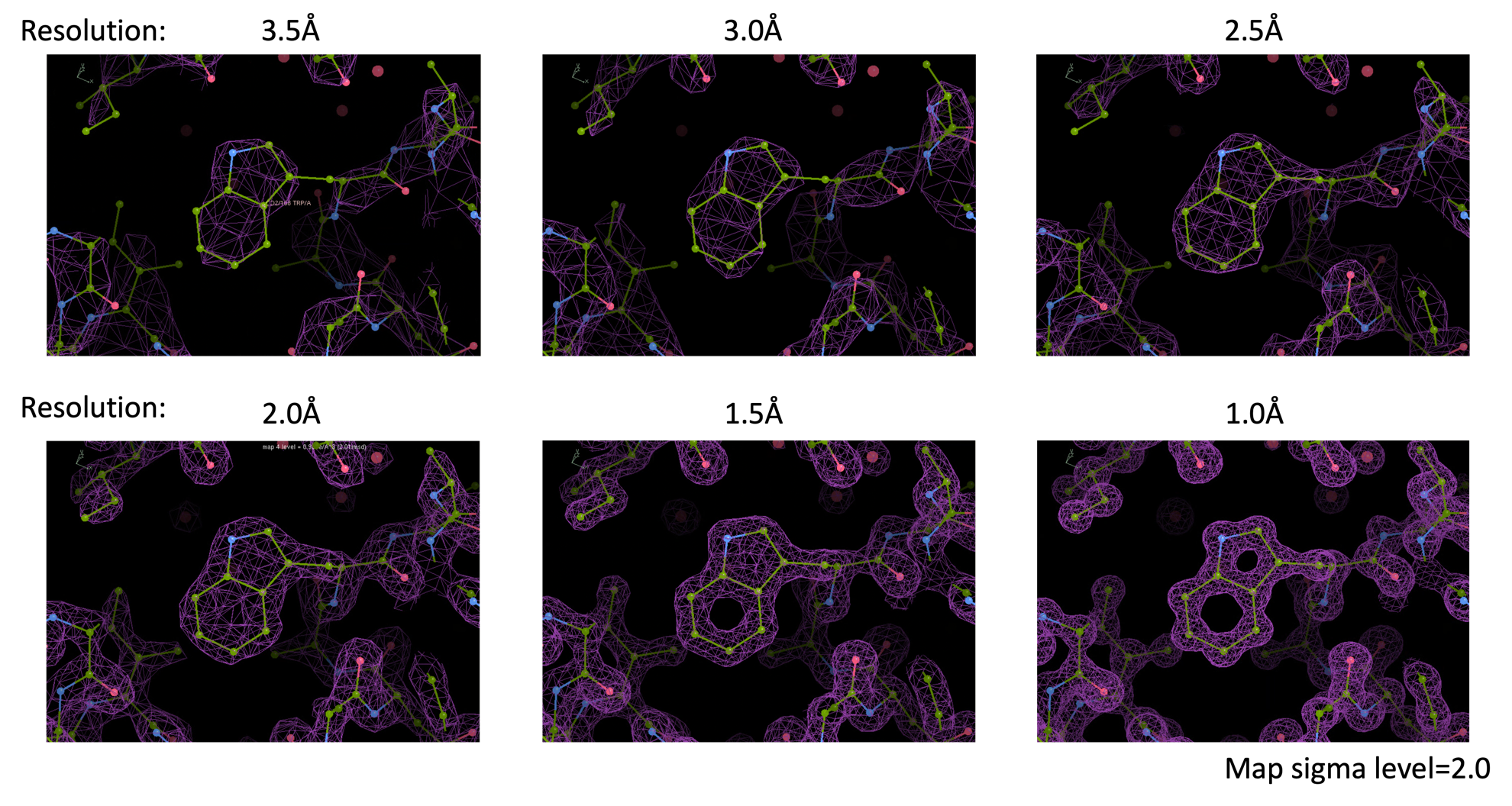
Step.2コラム:分解能について
上記で「分解能」という単語が出てきたが, どれくらいの分解能でどれくらいの電子雲になるか感覚的に理解することは今後の解析において非常に重要である. そこで, 以下に計算的に導出した各分解能別の電子雲マップを示す. 分解能3.0Å程度でほぼすべてのペプチドがトレースでき, 側鎖が同定できる物も多い. 分解能2.5Å以上で簡便に構造解析ができる. 分解能1.5Å以上が出れば各原子がほぼ分離される. 実際のところ, 3.0Å ~ 1.5Å程度の分解能で構造解析されることが多い.
あとがき
本タンパク質X線結晶構造解析チュートリアル第2回目では, 回折画像と構造因子$F(hkl)$の計算を行った. 得られた構造因子$F(hkl)$ファイルを用いて、次回は初期位相の決定を行う。
他のStepへのリンク
- Step.0 構造決定の流れ
- Step.1 解析ソフトのインストール
- Step.2 回折画像の確認と構造因子の計算 (本記事)
- Step.3 初期位相を求める
- Step.4 構造精密化とバリデーション
-
Step.4で解説するが, 電子雲マップ$\rho (xyz)$とタンパク質モデルの同一性を推し量る指標($R$, $R_{free}$など)があり, それがあまりにも悪いと信頼できる構造とは認められない. ↩
-
筆者は8時間のビームタイムで130結晶のデータセット測定したことがある.仮に1日このペースで測定した場合, 140.4万枚のファイルが生成されることになり, これが連日続くとなるとシンクロトロンやビームライン管理者にとって、ファイル管理は非常に困難となる. ↩
-
俗に言う"良い質のタンパク質結晶"とは,「結晶中にタンパク質が密に詰まっており, X線を高分解能まで回折するもの」である. 結晶を扱う際に「小さくて重い」結晶であればその確率は高いが, 結晶溶液中の他の低分子化合物である可能性もある. 1.0~3.5Åの分解能で回折点が数多く確認されれば, タンパク質の結晶であると判断できる. ↩
-
おそらく最初の数枚はなにかの要因でX線そのものが照射されていない. これが原因でIndexingがうまく行かないが, 本チュートリアルではこのまま進む. ↩
-
あまりにも簡単すぎる説明なので, X線結晶構造解析学を専攻とする方はきちんと導出式まで確認されることをおすすめしたい. ↩