はじめに
AlphaFold2の登場でタンパク質構造はこれまでにないほど全世界から注目されている. 筆者のようにタンパク質X線結晶構造解析で学位を得た人間だけではなく, 他分野の研究者がタンパク質構造を活用して新たな科学を切り開こうとしている. 生命現象・病気の解明, 革新的な医薬品・農薬・食品の開発, 素材としてのタンパク質の開発などなど, タンパク質が関わる分野にとってその構造はより一層重要性が増しており, 高精度な予測構造を出力するAlphaFold2は科学の歩みをより一層加速させるであろう.
しかし, いくらAlphaFold2が高精度な構造を出力するといってもそれはあくまで「予測構造」でしかない. 「予測構造」で仮説を立てたあとは, 多くの場合「実験による構造決定」での検証が待っている. 皮肉なことであるが, AlphaFold2の登場により, 今後多くの専門外の研究者が実験的に構造決定する必要が出てくるであろう.
本記事ではそんな未来を先取りし, 最も代表的なタンパク質構造決定法である「X線結晶構造解析法」を取り上げ, それを用いた構造解析法の基礎的なチュートリアルを示す. ぜひ手を動かしてX線結晶構造解析法の基礎をマスターしていただければ幸いである.
この記事の対象と目的
タンパク質X線結晶構造法を専門外とする研究者・大学院生・学部生を対象とし, タンパク質X線結晶構造解析について回折画像から構造決定まで一通りできるようになることを目的とする. 前提として, Linux環境でBashコマンドをある程度打てる方を想定している.
本記事では, タンパク質の構造解析のチュートリアルを全4回の連載でお届けする. 今回は第1回目である.
また, 本記事は完全にオープンである. 学校でも研究機関でも企業でも, ぜひこの記事を広めて, 教育に活用していただければ幸いである. しかし, 著作権は放棄していない.
目次
- Step.0 構造決定の流れ (本記事)
- Step.1 解析ソフトのインストール (本記事)
- Step.2 回折画像の確認と構造因子の計算
- Step.3 初期位相を求める
- Step.4 構造精密化とバリデーション
タンパク質のX先結晶構造解析に必要な計算機環境
筆者の経験から, タンパク質のX線結晶構造解析には以下の計算機環境が適していると考えている. コンピューターパワーが必要であるため, 間違ってもRaspberry Piなどでやってはいけない.
- OS
- Ubuntu, CentOSなどのLinux (強く推奨)
- MacOSX
- Windows 10/11 (ネイティブ環境もしくはWSL2上のLinux環境を使用)
- CPU: > 4 core (推奨:> 16 core)
- Main Memory: > 16 GB (推奨: > 32 GB)
- Storage: > 50 GB (ソフトウェアインストールに約 12 GB.データセット 20 - 30 GB/crystal)
- GPU:必須ではない(推奨: GPU搭載)
Githubレポジトリ
本チュートリアルに関連する代表的なファイルはGithubにアップロードしている. 適宜ご参考いただければ幸いである.
Step.0 構造決定の流れ
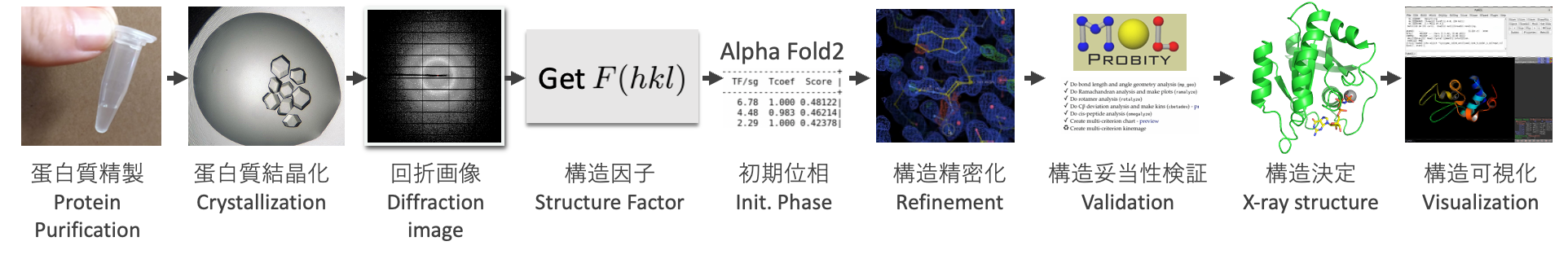
タンパク質X線結晶構造解析では, どのような流れで構造を決めているのだろうか. 1959年にMax Perutzがタンパク質のX先結晶構造解析法を確立して以来, その基本的な方法は実は全く変わっていない1. タンパク質X線結晶構造解析法を端的に言い表すならば「X線解析の実験データ(= 構造因子, $F(hkl)$)から電子雲マップ$\rho (xyz)$を求め, そこにアミノ酸を当てはめてタンパク質の構造を決定する方法」である. タンパク質のX線結晶構造解析法の基本である構造因子$F(hkl)$と電子雲$\rho (xyz)$を関連付ける基本式を以下に示す.
\rho (xyz) = \Sigma_h \Sigma_k \Sigma_l \frac{1}{V} | F(hkl) | \exp [ -2 \pi i (hx + ky + lz) + i \alpha (hkl) ]
式中の$F(hkl)$が回折画像から導出される構造因子である. Vは結晶の単位格子の体積であり, これも実験データから導出される. $hkl$はミラー指数, $xyz$は実空間上の座標である2. 一方, 問題は位相$\alpha (hkl)$である. タンパク質モデルの構築に必要な電子雲マップ$\rho (xyz)$を導くには, 位相$\alpha (hkl)$が必要であるが, 回折実験で使用される最新の2次元検出器でも位相$\alpha (hkl)$の情報を直接捉えることは出来ない. 故に, 計算や別の実験で位相$\alpha (hkl)$を導出する必要があり, これは位相問題として知られている.
位相問題はタンパク質X線結晶構造解析において頭の痛い問題の一つである. その壁を突破するためいくつかの方法が考案されてきたが, 最も一般的で簡便な方法は, 類似した蛋白質構造を用いる分子置換法(Molecular Replacement法, MR法)である. MR法ではPDBに登録されているタンパク質構造を類似構造として入力することが多いが, PDBに登録してあるタンパク質構造の増加によって, 近年ますます適用できる範囲が広がってきている. 更に, 昨年のAlphaFold2の登場により, その予測構造をMR法の入力とすることで, かなりの確率で精度良い初期位相(タンパク質モデル構造を構築するための粗い位相情報)を導くことが可能になっている3. 本チュートリアルでもAlphaFold2の予測構造を用いる.
初期位相を求めた後は, 3次元空間上でパズルのように電子雲マップ$\rho (xyz)$にタンパク質モデル構造を当てはめる. しかし, ただ当てはめれば良いものではなく, モデル構造の構築とともに位相$\alpha (hkl)$が改善(=電子雲マップ$\rho (xyz)$が改善)され, それをもとにタンパク質構造を徐々に修正してゆく. このように, アミノ酸の当てはめと位相計算を繰り返し, 位相$\alpha (hkl)$のベストな値を求めつつ, 構造をブラッシュアップしてゆく工程を「構造精密化」と呼称する.
最後に, 導出された構造が蛋白質構造として適しているかチェック(構造妥当性検証)して, 構造決定となる.
本チュートリアルでは, 上図における回折画像から構造決定までを取り扱う. それより前, 蛋白質精製〜回折画像までは, 生化学・蛋白質結晶作成・放射光などなど, 別の専門性が求められる. そのため, 本チュートリアルでは取り扱わない4.
構造解析に必要なデータは, ①回折画像のデータセットと②タンパク質の配列情報の2つである. 本チュートリアルでは, 構造生物界のHello World構造(?)とも言うべき, Lysozymeの構造5を明らかにする. 各データの入手は以下の通りである.
| データ | Download URL | 備考 |
|---|---|---|
| 回折画像 | Integrated Resource for Reproducibility in Macromolecular Crystallography, NIH, nsls2_fmx_20161122_lys_266 | 米国にある放射光施設 National Synchrotron Light Sourceのビームライン17-ID-2で, ビームラインの調整目的で取得されたデータ. 約13GB. |
| アミノ酸配列 | UniProt, P00698 LYSC_CHICK | "Sequence"セクションの"Download"ボタンをクリックし, 出てきた配列の19残基目から最後までの配列を, 適当なテキストファイル名(例:lysozyme.fasta)で保存する. 以下に19残基目から最後までのLysozymeの配列を載せるので適宜使用していただきたい. |
KVFGRCELAAAMKRHGLDNYRGYSLGNWVCAAKFESNFNTQATNRNTDGSTDYGILQINSRWWCNDGRTPGSRNLCNIPCSALLSSDITASVNCAKKIVSDGNGMNAWVAWRNRCKGTDVQAWIRGCRL
Step.1 解析環境の構築
Step.1-1 X線結晶構造解析に使用するソフトウェア
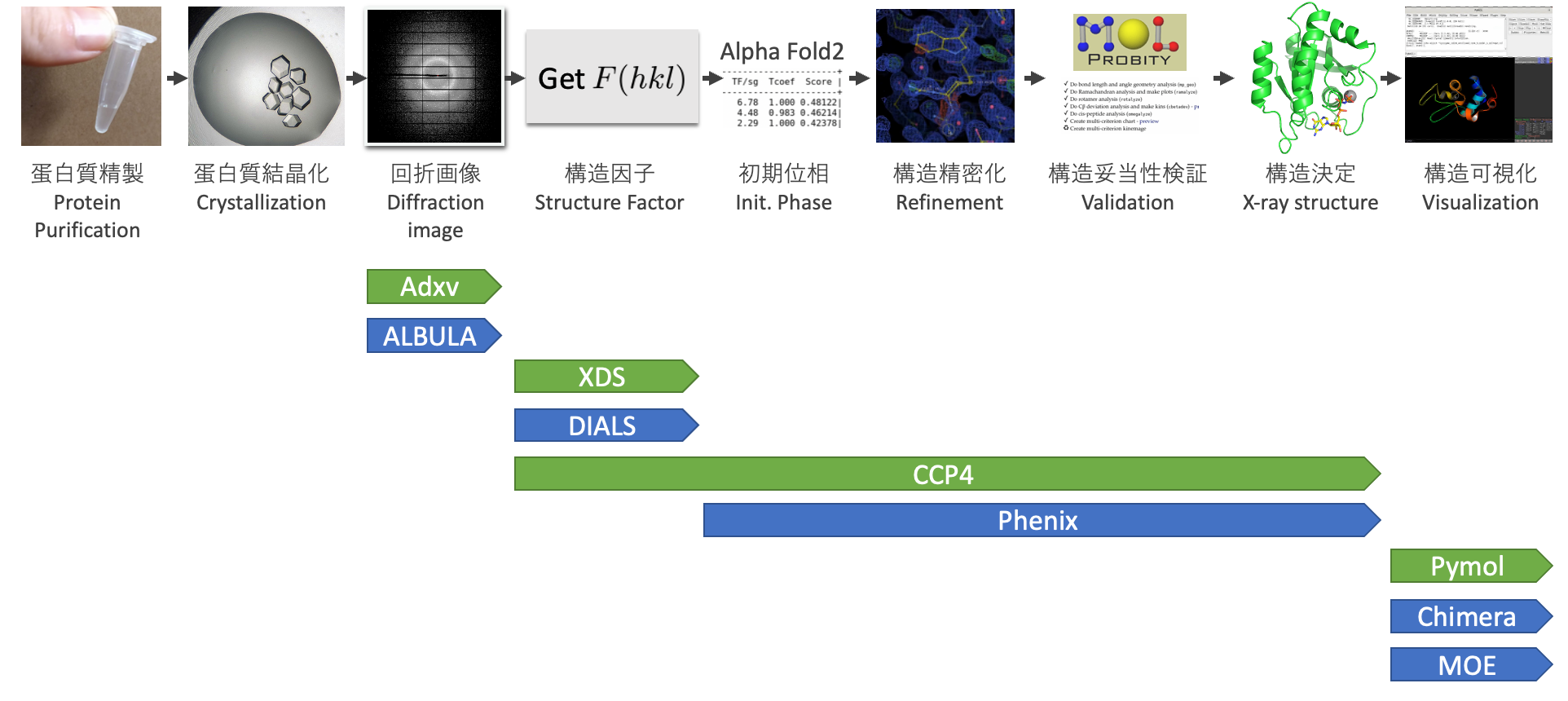
歴史のある研究分野ほど数多くの定番ソフトウェアが存在するのが常である. その例に漏れず, タンパク質のX先結晶構造解析法も一つですべてをカバーできるソフトウェアは存在しない. 数多く存在するソフトウェアの中から, 本チュートリアルではゴールデンスタンダードと思われる4つの必須のソフトウェアを用いる. 基本的に非商用利用は無償である6.
以下に, 本チュートリアルで使用するソフトウェア一覧を示す. 必須ソフトウェア(緑ハイライト)のみならず, すべてのソフトウェアをインストールしておけば, 実践で構造解析する上でも困ることはほぼないであろう7. タンパク質のX線結晶構造解析の各工程における各ソフトのカバー範囲, そして各ソフトウェアの詳細を記す.
| 必須 | 使用ステップ | ソフト名称 | 機能 | ライセンス | URL |
|---|---|---|---|---|---|
| ✓ | 回折画像 | Adxv | 回折画像のViewer (各検出器メーカー対応) | 無償 | https://www.scripps.edu/tainer/arvai/adxv.html |
| 回折画像 | ALBULA | 回折画像のViewer (業界スタンダード検出器メーカーのDectris社純正) | 確認必要 | https://www.dectris.com/support/downloads/software/ | |
| ✓ | 構造因子 | XDS | 回折画像から構造因子$F(hkl)$を計算 | 非営利:無償. 営利:ライセンス必要. | https://xds.mr.mpg.de/ (Wiki: https://strucbio.biologie.uni-konstanz.de/xdswiki/index.php/Installation) |
| 構造因子 | DIALS | 回折画像から構造因子$F(hkl)$を計算 | BSD-3 | https://dials.github.io/ | |
| ✓ | 構造因子, 初期位相, 精密化, バリデーション | CCP4 | 事実上のタンパク質構造デフォルトスタンダードソフトウェアスイート. | アカデミック:フリー, 営利企業:有償 | https://www.ccp4.ac.uk/ |
| 初期位相, 精密化, バリデーション | Phenix | CCP4と対をなす事実上のデフォルトスタンダードソフトウェアスイート. ダウンロードには所属情報が必要4. | アカデミック:フリー, 営利企業:有償 | https://phenix-online.org/ | |
| ✓ | 構造可視化 | Pymol | 定番の蛋白質構造可視化プログラム. 公式版は学生・教師以外はサブスク式の有料. OSS版は企業利用もFreeなので, とりあえずはOSS版インストを推奨. | 公式版:基本有償, OSS版:フリー | 公式版:https://pymol.org/2/ , OSS版:https://github.com/schrodinger/pymol-open-source |
| 構造可視化 | Chimera | 定番の蛋白質構造可視化プログラム. Pymolとは違う味が出せる. | 非商用:フリー, 商用:有償 | https://www.cgl.ucsf.edu/chimera/ | |
| 構造可視化 | MOE | 蛋白を扱うバイオインフォ屋さんがよく使う可視化・編集・分析ソフトウェア. 可視化だけにこのソフトを使うのはオーバースペック. | 有償 | https://www.chemcomp.com/Products.htm |
Step.1-2 計算環境
タンパク質X線結晶構造解析法は伝統的にLinuxベースの計算機環境で発展してきた. 現在はWindowsで動くソフトも多く出てきているが, 残念ながらそれでもGUI環境で不具合が起きる場合も多く8, 筆者的にはピュアなLinux環境(Ubuntuなど)もしくはMacで動かす事をおすすめしたい. もちろん, WindowsのWSL2上のLinux環境でもある程度は動くが, 筆者環境ではGUIソフトのCootで不具合が発生した.
今回のチュートリアルでは, 環境構築トラブルの低減と筆者の計算機環境の関係で, クラウド環境にVM(Virtural Machine)のLinux環境を立ててその上で各種処理を行う. もちろん, お使いのPCにLinuxが入っていれば, 本チュートリアルと同様にやっていただくことで問題なく構造解析できるはずである. むしろ, 構造精密化のステップで3D構造を扱うので, できればGPUが装着されたローカルPCにLinuxをインストールされることを強くおすすめしたい9.
Step.1-3 Google Cloud Platform(GCP)上に計算機環境を構築する (Skip可能)
筆者の個人的な好み10で, クラウドはGCPを使うことが多い. よって, 今回はGCPでVMを構築するが, AWSなど他のVMでもインスタンスを作ってしまえば全く同様に環境構築が可能である. なお, すでにローカルPC等でLinux環境をお持ちの方は本ステップをスキップしていただきたい.
- GCPに登録し, Compute Engineで以下のスペックのVMを立てる. 回折画像から構造因子を得る計算でCPUパワーを使うため, 24 coreと多めにしている.
- N1インスタンス(vCPU:24core, Memory:32GB)
- ストレージ: 200GB バランス
- インスタンスタイプ: スポット
- OS: Ubuntu20.04LTE
- (利用料:1ヶ月約100ドル程度)
はじめてGCPに触れる方は無料で90日間$300は使えるのでそれを利用するのが良いだろう. 基本的に1日で終了するため, 500円もかからないと思われる(利用料は実施者に依存).
2. インスタンスが立ち上がったら, 以下コマンドでUbuntuのDesktop環境をインストールする.
# aptを更新し最新にアップデート
$ sudo apt update
$ sudo apt upgrade
# Desktop環境をインストール
$ sudo apt install ubuntu-desktop
...
3. ChromeのRemote Desktopを用いて, GUI環境にアクセスする方法を確立する (参考: GCPの公式ページ).
4. このようにChromeからUbuntuのデスクトップが見えたらインスタンスのセットアップは完了.
Step.1-4 Adxvのインストール
以下, 各種ソフトウェアを本チュートリアルを実行する計算機環境にインストールする. 上記Step.1-3でGCP Compute Engine環境を構築した場合は, その環境に以下のソフトウェア群をインストールする.
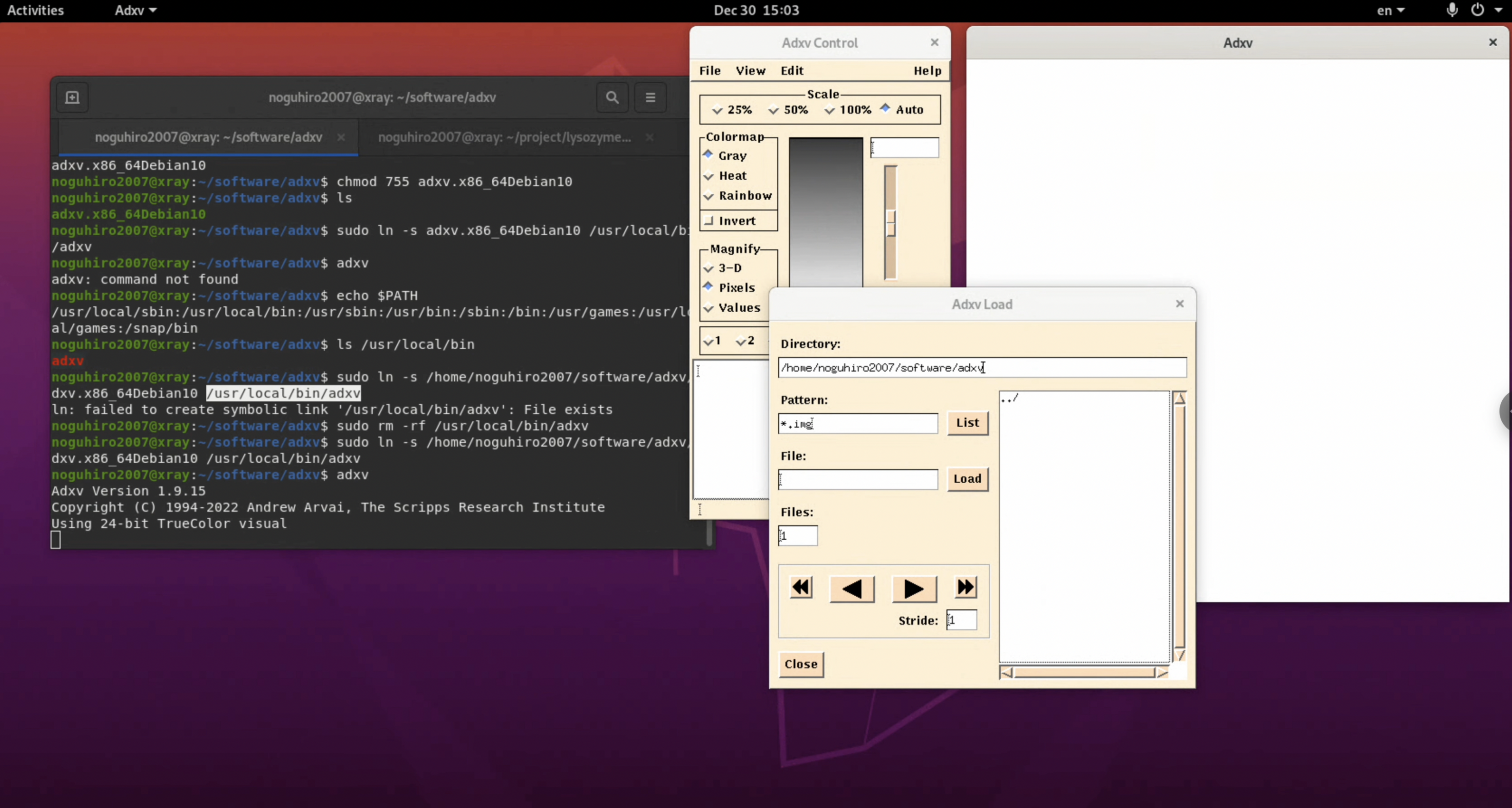
1.Adxvのページから, 自らの環境に適したAdxvをダウンロードする. 本チュートリアル環境では, GCP上に構築したUbuntu 20.04 LTE インスタンスを使用するので, 「adxv.x86_64Debian10」を選択してダウンロードする.
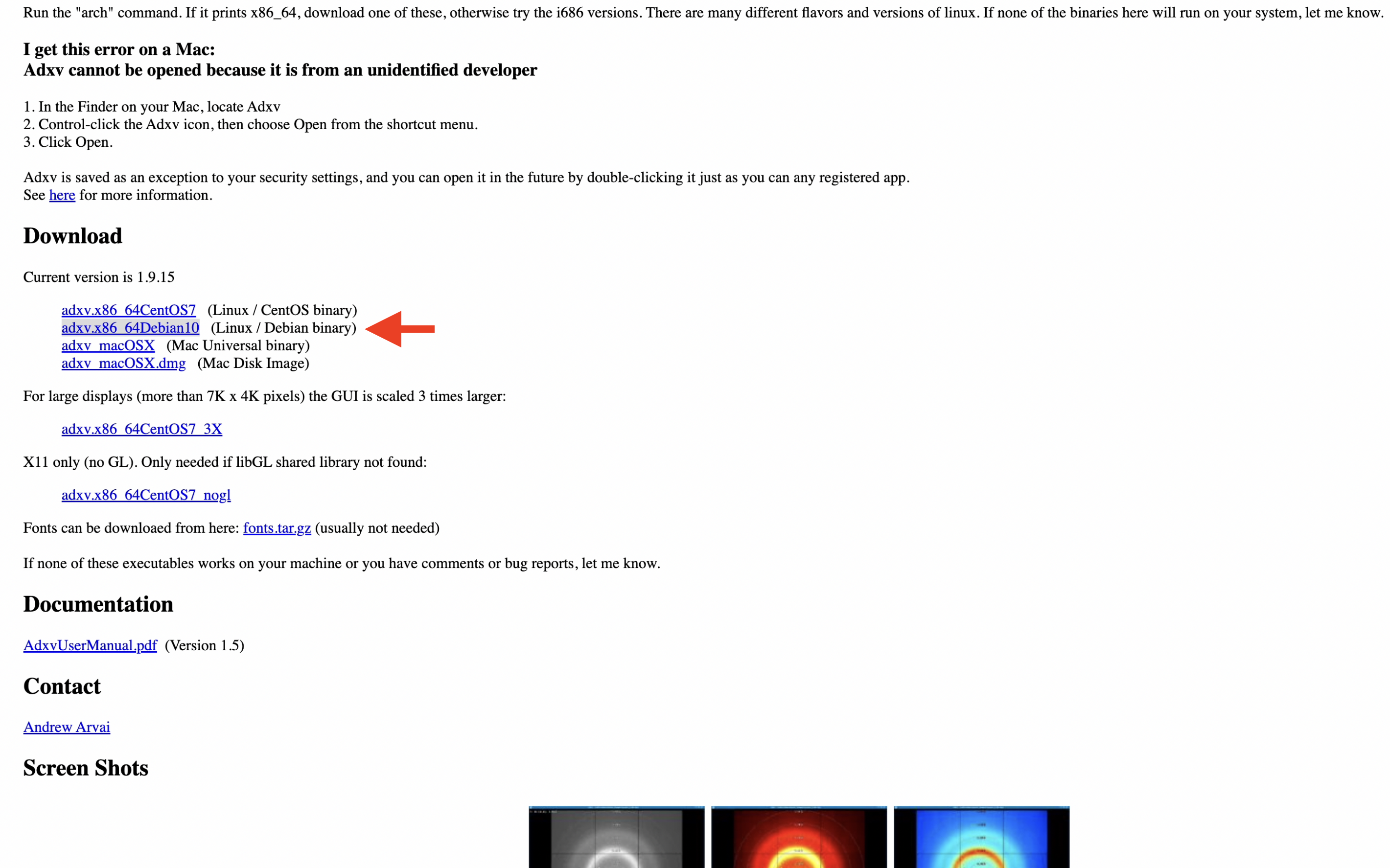
2. 「adxv」とタイプしたら起動するように, すでにpathが通っているフォルダ(例えば /usr/local/bin)にシンボリックリンクを追加しておく.
# ダウンロードフォルダからお好みのフォルダにadxvのバイナリーファイルを移動する.
$ mv <Download_folder_path>/adxv.x86_64Debian10 <your_favorite_folder_path>/adxv.x86_64Debian10
# adxvのバイナリーファイルに実行権限を付与する.
$ chmod u+x <your_favorite_folder_path>/adxv.x86_64Debian10
# pathが通っているフォルダにadxvのバイナリーファイルのシンボリックリンクを追加する.
$ sudo ln -s <your_favorite_folder_path>/adxv.x86_64Debian10 /usr/local/bin/adxv
3. 「adxv」とタイプし, adxvが無事に起動するか確認する. 図のように起動したらインストールが完了である.
$ adxv
Step.1-5 XDSのインストール
XDSは非常に長い歴史を持つCUIベースのX線構造解析の回折画像処理プログラムである. 処理指示ファイル(XDS.INP)に処理内容を書いて実行するが, XDS.INPには検出器等に関する詳細な回折実験情報も含むため, 初心者がそれをScratchで作成するのはかなり大変である. 故に, それを自動作成してくれるスクリプトをインストール・利用するのが一般的であり, またその他の使い勝手を良くする補助プログラムも含めて一括でインストールすることが多い. 本チュートリアルでも, 補助スクリプト・プログラムもまとめてインストールする.
- 基本的にXDSのInstall wikiに従って, XDSやその他の追加機能などを含む関連するソフトウェア群を一括でインストールする. 本チュートリアル環境の場合Ubuntu20.04LTEなので, Linuxのインストール手順に従う.
## root権限を獲得する
$ sudo -i
## XDSをインストール (root権限で実行)
cd /usr/local/bin
wget -O- https://xds.mr.mpg.de/XDS-INTEL64_Linux_x86_64.tar.gz | tar xzvf -
ln -sf XDS-INTEL64_Linux_x86_64/* .
## その他のXDS追加ソフトを一括インストール(root権限で実行)
wget -N https://strucbio.biologie.uni-konstanz.de/pub/linux_bin/get_folder.sh
chmod +x get_folder.sh
./get_folder.sh
本チュートリアルの環境では, get_folder.shにおいて, 以下のエラーが発生した.
check Unix tools that generate_XDS.INP may need -
python not found
strings not found
指示に従い, Pythonおよびstringsをインストールする.
# Pythonのインストール
$ sudo apt install python3 python3-dev
# Stringsのインストール
$ sudo apt install binutils
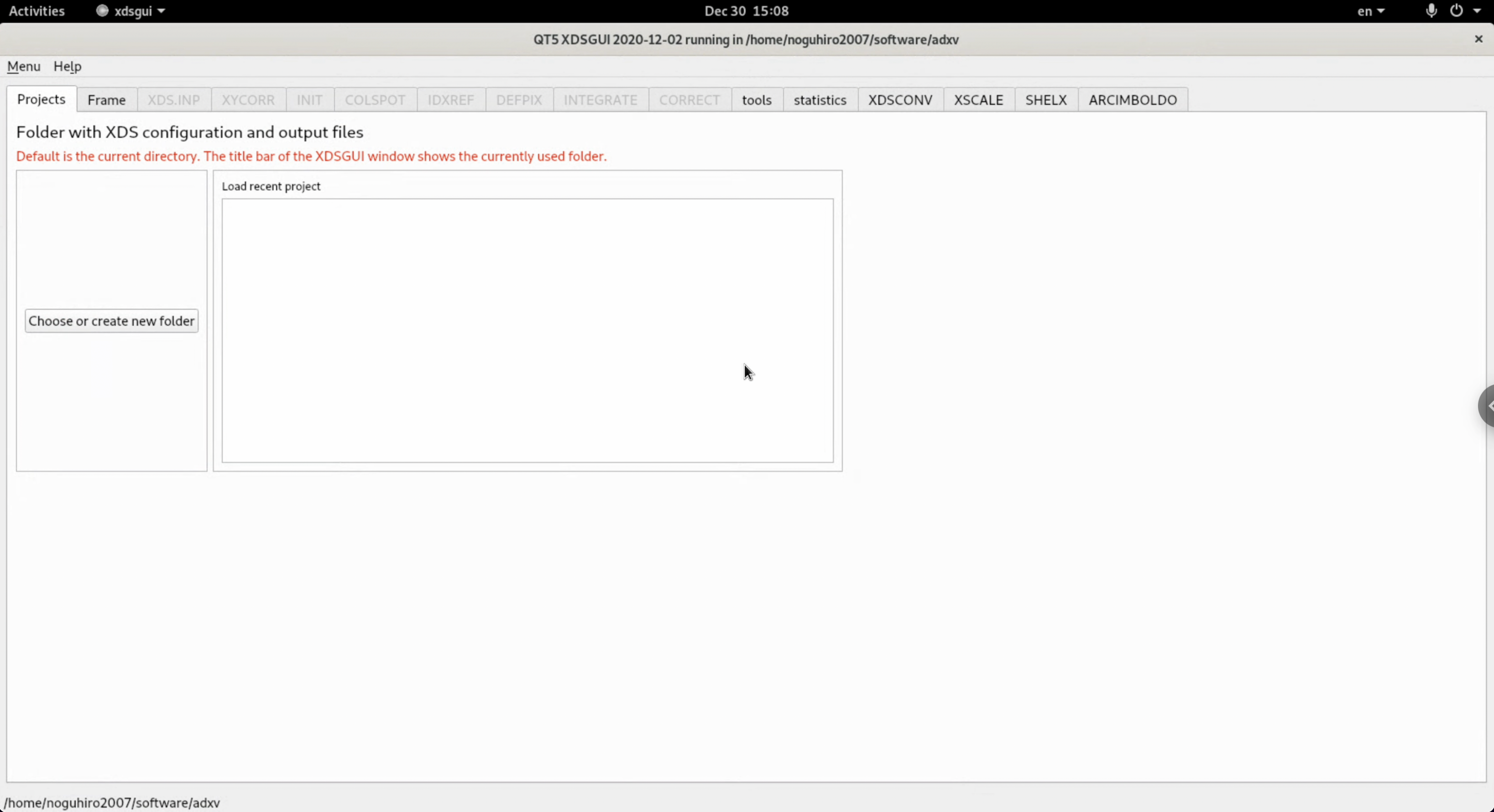
2.xdsguiと入力し, 起動したらインストール成功である.
$ xdsgui
このとき, 本チュートリアル環境では「libQt5OpenGL.so.5がない」と怒られたので, XDSのInstall wikiに従って, 該当ライブラリーをインストールする.

$ sudo apt install libqt5gui5 libqt5printsupport5 libqt5opengl5
Step.1-6 CCP4のインストール
-
CCP4のダウンロードサイトから, 計算機環境に適したものをダウンロードする. 本チュートリアルでは, Ubuntu20.04の環境なので, "GNU/Linux"の"Package Manager"を選択し, インストーラーをダウンロードする.
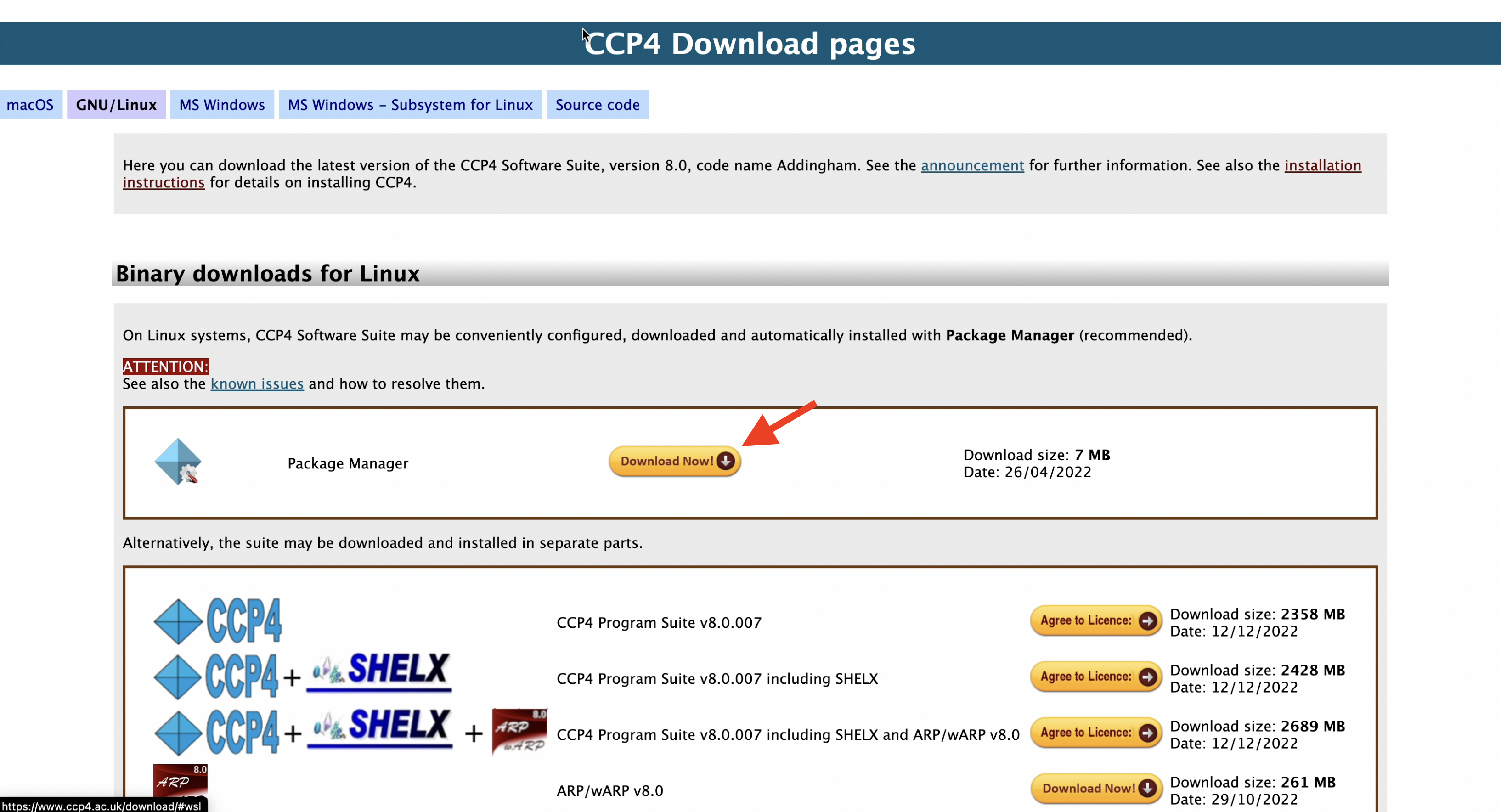
2. Package Managerを起動し, インストールを開始します.
# ダウンロードしたファイルを解凍します.
$ tar -zxvf <path_for_download_folder>/linux-x86_64_ccp4-<version>-setup.tar.gz
# 例: $ tar -zxvf ~/Download/linux-x86_64_ccp4-8.0-setup.tar.gz
# 解凍したファイルを実行します
$ <path_for_download_folder>/ccp4-<version>-setup
# 例: $ ~/Download/ccp4-8.0-setup
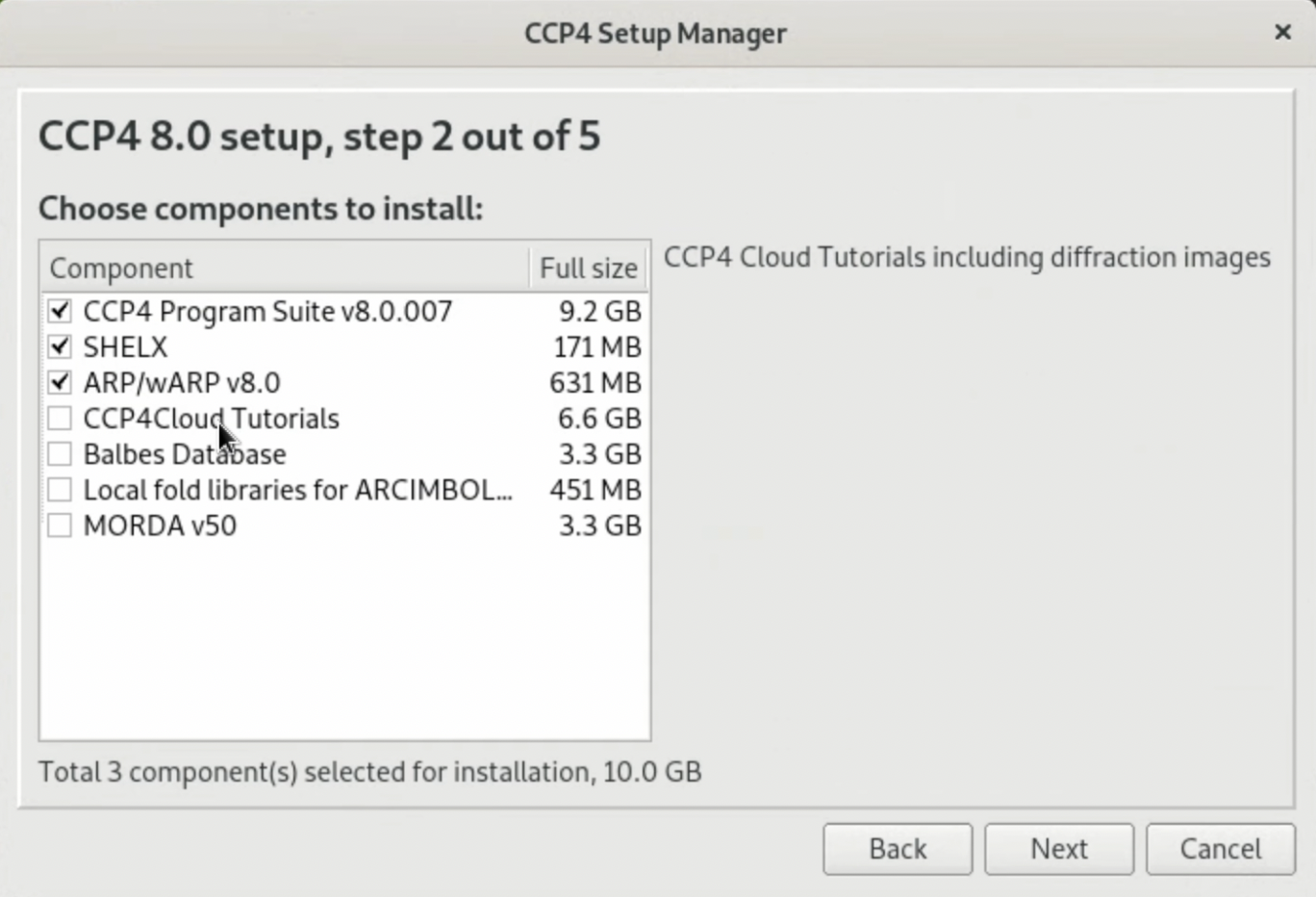
3. Package Managerの指示に従ってインストールを進行する. 途中出でてくる"Choose components to install:"は, 本チュートリアルでは一番上の"CCP4 Program Suite v???"のみ必要なので, これだけ選んでおけば問題ない. なお, SHELXは重原子等を用いた初期位相の決定(MIR, SAD, MAD法など)を行う際によく用いるソフトウェアであり, ARP/wARPは蛋白質モデルの自動構築を行うソフトウェアの一つである. タンパク質X結晶構造解析を本格的に学んでいる方や, X先結晶構造解析を業務・研究に使用する予定のある方はこれら2つもインストールしておくと良いかもしれない.
なお, 筆者の環境では, "tcshが入っていない"というエラーが出た. よって, "tcsh"をaptでインストールし, インストールを進めた.
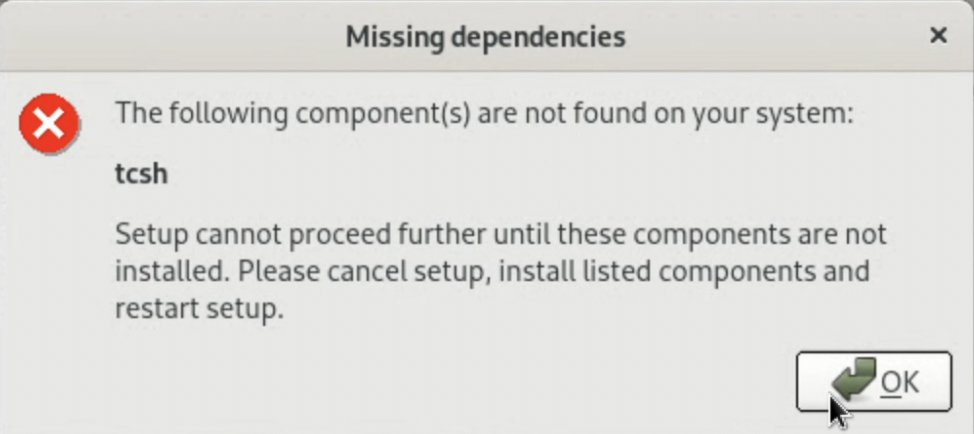
$ sudo apt install tcsh
4. ライセンス条項に同意したら, ファイルのダウンロードが始まり, インストールが始まる. 環境にもよるが30~60分以上かかるので暫く待つ.
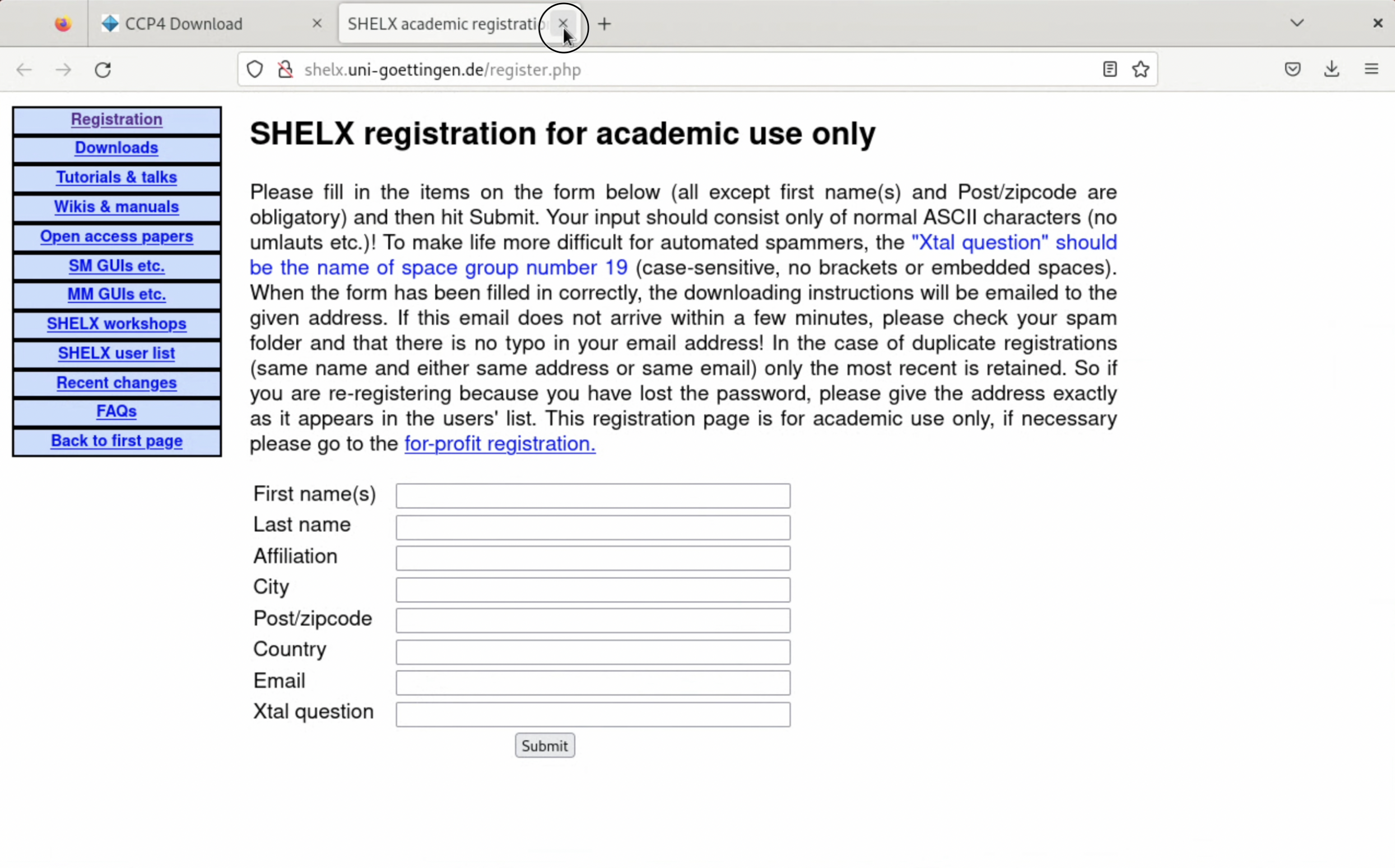
5. SHELXをインストールした際はユーザー登録を求められるページが表示されるので, 適切にユーザー登録を行う. なお"Xtal equestion"の答えは, Google検索ですぐに答えが出てくる.
6. インストール終了後, 最後の仕上げとしてccp4のpathを設定する. ccp4が用意しているbashスクリプトファイルをsourceコマンドで実行するが, ターミナル起動時に読み込まれる"~/.bashrc"などに記述しておくと便利である.
# ccp4は標準では/opt/xtal/ccp4-8.0にインストールされる.
# この場合, 読み込むsetupスクリプトは/opt/xtal/ccp4-8.0/bin/ccp4.setup-shである.
$ vi ~/.bashrc
# 以下の画像のように~/.bashrcの末尾に
# "source /opt/xtal/ccp4-8.0/bin/ccp4.setup-sh"を追加する.
# sourceコマンドでsetupスクリプトを読み込む.
$ source ~/.bashrc
7. 以下のコマンドでccp4iが起動すればCCP4のインストールは成功している.
$ ccp4i
Step.1-7 Pymolのインストール
- Pymolは有償版(学生・教員は無償のEducation version)も存在するが, 今回はOSSの無償バージョンをインストールする.
- PyMol Wiki: Linux Installを参考にしてインストールする. 本チュートリアルの環境はUbuntu 20.04 LTEなので以下のコマンドでインストールを行う.
$ sudo apt install pymol
3. pymolを起動しインストールの確認を行う.
$ pymol
あとがき
今回はタンパク質X先結晶構造解析の概略と, 構造解析に使用する使用データセット, そして各種ソフトをインストールした. 次回は回折データセットから構造因子F(hkl)を計算する.
他のStepへのリンク
- Step.0 構造決定の流れ (本記事)
- Step.1 解析ソフトのインストール (本記事)
- Step.2 回折画像の確認と構造因子の計算
- Step.3 初期位相を求める
- Step.4 構造精密化とバリデーション
-
それぞれのステップにおいても技術革新がなされており, 当時に比べてタンパク質X先結晶構造解析はよりアクセスしやすい実験法になってきている. しかしながら, 本チュートリアルを長々書く程度には, まだタンパク質X先結晶構造解析には専門性が必要なのが現実である. ↩
-
本基本式の導出には基礎的な結晶学の知識が必要になるが, 原子一個の回折現象から徐々に範囲を広げることで本式の導出が可能となる. 基本式の導出については本チュートリアルのレベルを大幅に超えるため, 興味のある方は成書や各大学の公開資料などを参考にしてほしい. ↩
-
近年, 回折画像を得るステップまでは自動化が急速に進み, 限られた研究者のみで大量のサンプルをさばくことが可能になっている. 特に, 回折画像を記録する2次元検出器(ディテクター)の進化により, 大量の回折画像を短時間で測定可能になった事が大きい. ↩ ↩2
-
鶏の卵白由来の糖質加水分解酵素であり, 実験でも大腸菌の溶出によく使われる. 比較的安価で入手可能であり, 高分解能が出る結晶も作りやすく, なおかつ適度に解析難易度が高いことから放射光施設などで行われている初心者向けの講習会や, 装置のバリデーションなどで使われる機会が多い. タンパク質X先結晶構造解析業界における事実上のモデル構造である. ↩
-
企業や研究所で使用される方は, 各ソフトのライセンスを熟読されたい. ソフトの利用に関して生じた損害などに対して, 当方では一切責任を取らない. ↩
-
少なくとも筆者はこれらのソフトウェアを用いて60を超える構造を解明し, PDBに登録してきた. ↩
-
Cootなどで不具合が発生する確率が高く, 原因を見てみるとそのほとんどがGUIライブラリーに起因する. 筆者も多くの時間をこの本質的ではないトラブル解決に費やしたが, Linux環境ではそのトラブルが起きたことはない. ↩
-
Remote DesktopやChrome Remote Desktopなどのリモートデスクトップで3D構造をグリグリいじるのは非常にストレスがたまるので, 可能ならばローカル環境を推奨したい. ↩
-
AWSと比較して無料枠が多いので.... ↩