はじめに
XGBoostは,GBDTの一手法であり,pythonでも実装することが出来ます.
しかし,実装例を調べてみると,同じライブラリを使っているにも関わらずその記述方法が複数あり,混乱に陥りました.そのため,筆者の備忘録的意味を込めて各記法で同じことをやってみようというのがこの記事の趣旨です.
(XGBoostの詳しい説明等は省きますのでご了承ください)
実装環境
- Windows10
- Anaconda3
- Python3
- XGBoostは以下のライブラリをインストールして実装
pip install xgboost
実装内容
- sklearn付属データセットであるBoston house-pricesを用いて住宅価格を回帰する
- XGBoostによる回帰を2種類の記述方法で行う
- 回帰/評価/FeatureImportanceの表示 などをそれぞれできるだけ同じ条件で行う
実装~共通部~
データセットの読み込み
# 今回使うライブラリを先にインポートしておきます
import pandas as pd
import numpy as np
import xgboost
import matplotlib.pyplot as plt
from sklearn.datasets import load_boston
from sklearn.model_selection import train_test_split
from sklearn.metrics import mean_squared_error
boston = load_boston()
df_boston = pd.DataFrame(boston.data, columns=boston.feature_names)
# まとめて表示するために目的変数をdataframe末尾にpriceとしてくっつける
df_boston['PRICE'] = boston.target
print(df_boston.head())
| CRIM | ZN | INDUS | CHAS | NOX | RM | AGE | DIS | RAD | TAX | PTRATIO | B | LSTAT | PRICE | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0.00632 | 18.0 | 2.31 | 0.0 | 0.538 | 6.575 | 65.2 | 4.0900 | 1.0 | 296.0 | 15.3 | 396.90 | 4.98 | 24.0 |
| 1 | 0.02731 | 0.0 | 7.07 | 0.0 | 0.469 | 6.421 | 78.9 | 4.9671 | 2.0 | 242.0 | 17.8 | 396.90 | 9.14 | 21.6 |
| 2 | 0.02729 | 0.0 | 7.07 | 0.0 | 0.469 | 7.185 | 61.1 | 4.9671 | 2.0 | 242.0 | 17.8 | 392.83 | 4.03 | 34.7 |
| 3 | 0.03237 | 0.0 | 2.18 | 0.0 | 0.458 | 6.998 | 45.8 | 6.0622 | 3.0 | 222.0 | 18.7 | 394.63 | 2.94 | 33.4 |
| 4 | 0.06905 | 0.0 | 2.18 | 0.0 | 0.458 | 7.147 | 54.2 | 6.0622 | 3.0 | 222.0 | 18.7 | 396.90 | 5.33 | 36.2 |
trainデータとtestデータの作成
# データを特徴量xと目的変数yに分割
x = df_boston.loc[:,'CRIM':'LSTAT']
y = df_boston['PRICE']
# 7:3でtrainデータとtestデータに分割
trainX, testX, trainY, testY = train_test_split(x, y, test_size=0.3)
# それぞれの形状を出力してみる
print('x.shape = {}'.format(x.shape))
print('y.shape = {}'.format(y.shape))
print('trainX.shape = {}'.format(trainX.shape))
print('trainY.shape = {}'.format(trainY.shape))
print('testX.shape = {}'.format(testX.shape))
print('testY.shape = {}'.format(testY.shape))
# x.shape = (506, 13)
# y.shape = (506,)
# trainX.shape = (354, 13)
# trainY.shape = (354,)
# testX.shape = (152, 13)
# testY.shape = (152,)
実装~訓練~
手法1
手法1はscikit-learn互換のAPIを持ったインタフェースを使った手法です.
馴染みがあるであろうこちらから先に記述していきます.
まずは特にパラメータの指定をせず最も簡単な実装から.
# 回帰なのでXGBRegressorを利用
reg = xgb.XGBRegressor()
# eval_setには検証用データを設定する
reg.fit(trainX, trainY,
eval_set=[(trainX, trainY),(testX, testY)])
# [0] validation_0-rmse:21.5867 validation_1-rmse:21.7497
# [1] validation_0-rmse:19.5683 validation_1-rmse:19.7109
# [2] validation_0-rmse:17.7456 validation_1-rmse:17.8998
# 省略
# [97] validation_0-rmse:1.45198 validation_1-rmse:2.7243
# [98] validation_0-rmse:1.44249 validation_1-rmse:2.72238
# [99] validation_0-rmse:1.43333 validation_1-rmse:2.7233
# 予測実行
predY = reg.predict(testX)
# MSEの表示
print(mean_squared_error(testY, predY))
# 7.4163707577050655
パラメータをもう少しちゃんと指定してやると以下になります.
reg = xgb.XGBRegressor(#目的関数の指定 初期値も二乗誤差です
objective='reg:squarederror',
#学習のラウンド数 early_stoppingを利用するので多めに指定
n_estimators=50000,
#boosterに何を用いるか 初期値もgbtreeです
booster='gbtree',
#学習率
learning_rate=0.01,
#木の最大深さ
max_depth=6,
#シード値
random_state=2525)
# 学習過程を表示するための変数を用意
evals_result = {}
reg.fit(trainX, trainY,
eval_set=[(trainX, trainY),(testX, testY)],
#学習に用いる評価指標
eval_metric='rmse',
#目的関数の改善が見られない場合に学習を打ち切るラウンド数を指定
early_stopping_rounds=15,
#学習過程の記録をするためにコールバックAPIを利用し先ほどの変数を指定
callbacks=[xgb.callback.record_evaluation(evals_result)])
# [1] validation_0-rmse:19.5646 validation_1-rmse:19.7128
# [2] validation_0-rmse:17.7365 validation_1-rmse:17.9048
# [3] validation_0-rmse:16.0894 validation_1-rmse:16.2733
# 省略
# [93] validation_0-rmse:0.368592 validation_1-rmse:2.47429
# [94] validation_0-rmse:0.3632 validation_1-rmse:2.47945
# [95] validation_0-rmse:0.356932 validation_1-rmse:2.48028
# Stopping. Best iteration:
# [80] validation_0-rmse:0.474086 validation_1-rmse:2.46597
predY = reg.predict(testX)
print(mean_squared_error(testY, predY))
# 6.080995445035289
何にも指定しなかった時と比べて多少lossが改善されました.
他にも指定できるパラメータはあるんですが詳しくは
XGBoost の Python API Reference
の中のScikit-Learn APIを参照してください.
ただし,このAPIにも書かれていないパラメータが存在しておりたびたび混乱しています.
例えば何も指定しなかった場合のモデルを出力すると
reg = xgb.XGBRegressor()
print(reg)
# XGBRegressor(base_score=0.5, booster='gbtree', colsample_bylevel=1,
# colsample_bynode=1, colsample_bytree=1, gamma=0,
# importance_type='gain', learning_rate=0.1, max_delta_step=0,
# max_depth=3, min_child_weight=1, missing=None, n_estimators=100,
# n_jobs=1, nthread=None, objective='reg:linear', random_state=0,
# reg_alpha=0, reg_lambda=1, scale_pos_weight=1, seed=None,
# silent=None, subsample=1, verbosity=1)
といった具合にモデルの初期値を確認できます.
しかし,最下段にある
silent=None
をAPIで確認してもそもそもそのようなパラメータが存在していません.
一部サイトでは訓練時の出力の有無のパラメータであると記述されていますが,指定しても特に何も変化しませんでした.
手法2
次は2つ目の手法.こちらはxgboostライブラリのオリジナルAPIになります.
よって少しだけデータセットの扱いに違いがあります.
# xgb.DMatrixによってオリジナルAPIで使用できる形にします
# feature_namesは指定しなくても構いませんが後々便利なのでつけておいた方が無難です
dtrain = xgb.DMatrix(trainX, label=trainY, feature_names = x.columns)
dtest = xgb.DMatrix(testX, label=testY, feature_names = x.columns)
reg = xgb.train(#わざとパラメータを指定せずに初期値で訓練するので空リストを指定
params=[],
dtrain=dtrain,
#evalには検証用データを設定
evals=[(dtrain, 'train'), (dtest, 'eval')])
# [0] train-rmse:17.1273 eval-rmse:17.3433
# [1] train-rmse:12.3964 eval-rmse:12.7432
# [2] train-rmse:9.07831 eval-rmse:9.44546
# [3] train-rmse:6.6861 eval-rmse:7.16429
# [4] train-rmse:5.03358 eval-rmse:5.70227
# [5] train-rmse:3.88521 eval-rmse:4.7088
# [6] train-rmse:3.03311 eval-rmse:4.09655
# [7] train-rmse:2.44077 eval-rmse:3.6657
# [8] train-rmse:2.0368 eval-rmse:3.40768
# [9] train-rmse:1.72258 eval-rmse:3.29363
predY = reg.predict(dtest)
print(mean_squared_error(testY, predY))
# 10.847961069710934
初期値では学習のラウンド数がかなり少なく設定されていることが分かります.
そのせいで手法1よりも良い結果ではありません.
では同じようにパラメータ等を設定して実行してみます.
# xgb.DMatrixによってオリジナルAPIで使用できる形にします
dtrain = xgb.DMatrix(trainX, label=trainY, feature_names = x.columns)
dtest = xgb.DMatrix(testX, label=testY, feature_names = x.columns)
# 先にxgb_paramsとしてパラメータを設定しておきます
xgb_params = {#目的関数
'objective': 'reg:squarederror',
#学習に用いる評価指標
'eval_metric': 'rmse',
#boosterに何を用いるか
'booster': 'gbtree',
#learning_rateと同義
'eta': 0.1,
#木の最大深さ
'max_depth': 6,
#random_stateと同義
'seed': 2525}
# 学習過程を取得するための変数を用意
evals_result = {}
reg = xgb.train(#上で設定した学習パラメータを使用
params=xgb_params,
dtrain=dtrain,
#学習のラウンド数
num_boost_round=50000,
#early stoppinguのラウンド数
early_stopping_rounds=15,
#検証用データ
evals=[(dtrain, 'train'), (dtest, 'eval')],
#上で用意した変数を設定
evals_result=evals_result)
# [1] train-rmse:19.5646 eval-rmse:19.7128
# [2] train-rmse:17.7365 eval-rmse:17.9048
# [3] train-rmse:16.0894 eval-rmse:16.2733
# 省略
# [93] train-rmse:0.368592 eval-rmse:2.47429
# [94] train-rmse:0.3632 eval-rmse:2.47945
# [95] train-rmse:0.356932 eval-rmse:2.48028
# Stopping. Best iteration:
# [80] train-rmse:0.474086 eval-rmse:2.46597
predY = reg.predict(dtest)
print(mean_squared_error(testY, predY))
# 6.151798278561384
学習過程で表示されるlossに関しては手法1と全く同じ値が出ています.
しかしその後predictを行ってMSEを出力すると値が違いますね…ちょっとこれは原因わからないのでスルーします.
コードを見比べて頂けると分かる通り,パラメータの名前や表記する場所が若干異なるので注意しなければなりません.
例えば学習率etaを手法1と同じようにlearning_rateとして設定すると,実行は可能ですが値は反映されません.
こちらのパラメータについては
XGBoost Parameters
と
XGBoost の Python API Reference
のLearning APIを確認してください.
実装~学習過程のグラフ表示~
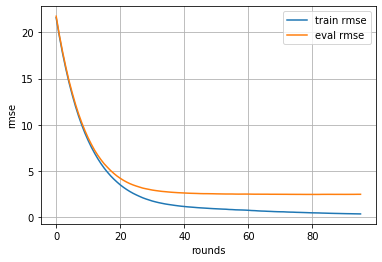
学習時に用意したevals_resultに値が格納されていますのでそれを用いて推移をグラフにします.
手法1
# trainデータに対してのloss推移をplot
plt.plot(evals_result['validation_0']['rmse'], label='train rmse')
# testデータに対してのloss推移をplot
plt.plot(evals_result['validation_1']['rmse'], label='eval rmse')
plt.grid()
plt.legend()
plt.xlabel('rounds')
plt.ylabel('rmse')
plt.show()
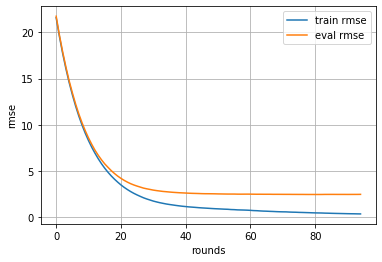
手法2
手法1の時と比べて何故かevals_resultに格納される際の名前が違います.
# trainデータに対してのloss推移をplot
plt.plot(evals_result['train']['rmse'], label='train rmse')
# testデータに対してのloss推移をplot
plt.plot(evals_result['eval']['rmse'], label='eval rmse')
plt.grid()
plt.legend()
plt.xlabel('rounds')
plt.ylabel('rmse')
plt.savefig("img.png", bbox_inches='tight')
plt.show()
実装~FeatureImportanceの表示~
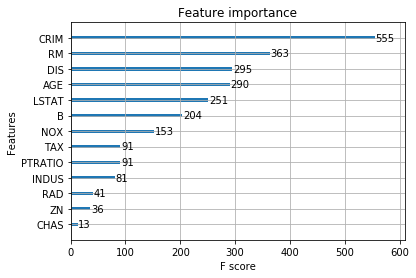
xgboostには**xgb.plot_importance()**というFeatureImportanceをplotしてくれるメソッドが用意されており,手法1・2両方で使用することができます.
xgb.plot_importance(reg)
また,引数にimportance_typeを指定することができ
API のimportance_typeの記述と
こちらの記事 を参考にさせて頂くと
- weight
- 全ての木の分岐でその特徴量が利用される階数
- gain
- 分岐でその特徴量が利用されたときどれだけ評価基準を改善させたかの平均値
- cover
- 葉に分類されたtrainデータの二次勾配の合計値(二乗誤差の時は単純に分類に登場した回数のようです)
の3つを利用できます.(APIを読むにgainとcoverについて平均値ではなく合計値を用いることでもできるようです)
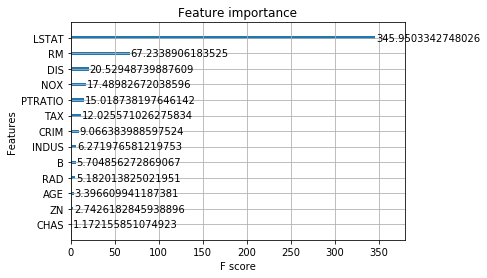
初期値はweightとなっているようで,例えばgainを指定して出力すると以下になります.
xgb.plot_importance(reg,importance_type='gain')
また,xgb.get_score()というメソッドも用意されており,グラフに利用している値をディクショナリとして取得できます.
ただしこのメソッドは手法2でしか利用できず,手法1で同じようなことを実行する方法があるのかは今のところわかりません…この辺の不統一具合が気になるところです.
print(reg.get_score(importance_type='weight'))
# {'LSTAT': 251,
# 'RM': 363,
# 'CRIM': 555,
# 'DIS': 295,
# 'B': 204,
# 'INDUS': 81,
# 'NOX': 153,
# 'AGE': 290,
# 'PTRATIO': 91,
# 'RAD': 41,
# 'ZN': 36,
# 'TAX': 91,
# 'CHAS': 13}
print(reg.get_score(importance_type='gain'))
# {'LSTAT': 345.9503342748026,
# 'RM': 67.2338906183525,
# 'CRIM': 9.066383988597524,
# 'DIS': 20.52948739887609,
# 'B': 5.704856272869067,
# 'INDUS': 6.271976581219753,
# 'NOX': 17.48982672038596,
# 'AGE': 3.396609941187381,
# 'PTRATIO': 15.018738197646142,
# 'RAD': 5.182013825021951,
# 'ZN': 2.7426182845938896,
# 'TAX': 12.025571026275834,
# 'CHAS': 1.172155851074923}
DMatrixを作成する際にfeature_namesを指定したと思いますが,そちらを指定しなかった場合グラフなどに特徴量の名前が表示されないので非常にわかりにくくなります.設定するのがよしです.
その他各手法の特徴
手法1
手法1の最も便利な点はsklearnのGridSerchCV等を用いてパラメータサーチが可能なことではないかと思います.
XGBoostを用いてパラメータサーチを行う記事では調べた限り全て手法1が使われていました.
参考程度にRandomizedSearchCVのコードを載せます.
from sklearn.model_selection import RandomizedSearchCV
params = {
'n_estimators':[50000],
'objective':['reg:squarederror'],
'eval_metric': ['rmse'],
'booster': ['gbtree'],
'learning_rate':[0.1,0.05,0.01],
'max_depth':[5,7,10,15],
'random_state':[2525]
}
mod = xgb.XGBRegressor()
# n_iterにはランダムサーチする回数をしていします
rds = RandomizedSearchCV(mod,params,random_state=2525,scoring='r2',n_jobs=1,n_iter=50)
rds.fit(trainX,
trainY,
eval_metric='rmse',
early_stopping_rounds=15,
eval_set=[(testX, testY)])
print(rds.best_params_)
# {'seed': 2525,
# 'objective': 'reg:squarederror',
# 'n_estimators': 50000,
# 'max_depth': 5,
# 'learning_rate': 0.1,
# 'eval_metric': 'rmse',
# 'booster': 'gbtree'}
手法2
正直オリジナルのAPIの方は馴染みもなくわざわざわざ使う必要はない気もします.
ただし,上記でこちらの手法でしか使えないメソッドがあったように,他にもそのような例が存在する可能性があります.
基本は手法1で実装してあれ?これできないなってやつをこちらで実装してみるのがいいのではないでしょうか.
おわりに
今回はXGBoostについて調べていく中で手法の混在による不明瞭な点をできるだけわかりやすくまとめました.
同じような状況に陥っている人がここにたどり着いて問題を解消されることを祈っています.
xgboostライブラリでは他にも生成した木を図として表示できるメソッドなど便利なものが多いので一度APIにざっと目を通して色々と実装してみることをお勧めします.
人生初めてのQiita記事であり,至らぬ点ばかりと思いますがここまで読んでいただきありがとうございました.
参考記事
Python: XGBoost を使ってみる
xgboostの使い方:irisデータで多クラス分類
PythonでXGBoostを使う
Xgboost : feature_importanceのimportance_type算出方法
xgboost: テーブルデータに有効な機械学習モデル