ASE(Atomic Simulation Environment)で、モデルの保存、可視化を行う
・Jupyter上でモデルを可視化する
・trajファイルのをJupyter上で図を描写する
・pngで2次元的な図を出力
・cifファイルで出力 (→ VESTAで開く)
・povを介して、pngで3次元的な図を出力
まずは、描写させるモデル。slabという名前に格納する
(ASE公式サイト参照)
from ase import Atoms
from ase.build import fcc111, add_adsorbate, bulk
adsorbate = Atoms('CO')
adsorbate[1].z = 1.1
slab = fcc111('Cu', (2, 2, 3), a=3.61, vacuum=7.0)
add_adsorbate(slab, adsorbate, 1.8, 'ontop')
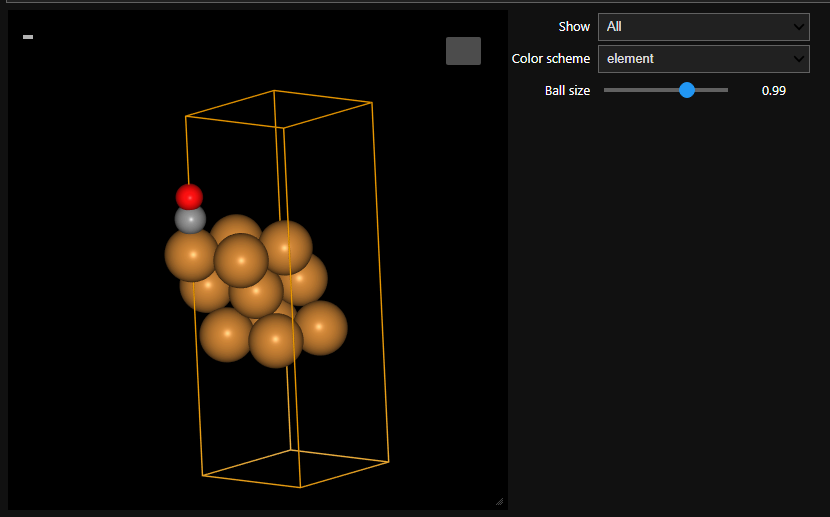
Jupyter上で可視化する
from ase.visualize import view
view(slab, viewer='ngl')
traj (&jupyter上でステップを確認する)
from ase.io import write
write('slab_x3.traj',slab* (3, 3, 1))
# ASE@jupyterで再度読み込んで描写させる場合
from ase.io.trajectory import Trajectory
traj_slab = Trajectory('slab_x3.traj')
from ase.visualize import view
view(traj_slab, viewer='ngl')
Jupyter Lab上で表示させた場合。グリグリ動かせます。
最適化ステップなども保存されていれば再生も可能
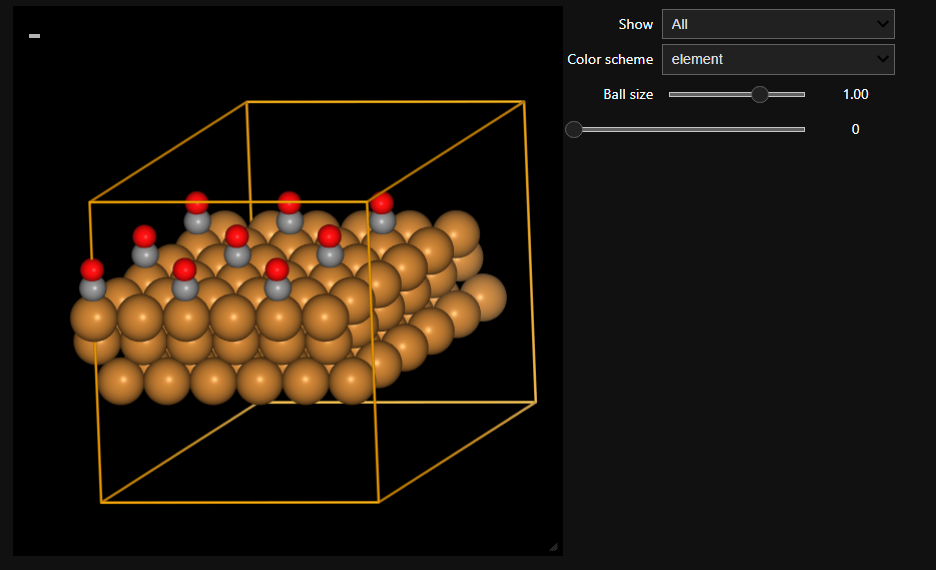
png (2D)
from ase.io import write
write('slab_x3.png', slab* (3, 3, 1), rotation='10z,-80x')
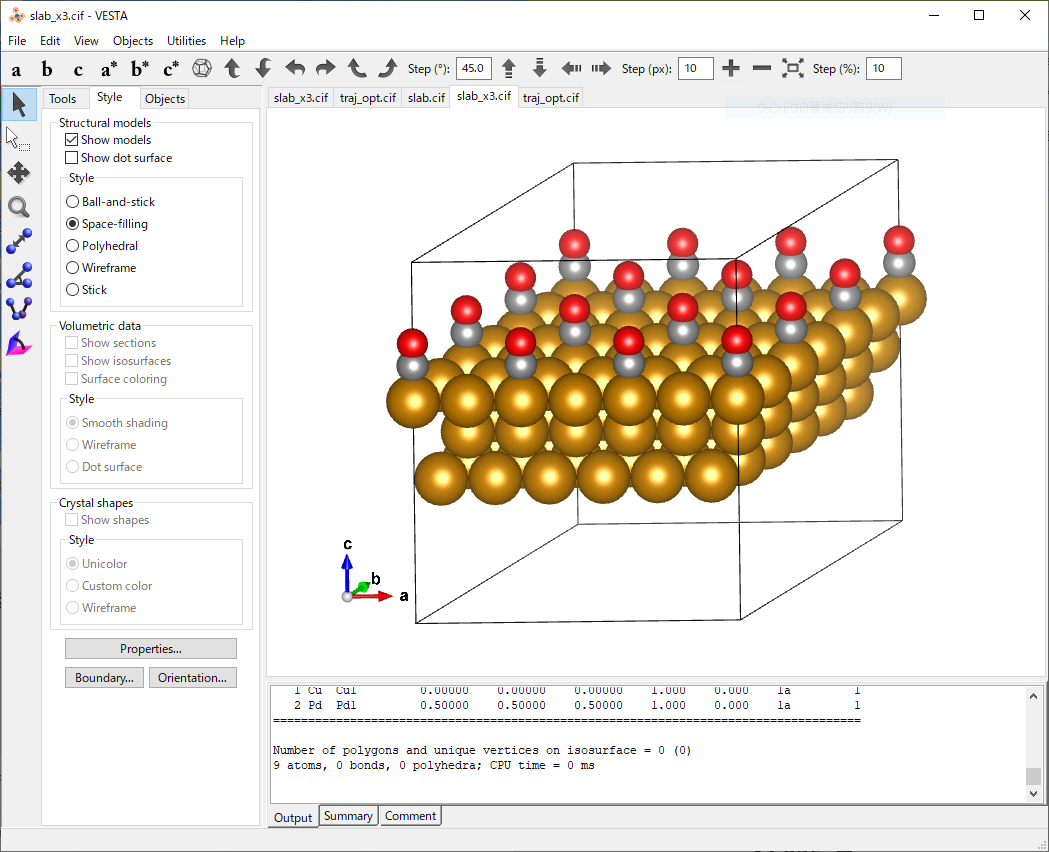
cif
from ase.io import write
write('slab_x3.cif', slab* (3, 3, 1))
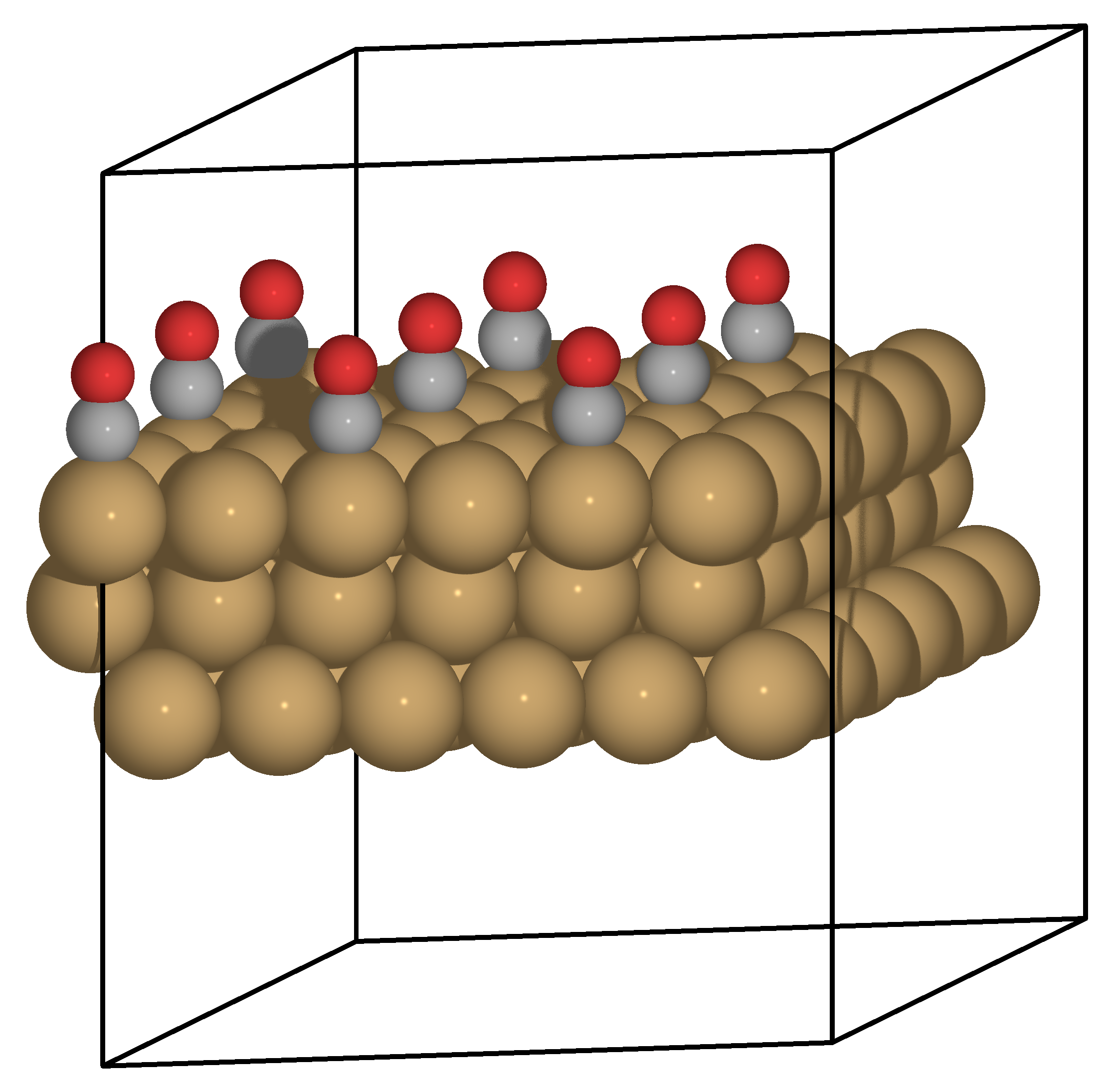
pov →png(3D)
from ase.io import write
write('pov_slab_x3.pov', slab* (3, 3, 1), rotation='10z,-80x')
- povからpngに変換する
ここから先は、ターミナル上を想定。Jupyter上でやるならXwindows必要かも。
#povrayをインストールする (ubuntu ver.)
$ sudo apt install povray
$ cat pov_slab_x3.pov
...
background {color White transmit 0.0}
camera {orthographic
right -22.99*x up 21.90*y
direction 1.00*z
...
# povファイルのrightの行のxとyの比を維持して下記のコマンドで変換する
# 背景白なら、transmit 1.0を0.0に書き換える。
$ !povray -W2230 -H2190 pov_slab_x3.pov
ASEとQuantum Espresso関連の投稿記事リンク
ASEとQuantum Espressoを使って、
- 手動でCuの単位格子最適化して、平衡体積弾性率を見積る
- Ptが単純立方構造、fcc、hpcのいずれの構造をとるかを調べる
- 六方晶Hfの格子定数aとcを最適化する
- H2Oなどの3原子分子を構造を最適化して、安定構造を見つける
他