Overview
この記事は
pandasクックブック-―Pythonによるデータ処理のレシピ の著者である Ted Petrou 氏の以下の記事、
を、許諾を得て翻訳したものです。
https://twitter.com/arc279/status/1095511875050033152
不自然な点、間違っている点などがありましたら指摘してもらえると助かります。
リポジトリはここにあります。
また、長文のため、具体例だけざっくり訳した版も用意しました。
以下、翻訳です。
Minimally Sufficient Pandas(必要最低限の Pandas)
この記事では、データ分析にpandasを使用する際の、私の最適と思う意見を提案します。
私の目的は、データ分析のほとんどのケースでは、ライブラリの一部の機能だけで十分であることを主張することです。
ここで紹介する必要十分な最低限の機能は、 pandas の初心者からプロフェッショナルまで役に立つでしょう。
誰もが私の提案に同意するわけではないでしょうが、それらは私の実際のライブラリの使い方、あるいは教えている手法です。
もし、あなたが同意しないか、別の提案があるなら、以下のコメントに残してください。
(訳注:元記事のコメント欄)
この記事を読み終わると
- Pandas の構文などではなく、実際のデータ分析に集中するためには、一部の機能に限定しても十分であることがわかります
- Pandas を用いた一般的な、多岐にわたるデータ分析のタスクに対する、具体的な単一アプローチの指針を持つことができます
100時間以上の無料のチュートリアル動画
私は、100時間以上にも及ぶ、Pandas, Matplotlib, Seaborn, そして Scikit-Learn の非常に詳細なチュートリアル動画を公開しています。(2019年の2月より)
ぜひ Dunder Data YouTube channel を購読して更新をチェックしてください。
Upcoming Classes
よりパーソナライズされた授業が必要ならば、今年の3月に予定されている Machine Learning Bootcamps in NYC の私の講義に参加してください。
Pandas は強力だが扱いが難しい
Pandas はデータ分析者の間で最もポピュラーな python のライブラリです。
かなり多くの機能を提供していますが、学習コストが高いライブラリでもあります。
これにはいくつかの理由があります:
- 一般的なタスクをこなすのに複数の方法がある
- DataFrame には240を超えるメソッドと属性がある
- (基本的に同じコードを参照している)お互いのエイリアスであるメソッドがある
- ほぼ同じ機能のメソッドがある
- 同じ目的を達するのに、異なる人によって書かれたさまざまな方法によるチュートリアルが氾濫している
- 一般的なタスクを、慣用的な方法でこなす公式のガイドラインが存在しない
- 公式のドキュメントにも、慣用的でないコードが存在する
Pandas の必要最低限の機能はなにか?
データ分析ライブラリの目的は、分析者がデータ分析に集中できるツールを提供することです。
Pandas はまさにうってつけのツールですが、分析に集中できるようにはなっていません。
代わりに、ユーザは複雑で過剰な構文を覚えることを強要されます。
私は Minimally Sufficient Pandas の定義として、以下を提案します。
- たいていの要求をこなすことができるのに十分な、ライブラリの機能の一部
- コードの書き方ではなくデータの分析に集中することができること
この Pandas の必要最低限のサブセットでは:
- 単純、明白、直接的で、退屈なコードになるでしょう
- あるタスクをこなすために、ただ1つの明白な方法を選ぶようになるでしょう
- そして毎回、明白な方法を選ぶでしょう
- 脳内の短期記憶に多くのコマンドを覚えておく必要はありません
- 他人やあなたにとって、より理解しやすいコードになるでしょう
よくあるタスクの標準化
Pandas はしばしば、同じタスクをこなすのに複数のアプローチを提供します。
これは、あなたの取ったアプローチが、他人のそれとは異なるかもしれないことを意味します。
これは、1列のカラムを選択する、などの最も基本的なタスクでも発生する可能性があります。
複数の異なるアプローチを採用しても、個人による単発の分析では、多くは問題にはならないでしょう。
しかし、多人数で長期間かかる分析において、それぞれが異なるアプローチを採用した場合、大混乱を引き起こす可能性があります。
一般的なタスクに対して、標準的なアプローチを採用しないことによって、各自のアプローチの僅かな違いをすべて覚えておくための、大きな認知的負荷が開発者にかかります。
各人が一般的なタスクをこなすために、それぞれの好む方法を採用することは、エラーと非効率を引き起こします。
Stack Overflow の雪崩
Stack Overflow で Pandas に関する質問を検索すると、よくある一般的なタスクに対して、複数の競合するさまざまな結果が得られることは、珍しくありません。
DataFrameの列名の変更に関するこの質問には28の回答があります。
あるタスクをこなすために慣用的な方法を1つ知りたい人にとって、この大量の情報に目を通すことは非常に困難です。
No Tricks
ライブラリの大部分を削ることには、いくつかの(良い)制限があります。
あいまいな Pandas のトリックをたくさん知っていることは、あなたの友達にドヤれるかもしれませんが、たいてい良いコードに繋がることはありません。
それどころか、理解しがたく、そしてデバッグがより困難な長大なコードを生み出すでしょう。
Pandas の具体例
では、タスクをこなすために複数のアプローチが存在する Pandas の、具体例について説明します。
異なるアプローチを比較対照して、どちらのアプローチが望ましいかについての指針を示します。
トピックは以下のとおりです:
- 単一の列を選択する
-
ixインデクサは非推奨 -
atiatによるセルの選択 -
read_csvとread_tableは重複 -
isnaとisnull、notnaとnotnull - 算術演算子および比較演算子と、それらに対応するメソッド
- python の組み込み関数と、同名の Pandas の同名のメソッド
-
groupbyによる集計方法の統一 - MultiIndex の扱いかた
-
groupbypivot_tablecrosstabの違い -
pivotとpivot_table -
meltとstackの類似点 -
pivotとunstackの類似点
必要最低限のガイドライン
具体例は全て以下の原則に則っています。
あるメソッドが他のメソッドよりも追加の機能を提供していない場合(つまり、その機能が他のメソッドのサブセットである場合)、それを使用するべきではありません。メソッドは、追加の独自の機能がある場合にのみ検討する必要があります。
単一の列を選択
Pandas の DataFrame から1列のデータを選択することは、最も簡単なタスクにでありながら、残念ながら、Pandas の提供する複数の手法にユーザが最初に直面するケースです。
ブラケット ([]) またはドット表記を使用して、単一の列を Series として選択できます。小さな DataFrame から、両方の方法を使用して列を選択してみましょう。
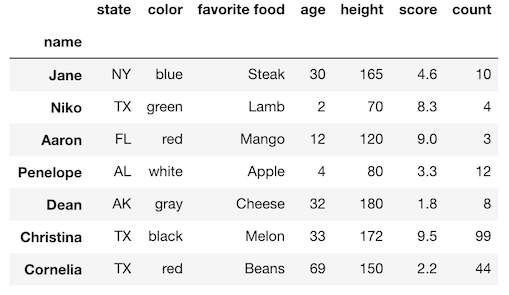
>>> import pandas as pd
>>> df = pd.read_csv('data/sample_data.csv', index_col=0)
>>> df
ブラケット表記での選択
DataFrame の括弧の中に列名を指定すると、単一の列が Series として選択されます。
>>> df['state']
name
Jane NY
Niko TX
Aaron FL
Penelope AL
Dean AK
Christina TX
Cornelia TX
Name: state, dtype: object
ドット表記での選択
あるいは、ドット表記を使用して単一の列を選択することも出来ます。出力は上記と全く同じです。
>>> df.state
ドット表記の問題点
ドット表記には3つの問題点があります。以下の状況では機能しません:
- 列名にスペースが入っているとき
- 列名が DataFrame のメソッドと同じ名前であるとき
- 列名を変数で指定したいとき
列名にスペースが入っているとき
もし必要なカラム名にスペースが入っていると、ドット表記では指定することが出来ません。
Python はスペースを変数名、演算子の区切りとして扱うため、したがって、スペースを含む列名は正しい構文として扱われません。
エラーを起こしてみましょう。
df.favorite food
列名にスペースを含む列の取得はブラケット表記を用いる必要があります。
df['favorite food']
列名が DataFrame のメソッドと同じ名前であるとき
列の名前と DataFrame のメソッド名が競合した場合、 Pandas は常に列名ではなくメソッドを参照します。
例えば、列名 count はメソッドなので、ドット表記で参照されます。
Python はメソッド自体を参照できるため、実際にはエラーになりません。
(訳注:python の関数は first-class object)
では、メソッドを参照してみましょう。
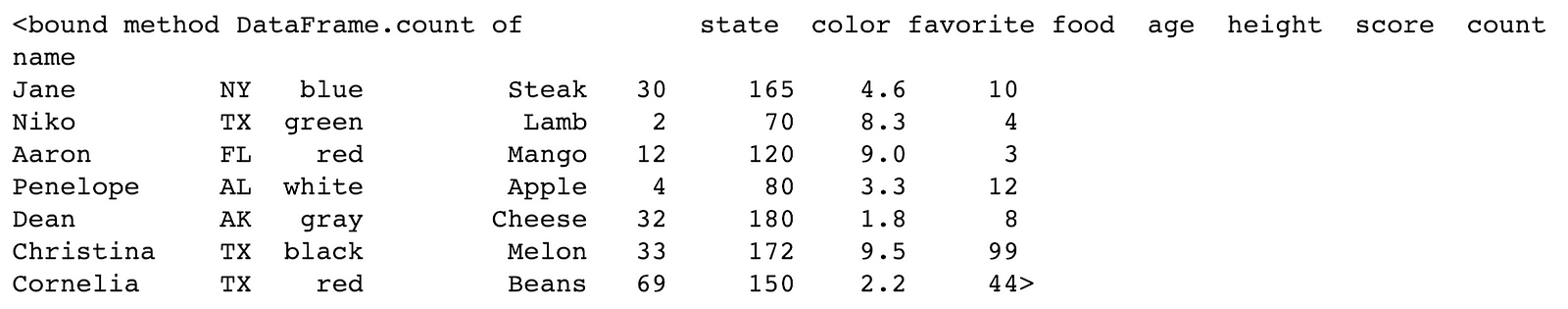
df.count
初めて遭遇した場合、非常に混乱するでしょう。
1行目に bound method DataFrame.count of と記載されています。
これは DataFrame の object の保持する何らかのメソッドである、とPythonは教えてくれます。
メソッド名の代わりに、公式な文字列表現(訳注: repr()を適用したもの)を出力します。
多くの人は、この出力を見て、何らかの分析を行った結果だと勘違いするでしょう。
しかしこれは正しくなく、ほとんど何も起こっておらず、ただオブジェクトの表現を出力するメソッドへの参照が作成さました。
それだけです。
ドット表記を使用して、列が Series として選択されなかったことは明らかです。
繰り返しますが、DataFrame メソッドと同じ名前の列を選択するときは、ブラケット表記を使用する必要があります。
df['count']
列名を変数で指定したいとき
選択したい列名を変数に保持していたとしましょう。
この場合もブラケットを使用することが唯一の手法です。
以下は、列名の値を変数に代入してから、この変数をブラケットに渡す単純な例です。
>>> col = 'height'
>>> df[col]
ブラケット表記はドット表記のスーパーセットである
ブラケット表記は、単一の列を指定する機能としては、ドット表記のスーパーセットです。
ドット表記では扱えない3つのケースを紹介しました。
多くの Pandas はドット表記で書かれています。なぜ?
多くのチュートリアルで、単一の列を選択するのにドット表記が使われています。
ブラケット表記のほうが明らかに優れているのに、なぜでしょうか?
公式のドキュメントの多くの例に使われていたりしますし、3文字多いタイピングが面倒に感じるからかもしれません。
ガイダンス:データの列を選択する際にはブラケット表記を使用する
ドット表記はブラケット表記よりも多くの機能を追加せず、使えないシチュエーションも存在します。
そのため、私は決して使いません。
唯一の利点は、3文字少ないキーストロークです。
単一のデータ列を選択する際にはブラケット表記のみを使用することをお勧めします。
この非常に一般的なタスクに対して、アプローチを統一するだけで、 Pandas のコードは遥かに一貫性のあるものになります。
ix インデクサは非推奨です、使ってはいけません
Pandas は行を選択する際に、ラベルでの指定と、整数での位置指定を提供しています。
この一見柔軟な2通りの方法は、初学者にとって大きな混乱の原因になります。
ix インデクサは、ラベルと整数位置の両方で行と列を選択するために、Pandas の初期に実装されましたが、
Pandas の行と列の名前は、整数と文字列どちらにもなり得ることがあるため、これはかなり曖昧で有ることが判明しました。
これを明示的に選択するために、 loc と iloc インデクサが実装されました。
loc インデクサーはラベルのみ、 iloc インデクサーは整数位置のみに対して使用できます。
ix インデクサは多目的でしたが、loc iloc インデクサの登場により推奨されなくなりました。
ガイダンス: 全ての ix の痕跡を削除し、 loc および iloc に置き換えてください
at iat による選択
DataFrame 内の単一のセルをを選択するために、さらなる2つのインデクサである at iat が存在します。
これらは、類似の loc iloc インデクサに比べて、わずかにパフォーマンスの面で優れています。
しかし、それらの機能を追加でさらに覚えておかなければならない負担ももたらします。
また、ほとんどのデータ分析において、大規模でない限りは、パフォーマンスの向上は役に立ちません。
そして、本当にパフォーマンスが問題になるケースでは、DataFrame から NumPy 配列に変換することで、パフォーマンスは大幅に向上します。
パフォーマンス比較: iloc vs iat vs NumPy
単一セルを選択するケースで、 iloc、 iat、 NumPy配列のパフォーマンスを比べてみましょう。
ランダムデータを含む 100k行 5列 のNumPy配列を作成します。
それを元に DataFrame に変換して、試してみましょう。
>>> import numpy as np
>>> a = np.random.rand(10 ** 5, 5)
>>> df1 = pd.DataFrame(a)
>>> row = 50000
>>> col = 3
>>> %timeit df1.iloc[row, col]
13.8 µs ± 3.36 µs per loop
>>> %timeit df1.iat[row, col]
7.36 µs ± 927 ns per loop
>>> %timeit a[row, col]
232 ns ± 8.72 ns per loop
iloc に比べて iat は2倍の速度も出ない一方、 NumPy配列では約60倍も速度が向上します。
パフォーマンス要件を満たす必要のあるアプリケーションが本当にあるのであれば、Pandas ではなく NumPy を直接使用するべきです。
ガイダンス:単一セル選択に本当にパフォーマンスが必要な場合は、at iat ではなく、 NumPy配列を使用してください
メソッドの重複
Pandas には全く同じことをするメソッドが複数あります。
2つのメソッドが内部で全く同じ機能を使用している場合は、常に、それらは互いのエイリアスであると言います。
ライブラリ内で同じ機能の重複は完全に不要であり、名前空間を汚染し、分析者に余計な情報を覚えておくよう強制します。
次のセクションでは、いくつかの重複したメソッド、あるいはよく似たメソッドについて説明します。
read_csv と read_table の重複
重複の例として、read_csv と read_table 関数を挙げましょう。
どちらもテキストファイルからデータを読み込んで、まったく同じことを行います。
唯一の違いは、 read_csv がデフォルトのセパレータがカンマであるのに対して、 read_table はタブ文字であることです。
read_csv と read_table が同じ結果になることを確認しましょう。
サンプルデータとして、public な College Scoreboard dataset を使用します。
equals メソッドは、2つの DataFrameがまったく同じ値を持つかどうかを確認します。
>>> college = pd.read_csv('data/college.csv')
>>> college.head()
>>> college2 = pd.read_table('data/college.csv', delimiter=',')
>>> college.equals(college2)
True
read_table は非推奨です
私は、非推奨としたほうが良いと思う関数とメソッドに関して、
Pandas の Githubリポジトリ に Issue を出しています。
read_table メソッドは非推奨のため、使用しないでください。
ガイダンス:区切り文字付きテキストファイルを読み込む場合は read_csv のみを使用してください
isna vs isnull と notna vs notnull
isna isnull メソッドはどちらも、DataFrame内に欠損値があるかどうかを判別します。
結果は常に bool 値の DataFrame (あるいは Series)になります。
これらのメソッドは完全に同じです。
一方が他方のエイリアスである、と前の段落で言った通り、両方は必要ありません。
na という欠損値を表す文字列が、他に欠損値を扱う dropna fillna といったメソッド名に追加されたため、 それに合わせて isna メソッドも追加されました。
紛らわしいことに、Pandas は欠損値の表現として NaN None NaT を使用していますが、 NA は使用しません。
notna と notnull はお互いのエイリアスであり、単に isna の反対を返します。これも両方は必要ありません。
isna が isnull のエイリアスであることを確認しましょう。
>>> college_isna = college.isna()
>>> college_isnull = college.isnull()
>>> college_isna.equals(college_isnull)
True
私は isna notna しか使いません
サフィックスに na が付いているメソッドを使用することで、他の欠損値メソッド dropna fillna の名前と整合が取れます。
Pandasは bool 値の DataFrame を反転するための演算子 ~ を提供しているため、notna の使用を避けることもできます。
ガイダンス: isna と notna を使いましょう
算術演算子、比較演算子とそれに対応するメソッド
全ての算術演算子には、同等の機能を提供するメソッドがあります。
(訳注:表にしました)
| op | method |
|---|---|
| + | add |
| - |
sub subtract
|
| * |
mul multiply
|
| / |
div divide truediv
|
| ** | pow |
| // | floordiv |
| % | mod |
全ての比較演算子にも、同等の機能のメソッドがあります。
| op | method |
|---|---|
| > | gt |
| < | lt |
| >= | ge |
| <= | le |
| == | eq |
| != | ne |
ugds 列(undergraduate population の略)のSeriesを選択し、100を加えることで、
+ 演算子とそれに対応するメソッドの両方で同じ結果が得られることを確認しましょう。
>>> ugds = college['ugds']
>>> ugds_operator = ugds + 100
>>> ugds_method = ugds.add(100)
>>> ugds_operator.equals(ugds_method)
True
各学校の z-score を計算する
もう少し複雑な例を見てみましょう。
以下では、 institution 列をインデックスにセットして、SAT 列を選択します。
これらのスコアを提供していない学校は dropna で落とします。
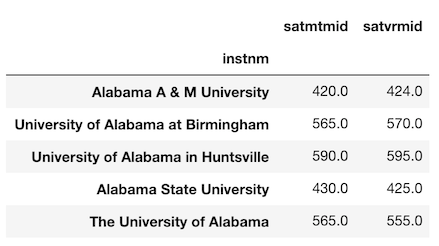
>>> college_idx = college.set_index('instnm')
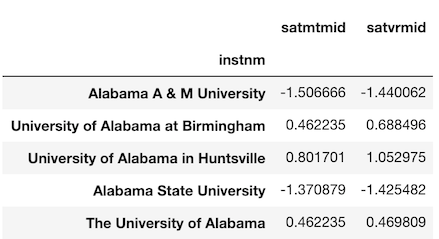
>>> sats = college_idx[['satmtmid', 'satvrmid']].dropna()
>>> sats.head()
各大学の SATスコアの z-score に興味があるとしましょう。
(訳注:名前が紛らわしいですが z-score はいわゆる偏差値で、SATスコアが計算対象のスコアです)
これを計算するには、平均値を引き、標準偏差で割る必要があります。
まず各列の平均と標準偏差を計算しましょう。
>>> mean = sats.mean()
>>> mean
satmtmid 530.958615
satvrmid 522.775338
dtype: float64
>>> std = sats.std()
>>> std
satmtmid 73.645153
satvrmid 68.591051
dtype: float64
算術演算子を使った計算結果を見てみます。
>>> zscore_operator = (sats - mean) / std
>>> zscore_operator.head()
メソッドを使った方法も算出して、結果を比べてみましょう。
>>> zscore_methods = sats.sub(mean).div(std)
>>> zscore_operator.equals(zscore_methods)
True
実際にメソッドが必要になる場合
いまの所、演算子よりもメソッドを使ったほうが良い場面には出会ったことがありません。
メソッドを使用する場合でしか対応できないケースを見てみましょう。
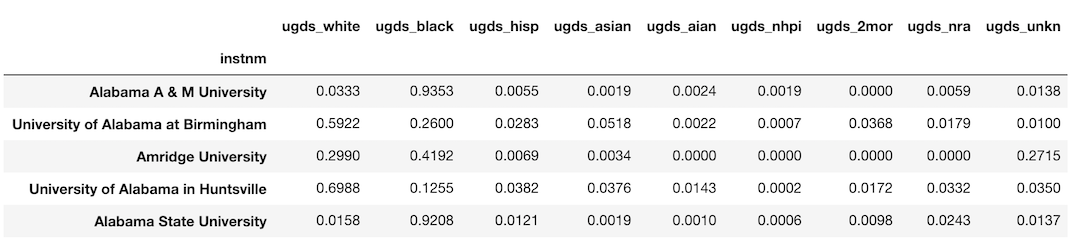
college データセットには、学部生の人種の相対頻度である連続値の列が9つ含まれています。
ugds_white から ugds_unkn までです。
これらの列を新たな DataFrame に取り出しましょう。
>>> college_race = college_idx.loc[:, ‘ugds_white’:’ugds_unkn’]
>>> college_race.head()
学校ごとに人種別の生徒数に関心があるとしましょう。
学部全体の人口に各列を掛ける必要があります。
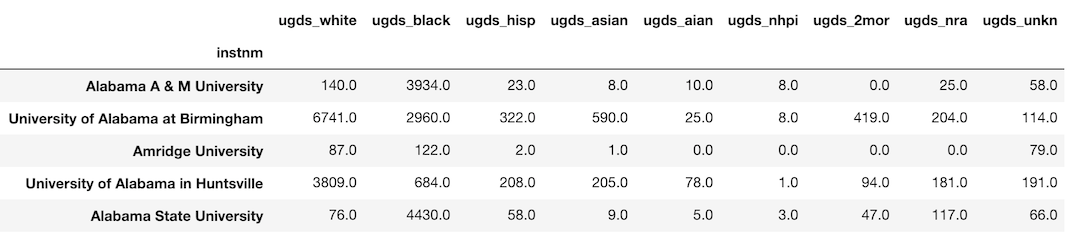
Series として ugds 列を選択しましょう。
>>> ugds = college_idx['ugds']
>>> ugds.head()
instnm
Alabama A & M University 4206.0
University of Alabama at Birmingham 11383.0
Amridge University 291.0
University of Alabama in Huntsville 5451.0
Alabama State University 4811.0
Name: ugds, dtype: float64
そして、先程の college_race にこの Series を掛けます。
直感的にはこれでうまくいきそうですが、そう上手くはいきません。
代わりに、7544列の巨大な DataFrame になります。
>>> df_attempt = college_race * ugds
>>> df_attempt.head()
>>> df_attempt.shape
(7535, 7544)
インデックス あるいは 列の自動調整
Pandas のオブジェクト同士の計算は常に、互いのインデックスあるいは列の間で調整が行われます。
上記の操作では、 college_race(DataFrame) と ugds(Series) を掛け合わせました。
Pandasは自動的に(そして暗黙的に) college_race の列を ugds のインデックスの値に揃えました。
college_race 列の値はどれも ugds のインデックス値と一致していません。
Pandasは、一致するかどうかに関わらず、 outer join することによって調整します。
結果は、全ての値が欠損値である馬鹿げた DataFrame になります.
college_race DataFrameの元の列名を表示するには、右端までスクロールします。
メソッドを使うことで調整の方向を変更する
全ての演算子の機能は単一であり、乗算演算子 * の機能を私達が変更することは出来ません。
一方メソッドは、パラメータによって操作を制御できるようにすることが出来ます。
mul メソッドの axis パラメータを使います
上記で述べた演算子に対応する全てのメソッドには、計算の方向を変更できる axis パラメータがあります。
DataFrame の列を Series のインデックスに合わせる代わりに、DataFrame のインデックスを Series のインデックスに合わせることができます。
今からやり方をお見せしましょう。
>>> df_correct = college_race.mul(ugds, axis='index').round(0)
>>> df_correct.head()
axis パラメータのデフォルト値は columns に設定されています。
適切な調整が行われるように、それを index に変更しました。
ガイダンス:どうしても必要なときだけ算術/比較メソッドを使い、そうでなければ演算子を使う
算術演算子と比較演算子の方がより一般的であり、こちらを最初に試しましょう。
どうしても演算子では解決できない場合に限り、同等のメソッドを使用しましょう。
Python 組み込み関数と同名のPandas のメソッド
Python のビルトイン関数と同じ名前、同じ結果を返す DataFrame / Series のメソッドがいくつかあります。これらです:
- sum
- min
- max
- abs
1列のデータでテストして、同じ結果が得られることを確認しましょう。
まず、学部生の人口の列 ugds から欠損値を除外します。
>>> ugds = college['ugds'].dropna()
>>> ugds.head()
0 4206.0
1 11383.0
2 291.0
3 5451.0
4 4811.0
Name: ugds, dtype: float64
Verifying sum
>>> sum(ugds)
16200904.0
>>> ugds.sum()
16200904.0
Verifying max
>>> max(ugds)
151558.0
>>> ugds.max()
151558.0
Verifying min
>>> min(ugds)
0.0
>>> ugds.min()
0.0
Verifying abs
>>> abs(ugds).head()
0 4206.0
1 11383.0
2 291.0
3 5451.0
4 4811.0
Name: ugds, dtype: float64
>>> ugds.abs().head()
0 4206.0
1 11383.0
2 291.0
3 5451.0
4 4811.0
Name: ugds, dtype: float64
それぞれの処理時間を測ってみます
sum performance
>>> %timeit sum(ugds)
644 µs ± 80.3 µs per loop
>>> %timeit -n 5 ugds.sum()
164 µs ± 81 µs per loop
max performance
>>> %timeit -n 5 max(ugds)
717 µs ± 46.5 µs per loop
>>> %timeit -n 5 ugds.max()
172 µs ± 81.9 µs per loop
min performance
>>> %timeit -n 5 min(ugds)
705 µs ± 33.6 µs per loop
>>> %timeit -n 5 ugds.min()
151 µs ± 64 µs per loop
abs performance
>>> %timeit -n 5 abs(ugds)
138 µs ± 32.6 µs per loop
>>> %timeit -n 5 ugds.abs()
128 µs ± 12.2 µs per loop
sum max min のパフォーマンスの違い
sum max min は明確に差が出ています。
Pandas のメソッドが呼び出される時とは違い、Python 組み込み関数が呼び出されるときにはまったく異なるコードが実行されます。
sum(ugds) を呼び出すと、各値を1つずつ繰り返すための Python の forループが作成されるのと本質的に同等です。
一方、 ugds.sum() を呼び出すと、Cで書かれた Pandas 内部の sumメソッドが実行され、forループで反復するよりもはるかに高速です。
そこまで差が大きくない理由は、Pandas には多くのオーバーヘッドがあることです。
上記の代わりに NumPy array を使用して再度検証すると、10000要素の float の配列で、 Numpy array の sum はPython組み込みのsum関数を200倍ほど上回ります。
abs では結果に差が出ない理由
abs関数と Pandasの absメソッドにパフォーマンスの違いがないことに注意してください。
これは内部でまったく同じコードが呼び出されているためです。
Python は abs関数の挙動を変更する手段を提供しており(訳注: __abs__ マジックメソッドのこと)
、開発者はabs関数が呼び出されるたびに実行されるカスタムメソッドを実装することができます。
そのため、 abs(ugds) と書いても ugds.abs() を呼び出すことと同等です。それらは文字通り同じです。
ガイダンス:python 組み込みの同名の関数よりも、 Pandas のメソッドを使う
groupby による集約方法の統一
集約を実行する際の groupby メソッドにはいくつかの書き方がありますが、コードの統一感の観点から、単一の書き方を採用することをお勧めします。
groupby の3つの要素
通常、 groupby メソッドを呼び出すときは集約を実行します。
最も一般的な使用法でしょう。
groupby での集約処理は、3つの要素に分解できます。
- 列のグループ化
- 列内のユニークな値の抽出
- 列の集約
- 値を集計される列。通常は数値型。
- 集約関数
- 値の集計方法(合計、最小、最大、平均、中央値など)
私の groupby の書き方
Pandas は groupby で集約する際の書き方を何パターンか用意していますが、私は以下の書き方をしています。
df.groupby('grouping column').agg({'aggregating column': 'aggregating function'})
州ごとの SAT の最大値を算出する groupby の書き方のビュッフェ
以下では、州ごとの最大SATスコアを見つけるために、同じ(または類似の)結果を返す、いくつかの異なる書き方について説明します。
まず、使用するデータを見てみましょう。
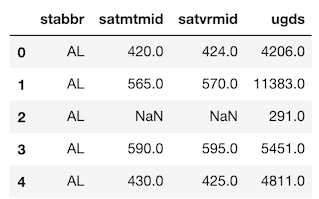
>>> college[['stabbr', 'satmtmid', 'satvrmid', 'ugds']].head()
Method 1:
私の推奨する集約によるグループ化のです。複雑なケースを扱います。
>>> college.groupby('stabbr').agg({'satmtmid': 'max'}).head()
Method 2a:
集約対象のカラムはブラケット記法で選択することもできます。
この場合、戻り値は DataFrame ではなく Series になることに気を付けてください。
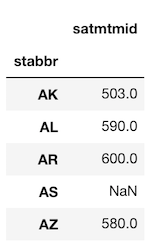
>>> college.groupby('stabbr')['satmtmid'].agg('max').head()
stabbr
AK 503.0
AL 590.0
AR 600.0
AS NaN
AZ 580.0
Name: satmtmid, dtype: float64
Method 2b:
agg のエイリアスである aggregate メソッドを使うことも出来ます。
結果は上記と同じ Series になります。
>>> college.groupby('stabbr')['satmtmid'].aggregate('max').head()
Method 3:
agg メソッド経由ではなく、集約関数を直接呼ぶことも出来ます。
結果は上記と同じ Series になります。
>>> college.groupby('stabbr')['satmtmid'].max().head()
私の提案する方法の主な利点
この書き方だと、より複雑な集約の場合にも同じ書き方で対応できるのが、私がお勧めする理由です。
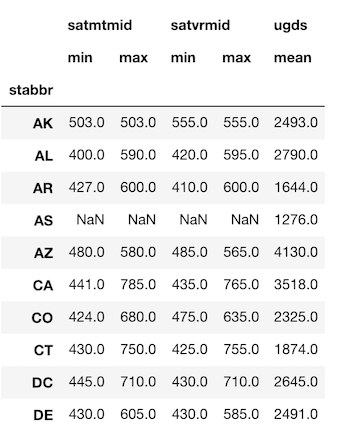
たとえば、州ごとの学部生数の平均とともに、数学と母国語の科目のSATスコアの最大値と最小値を求めたい場合は、次のようにします。
>>> df.groupby('stabbr').agg({'satmtmid': ['min', 'max'],
'satvrmid': ['min', 'max'],
'ugds': 'mean'}).round(0).head(10)
この手段は、他の方法では対応できません。
ガイダンス: まずは df.groupby('grouping column').agg({'aggregating column': 'aggregating function'}) 方式の書き方を検討してください
(訳注)dictの順序を保持しない python3.6 未満だとカラムの追加される順序が不定になるので注意
MultiIndex の扱い方
MultiIndex、またはマルチレベルインデックスは、DataFrame に追加しづらい一方、データを見やすくする事がたまにはあるもの、
しかし多くの場合、扱いがさらに難しくなります。
groupby の集約に複数のカラムを指定している場合、あるいは複数のカラムを集約している場合は、たいてい MultiIndex に遭遇するでしょう。
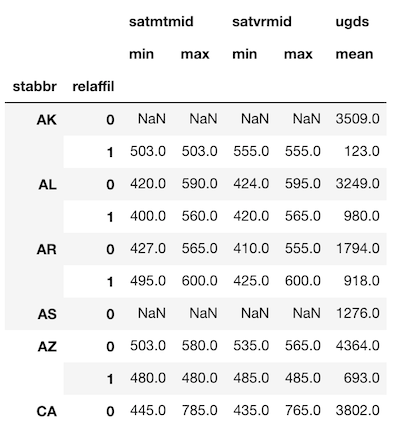
前の章の groupby の最後のサンプルと同じ結果を作成しましょう。
ただし、今回は州と宗教の両方で集約します。
>>> agg_dict = {'satmtmid': ['min', 'max'],
'satvrmid': ['min', 'max'],
'ugds': 'mean'}
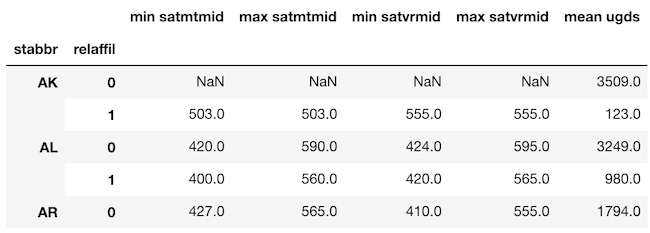
>>> df = college.groupby(['stabbr', 'relaffil']).agg(agg_dict)
>>> df.head(10).round(0)
インデックスと列が両方 MultiIndex
行と列、共に2レベルのMultiIndexになりました。
MultiIndex では、選択とさらなる処理は困難です
MultiIndex が DataFrame に追加する機能は殆どありません。
インデックスの一部を選択するための構文が異なっており、他のメソッドと共に用いるのが難しいです。
もしあなたが Pandas のエキスパートなら、インデックスの一部の選択を利用することで若干のパフォーマンス向上が見込めるでしょうが、私は複雑さが増すことを好みません。
よりシンプルな、シングルインデックスの DataFrame を扱うことをお勧めします。
シングルインデックスに変換 - 列名を変更してインデックスをリセットする
この DataFrame を変換して、単一のインデックスだけが残るようにしてみましょう。
groupby の処理中に直接 DataFrame の列名を変更することはできません(そう、Pandas は非常に単純なことはできないのです)。
なので、手動で上書きする必要があります。
やってみましょう。
>>> df.columns = ['min satmtmid', 'max satmtmid', 'min satvrmid',
'max satvrmid', 'mean ugds']
>>> df.head()
ここから、reset_index メソッドを使用して、各レベルごとのインデックスを列に移動させることが出来ます。
>>> df.reset_index().head()
ガイダンス: MultiIndex の使用を避ける。groupby の呼び出し後は、列名を変更してインデックスをリセットすることによってフラット化します。
groupby pivot_table crosstab の類似点
集約時の groupby、pivot_table、および pd.crosstab は本質的に同じ処理をしている、ということを知って、驚く人もいるかも知れません。ただし、それぞれに固有の使用例があるため、すべてが Minimally Sufficient Pandas に含まれるための条件を満たしています。
groupby の集約と pivot_table の等価性
groupby メソッドを用いて集約をすることは、 pivot_table メソッドが行っていることと本質的には同じです。
どちらのメソッドもまったく同じデータを返しますが、 shape が異なります。
これが事実であることを証明する、簡単な例を見てみましょう。
新たに、ヒューストン市の従業員の人口統計情報を含むデータセットを使用します。
>>> emp = pd.read_csv('data/employee.csv')
>>> emp.head()
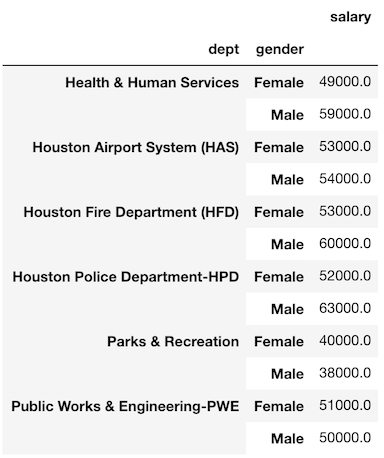
部門別の性別による平均給与を groupby で集計してみましょう。
>>> emp.groupby(['dept', 'gender']).agg({'salary':'mean'}).round(-3)
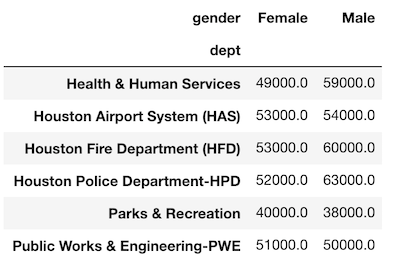
pivot_table を使ってこのデータを複製することができます。
>>> emp.pivot_table(index='dept', columns='gender',
values='salary', aggfunc='mean').round(-3)
値がまったく同じことに注意してください。
唯一の違いは、gender 列がピボットされているため、ユニークな値が列名になっていることです。
groupby の3要素が pivot_table にもあります。
- 集約のキーになる列は
indexcolumnsパラメータで指定され、 - 集約対象の列は
valuesパラメータに渡され、 - 集計関数は
aggfuncパラメータに渡されます。
両方の集約のキー列を list として index パラメーターに渡すことで、データと shape の両方を正確に複製することが実際に可能です。
>>> emp.pivot_table(index=['dept','gender'],
values='salary', aggfunc='mean').round(-3)
通常、pivot_table は2つの集約のキーを使用します。1つはインデックスとして、もう1つは列としてです。
ただし、単一の集約キー列を使用することは出来ます。
以下は、groupby を使用して単一の集約キー列を正確に複製したものです。
>>> df1 = emp.groupby('dept').agg({'salary':'mean'}).round(0)
>>> df2 = emp.pivot_table(index='dept', values='salary',
aggfunc='mean').round(0)
>>> df1.equals(df2)
True
ガイダンス: グループ間を比較する際には pivot_table を使う
私は、グループ間で値を比較する際には pivot_table を好み、分析を継続したい場合は groupby を使用します。
上記の通り、pivot_table の結果を見ると、男性と女性の給料が用意に比較できます。
要約することで人間に見やすくなり、記事やブログ投稿に表示されるデータの種類も少なくなります。
私はピボットテーブルを集計の結果と見なしています。
groupby の結果はきちんとした形式になるでしょうし、
それはその後の分析を容易にするのに役立ちますが、解釈可能ではありません。
pivot_table と pd.crosstab の等価性
pivot_table メソッドと crosstab 関数はどちらも、同じ shape でまったく同じ結果を生成できますし、
両方とも index、columns、values、および aggfunc といったパラメータを共有しています。
表面上の主な違いは、 crosstab は関数であり、DataFrame のメソッドではないということです。
そのため、パラメータとして列名を指定するのではなく、Series として列を使用することを強制されます。
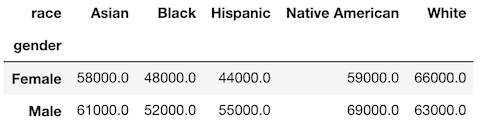
性別と人種による平均給与の集計結果を見てみましょう。
>>> emp.pivot_table(index='gender', columns='race',
values='salary', aggfunc='mean').round(-3)
crosstab とまったく同じ結果を取得するには、以下のようにします。
>>> pd.crosstab(index=emp['gender'], columns=emp['race'],
values=emp['salary'], aggfunc='mean').round(-3)
crosstab は度数を見るために用意されています
クロス集計(分割表とも言う)は、2つの変数間の度数を示します。
これは、2つの列が指定されている場合の crosstab のデフォルトの機能です。
すべての人種と性別の組み合わせの度数を表示してみましょう。
aggfunc を指定する必要がないことに注意してください。
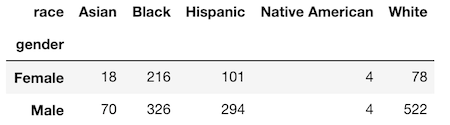
>>> pd.crosstab(index=emp['gender'], columns=emp['race'])
pivot_table メソッドでも同じ結果を複製することができますが、 size 集約関数を使用しなければなりません。
>>> emp.pivot_table(index='gender', columns='race', aggfunc='size')
相対頻度 - crosstab 独自の機能
この時点では crosstab は pivot_table メソッドの一部に過ぎません。
しかし、 crosstab にしか存在しない唯一の機能があり、そのため Minimally Sufficient Subset に追加することは潜在的に価値があります。
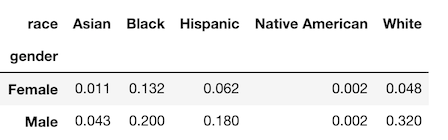
normalize パラメータを使用して、グループ間の相対頻度を計算することができます。
たとえば、race ごとに gender のパーセンテージの内訳が必要な場合は、normalize パラメータを columns に設定できます。
>>> pd.crosstab(index=emp['gender'], columns=emp['race'],
normalize='columns').round(2)
また以下のように、 index を指定して行を正規化したり、all を指定して DataFrame 全体を正規化することもできます。
>>> pd.crosstab(index=emp['gender'], columns=emp['race'],
normalize='all').round(3)
ガイダンス: 相対頻度を見たい場合のみ crosstab を使う
それ以外の crosstab が必要なすべての状況は、 pivot_table で代用できます。
pivot_table を実行した後に手動で相対頻度を計算することも可能ではあるので、 crosstab はそれほど必要ではないでしょう。
しかし、読みやすく1行のコードで計算処理を書けるため、私は今後も使用していきます。
pivot と pivot_table
ほとんど役に立たず、基本的に無視しても構わない pivot メソッドがあります。
この関数は pivot_table に似ていますが、一切集約処理をせず、index columns values の3つのパラメータしかありません。
これらの3つのパラメータはすべて pivot_table にあります。
集約せずにデータを再整形します。
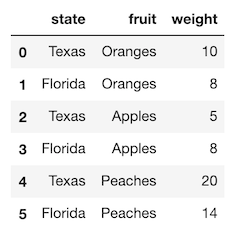
新しい簡単なデータセットを使った例を見てみましょう。
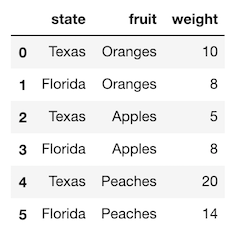
>>> df = pd.read_csv('data/state_fruit.csv')
>>> df
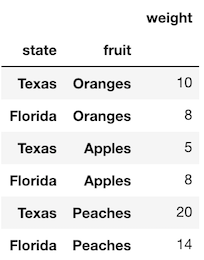
pivot メソッドを使用してこのデータを変形し、 fruit が列になり、weight が値になるようにしましょう。
>>> df.pivot(index='state', columns='fruit', values='weight')
pivot メソッドを使うことで、集約、あるいは他の何かをせずにデータの再整形だけが出来ます。
一方、pivot_table では集計を行う必要があります。
この場合だと、state と fruit ごとに1つの値しかあないため、非常に多くの集約関数が同じ値を返します。
max 集計関数を使用して、これとまったく同じテーブルを再作成しましょう。
>>> df.pivot_table(index='state', columns='fruit',
values='weight', aggfunc='max')
pivot の問題点
pivot メソッドにはいくつかの大きな問題があります。
まず、 index と columns の両方が単一の列に設定されている場合にのみ処理できます。
インデックス内に複数の列を保持したい場合は、 pivot を使用することはできません。
また、index と columns の同じ組み合わせが複数回現れると、集計が実行されないため、エラーが発生します。
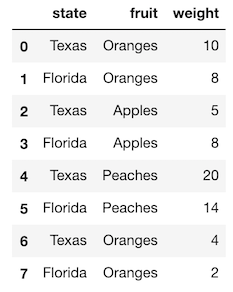
上記と似ていますが、2行追加されているデータセットを使用して、このエラーを見てましょう。
>>> df2 = pd.read_csv('data/state_fruit2.csv')
>>> df2
これを pivot しようとしても、 Oranges と Florida Texas の両方の組み合わせに重複があるため、うまくいきません。
>>> df2.pivot(index='state', columns='fruit', values='weight')
ValueError: Index contains duplicate entries, cannot reshape
このデータを再整形する場合は、値をどのように集計するかを決めなければなりません。
ガイダンス: pivot_table のみを使用し、pivotを使用しないことを検討してください。
pivot でできることは全て pivot_table でもできます。
この場合、集計を実行する必要がない場合でも、集約関数を指定する必要があります。
melt と stack の類似点
melt メソッドと stack メソッドはまったく同じ方法でデータを変形します。
主な違いは、melt メソッドはインデックスにあるデータに対しては使えないことです。stack メソッドは可能です。
具体例を見たほうが理解しやすいでしょう。
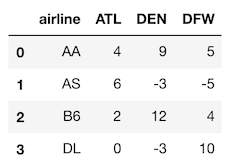
では、航空会社の到着遅延の小さなデータセットを読んでみましょう。
>>> ad = pd.read_csv('data/airline_delay.csv')
>>> ad
このデータを再構成して、airline airport arrival delay の3つの列を作成します。
melt メソッドに指定する2つの主なパラメータから始めましょう、
垂直方向のままにする(再形成しない)列名である id_vars と、単一の列に再形成する列名である value_vars です。
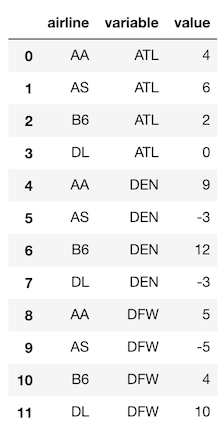
>>> ad.melt(id_vars='airline', value_vars=['ATL', 'DEN', 'DFW'])
stack メソッドでもほぼ同じデータを作成できますが、それは再整形された列をインデックスに入れます。
また、現在のインデックスは保持されます。
上記と同じデータを作成するには、最初に再整形されない列にインデックスを設定する必要があります。
やってみましょう。
>>> ad_idx = ad.set_index('airline')
>>> ad_idx
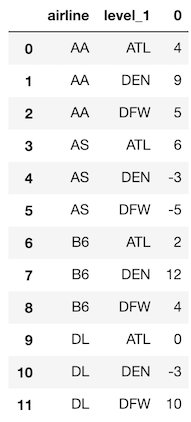
これで、パラメータを設定せずに stack を使用して、melt とほぼ同じ結果を得ることができます。
>>> ad_idx.stack()
airline
AA ATL 4
DEN 9
DFW 5
AS ATL 6
DEN -3
DFW -5
B6 ATL 2
DEN 12
DFW 4
DL ATL 0
DEN -3
DFW 10
dtype: int64
結果は、2レベルの MultiIndex を持つSeriesになります。
データの値は同じですが、順序が異なります。
reset_index を呼ぶことで、単一インデックスの DataFrame に戻ります。
>>> ad_idx.stack().reset_index()
melt でカラム名を変更する
カラム名を直接変更することができ、MultiIndex を避けることができるため、melt を使用することをお勧めします。
var_name および value_name パラメータは、再整形された列の名前を変更するための melt のパラメータです。
また、id_vars にないすべての列が再形成されるので、残りの全ての列を指定する必要もありません。
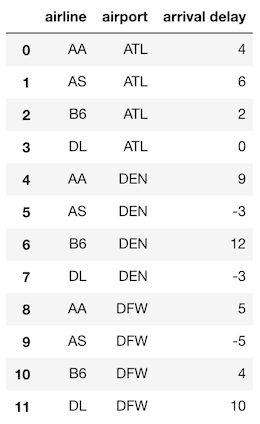
>>> ad.melt(id_vars='airline', var_name='airport',
value_name='arrival delay')
ガイダンス: 列の名前を変更することができ、MultiIndex を避けることができるため、stack より melt を使用する
pivot と unstack の類似点
pivot メソッドがどのような処理を行うかは、すでに説明しました。
unstack は、インデックスを値として扱う、似たような処理です。
pivot で使用した単純なDataFrameを見てみましょう。
>>> df = pd.read_csv('data/state_fruit.csv')
>>> df
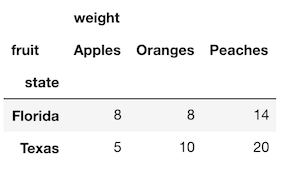
unstack メソッドはインデックス内の値を転置します。
pivot メソッドで index および columns パラメータとして使用する列に対応するように、インデックスを設定する必要があります。
やってみましょう。
>>> df_idx = df.set_index(['state', 'fruit'])
>>> df_idx
これで、パラメータなしで unstack を使用できます。
これにより、インデックスレベルが実際のデータ(fruit 列)に最も近くなり、その一意の値が新しい列名になります。
>>> df_idx.unstack()
結果は、pivot メソッドで返された結果とほぼ同じになりますが、列が MultiIndex になります。
ガイダンス: unstack pivot よりも pivot_table を使う
pivot と unstack はどちらも同じように動作しますが、
上述した通り、 pivot_table は pivot が扱うことができるすべてのケースを扱うことができるため、 pivot_table を使うことをお勧めします。
以上で具体例を終わります
上記の具体的な例で、Pandasで最も一般的なタスクの中で、アプローチの定型手段が無い方法を、だいたいカバーできたと思います。
いずれの例でも、私は単一のアプローチを採用することを提案しました。
これは、私がPandasを使用してデータ分析を行うときに使用するアプローチであり、私が生徒に教えるアプローチです。
The Zen of Python
Minimally Sufficient Python は Tim Peters による Python の19の格言 The Zen of Python に触発されました。
特に注目に値する格言はこれです:
There should be one-- and preferably only one --obvious way to do it.
何かいいやり方があるはずだ。誰が見ても明らかな、たったひとつのやり方が。
私は Pandas が、今まで見てきたどのライブラリよりも、この手引きに従っていないことに気付きました。
Minimally Sufficient Pandas は、この原則に沿うようにユーザーを誘導する試みです。
Pandas Style Guide
上記の具体例では、多くのタスクに対するガイダンスを提供しましたが、ライブラリ内の全てを網羅しているわけではありません。
同意できないガイダンスもあるでしょう。
あなたがライブラリを使うのを助けるために、私は Pandas style guide を作成することを勧めます。
これは、コードベースを統一させるために作成されるための、コーディングスタイルガイドと大差ありません。
すべての Pandas を使用している分析者のチームにとって有益なものです。
Pandasスタイルガイドを強制すると、以下のことに役立ちます:
- すべての一般的なデータ分析タスクに同じ構文を使用させることができます
- Pandas のコードを本番環境に投入するのを用意にします
- Pandasのバグに遭遇する可能性を減らします。
大量の未解決のIssue があるため、ライブラリの一部機能に限って使用することで、これらを回避するのに役立ちます。
Best of the API
Pandas DataFrame APIは膨大です。
ほとんど、あるいはまったく使用されていないか、エイリアスであるメソッドが大量にあります。
以下は、ほぼすべてのタスクを完了するのに、これだけ十分だと私が考える、 DataFrame の属性とメソッドのリストです。
属性
- columns
- dtypes
- index
- shape
- T
- values
集約関数
- all
- any
- count
- describe
- idxmax
- idxmin
- max
- mean
- median
- min
- mode
- nunique
- sum
- std
- var
集約関数ではない統計メソッド
- abs
- clip
- corr
- cov
- cummax
- cummin
- cumprod
- cumsum
- diff
- nlargest
- nsmallest
- pct_change
- prod
- quantile
- rank
- round
部分選択
- head
- iloc
- loc
- tail
欠損値の取り扱い
- dropna
- fillna
- interpolate
- isna
- notna
Grouping
- expanding
- groupby
- pivot_table
- resample
- rolling
データの結合
- append
- merge
その他
- asfreq
- astype
- copy
- drop
- drop_duplicates
- equals
- isin
- melt
- plot
- rename
- replace
- reset_index
- sample
- select_dtypes
- shift
- sort_index
- sort_values
- to_csv
- to_json
- to_sql
関数群
- pd.concat
- pd.crosstab
- pd.cut
- pd.qcut
- pd.read_csv
- pd.read_json
- pd.read_sql
- pd.to_datetime
- pd.to_timedelta to_sql
結論
Minimally Sufficient Pandas は、構文を見失わずに、より効果的にデータ分析で効果を上げたいという方に役立つガイドであると、強く感じます。