前回の記事ではデータフレーム内の分子群に対して化学反応を適用しました。
今回は化学反応を適用することで得られるmolオブジェクトが元のデータファイルに
含まれているか(=完全一致するか)どうかの判定を行います。
これまでの記事のシリーズ
RDKit入門①:まずは環境構築
RDKit入門②:1分子の読み込みと部分構造検索
RDKit入門③:複数分子の読み込み (前編)と分子群の描画
RDKit入門④:複数分子の読み込み (後編)とデータフレームの加工
RDKit入門⑤:データフレーム内の分子群に対する部分構造検索
RDKit入門⑥:Morganフィンガープリントの作成とそれを用いたタニモト係数の計算による分子類似性評価
RDKit入門⑦:反応式の取り扱い(前編)
RDKitで化学反応を扱った際の生成物群を整理し図示する方法
RDKit入門⑧:反応式の取り扱い(後編)
RDKitで化学反応を繰り返しつつ分子の重複数をカウント・記載する
RDKit入門⑨:データフレーム内の分子群に対する化学反応の適用とExcelファイルへの出力
環境
- Windows 10
- python 3.8.8
- RDKit 2021.03.1
下準備
今回もAsinex社のビルディングブロックを題材として扱います。
まずは前回の記事中で作成したpickleファイルを読み込みます。
from rdkit import Chem
from rdkit.Chem import AllChem, Draw, PandasTools
import pandas as pd
# パッケージの読み込み
df = pd.read_pickle("Asinex_Building_Blocks.pkl")
# Asinex_Building_Blocks.pklを読み込み、得られたデータフレームをdfと定義した。
今回はAr-X (X = Cl, Br, I) → Ar-OHという反応を題材とし、手持ちのデータ内にある
Ar-OHを合成するのに必要な原料Ar-Xが手元にあるかどうか探す作業を行います。
まず原料・生成物それぞれに対応する化合物群をピックアップします。
query_1 = Chem.MolFromSmarts("c[Oh]")
# 検索したい構造をSMARTS記法で表し、query_1というmolオブジェクトにした。
df_1 = df[df['ROMol'] >= query_1]
# query_1の部分構造を有する化合物をピックアップしたデータフレームをつくり、df_1と定義した。
query_2 = Chem.MolFromSmarts("c[Cl,Br,I]")
# 検索したい構造をSMARTS記法で表し、query_2というmolオブジェクトにした。
df_2 = df[df['ROMol'] >= query_2]
# query_2の部分構造を有する化合物をピックアップしたデータフレームをつくり、df_2と定義した。
続いて反応を定義します。
今回はAr-OH → Ar-Cl, Ar-OH → Ar-Br, Ar-OH → Ar-Iとなる反応をそれぞれ個別に定義します。
rxn_Cl = AllChem.ReactionFromSmarts("[c:1][Oh:2]>>[c:1]Cl")
rxn_Cl.Initialize()
def Chlorination(mol):
while rxn_Cl.IsMoleculeReactant(mol) == True:
mol = rxn_Cl.RunReactants([mol])[0][0]
mol.UpdatePropertyCache(strict = False)
return mol
# 分子内の全てのフェノール性水酸基をClに変換する反応をChlorinationと定義した。
rxn_Br = AllChem.ReactionFromSmarts("[c:1][Oh:2]>>[c:1]Br")
rxn_Br.Initialize()
def Bromination(mol):
while rxn_Br.IsMoleculeReactant(mol) == True:
mol = rxn_Br.RunReactants([mol])[0][0]
mol.UpdatePropertyCache(strict = False)
return mol
# 分子内の全てのフェノール性水酸基をBrに変換する反応をBrominationと定義した。
rxn_I = AllChem.ReactionFromSmarts("[c:1][Oh:2]>>[c:1]I")
rxn_I.Initialize()
def Iodation(mol):
while rxn_I.IsMoleculeReactant(mol) == True:
mol = rxn_I.RunReactants([mol])[0][0]
mol.UpdatePropertyCache(strict = False)
return mol
# 分子内の全てのフェノール性水酸基をIに変換する反応をIodationと定義した。
定義した反応をそれぞれ実施し、生成物を新しい列に格納します。
df_1["Chloride"] = df_1['ROMol'].map(Chlorination)
df_1["Bromide"] = df_1['ROMol'].map(Bromination)
df_1["Iodide"] = df_1['ROMol'].map(Iodation)
# df_1のROMolの列に格納されているMolオブジェクトにChlrination, Bromination, Iodationを適用した。
# 出力結果をそれぞれChloride, Bromide, Iodideに格納した。
化合物の完全一致検索について
反応の定義・適用により生成物のmolオブジェクトが用意できたため、
後はdf_1のChloride, Bromide, Iodide内に格納されている化合物と完全一致するものが
df_2の中に含まれているかどうかを調べるだけなのですが、一点注意すべきポイントがあります。
以下、トルエンを例に検証します。
test_1 = Chem.MolFromSmiles("Cc1ccccc1")
test_2 = Chem.MolFromSmiles("c1ccc(C)cc1")
test_1 >= test_2 #True
test_1 <= test_2 #True
test_1 == test_2 #False
test_1, test_2はそれぞれトルエンのmolオブジェクトを発生させたものです。
SMILES構造式の書き方を変えているだけなので、test_1の部分構造としてtest_2が含まれますし、
同様にtest_2の部分構造としてtest_1が含まれることが確認できます。
しかしtest_1とtest_2はmolオブジェクトの用意の仕方が異なるせいなのか、
==(eq比較演算子)で完全一致かどうか確認するとFalseを返します。
この問題を解決するにはどうすればよいでしょうか。
一つの手法としてcanonical SMILESの生成と比較が利用可能です。
Chem.MolToSmiles関数にはデフォルトでcanonical = Trueが設定されているため、
同一分子から生成されるSMILES構造式は必ず同一になります。
Chem.MolToSmiles(test_1) == Chem.MolToSmiles(test_2) #True
化合物の完全一致検索の方法を確認したので実際の作業に移ります。
以下、与えたmolオブジェクトがdf_2に含まれる場合に対応する化合物のIDNUMBERを返し、
含まれない場合に'Not Exist'というメッセージを返す関数を定義します。
def Halide_search(mol):
halide_row = df_2[df_2['SMILES'] == Chem.MolToSmiles(mol)]
if len(halide_row) >= 1:
return str(halide_row['IDNUMBER'].values)
else:
return 'Not Exist'
なお、上で定義した関数ではdf_2のSMILES列内の文字列との比較をおこなっていますが、
これは前回の記事で生成したSMILES構造式がcanonicalであることを利用しています。
そうでない題材を使用する際は一旦canonical SMILESの生成をおこなってください。
また、上で定義した関数の3行目がif len(halide_row) == 1ではなく >= 1となっているのは
対応する化合物が複数個ある場合を想定しています(今回の題材では== 1でも結果変わらず)。
定義した関数を用いて化合物の完全一致検索を行います。
df_1['Cl_Product'] = df_1['Chloride'].map(Halide_search)
df_1['Br_Product'] = df_1['Bromide'].map(Halide_search)
df_1['I_Product'] = df_1['Iodide'].map(Halide_search)
# Halide_searchをChloride, Bromide, Iodide列内のMolオブジェクトにそれぞれ適用した。
# 適用結果をそれぞれCl_Product, Br_Product, I_Product列に格納した。
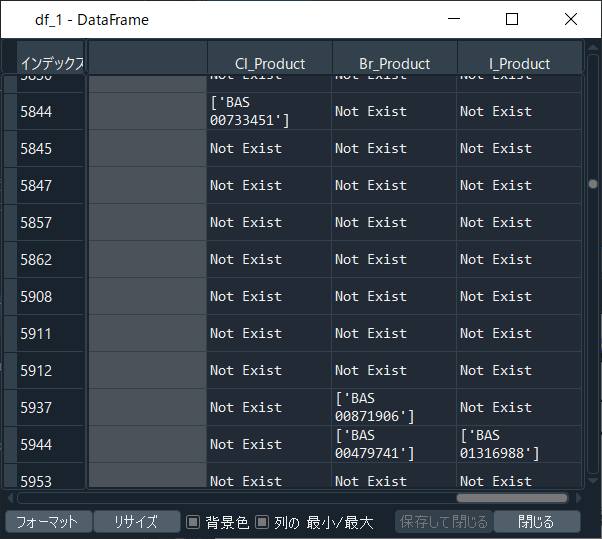
このように検索結果が格納されました。
中身を確認してみると殆どは'Not Exist'であったため、データを整理して見てみましょう。
df_3 = df_1.drop(['SMILES', 'Chloride', 'Bromide', 'Iodide'], axis = 1)
# df_1から上記4つの列を削除したものをdf_3と定義した。
drop_index = df_3.index[(df_3['Cl_Product'] == 'Not Exist') & (df_3['Br_Product'] == 'Not Exist') & (df_3['I_Product'] == 'Not Exist')]
# df_3のうちCl_Product, Br_Product, I_Productいずれの値も'Not Exist'の列の番号をdrop_indexに格納した。
df_4 = df_3.drop(drop_index)
# df_3からdrop_indexに当てはまる行を削除し、df_4と定義した。
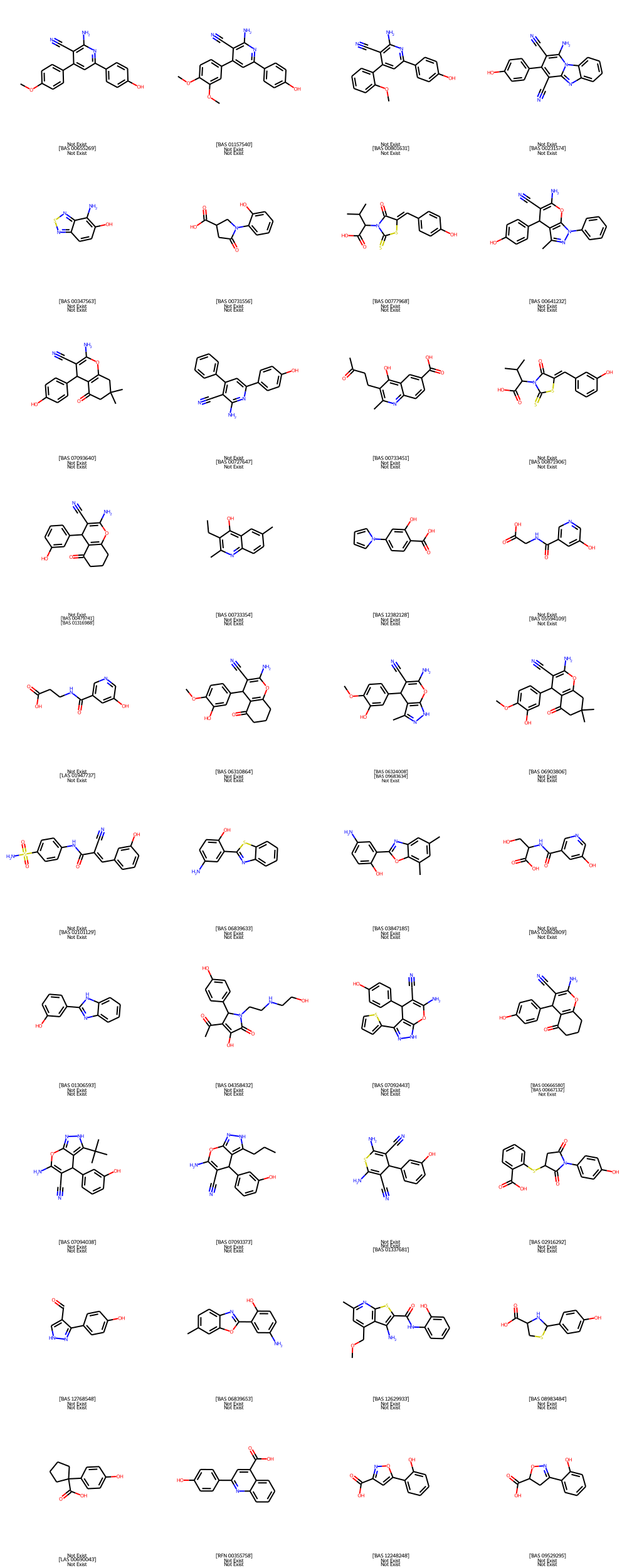
df_4には40個の化合物が残りました。
最後にこの40個の中身と対応する原料のIDNUMBERを描画してみましょう。
Draw.MolsToGridImage(df_4['ROMol'], molsPerRow = 4, legends = [str(x+'\n'+y+'\n'+z) for x,y,z in zip(df_4['Cl_Product'], df_4['Br_Product'], df_4['I_Product'])], subImgSize = (250,250))
# df_4内の分子を描画しつつ、対応する原料のIDを記載した。
おわりに
今回はRDKitを用いて以下の作業を実施いたしました。
- canonical SMILESの比較による完全一致検索