今回はデータフレーム内の分子群に対して化学反応を適用したり、
molオブジェクトの含まれるデータフレームを画像つきのExcelファイルとして出力したりします。
これまでの記事のシリーズは以下をご覧ください。
RDKit入門①:まずは環境構築
RDKit入門②:1分子の読み込みと部分構造検索
RDKit入門③:複数分子の読み込み (前編)と分子群の描画
RDKit入門④:複数分子の読み込み (後編)とデータフレームの加工
RDKit入門⑤:データフレーム内の分子群に対する部分構造検索
RDKit入門⑥:Morganフィンガープリントの作成とそれを用いたタニモト係数の計算による分子類似性評価
RDKit入門⑦:反応式の取り扱い(前編)
RDKitで化学反応を扱った際の生成物群を整理し図示する方法
RDKit入門⑧:反応式の取り扱い(後編)
RDKitで化学反応を繰り返しつつ分子の重複数をカウント・記載する
環境
- Windows 10
- python 3.8.8
- RDKit 2021.03.1
まずは今回の題材となるsdfファイルを入手します。
今回はAsinex社が所有するビルディングブロック22,525化合物のsdfファイルを使用します。
以下のサイトよりzipファイルをダウンロードし、sdfファイルを作業ディレクトリに格納してください。
http://www.asinex.com/?page_id=97
下準備
まずはsdfファイルを読み込み、使用しやすい状態へ加工します。
加工したデータについては次回以降も使いやすいようにpickleファイルとして保存しておきます。
(sdfの読み込み・加工については以前の記事をご覧ください。)
from rdkit import Chem
from rdkit.Chem import AllChem, Draw, PandasTools
# パッケージの読み込み。
df = PandasTools.LoadSDF("2021-02 Asinex BB - 22525.sdf")
df['SMILES'] = df['ROMol'].map(Chem.MolToSmiles)
df = df.drop(['ID', 'ROMol'], axis = 1)
PandasTools.AddMoleculeColumnToFrame(df, smilesCol = 'SMILES')
df.to_pickle("Asinex_Building_Blocks.pkl")
# 2021-02 Asinex BB - 22525.sdfを読み込み、dfと定義した。
# 格納されていた分子からSMILES構造式を作成し、SMILESという名の列に格納した。
# IDの列とROMolの列を削除した。
# SMILES列に格納されたSMILES構造式からMolオブジェクトを作成した。
# dfをAsinex_Building_Blocks.pklという名のpickelファイルで保存した。
一旦ROMolの列を削除してSMILESから再度作り直しているのは見やすさのためなので
この作業をすっ飛ばしても以降の作業に大きな影響はありません。
(座標情報を消すことで構造式の描画がすっきりする)
ビルディングブロックメーカーに勤務していると以下のような気持ちが生まれるかもしれません。
「良い官能基変換反応が見つかったので、社内試薬に対して適用したい!
基質となりうる社内試薬と、想定しうる生成物をそれぞれリストアップしてみよう!」
今回はそういうシチュエーションを仮定し、実際に作業してみます。
題材とする反応はAr-X (X = Cl, Br, I) → Ar-Bpin とします。
まずは基質となる化合物群をピックアップします。
(部分構造検索の方法については以前の記事をご覧ください。)
query = Chem.MolFromSmarts("c[Cl,Br,I]")
# 検索したい構造をSMARTS記法で表し、queryというmolオブジェクトにした。
df_1 = df[df['ROMol'] >= query]
# queryの部分構造を有する化合物をピックアップしたデータフレームをつくり、df_1と定義した。
PandasTools.SaveXlsxFromFrameについて
先の操作でピックアップした2,826個のハロゲン化アリールをExcelファイルに出力します。
PandasTools.SaveXlsxFromFrame(df_1, "aryl_halides.xlsx", size = (200,200))
# df_1をaryl_halides.xlsxという名のExcelファイルとして出力した。
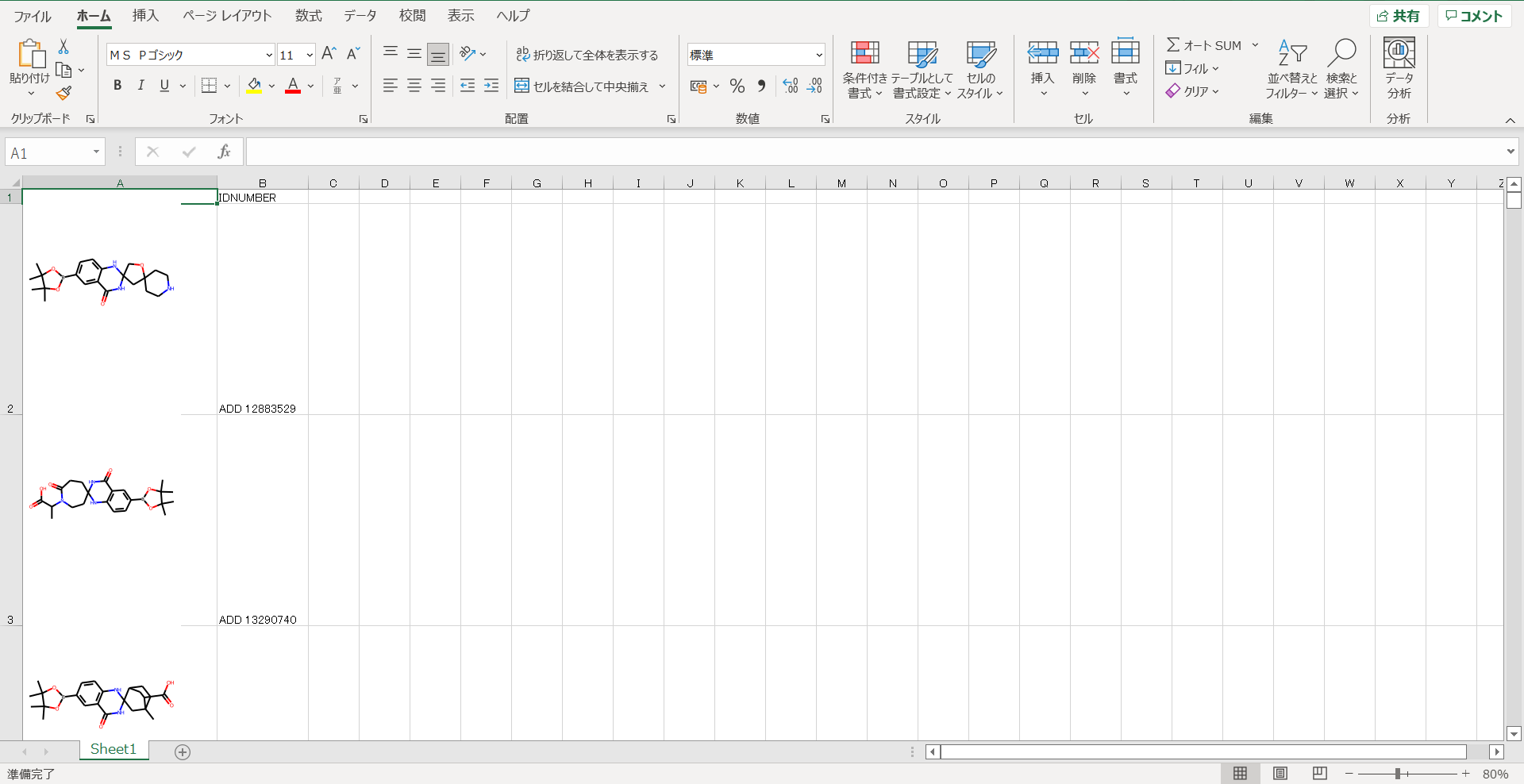
上記の操作で作業ディレクトリに以下のようなExcelファイルが保存されます。

molオブジェクトが画像として埋め込まれたデータフレームがExcel形式で出力されました。
この形式で出力すると非RDKitユーザーにも見やすい形式でデータを渡すことができて便利です。
この関数を使用する際は以下の引数を指定します。
- 出力したいデータフレーム
- 出力するExcelファイルのファイル名
- molオブジェクトが格納された列名(デフォルトではmolCol='ROMol')
- 埋め込む加増のサイズ(デフォルトではsize=(300, 300))
反応の定義と実行、Excelファイル出力
続いてAr-X (X = Cl, Br, I) → Ar-Bpinの反応を定義し、実行します。
なお分子内の反応点は全て変換するように関数を自作します。
(反応の定義については以前の記事をご覧ください。)
rxn = AllChem.ReactionFromSmarts("[c:1][Cl,Br,I:2]>>[c:1]B1OC(C)(C)C(C)(C)O1")
rxn.Initialize()
def Borylation(mol):
while rxn.IsMoleculeReactant(mol) == True:
mol = rxn.RunReactants([mol])[0][0]
mol.UpdatePropertyCache(strict = False)
return mol
# 分子内の全てのハロゲン化アリールをAr-Bpinに変える反応をBorylationと定義した。
定義した反応をデータフレーム内の分子群に対して実行し、新たな列に格納します。
その後データフレームから基質とSMILES列を削除し、別名のデータフレームとして保存します。
df_1["Product"] = df_1['ROMol'].map(Borylation)
# df_1のROMolの列に格納されているMolオブジェクトにBorylationを適用し、Product列に格納。
df_2 = df_1.drop(['SMILES', 'ROMol'], axis = 1)
# df_1からSMILESとROMolの列を削除したものをdf_2と定義した。
新しく作成したデータフレームについてもExcelファイルとして出力してみましょう。
PandasTools.SaveXlsxFromFrame(df_2, "aryl_Bpin.xlsx", molCol = 'Product', size = (200,200))
# df_2をaryl_Bpin.xlsxという名のExcelファイルとして出力した。
おわりに
今回はRDKitを用いて以下の内容を実施いたしました。
- データフレーム内の化合物群に対する化学反応の適用
- PandasTools.SaveXlsxFromFrame関数を用いたデータフレームの出力