この記事は以下の記事の続きとなります。
[RDKit入門②:1分子の読み込みと部分構造検索][1]
[1]:https://qiita.com/Ru-PNP/items/58d733ef327677d273fd
今回はsdfファイルから複数の分子を一度に読み込み、描画を行います。
環境
- windows 10
- python 3.8.5
- RDKit 2020.09.4
まずは今回の題材となるsdfファイルを入手します。
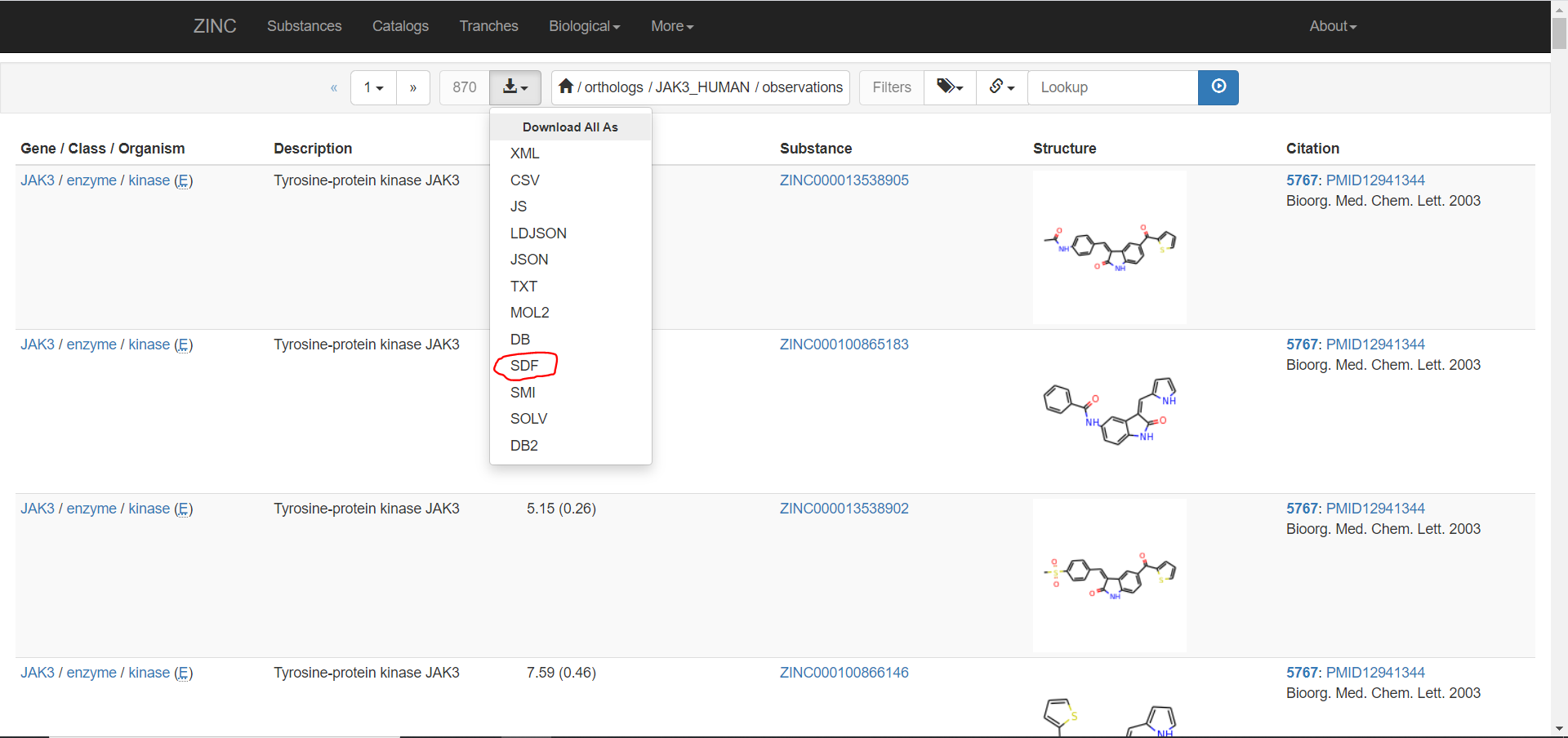
[ZINC15][2]のサイトからヒトJAK3のリガンド一覧のsdfファイルを入手して使用することとします。
[2]:https://zinc15.docking.org/
入手手順:
ZINC15→上部タブからBiologicalを選択→[Major Target Classes][3]を選択
→[Browse][4]を選択→[enzymeの#Sub Classes][5]を選択→[kinaseの#Genes][6]を選択
→[JAK3のOrthologs][7]を選択→[JAK3_HUMANの#Obs.][8]を選択します。
下矢印マークをクリック→SDFを選択し、870分子のSDFファイルをダウンロードしましょう。
[3]:https://zinc15.docking.org/majorclasses/home/
[4]:https://zinc15.docking.org/majorclasses/
[5]:https://zinc15.docking.org/majorclasses/enzyme/sub_classes/
[6]:https://zinc15.docking.org/subclasses/kinase/genes/
[7]:https://zinc15.docking.org/genes/JAK3/orthologs/
[8]:https://zinc15.docking.org/orthologs/JAK3_HUMAN/observations/
ファイル名はJAK3_HUMAN-observations.sdfとしておきます。
ダウンロードしたsdfファイルは作業ディレクトリに保存しておいてください。
sdfファイルの読み込み
sdfファイル読み込みの方法として以下の2つを紹介します。
Chem.SDMolSupplier(path-to-sdf)
PandasTools.LoadSDF(path-to-sdf)
path-to-sdfの部分には読み込みたいsdfファイルの名前を入力します。
今回は前者の使い方を紹介します(後者は次回)。
先に今回使うパッケージを読み込んでおきましょう。
from rdkit import Chem
from rdkit.Chem import Draw
# パッケージの読み込み
Chem.SDMolSupplierについて
今回紹介するChem.SDMolSupplierはsdfファイルを読み込み、中に格納されている分子を
molオブジェクトの集合体として与えるメソッドです。
以下の手順を踏みmolオブジェクトのリストを作成します。
suppl = Chem.SDMolSupplier("JAK3_HUMAN-observations.sdf")
# JAK3のリガンドのsdfファイルを読み込み、supplと定義した。
mols = [x for x in suppl if x is not None]
# suppl内のオブジェクトをリストに格納し、molsと名付けた。
なお、sdfファイルに何らかの不具合があるとRDKitがmolオブジェクトを与えないことがあるため
リストに格納する際にNoneオブジェクトを除外するようにしておきます。
読み込んだ後は念のために読み込んだ分子の数を数えておきましょう。
print(len(mols))
# 870
今回は特にエラーなく870分子全てを読み込むことができました。
なお、今回の方法でsdfファイルを読み込む場合、標準では水素原子は自動的に除外されます。
指定しない場合はremoveHs = Trueが自動的に設定されているためです。
もし水素原子も読み込んでおきたい場合は以下のようにremoveHs = Falseを指定しましょう。
suppl_Hs = Chem.SDMolSupplier("JAK3_HUMAN-observations.sdf", removeHs = False)
# 水素原子を除去せずにJAK3リガンドのsdfファイルを読み込み、suppl_Hsと定義した。
mols_Hs = [x for x in suppl_Hs if x is not None]
## suppl_Hs内のオブジェクトをリストに格納し、mols_Hsと名付けた。
print(len(mols_Hs))
# 870
なお、sdfファイルには分子のプロパティが載っていますが、
今回読み込んだmolオブジェクトにはsdfファイルに入っていたプロパティが格納されています。
Mol.GetPropNames()でどのようなプロパティが設定されるか取得することが可能です。
またMol.GetProp(Names)で実際のプロパティの値を取得することができます。
試しにmolsの1番目の分子について情報を取得してみましょう。
for prop in mols[0].GetPropNames():
print('{}: {}'.format(prop, mols[0].GetProp(prop)))
取得結果は以下の通りです。
zinc_id: ZINC000000009851
ortholog_name: JAK3_HUMAN
gene_name: JAK3
affinity: 5.04
chembldocid: 81718
title: None
reference.pubmed_id: None
reference.doi: 10.1039/C0MD00115E
reference.chembl_id: CHEMBL3217494
reference.journal: MedChemComm
reference.year: 2010
分子群の描画
[前回の記事][1]で1分子の画像を描画する方法について少しだけ触れました。
今回の記事では複数分子を一度に表示する方法について簡単に紹介します。
Draw.MolsToGridImageについて
複数の分子の画像を表示するにはDraw.MolsToGridImageメソッドを使用します。
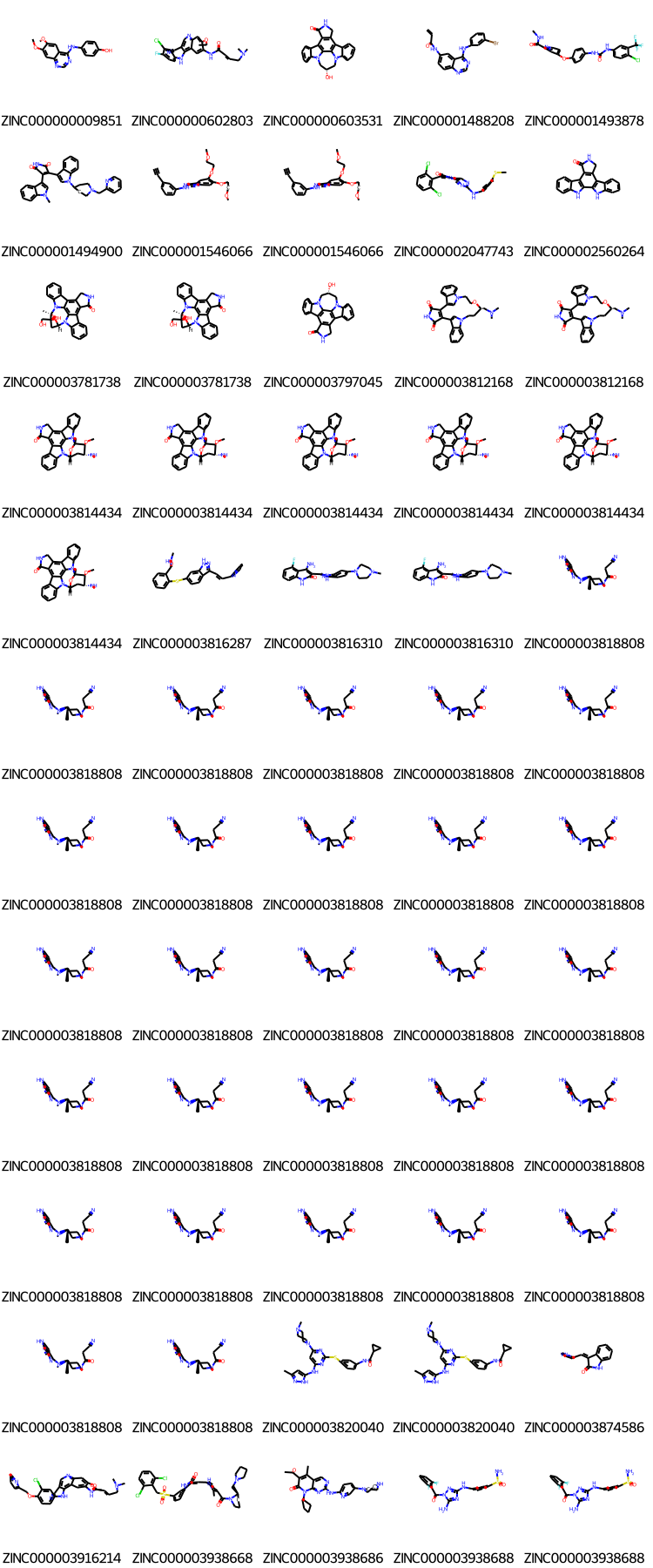
Draw.MolsToGridImage(mols[:60], molsPerRow = 5, subImgSize = (150,150),
legends = [mol.GetProp("zinc_id") for mol in mols], maxMols = 60)
# molsのうち最初の60分子を描画した。
# 1行あたりに表示する分子数をmolsPerRowで指定した(今回は5個とした)
# 画像の大きさをsubImgSizeで指定した(今回は150×150とした)
# 分子のラベルをlegendsで指定した(今回はzinc_idのリストを取得して指定した)
# 表示する最大分子数をmaxMolsで指定した(今回は60分子とした)
なお、Draw.MolsToGridImageで最低限指定しなくてはならないのは分子のリストのみです。
その他のパラメーターについては指定しない場合デフォルトで指定された値が適用されます。
以下、デフォルトで指定されている値の一部を紹介します。
- molsPerRow = 3 (1行あたりに表示する分子は3分子)
- subImgSize = (200,200) (画像の大きさは200×200)
- legends = None (分子にラベルはつけない)
- maxMols = 50 (最大で50分子まで表示)
一番最後のmaxMolsについて、デフォルトだと51分子目移行はカットされてしまいます。
それ以上の分子数を一度に表示したい場合はmaxMolsの値を十分大きな値に設定してください。
忘れがちな割には重要なパラメーターだと思いますので注意しましょう。
おわりに
今回はRDKitを用いて以下の内容を実施いたしました。
- sdfファイルの読み込みとmolオブジェクトの取り出し
- 取り出したmolオブジェクトからのプロパティの取得
- 複数のmolオブジェクトの画像をまとめて描画
次回はPandasToolsを用いたsdfファイルの読み込みと加工について紹介する予定です。
参考
今回の記事を書くにあたっては化学の新しいカタチさんの以下の記事を参考にいたしました。
また、関連する内容として以下の記事を紹介いたします。