BigQueryのサンプルデータにCOVID-19のオープンデータがありましたので、このデータでBigQueryとデータ可視化ツールMetabaseを接続してみました。
前回の記事
Google BigQueryとGoogleデータポータル(Data Studio)を使い始めてCOVID-19感染者数のグラフを作るまで
データ準備
前回の記事のとおり、BigQueryのサンプルデータセットにCOVID-19の感染者数のデータがあります。
そのデータを自分のGCPプロジェクトにコピーします。
以下のコマンドを実行するとコピーできます。サンプルデータの covid19_open_data というデータセットの covid19_open_data というテーブルを自分の mydataset というデータセットに同じ名前のテーブルとしてコピーします。
$ bq mk mydataset
$ bq cp bigquery-public-data:covid19_open_data.covid19_open_data mydataset.covid19_open_data
BigQueryの認証準備
MetabaseをAPIでBigQueryに接続するための認証の準備をします。
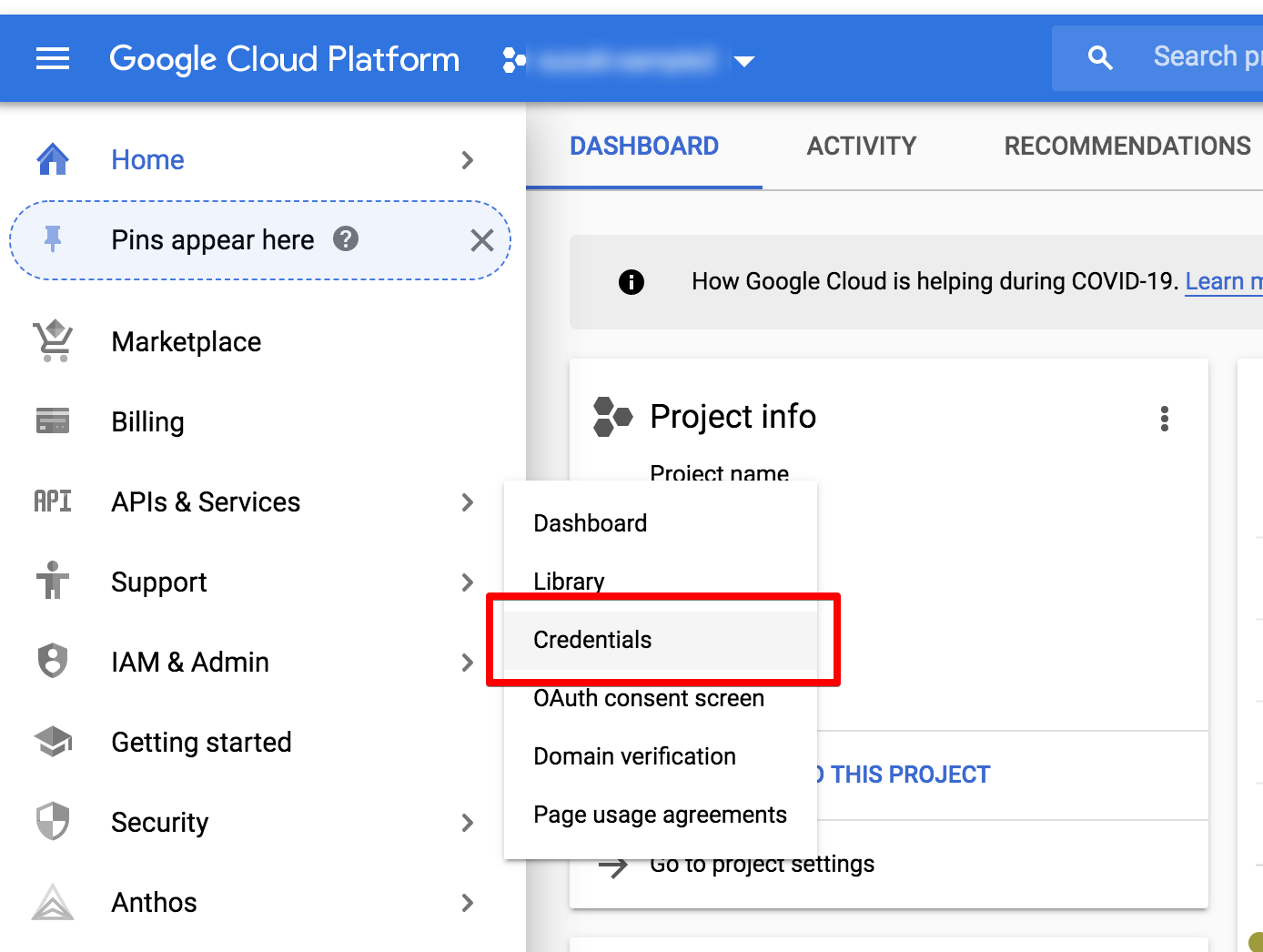
GCPの左のメニューの「APIs & Services」→「Credentials」をたどります。
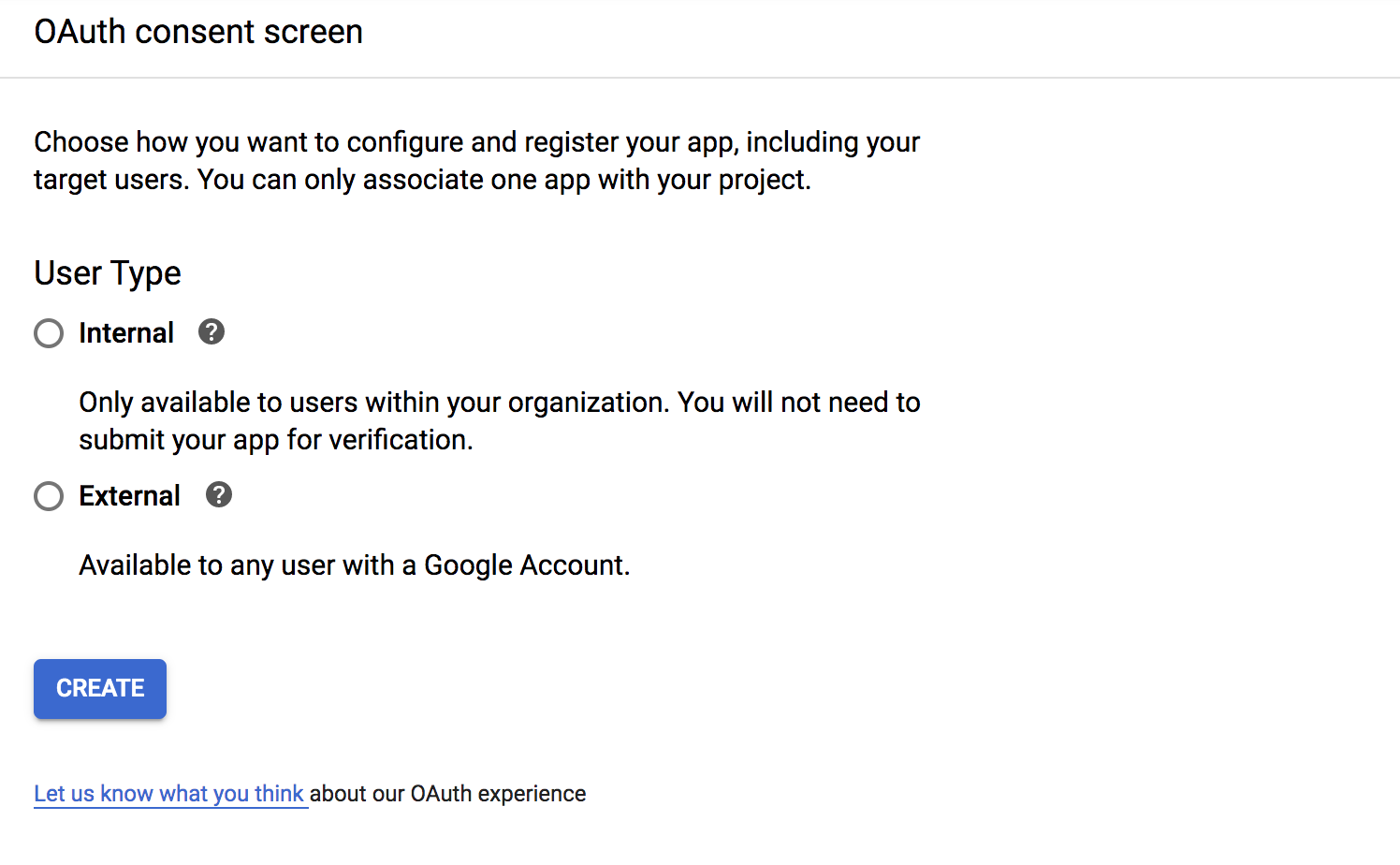
次のような画面になります。初めてBigQueryにAPI接続する場合には同意画面を設定(configure consent screen)するように求められます。
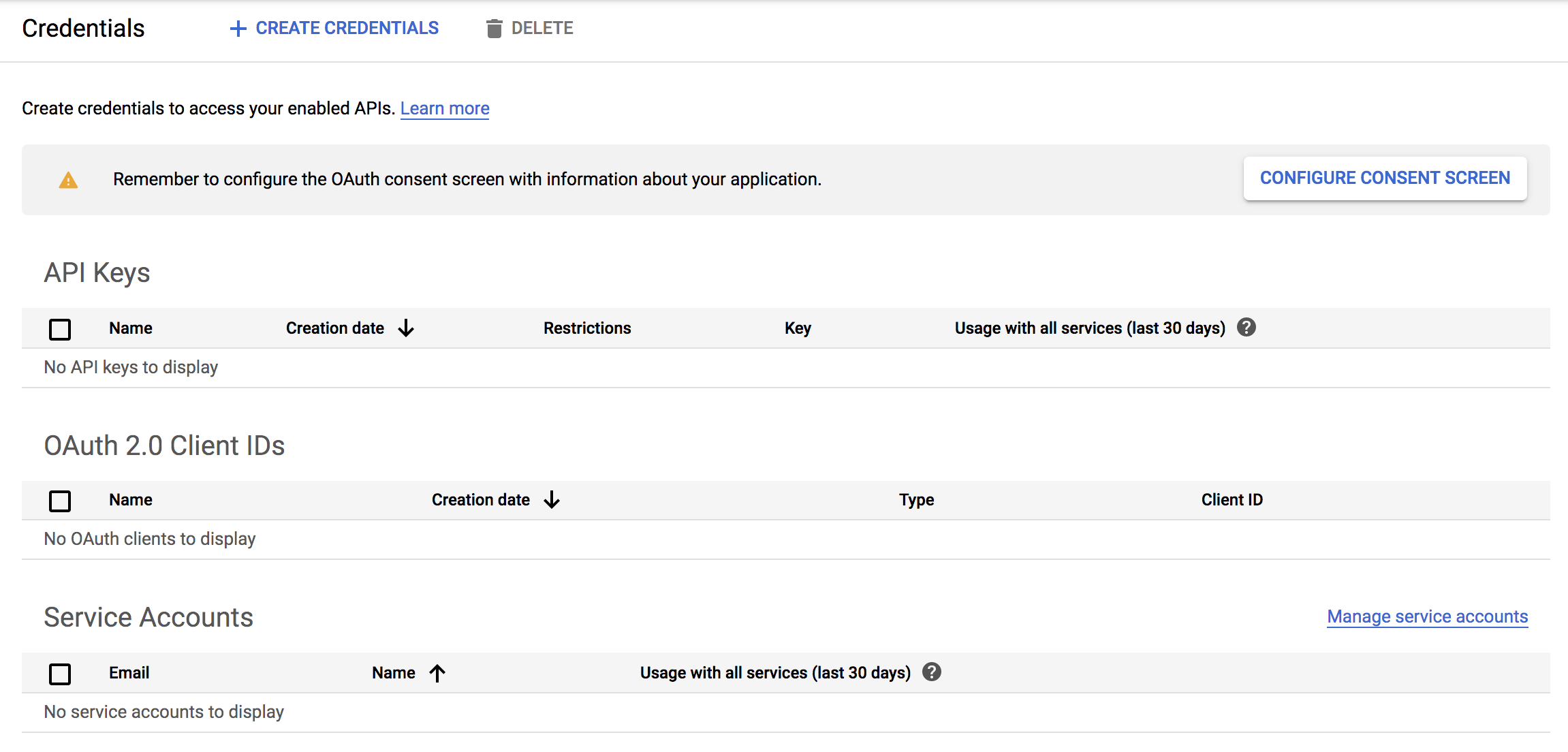
「CONFIGURE CONSENT SCREEN」をクリックすると設定画面になります。
Externalはアプリ配布などでだれでもアクセスできるようにするものです、たぶん。関係者だけで使うのであれば、Internalを選択し、「CREATE」をクリックします。
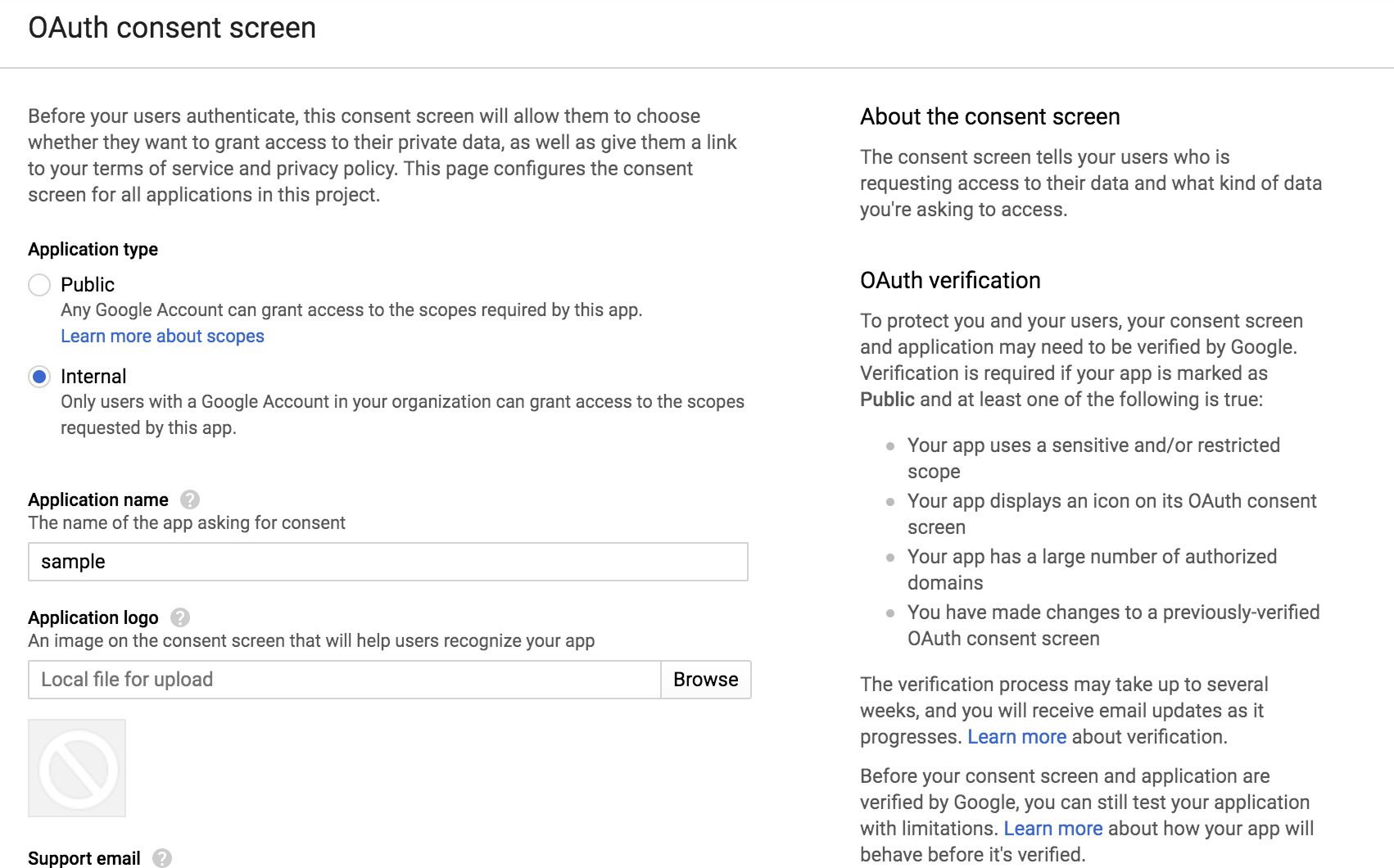
次のようなごちゃごちゃした設定画面になります。この設定画面はGCPのプロジェクトごとに設定するものです。
Application typeはInternalを選択します。Application nameはこのあとMetabaseと接続するときのGoogleアカウントでの接続承認画面に表示されます。とりあえずなんでもいいと思います。この他にメールアドレスとかいろいろ設定項目がありますが、とりあえずデフォルトのままにしておきました。
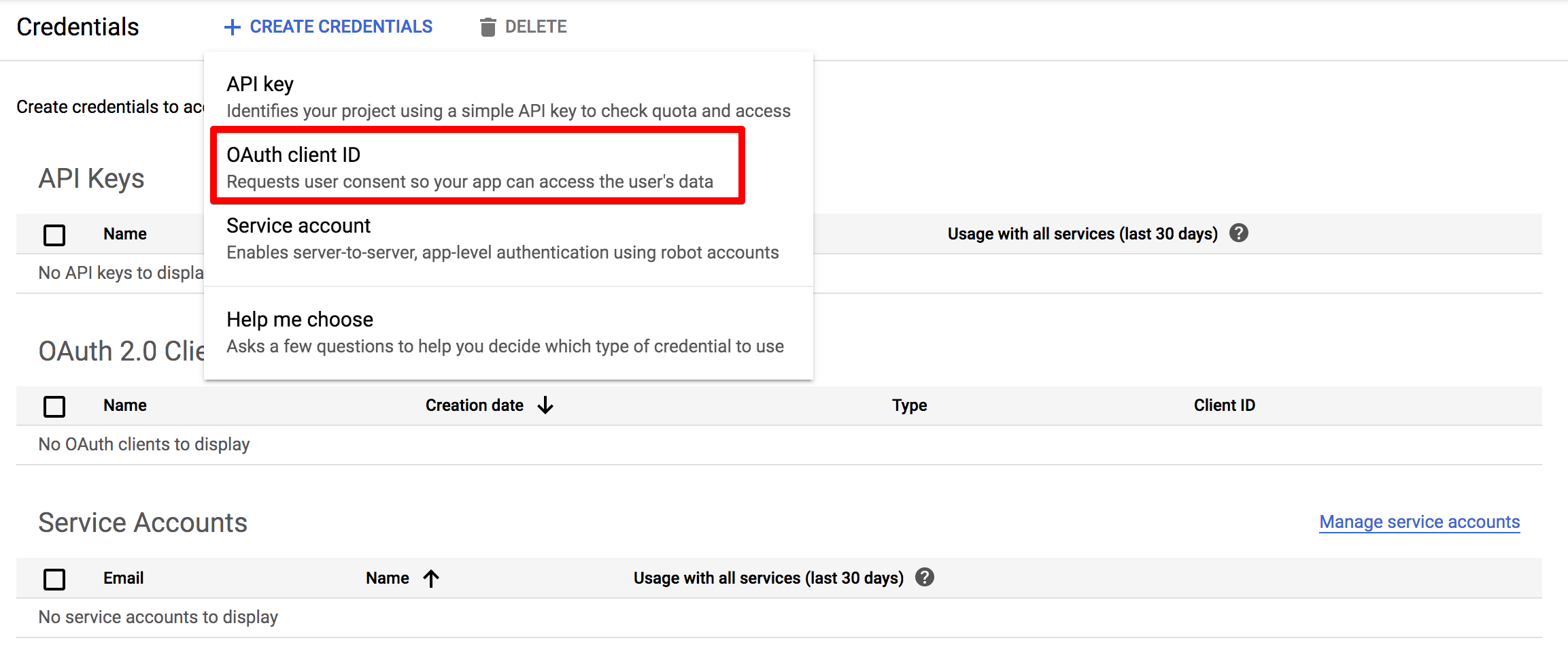
一番下の「SAVE」をクリックすると、この画面に戻ります。上の「CREATE CREDENTIALS」をクリックすると「OAuth client ID」作成のメニューがあります。
「OAuth client ID」をクリックすると、次の画面になります。
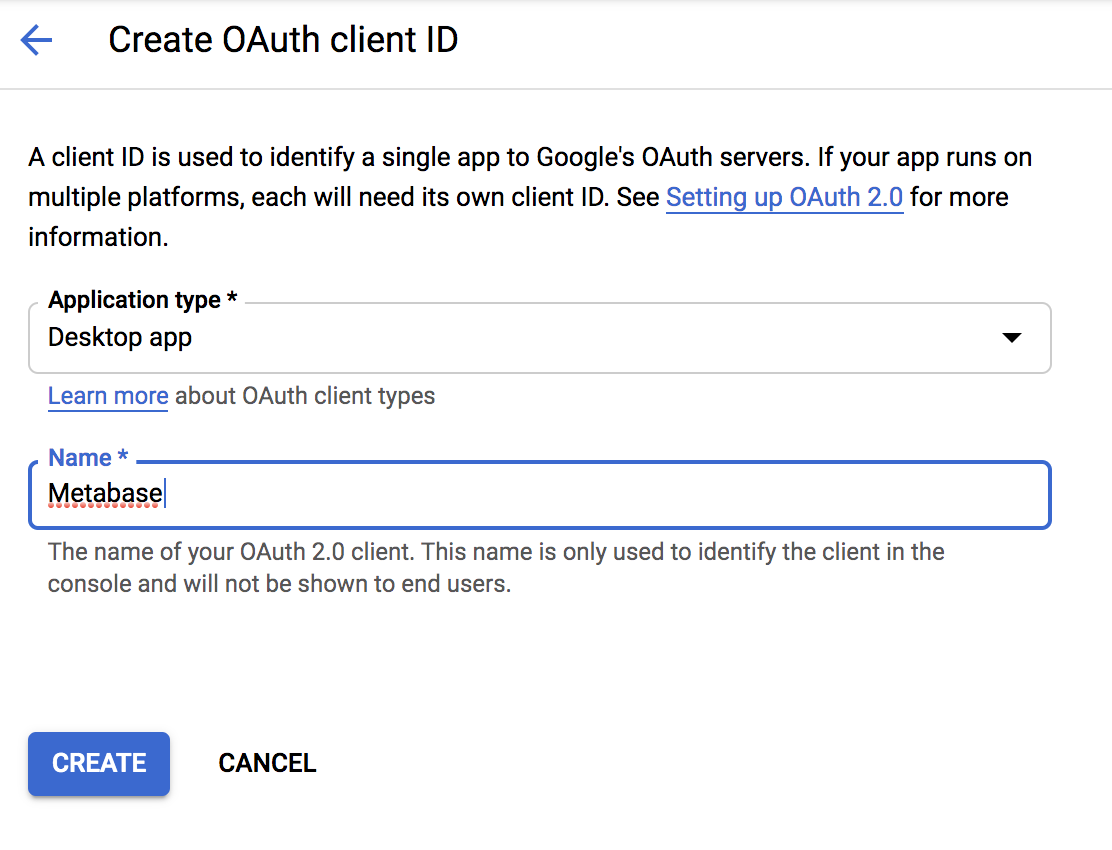
Application typeは以下の選択肢がありました。「Desktop app」を選択します。
- Web application
- Android
- Chrome app
- iOS
- TVs and Limited Input devices
- Desktop app
- Universal Windows Platform (UWP)
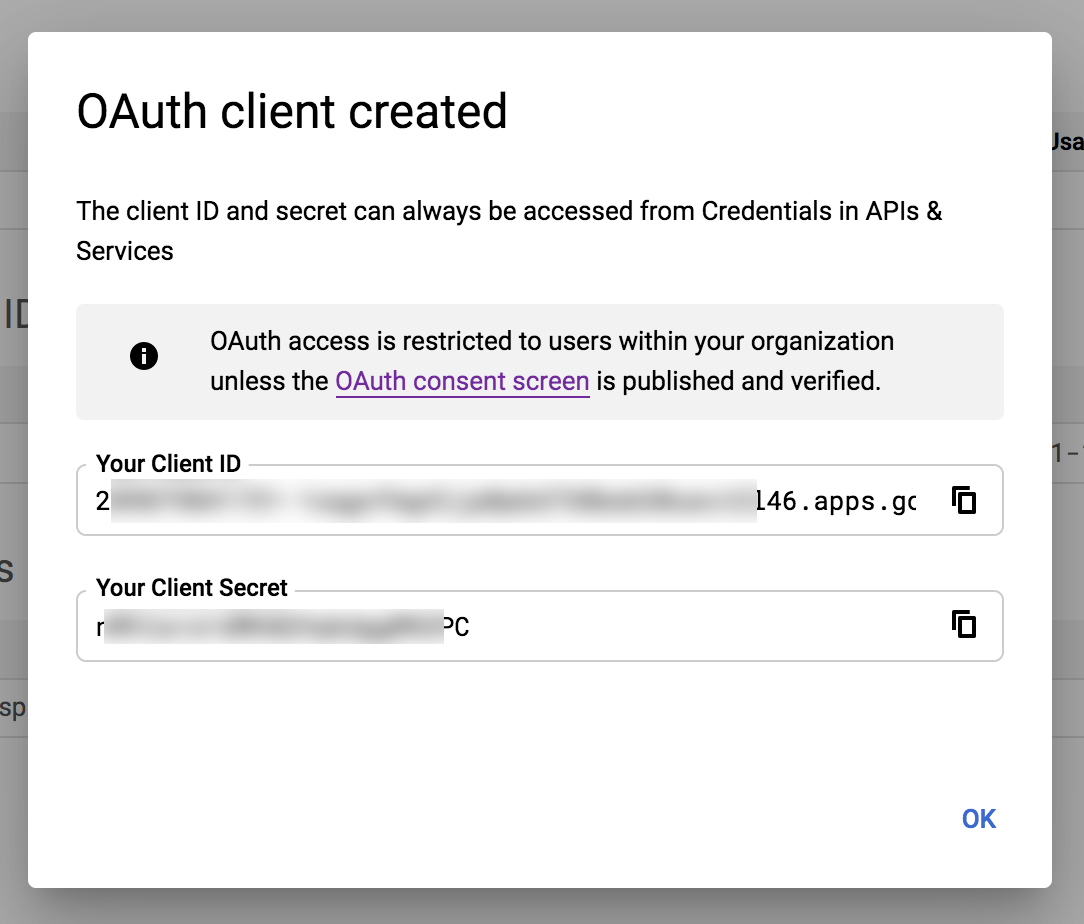
Nameも適当に入力して「CREATE」をクリックします。すると次の画面になります。
このClient IDとClient SecretというのがこのあとMetabaseからの接続の際に必要になってきます。この画面を閉じてしまっても、Credentialsのページに来ればOAuth 2.0 Client IDsの表示から確認することができます。
Metabaseからの接続の際にはProject IDというものも必要ですが、GCPのコンソールのダッシュボードからも確認できますし、コンソールのURLのところにも書かれています。 https://console.cloud.google.com/apis/credentials?project=XXXX のXXXXの部分です。
Metabaseインストール
GCPのCompute Engineのインスタンス上にMetabaseをインストールします。インスタンスのマシンタイプは最小のf1-microだとメモリが少なくてMetabaseには辛いです。g1-smallで試しました。
先にJavaをなんらかの方法でインストールします。ここではもうJavaはインストール済みとします。
$ java -version
openjdk version "13.0.1" 2019-10-15
OpenJDK Runtime Environment (build 13.0.1+9)
OpenJDK 64-Bit Server VM (build 13.0.1+9, mixed mode, sharing)
Metabaseのjarファイルをダウンロード。
$ wget https://downloads.metabase.com/v0.35.4/metabase.jar
Metabase実行。
$ java -jar metabase.jar
起動時のログが流れます。
ログが落ち着いてから3000番ポートにブラウザでアクセスすると、Welcomeの画面になります。
「Let's get started」をクリックすると、最初のID/PW登録と接続するデータベースの設定をする画面になります。
MetabaseをBigQueryに接続
「Let's get started」の画面でMetabaseにログインするID/PWを登録します。ここで登録する内容は、今回のMetabase起動方法ですと起動したカレントディレクトリにファイルで保存されるのみです。自分でちょっと試すだけであればメールアドレスも名前も会社名もなんでもいいです。
「Next」をクリックすると、接続するデータベースの設定画面になります。
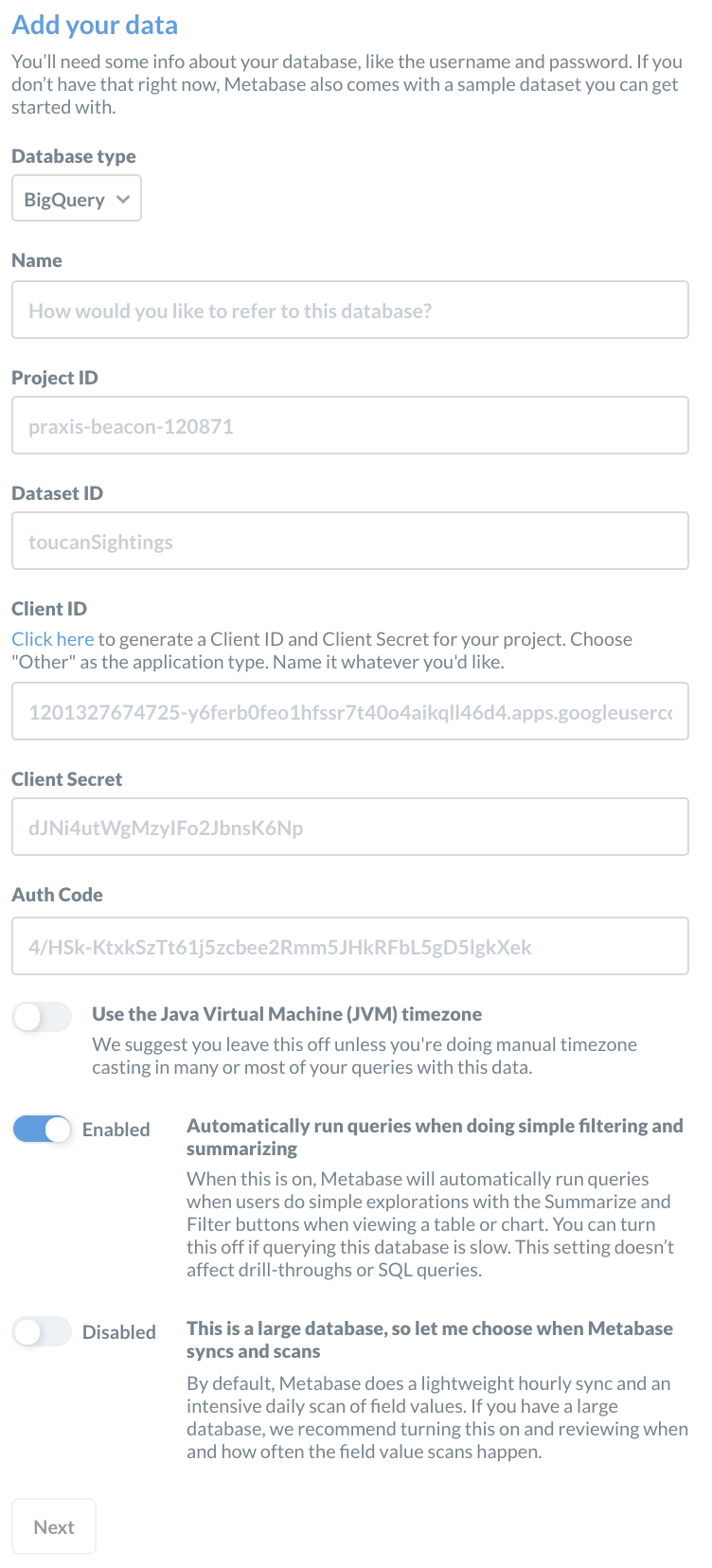
ここでBigQueryを選択します。
- Name: Metabaseの中で接続するデータベースを区別するための名前です。Metabaseは複数のデータベースに接続できますので、その場合は区別できるような適当な名前を付けます。
- Project ID: GCPのProject ID。
- Dataset ID: BigQueryで作成したデータセットの名前です。最初のコマンドの例では
mydatasetです。 - Client ID: GCPの画面で作成したOAuth clientのClient ID。
- Client Secret: GCPの画面で作成したOAuth clientのClient Secret。
- Auth Code: 承認画面のあとに出てくるコード。
Auth Codeの入力欄は、Client ID、Client Secretまでを入力すると次のような「Click here」というリンクが現れます。
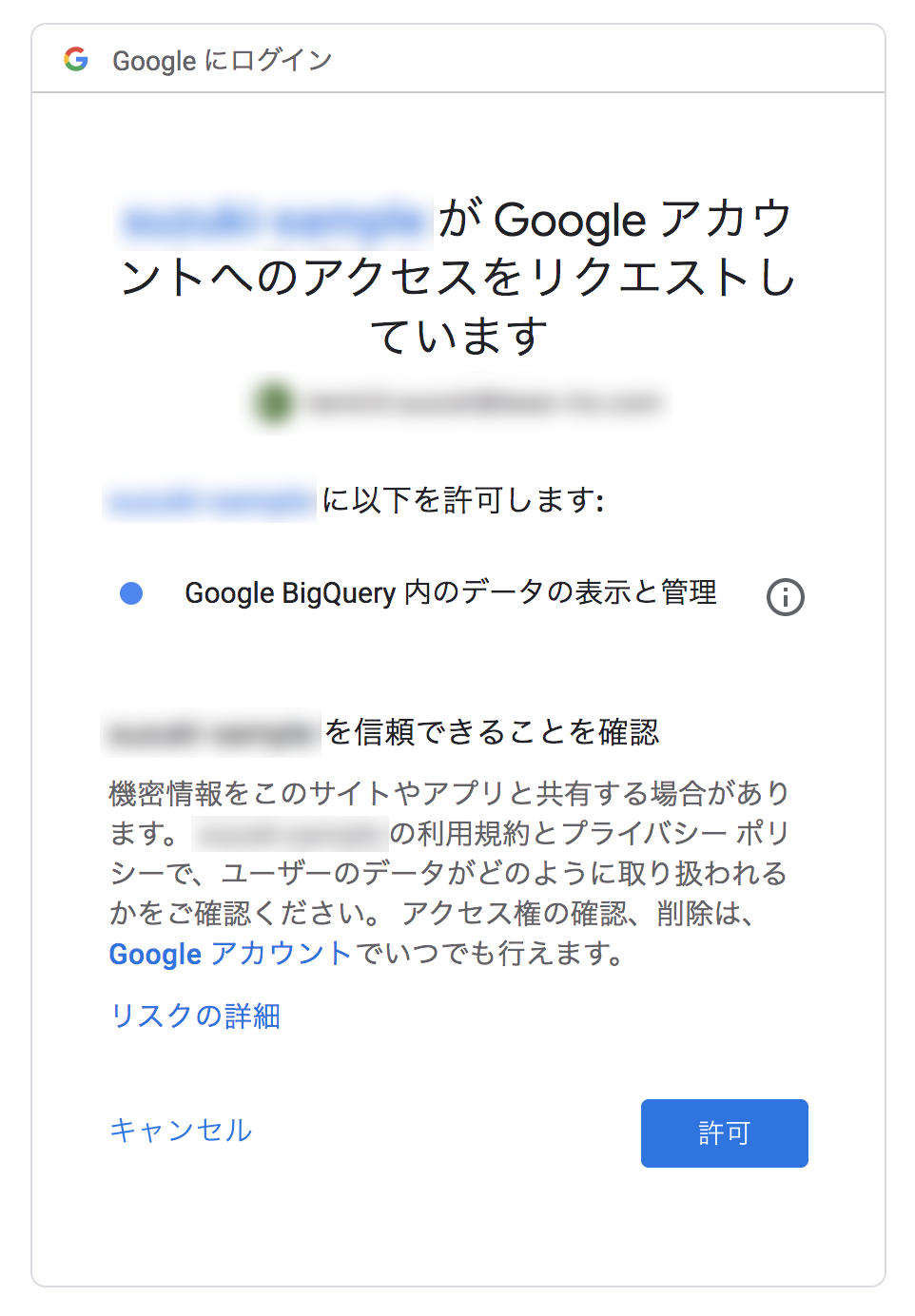
このリンクをGCPにログインしているブラウザで開くと、次のような承認を求める画面になります。
「XXXXが Google アカウントへのアクセスをリクエストしています」の「XXXX」の部分には、最初にGCPのプロジェクトのごちゃごちゃした画面でApplication nameとして設定した名前が入ってきます。
このコードをさきほどのAuth Codeの欄に貼り付けます。
あとはMetabaseの画面に従って「Next」を押していくと、Metabaseのトップページになります。
次のようにCOVID-19のテーブルが見えるはずですが、接続設定直後は見えないかもしれません。少ししてからリロードすれば現れます。
このスクリーンショットでの下の方の「BigQuery Sample」と書かれている箇所は、BigQuery接続設定を入力したときのNameに入力した名前が入ります。
「BigQuery Sample」→「Cov I D19 Open Data」(テーブル名が変)とクリックするとテーブルの中身がそのままテーブルで表示されます。
グラフ作成
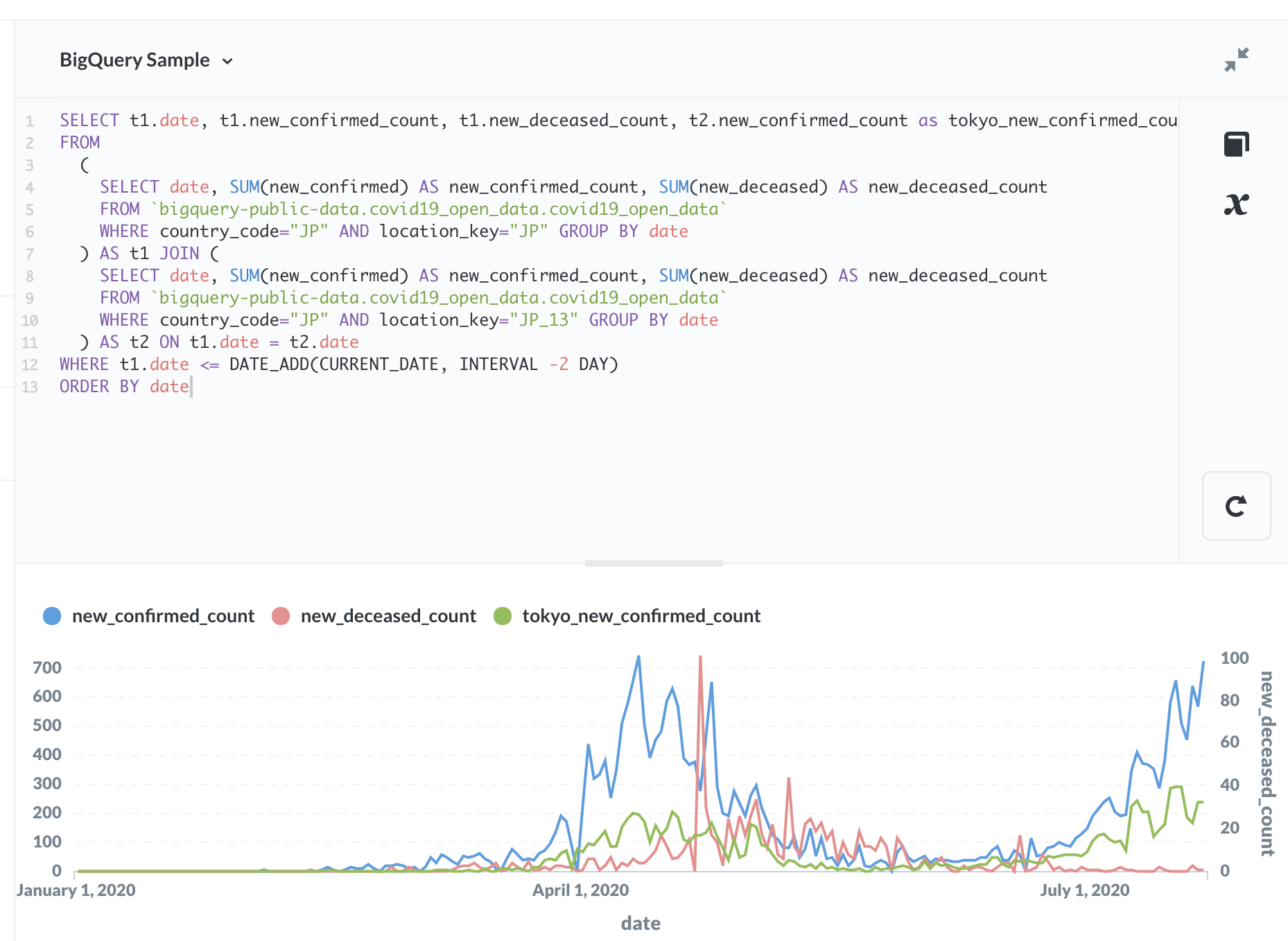
右上の「Ask a question」をクリックして、「Native query」をクリックして、「Select a database」で今回接続したデータベース(BigQuery Sample)を選択し、SQLを入力します。次のようなSQLを入れます。
SELECT t1.date, t1.new_confirmed_count, t1.new_deceased_count, t2.new_confirmed_count as tokyo_new_confirmed_count
FROM
(
SELECT date, SUM(new_confirmed) AS new_confirmed_count, SUM(new_deceased) AS new_deceased_count
FROM `bigquery-public-data.covid19_open_data.covid19_open_data`
WHERE country_code="JP" AND location_key="JP" GROUP BY date
) AS t1 JOIN (
SELECT date, SUM(new_confirmed) AS new_confirmed_count, SUM(new_deceased) AS new_deceased_count
FROM `bigquery-public-data.covid19_open_data.covid19_open_data`
WHERE country_code="JP" AND location_key="JP_13" GROUP BY date
) AS t2 ON t1.date = t2.date
WHERE t1.date <= DATE_ADD(CURRENT_DATE, INTERVAL -2 DAY)
ORDER BY date
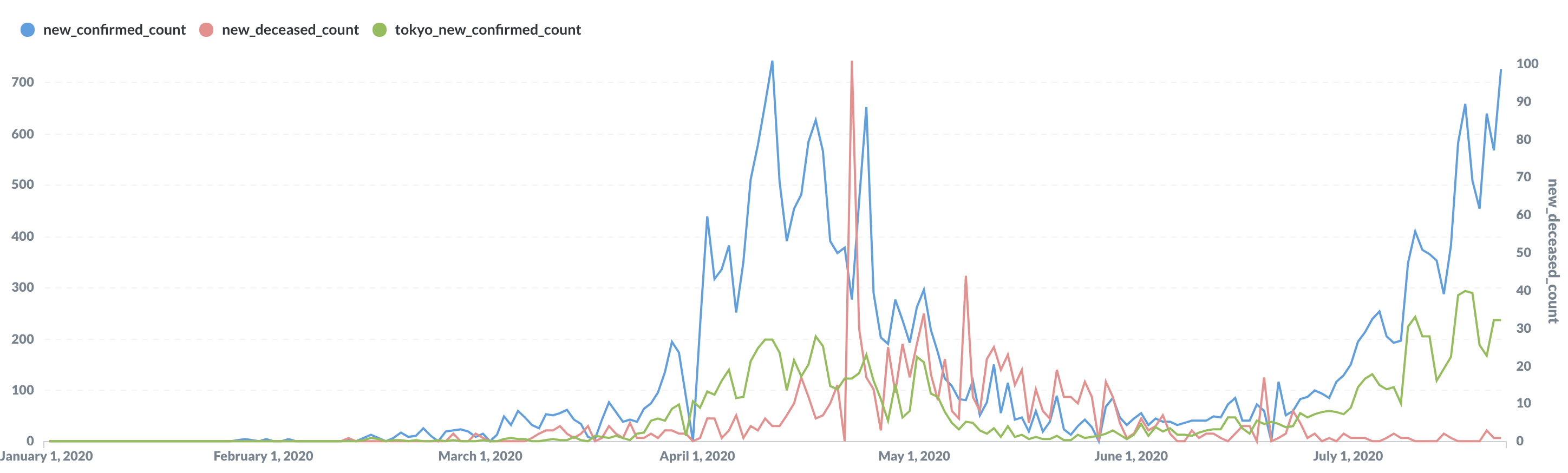
右下の矢印の実行ボタンをクリックして、Visualizationの設定をいろいろいじると、次のようなグラフを作れました。
- 青: 日本の新規感染者数
- 緑: 東京都の新規感染者数
- 赤: 日本の死者数
7月以降の流行は4月〜5月の流行に比べて死者数が少いまま推移しており、状況がなにか大きく違っていそうです。
リンク
私のMetabaseに関する記事
- オープンソースのデータ可視化ツールMetabaseのご紹介 (2020/07/16)
- MetabaseのQuestionの作成・保存方法 (2020/05/29)
- MetabaseにあるQuestionの可視化種類のまとめ (2020/05/23)