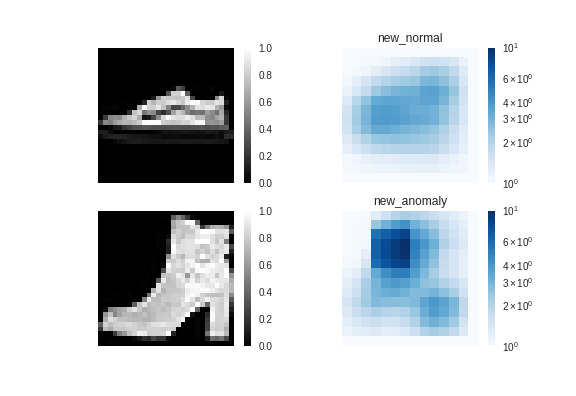
前編では、ヒートマップを使って異常個所を可視化しました。
後編では、従来手法と提案手法を数値的に比較してみます。
使うツールは、ROC曲線です。
ROC曲線
ROC曲線は、ラベル間のデータ数に差があるときに使われます。
ROC曲線について、詳しく知りたい方はこちら↓を参考にしてください。
http://www.randpy.tokyo/entry/roc_auc
今回は、ラベル間のデータ数に大きな差はありませんが、論文にならって
ROC曲線を描画します。最終的に、AUCを算出して、これが大きい方が優れた
異常検知器といえます。
コードの解説
コードの一部を解説します。
異常スコアの算出
論文によると、異常判定の基準は以下のとおりです。
このパッチ全てに関して異常度を算出し、少なくとも1枚が閾値を
超えてる場合、テストデータは異常であると判断した.
パッチというのは、前編でお伝えした小窓に相当します。
前編では、下記のヒートマップを作成するにあたり、元の画像(サイズ28×28)に
対し、小窓(サイズ8×8)を上下左右に走らせました。
このとき、1枚の画像から小窓のスコアは21×21=441個出てきます。
論文に従うと、441個の小窓のスコアの中から、その最大値(スコアが高いほど異常度が
高いです。)をその画像の異常スコアとします。
そして、閾値を決め、閾値より異常スコアが高いものを「異常画像」、
低いものを「正常画像」と判定します。
まとめると、以下のとおりです。
1枚の画像 → 小窓を走らせる → 441個のスコア → スコアの最大値 = 異常スコア → 閾値と比較
pythonのコードは以下のとおりです。関数には、テストデータを渡して、
小窓を走らせながら、画像1枚1枚に異常スコアを付けています。
nameの名前で、従来手法と提案手法を切り替えて評価しています。
# 最大異常値の計算
def result_score(model, x, name, height=8, width=8, move=2):
score = []
for k in range(len(x)):
max_score = -1000000000
if k%100 == 0:
print(k)
for i in range(int((x.shape[1]-height)/move)):
for j in range(int((x.shape[2]-width)/move)):
x_sub = x[k, i*move:i*move+height, j*move:j*move+width, 0]
x_sub = x_sub.reshape(1, height, width, 1)
#従来手法
if name == "old_":
#スコア
temp_score = model.evaluate(x_sub, batch_size=1, verbose=0)
if temp_score > max_score:
max_score = temp_score
#提案手法
else:
#スコア
mu, sigma = model.predict(x_sub, batch_size=1, verbose=0)
loss = 0
for o in range(height):
for l in range(width):
loss += 0.5 * (x_sub[0,o,l,0] - mu[0,o,l,0])**2 / sigma[0,o,l,0]
if loss > max_score:
max_score = loss
score.append(max_score)
return(score)
ROC曲線の描画
ROC曲線の描画は、以下の記事を参考にしました。
https://qiita.com/9pid/items/53946c3ec4b2489e7cb2
scikit-learnを使えば一発です。
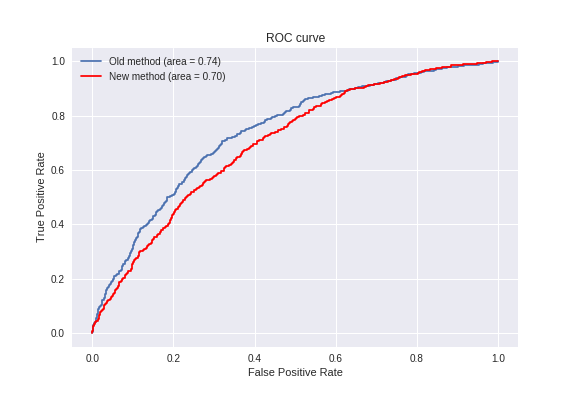
MNISTを使った結果
※11/21修正 コードにバグがあった関係で、図と文章を全面的に修正しました。
Colaboratoryの場合、計算時間は学習で10分、テストデータの評価で2時間ほど
かかります。
グラフの中のareaがAUCに相当します。そして、AUCが高いほうが優秀な異常検知器です。
予想に反して、従来手法の方が良い結果となりました。
提案手法の精度が落ちた理由は、後で考察します。
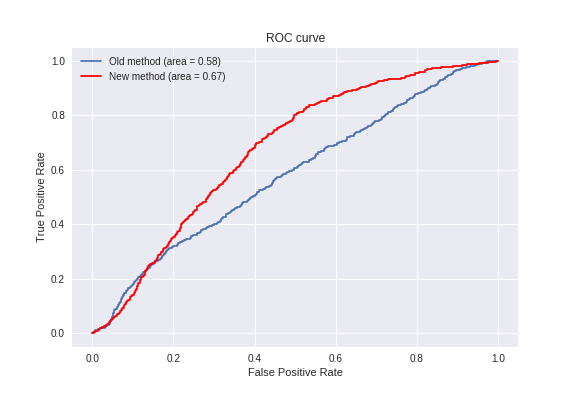
Fashion-MNISTを使った結果
※11/21修正 コードにバグがあった関係で、図と文章を全面的に修正しました。
計算時間はMNISTと同じくらいでした。
こちらはMNISTと逆で、提案手法の方が良い結果となりました。
この理由も後で考察します。
考察 11/21追記
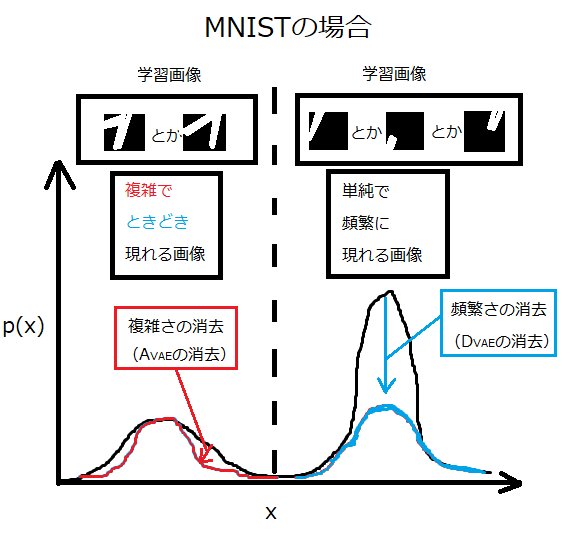
MNISTとFashion-MNISTで結果が変わった理由を考察してみると、
MNISTでは以下の図のような学習結果になっていると思われます。
そして、テストの際に1と9を入力すると、直線部分の画像は右の山に
位置してきます。一方、異常判別の材料となる画像(1の折れ曲がり部や
9の曲線部分の画像)は左の山に位置してきます。そして、1の折れ曲
がり部は左の山の「中央」付近に、9の曲線部分は左の山の「端」に
位置することになり、異常判別ができます。
ここで重要なのは、右の山は無視して良いということです。つまり、
異常か正常かを判断する材料は左の山だけで十分です。
ところが、提案手法では右の山を下げて、左の山と同じ土俵で勝負させます。
よって、提案手法では、本来無視してよい右の山(直線部分の画像)も判別し
異常と認識してしまっている可能性が高く、精度を下げる一因になっていると
思われます。
一方、Fashion-MNISTはブーツの かかと は複雑なため左の山に、
ブーツの口は単純なため右の山に属していると思われます。従って、
どちらの山も判断材料になるため、左右の山を同じ土俵で勝負させる
提案手法の方が精度が出たと思われます。
まとめますと、「複雑な」画像にしか異常が出ない場合は、従来手法の
方が優れていると思われます。工業製品のように「単純な」ところにも
「複雑な」ところにも異常が出る場合は、提案手法の方が優れていると
思われます。
ちなみに、Pythonでコードを組む場合、計算速度については、従来手法の方が
速く(model.evaluate()を使っているため)現場向きかと思われます。
一方、提案手法はfor文を駆使しているため、速度が遅くなります。
まとめ
※11/21修正 コードにバグがあった関係で、文章を修正しました。
前編と後編に分けて、VAEによる画像の異常検知を実装してきました。
見てお分かりのとおり、提案手法は視覚的にも能力的にも優れていること
が分かりました。
能力の面では、考察に示したとおり、異常検知の対象となる画像の性質を
考えて使い分けした方が良さそうです。
余裕があったら、本手法を使って画像以外の異常検知もやってみたいと思います。
2018/1/8追記
本手法より精度が良い論文の記事を書きました。
https://qiita.com/shinmura0/items/cfb51f66b2d172f2403b
コード全文
9/15 追記:コードにバグがありました。お詫びを申し上げ、修正させていただきます。
12/18 コード修正 (@gungiven さんご指摘ありがとうございます。)
from __future__ import absolute_import
from __future__ import division
from __future__ import print_function
from keras.layers import Lambda, Input, Dense, Reshape
from keras.models import Model
from keras.datasets import mnist
from keras.datasets import fashion_mnist
from keras.losses import mse
from keras.utils import plot_model
from keras import backend as K
from keras.layers import BatchNormalization, Activation, Flatten
from keras.layers.convolutional import Conv2DTranspose, Conv2D
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.colors as colors
import os
from sklearn import metrics
# 最大異常値の計算
def result_score(model, x, name, height=8, width=8, move=2):
score = []
for k in range(len(x)):
max_score = -1000000000
if k%100 == 0:
print(k)
for i in range(int((x.shape[1]-height)/move)+1):
for j in range(int((x.shape[2]-width)/move)+1):
x_sub = x[k, i*move:i*move+height, j*move:j*move+width, 0]
x_sub = x_sub.reshape(1, height, width, 1)
#従来手法
if name == "old_":
#スコア
temp_score = model.evaluate(x_sub, batch_size=1, verbose=0)
if temp_score > max_score:
max_score = temp_score
#提案手法
else:
#スコア
mu, sigma = model.predict(x_sub, batch_size=1, verbose=0)
loss = 0
for o in range(height):
for l in range(width):
loss += 0.5 * (x_sub[0,o,l,0] - mu[0,o,l,0])**2 / sigma[0,o,l,0]
if loss > max_score:
max_score = loss
score.append(max_score)
return(score)
# 8×8のサイズに切り出す
def cut_img(x, number, height=8, width=8):
print("cutting images ...")
x_out = []
x_shape = x.shape
for i in range(number):
shape_0 = np.random.randint(0,x_shape[0])
shape_1 = np.random.randint(0,x_shape[1]-height)
shape_2 = np.random.randint(0,x_shape[2]-width)
temp = x[shape_0, shape_1:shape_1+height, shape_2:shape_2+width, 0]
x_out.append(temp.reshape((height, width, x_shape[3])))
print("Complete.")
x_out = np.array(x_out)
return x_out
# reparameterization trick
# instead of sampling from Q(z|X), sample eps = N(0,I)
# z = z_mean + sqrt(var)*eps
def sampling(args):
z_mean, z_log_var = args
batch = K.shape(z_mean)[0]
dim = K.int_shape(z_mean)[1]
# by default, random_normal has mean=0 and std=1.0
epsilon = K.random_normal(shape=(batch, dim))
return z_mean + K.exp(0.5 * z_log_var) * epsilon
# dataset
(x_train, y_train), (x_test, y_test) = mnist.load_data()
# (x_train, y_train), (x_test, y_test) = fashion_mnist.load_data()
x_train = x_train.reshape(x_train.shape[0], 28, 28, 1)
x_test = x_test.reshape(x_test.shape[0], 28, 28, 1)
x_train = x_train.astype('float32') / 255
x_test = x_test.astype('float32') / 255
# 1と9のデータ抽出
x_train_1 = []
x_test_1 = []
x_test_9 = []
x_train_shape = x_train.shape
for i in range(len(x_train)):
if y_train[i] == 1:#スニーカーは7
temp = x_train[i,:,:,:]
x_train_1.append(temp.reshape((x_train_shape[1],x_train_shape[2],x_train_shape[3])))
x_train_1 = np.array(x_train_1)
x_train_1 = cut_img(x_train_1, 100000)
print("train data:",len(x_train_1))
for i in range(len(x_test)):
if y_test[i] == 1:#スニーカーは7
temp = x_test[i,:,:,:]
x_test_1.append(temp.reshape((x_train_shape[1],x_train_shape[2],x_train_shape[3])))
if y_test[i] == 9:
temp = x_test[i,:,:,:]
x_test_9.append(temp.reshape((x_train_shape[1],x_train_shape[2],x_train_shape[3])))
x_test_1 = np.array(x_test_1)
x_test_9 = np.array(x_test_9)
# network parameters
input_shape=(8, 8, 1)
batch_size = 128
latent_dim = 2
epochs = 10
Nc = 16
# build encoder model
inputs = Input(shape=input_shape, name='encoder_input')
x = Conv2D(Nc, kernel_size=2, strides=2)(inputs)
x = BatchNormalization()(x)
x = Activation('relu')(x)
x = Conv2D(2*Nc, kernel_size=2, strides=2)(x)
x = BatchNormalization()(x)
x = Activation('relu')(x)
x = Flatten()(x)
z_mean = Dense(latent_dim, name='z_mean')(x)
z_log_var = Dense(latent_dim, name='z_log_var')(x)
z = Lambda(sampling, output_shape=(latent_dim,), name='z')([z_mean, z_log_var])
encoder = Model(inputs, [z_mean, z_log_var, z], name='encoder')
# encoder.summary()
# build decoder model
latent_inputs = Input(shape=(latent_dim,), name='z_sampling')
x = Dense(2*2)(latent_inputs)
x = BatchNormalization()(x)
x = Activation('relu')(x)
x = Reshape((2,2,1))(x)
x = Conv2DTranspose(2*Nc, kernel_size=2, strides=2, padding='same')(x)
x = BatchNormalization()(x)
x = Activation('relu')(x)
x = Conv2DTranspose(Nc, kernel_size=2, strides=2, padding='same')(x)
x = BatchNormalization()(x)
x = Activation('relu')(x)
x1 = Conv2DTranspose(1, kernel_size=4, padding='same')(x)
x1 = BatchNormalization()(x1)
out1 = Activation('sigmoid')(x1)#out.shape=(n,28,28,1)
x2 = Conv2DTranspose(1, kernel_size=4, padding='same')(x)
x2 = BatchNormalization()(x2)
out2 = Activation('sigmoid')(x2)#out.shape=(n,28,28,1)
decoder = Model(latent_inputs, [out1, out2], name='decoder')
# decoder.summary()
# build VAE model
outputs_mu, outputs_sigma_2 = decoder(encoder(inputs)[2])
vae = Model(inputs, [outputs_mu, outputs_sigma_2], name='vae_mlp')
# VAE loss
m_vae_loss = (K.flatten(inputs) - K.flatten(outputs_mu))**2 / K.flatten(outputs_sigma_2)
m_vae_loss = 0.5 * K.sum(m_vae_loss)
a_vae_loss = K.log(2 * 3.14 * K.flatten(outputs_sigma_2))
a_vae_loss = 0.5 * K.sum(a_vae_loss)
kl_loss = 1 + z_log_var - K.square(z_mean) - K.exp(z_log_var)
kl_loss = K.sum(kl_loss, axis=-1)
kl_loss *= -0.5
vae_loss = K.mean(kl_loss + m_vae_loss + a_vae_loss)
vae.add_loss(vae_loss)
vae.compile(optimizer='adam')
# train the autoencoder
vae.fit(x_train_1,
epochs=epochs,
batch_size=batch_size)
#validation_data=(x_test, None))
vae.save_weights('vae_mlp_mnist.h5')
# 正常/異常のテストデータ
test_normal = x_test_1
test_anomaly = x_test_9
# 従来手法の評価
print("normal test data:",len(test_normal))
old_score_normal = result_score(vae, test_normal, "old_")
print("anomaly test data:",len(test_anomaly))
old_score_anomaly = result_score(vae, test_anomaly, "old_")
# 提案手法の評価
print("normal test data:",len(test_normal))
new_score_normal = result_score(vae, test_normal, "new_")
print("anomaly test data:",len(test_anomaly))
new_score_anomaly = result_score(vae, test_anomaly, "new_")
# 新旧手法のスコア可視化
path = 'images/'
if not os.path.exists(path):
os.mkdir(path)
plt.figure()
plt.plot(old_score_normal,label="normal")
plt.plot(old_score_anomaly,label="anomaly",c="red")
plt.title("Old method")
plt.xlabel("Test No")
plt.ylabel("Score")
plt.legend()
plt.savefig(path + "Old method.png")
plt.show
plt.figure()
plt.plot(new_score_normal,label="normal")
plt.plot(new_score_anomaly,label="anomaly",c="red")
plt.title("New method")
plt.xlabel("Test No")
plt.ylabel("Score")
plt.legend()
plt.savefig(path + "New method.png")
plt.show()
# ROC曲線の描画
y_true = np.zeros(len(test_normal)+len(test_anomaly))
y_true[len(test_normal):] = 1#0:正常、1:異常
old_score = np.array(old_score_normal)
old_score = np.hstack((old_score, np.array(old_score_anomaly)))
new_score = np.array(new_score_normal)
new_score = np.hstack((new_score, np.array(new_score_anomaly)))
# FPR, TPR(, しきい値) を算出
fpr_old, tpr_old, _ = metrics.roc_curve(y_true, old_score)
fpr_new, tpr_new, _ = metrics.roc_curve(y_true, new_score)
# AUC
auc_old = metrics.auc(fpr_old, tpr_old)
auc_new = metrics.auc(fpr_new, tpr_new)
# ROC曲線をプロット
plt.figure
plt.plot(fpr_old, tpr_old, label='Old method (area = %.2f)'%auc_old)
plt.plot(fpr_new, tpr_new, label='New method (area = %.2f)'%auc_new, c="r")
plt.legend()
plt.title('ROC curve')
plt.xlabel('False Positive Rate')
plt.ylabel('True Positive Rate')
plt.grid(True)
plt.savefig(path + "ROC curve.png")
plt.show()