TL;DR
- 書籍「効果検証入門 正しい比較のための因果推論/計量経済学の基礎」のRソースコードを、Pythonで(ほぼ)再現しました
- https://github.com/nekoumei/cibook-python
- 本記事では、主にRではライブラリどーん!で済むけどPythonではそうはいかない部分の解説をします
書籍の紹介
https://www.amazon.co.jp/dp/B0834JN23Y

上記Amazonに目次が載っているのでそれを見るのが早い気がしますが。。
とても良い本です。正確な意思決定を行うためにどうやってバイアスを取り除くか?に焦点を当てて種々の因果推論の手法(傾向スコア/DiD/RDDなど)をRソースコードによる実装とともに紹介されています。
全体を通して、現実問題の効果検証に因果推論を活用するにはどうすればいいか?という観点で書かれており、非常に実用的だなーと感じました。
Pythonで書きました
https://github.com/nekoumei/cibook-python
大元の公開されているRソースコード(https://github.com/ghmagazine/cibook )と対応する形で、Jupyter Notebookを作成しました。
また、本Python実装にあたって、グラフの可視化ライブラリは一部を除いてplotly.expressを使用しています。
GithubのNotebookレンダリングではplotlyのグラフは表示できないので、オンラインで確認したい場合はREADMEに記載しているGithub Pagesをご確認ください。(images配下のhtmlを表示しています)
plotly.expressに関しては、日本語だと下記記事がとても参考になります。
令和時代のPython作図ライブラリのデファクトスタンダードPlotlyExpressの基本的な描き方まとめ
Pythonで書くうえでの主要なポイント
回帰分析
statsmodelsのOLSおよびWLSを利用しています。
(ch2_regression.ipynbより抜粋)
## バイアスのあるデータでの重回帰
y = biased_df.spend
# R lmではカテゴリ変数は自動的にダミー変数化されているのでそれを再現
X = pd.get_dummies(biased_df[['treatment', 'recency', 'channel', 'history']], columns=['channel'], drop_first=True)
X = sm.add_constant(X)
results = sm.OLS(y, X).fit()
nonrct_mreg_coef = results.summary().tables[1]
nonrct_mreg_coef
ここで、Rのlm関数との違いが2つあります。
①lmではカテゴリカル変数のダミー変数化を自動で行っているようです。sm.OLSでは自動でできないので pd.get_dummies()を利用しています。
②lmではバイアス項が自動で追加されます。こちらも手動で追加します。(参考:統計: Python と R で重回帰分析してみる)
RData形式のデータセットの読込
実装内で利用されているデータセットの中には、experimentdatarのようにRパッケージとして公開されていたり、RData形式で公開されているデータセットがあります。
これらをPythonのみで読み込む方法はないです。(あったら教えてください)
コメント欄にて、upuraさんにご教示いただきました!
https://qiita.com/nekoumei/items/648726e89d05cba6f432#comment-0ea9751e3f01b27b0adb
.rdaファイルは、rdataというパッケージで、Rなしで読み込みが可能です。
(ch2_voucher.ipynbより抜粋)
parsed = rdata.parser.parse_file('../data/vouchers.rda')
converted = rdata.conversion.convert(parsed)
vouchers = converted['vouchers']
rpy2経由でRを使ってデータを読込、pandas DataFrameへの変換を行う場合は下記のとおりです。
(ch2_voucher.ipynbより抜粋)
from rpy2.robjects import r, pandas2ri
from rpy2.robjects.packages import importr
pandas2ri.activate()
experimentdatar = importr('experimentdatar')
vouchers = r['vouchers']
ch2_voucher.ipynbに記載しているとおり、Rパッケージのインストール等は事前にRの対話環境で行っています。
また、ch3_lalonde.ipynbにて使用するデータセットは.dtaファイルです。
こちらは、pandasのread_stata()によって読み込むことができます。
(ch3_lalonde.ipynbより抜粋)
cps1_data = pd.read_stata('https://users.nber.org/~rdehejia/data/cps_controls.dta')
cps3_data = pd.read_stata('https://users.nber.org/~rdehejia/data/cps_controls3.dta')
nswdw_data = pd.read_stata('https://users.nber.org/~rdehejia/data/nsw_dw.dta')
傾向スコアマッチング(最近傍マッチング)
傾向スコアマッチングのマッチング手法はいくつかありますが、書籍ではMatchItの最近傍マッチングを使っているようです。
Pythonにはちょうどいいライブラリはなさそうだったので愚直に実装しています。
(ch3_pscore.ipynbより抜粋)
def get_matched_dfs_using_propensity_score(X, y, random_state=0):
# 傾向スコアを計算する
ps_model = LogisticRegression(solver='lbfgs', random_state=random_state).fit(X, y)
ps_score = ps_model.predict_proba(X)[:, 1]
all_df = pd.DataFrame({'treatment': y, 'ps_score': ps_score})
treatments = all_df.treatment.unique()
if len(treatments) != 2:
print('2群のマッチングしかできません。2群は必ず[0, 1]で表現してください。')
raise ValueError
# treatment == 1をgroup1, treatment == 0をgroup2とする。group1にマッチするgroup2を抽出するのでATTの推定になるはず
group1_df = all_df[all_df.treatment==1].copy()
group1_indices = group1_df.index
group1_df = group1_df.reset_index(drop=True)
group2_df = all_df[all_df.treatment==0].copy()
group2_indices = group2_df.index
group2_df = group2_df.reset_index(drop=True)
# 全体の傾向スコアの標準偏差 * 0.2をしきい値とする
threshold = all_df.ps_score.std() * 0.2
matched_group1_dfs = []
matched_group2_dfs = []
_group1_df = group1_df.copy()
_group2_df = group2_df.copy()
while True:
# NearestNeighborsで最近傍点1点を見つけ、マッチングする
neigh = NearestNeighbors(n_neighbors=1)
neigh.fit(_group1_df.ps_score.values.reshape(-1, 1))
distances, indices = neigh.kneighbors(_group2_df.ps_score.values.reshape(-1, 1))
# 重複点を削除する
distance_df = pd.DataFrame({'distance': distances.reshape(-1), 'indices': indices.reshape(-1)})
distance_df.index = _group2_df.index
distance_df = distance_df.drop_duplicates(subset='indices')
# しきい値を超えたレコードを削除する
distance_df = distance_df[distance_df.distance < threshold]
if len(distance_df) == 0:
break
# マッチングしたレコードを抽出、削除する
group1_matched_indices = _group1_df.iloc[distance_df['indices']].index.tolist()
group2_matched_indices = distance_df.index
matched_group1_dfs.append(_group1_df.loc[group1_matched_indices])
matched_group2_dfs.append(_group2_df.loc[group2_matched_indices])
_group1_df = _group1_df.drop(group1_matched_indices)
_group2_df = _group2_df.drop(group2_matched_indices)
# マッチしたレコードを返す
group1_df.index = group1_indices
group2_df.index = group2_indices
matched_df = pd.concat([
group1_df.iloc[pd.concat(matched_group1_dfs).index],
group2_df.iloc[pd.concat(matched_group2_dfs).index]
]).sort_index()
matched_indices = matched_df.index
return X.loc[matched_indices], y.loc[matched_indices]
treatment群の1点と最も傾向スコアが近いcontrol群1点をマッチングさせる、を反復しています。その際、距離がstdの0.2倍に収まるペアのみ抽出するようしきい値を設けています。
このあたりの詳細は傾向スコアの概念とその実践が詳しかったです。
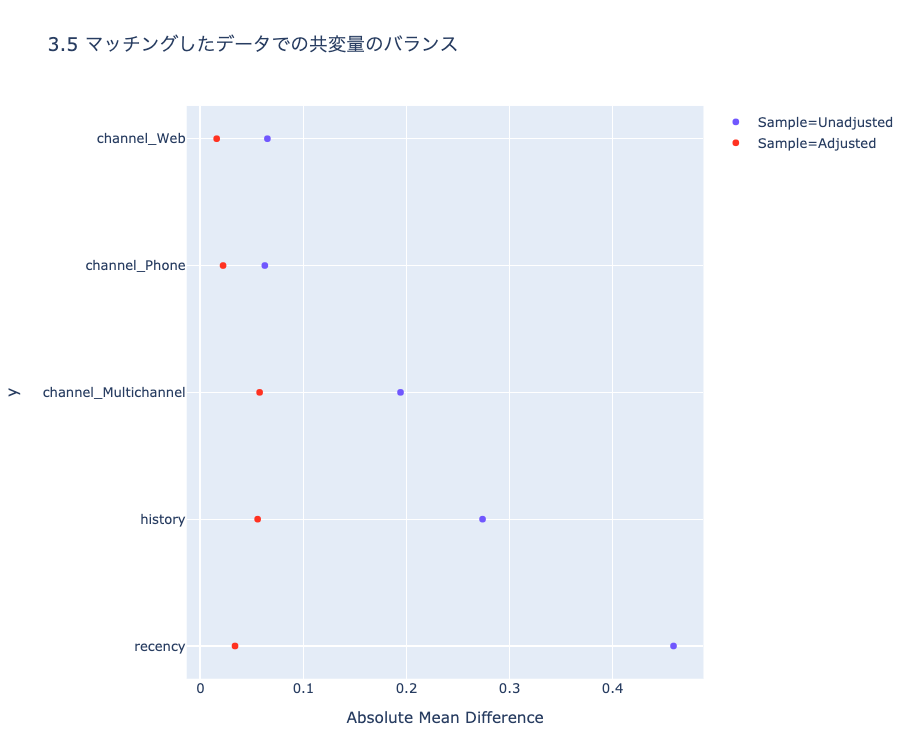
ch3_pscore.ipynbにてMatchItによるマッチング結果との比較をしていますが、完全に一致はしていません。結論が変わらない且つ共変量のバランスも良い感じ(下記参照)のため概ね良しとしています。
こちらも、Rのcobalt love.plot()のような便利ライブラリはないので自力で可視化しています。
(images/ch3_plot1.htmlより)
逆確率重み付き推定(IPW)
IPWは実装がシンプルなのがいいところですね。
こちらもWeightItと比較していますが、概ね正しそうです。
(ch3_pscore.ipynbより抜粋)
def get_ipw(X, y, random_state=0):
# 傾向スコアを計算する
ps_model = LogisticRegression(solver='lbfgs', random_state=random_state).fit(X, y)
ps_score = ps_model.predict_proba(X)[:, 1]
all_df = pd.DataFrame({'treatment': y, 'ps_score': ps_score})
treatments = all_df.treatment.unique()
if len(treatments) != 2:
print('2群のマッチングしかできません。2群は必ず[0, 1]で表現してください。')
raise ValueError
# treatment == 1をgroup1, treatment == 0をgroup2とする。
group1_df = all_df[all_df.treatment==1].copy()
group2_df = all_df[all_df.treatment==0].copy()
group1_df['weight'] = 1 / group1_df.ps_score
group2_df['weight'] = 1 / (1 - group2_df.ps_score)
weights = pd.concat([group1_df, group2_df]).sort_index()['weight'].values
return weights
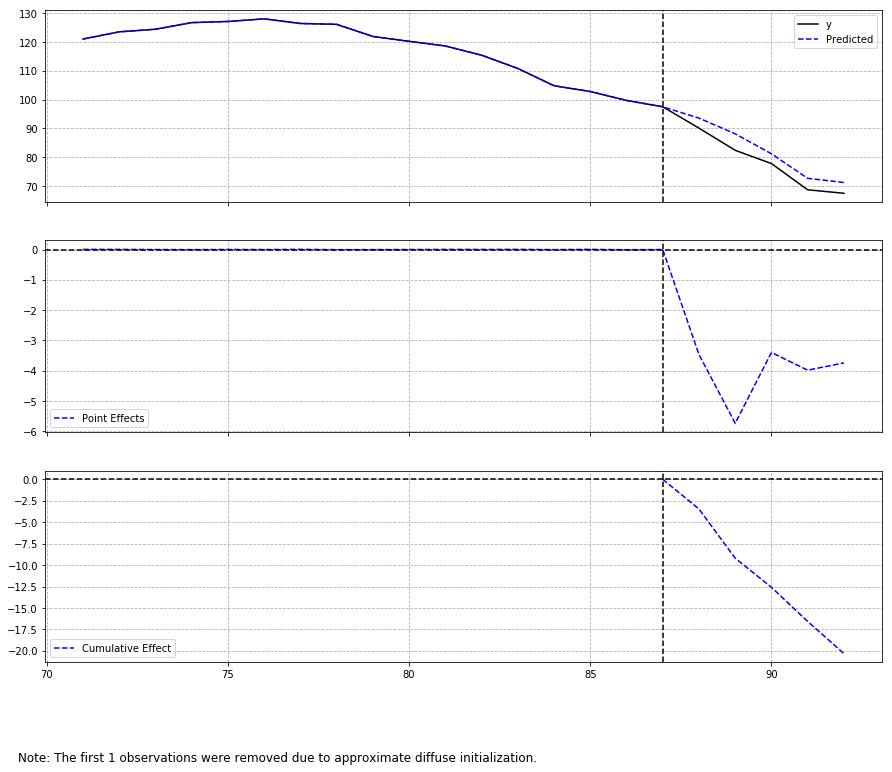
CausalImpact
[2021/09/11追記]
本家CausalImpactと同様、bstsを用いて推定するCausalImpactのPython実装 tfcausalimpactを使用します。
https://github.com/WillianFuks/tfcausalimpact

共変量が多いからかデフォルトのfit_method='vi'だと推定がやや不安定だったため、下記のとおりhmcを指定しています。
pre_period = [70, 87]
post_period = [88, 92]
ci = CausalImpact(data, pre_period, post_period, model_args={'fit_method': 'hmc'})
ci.plot()
[2021/09/11]tfcausalimpactに差し替えたため削除した内容
- dafiti/causalinpact: https://github.com/dafiti/causalimpact
- tcassou/causal_impact: https://github.com/tcassou/causal_impact
outputがキレイなのでいつもはdafitiのcausalimpactを使っているんですが、推定結果が書籍(Rの本家causalimpact)に近いのはtcassouのcausal_impactでした。
両者とも、statsmodelsの状態空間モデルで推定してるみたいですが、、ちょっと実装の違いはよくわからなかったです。誰か教えて下さい。。
どちらも、R実装と比較して推定誤差がめちゃめちゃ小さくなってるんですよね。共変量が多いのがあまりよくないのかもしれない。
dafiti/causalimpactのplot
tcassou/causal_impactのplot
回帰不連続デザイン(RDD)
Rだとrddtoolsでサクっと実行できますが、Pythonだとそうはいかないです。
まず、rddtoolsのrdd_reg_lmで使われている回帰式を確認します。
(参考:https://cran.r-project.org/web/packages/rddtools/rddtools.pdf P23)
Y = α + τD + β_1(X-c)+ β_2D(X-c) + ε
余談ですが、私はてっきりRDDって、cut-off値の左と右でそれぞれ回帰モデルつくって、cut-off値での推定値の差分を取るんだと思ってたんですが、1つの回帰式で表すんですね。意味的には同じなのかな。
ここで、DはXがcut-off値以降なら1, 以前なら0の値を持つバイナリ変数です。Dのcoefが効果量として確認したい値です。
また、cはcut-off値です。
以上を踏まえて、実装します。実装にあたってはrddtoolsのソースコードも参照しています。(https://github.com/MatthieuStigler/RDDtools/blob/master/RDDtools/R/model.matrix.RDD.R )
(ch5_rdd.ipynbより抜粋)
class RDDRegression:
# Rパッケージrddtoolsのrdd_reg_lmを再現する
# 参考:https://cran.r-project.org/web/packages/rddtools/rddtools.pdf P23
def __init__(self, cut_point, degree=4):
self.cut_point = cut_point
self.degree = degree
def _preprocess(self, X):
X = X - threshold_value
X_poly = PolynomialFeatures(degree=self.degree, include_bias=False).fit_transform(X)
D_df = X.applymap(lambda x: 1 if x >= 0 else 0)
X = pd.DataFrame(X_poly, columns=[f'X^{i+1}' for i in range(X_poly.shape[1])])
X['D'] = D_df
for i in range(X_poly.shape[1]):
X[f'D_X^{i+1}'] = X_poly[:, i] * X['D']
return X
def fit(self, X, y):
X = X.copy()
X = self._preprocess(X)
self.X = X
self.y = y
X = sm.add_constant(X)
self.model = sm.OLS(y, X)
self.results = self.model.fit()
coef = self.results.summary().tables[1]
self.coef = pd.read_html(coef.as_html(), header=0, index_col=0)[0]
def predict(self, X):
X = self._preprocess(X)
X = sm.add_constant(X)
return self.model.predict(self.results.params, X)
多項式回帰を行うための前処理として、scikit-learnのPolynomialFeaturesを利用しています。
(images/ch5_plot2_3.htmlより)
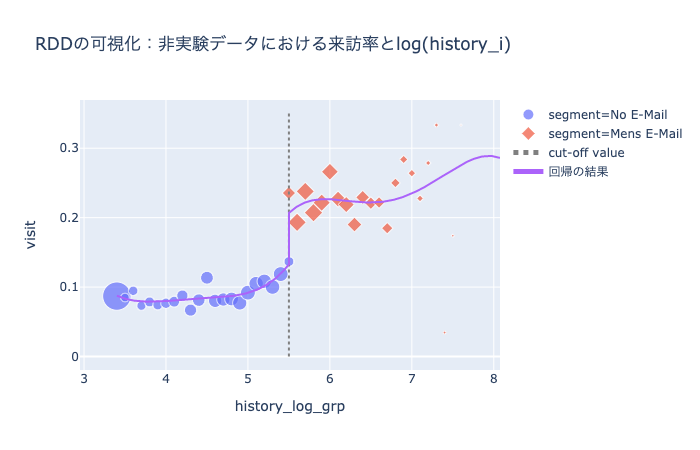
良いかんじに推定できました。Notebookを見ていただければわかりますが、効果量の推定も書籍とほぼ一致しています。よかった。
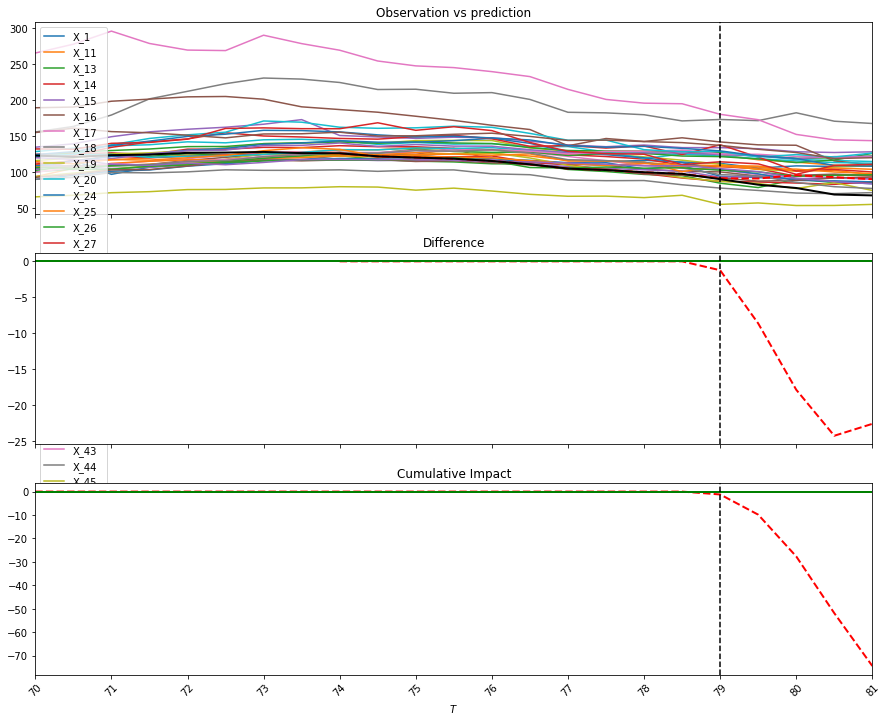
nonparametric RDD (RDestimate)
できませんでした、、、冒頭の「Pythonで(ほぼ)再現しました」のほぼの部分です。
RDestimateでは、Imbens and Kalyanaraman(2012) Optimal Bandwidth Choice for the Regression Discontinuity Estimator の手法を用いて、最適なバンド幅を選択するんですが、そのバンド幅の推定がどうやってるのかよくわかんなかったです。
ch5_rdd.ipynbでは、雑にバンド幅を変えたときのMSEが最適なバンド幅を見つけるように実装してみましたが、あまりうまく行っているように見えないです。
(ch5_plot4.htmlより)
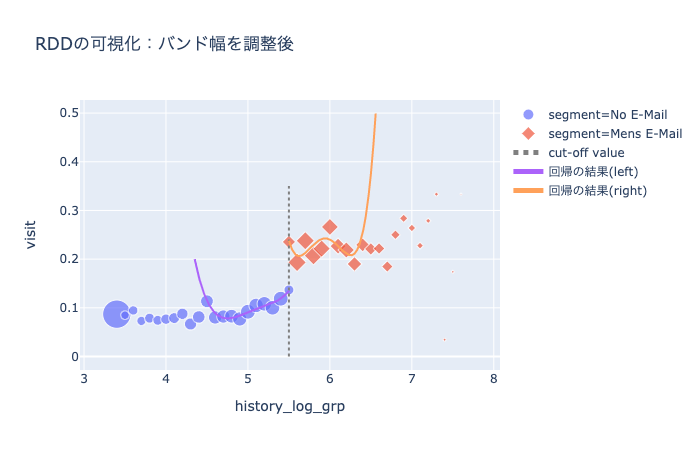
うーんってかんじですね。純粋にバンド幅を狭めて推定するとバンド幅外は予測できなくなるのはそれはそうな気がするんですが、実際どうやるんですかね。
あるいは、cut-off近傍の推定にだけ興味があるからあまり気にしなくていいのかな。
おわりに
理解や実装が間違っていたり、なんかおかしいところとかがあったらぜひ教えてほしいです。
Twitter