更新履歴
- 2018年12月17日 もっとTPOTで自動機械学習を試したという記事を書いたので、それに関する追記をしました。
- 2018年12月14日 「TPOTでは特徴選択までは自動化してくれない」なんて大嘘をついていたのを修正しました。
この記事の目的
その機械学習プロセス、自動化できませんか?という記事を読みました。
機械学習モデルの選定やパラメータチューニングなどを自動で行おうという試みがあるそうです。
この記事の中で、TPOTというパッケージが紹介されていました。
遺伝的プログラミングによって、機械学習を最適化してくれるもののようです。
さっそくTPOTで回帰学習を試したのですが、ちょっと躓いたりもしたので、記録を残します。
TPOTのHPに沿ってやっていきます。
環境
- Windows 10
- Python 3.7.2 (64bit)
- TPOT 0.9.5
下準備
パッケージのインストール
HPを見ながら。
いくつか必要なパッケージがあるようです。
pipでサクッとインストールします。
最後のpywin32はWindows環境のみ必要です。
pip install numpy scipy scikit-learn pandas deap update_checker tqdm stopit pywin32
さらに、xgboostというパッケージを入れると、XGBoostというものに対応させることもできるとか。
これは、Distributed Gradient Boostingというアルゴリズムを提供するものです。
いわゆるアンサンブル学習の一種ですね。
Windows未対応のようなので、今回は見送ります。
他にも、daskだとかscikit-mdrとかskrebateとかを入れてもいいよとありますが、見送ります。
そして、満を持して
pip install tpot
データの準備
回帰学習を試してみたいです。
HPのExampleを見ると、ボストンの住宅価格のデータで試してますね。
これでやってみます。
from sklearn.datasets import load_boston
housing = load_boston()
これで、住宅価格のデータセットがロードされました。
データの内容については、このあたりの記事をご覧ください。
housing.dataには、地域の犯罪発生率や大きな道路へのアクセスしやすさなどが入っています。
housing.targetには、教師信号となる住宅価格があります。
train_test_splitでトレーニングデータとテストデータに分けます。
from sklearn.model_selction import train_test_split
X_train, X_test, y_train, y_test = train_test_split(housing.data, housing.target,
train_size=0.75, test_size=0.25,
random_state=2018)
random_stateを設定して、結果が再現されるようにします。
TPOT回帰を実践
モジュールのインポート
まず、モジュールをimportします
from tpot import TPOTRegressor
…が、エラーが出てしまいました。
ImportWarning: Falling back to the python version of hypervolume module. Expect this to be very slow.
"module. Expect this to be very slow.", ImportWarning)
ググってみるといくつかディスカッションがされており、deapの?コンパイルの?問題らしいです。
こちらで、古いバージョンのdeapを入れてみろとありましたのでやってみます
(今のバージョンは1.2.2です)。
pip uninstall deap
pip install deap==1.0.2.post2
あらためて試したところ、エラーがなくなりました!
引数を入れてインスタンス生成
tpotは、sklearnと同じように使えます。
まず、tpotのインスタンスを作ります。
from tpot import TPOTRegressor
tpot = TPOTRegressor(generations=5, population_size=50, verbosity=2)
関数の詳しい使い方は、API Documentにあります。
が、理解するには遺伝的プログラミングについての基礎知識が必要そうですね…。
遺伝的プログラミングは遺伝的アルゴリズムを拡張したものだそうです。
遺伝的アルゴリズムについては、このページを見たらなんとなくわかったような気になりました。
generationsは、パイプライン最適化の繰り返し回数だそうです。
遺伝的アルゴリズムにおける「世代」ですね。
population sizeは、上記ページにおける個体群のことかと思います。
verbosityは、進捗報告のためのオプションです。
- 0: なにもprintしない
- 1: 最低限の情報をprintする
- 2: プログレスバーなんかも表示する
- 3: なんでもかんでもprintする
フィッティング実行
後は、このインスタンスにデータを与えてフィッティングするだけ。
せっかくなので実行時間をtimeで測ります。
import time
t0 = time.time()
tpot.fit(X_train, y_train)
print(time.time() - t0)
実行。
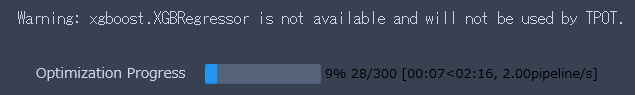
進捗状況がtqdmで表示されます。
私の環境では、126秒で終わりました。
テストデータに適用
スコアを見てみます。
注)再現性はありません(再現性を持たせる方法はこちら)。
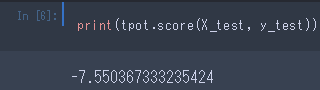
MSEだそうです。
平均二乗誤差なのに負数?とも思いますが、scikit-learnの仕様上、そういうもののようです(参照)。
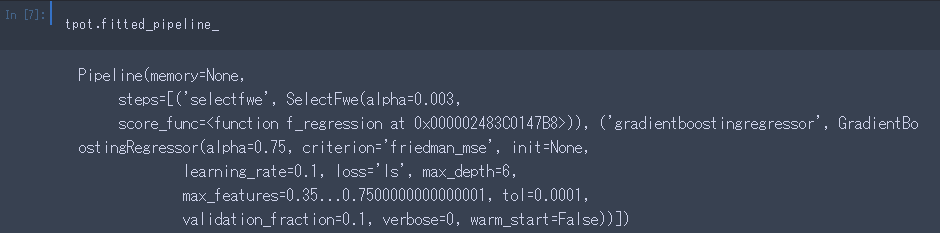
.fitted_pipeline_で、最終的に吐き出されたpipelineの情報を返してくれます。
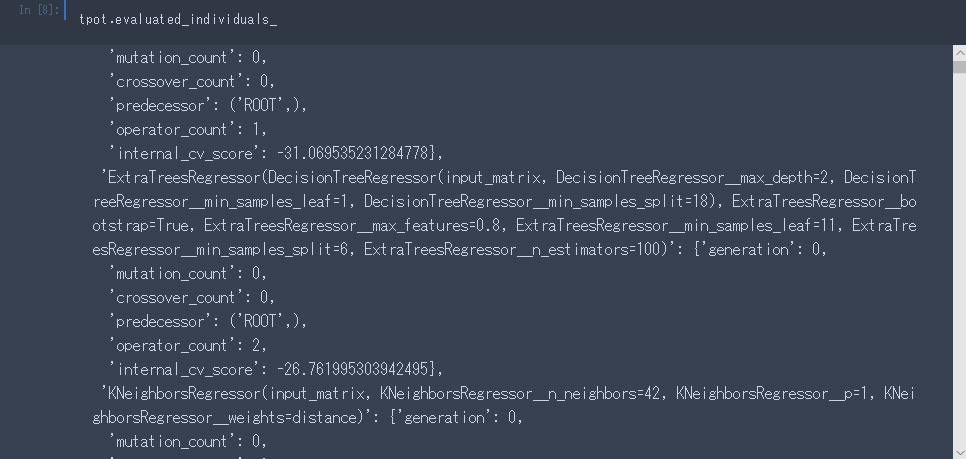
.evaluated_individuals_で、検討したモデルについての情報を返してくれます。
テストデータに適用して精度を見てみましょう。
精度は、わかりやすくPearsonの相関係数で見ます。
フィットされたpipelineから予測を行う場合は、.predictを使います。
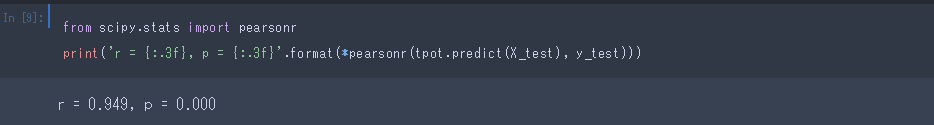
えげつない精度です。
決定係数R2も見てみましょう。
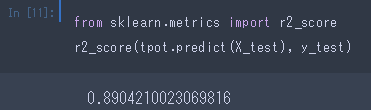
ちなみに、同じデータを使った記事では、重回帰分析によりR2=.635を示しています。
引数を変えて試す
先ほどの例では、公式ドキュメントのExampleにならい、generations=5, population_size=50でやっていました。
しかし、デフォルトの値はgenerations=100, population_size=100だったようです。
簡易的に試したということですね。
デフォルトのままの設定で動かしてみようと思います。
また、n_jobsを設定することで、並列演算してくれるようなので、これも試します。
並列演算してみる
generationsとpopulationsは先ほどと同じで、n_jobs=-1にして動かしてみます。
-1を設定すると、CPUのコアを最大限フル活動して並列演算してくれます。
tpot = TPOTRegressor(generations=5, population_size=50, verbosity=2, n_jobs=-1)
t0 = time.time()
tpot.fit(X_train, y_train)
print(time.time() - t0)
先ほどは126秒かかってたのが、45秒で済みました。
計算量が多くなれば、さらに効果が期待されそうですね。
世代と人口を増やしてみる
というわけで、generationsとpopulationsをデフォルトでやってみました。
結果…
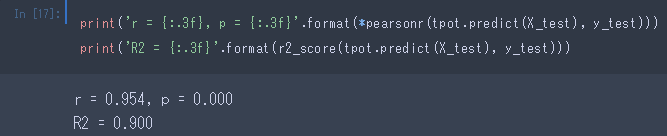
精度が上がってますね。
もともと高かったので、それほどの改善ではありませんが。
世代数を増やしたことが改善の主な要因かと思われます。
ではいったいどのくらいの世代数が求められるものでしょうか。
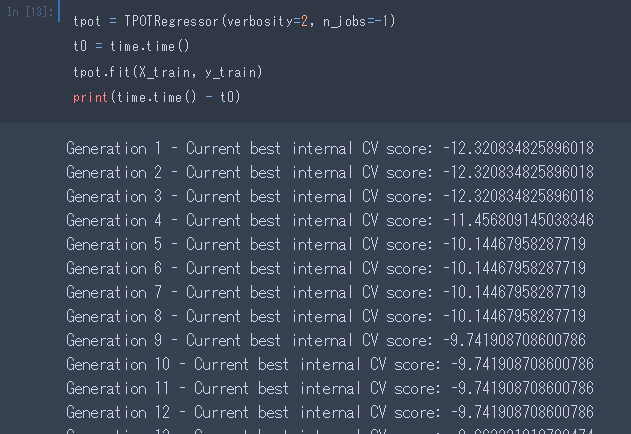
verbosityの設定により、世代ごとのスコアが出力されています。
こいつをプロットしてみましょう。
…と思ったのですが、世代ごとのCVを取得する方法が見つかりませんでした。
仕方ないので、上の出力をエクセルにコピペするというダッセェ方法でプロットしてみました
(こちらの記事に、もう少しまともな方法を載せました)。
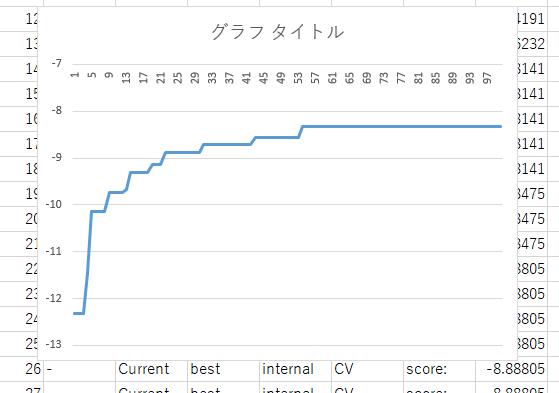
横軸が世代で、縦軸が精度です。
これをみると、53世代目くらいから精度が改善していませんね。
ちなみに、TPOTRegressorのearly_stopという引数があります。
これを指定すると、連続で~世代改善が見られなかったときに探索を打ち切ってくれるようです。
プロットを見ると、10世代くらい改善がないことがあるようです。
なので、指定するとしたら20くらいですかね(あくまでこのデータでは)。
(2018年12月18日 追記)
手持ちのデータで500世代ほど回したところ、階段状に精度が改善する様子が見られました。
遺伝的プログラミングというものの特性を(わからないなりに)考えると、当然かもしれませんね。
ですのでこのデータでも、もうしばらく世代を経るとまたグンと改善するかもしれません。
パイプラインの出力
tpot.export('tpot_boston_pipeline.py')
とやると、最適化されたpipelineを出力させることができるそうです。
出力された.pyファイルの中身を見てみましょう。
import numpy as np
import pandas as pd
from sklearn.ensemble import RandomForestRegressor
from sklearn.model_selection import train_test_split
# NOTE: Make sure that the class is labeled 'target' in the data file
tpot_data = pd.read_csv('PATH/TO/DATA/FILE', sep='COLUMN_SEPARATOR', dtype=np.float64)
features = tpot_data.drop('target', axis=1).values
training_features, testing_features, training_target, testing_target = \
train_test_split(features, tpot_data['target'].values, random_state=None)
# Average CV score on the training set was:-9.496544723555056
exported_pipeline = RandomForestRegressor(bootstrap=False, max_features=0.45, min_samples_leaf=1, min_samples_split=6, n_estimators=100)
exported_pipeline.fit(training_features, training_target)
results = exported_pipeline.predict(testing_features)
これを実行すると、与えたデータに最適化されたPipelineを適用して、交差検証で精度を返してくれるということかと思います。
'PATH/TO/DATA/FILE'と'COLUMN_SEPARATOR'に自分のデータ(.csv)のあるパスやセパレーターを入れる必要があります。
ちょっと.csvファイル用意するのが面倒なので、ここでは試しません。
まとめ
とっても簡単に使えました。
scikit-learnに慣れていれば、同じ要領で使えるのが嬉しいです。
今回は回帰をするためにTPOTRegressorを使いましたが、TPOTClassifierによる分類も同じようにできると思います。
ただ、TPOTでは特徴選択までは自動化してくれないようですね。
その点は、グリッドサーチなどと組み合わせる必要があるかもしれません。
↑大嘘です。大変失礼しました。
TPOTのHPを読むと、特徴選択や次元削減のような前処理も含めてやってくれるとあります。