EMAN2はクライオ電顕の解析ソフトの1つであるが、クライオ電子線トモグラフィーにおいてCNN (Convolutional neural network)によるセグメンテーション機能を有している。
本家HP:https://blake.bcm.edu/emanwiki/EMAN2
CNNによるsegmentation:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5623144/
また、UCSF Chimeraは分子描画プログラムの1つであるが、EMAN2の結果を描画するために使うため、同時にインストールする。
本家HP:https://www.cgl.ucsf.edu/chimera/
*UCSF Chimeraの無料版は非商用利用に限られる。企業での利用には有償ライセンスが必要なことに注意していただきたい
自分の実行環境
Windows11 Home
Ubuntu20.04.4 LTS
Nvidia GeForforce 1070Ti
UCSF-Chimeraのインストール
(参考) https://www.youtube.com/watch?v=v3fIS_6BU44
https://www.cgl.ucsf.edu/chimera/download.html からLinux用のインストラーをダウンロードする。(下ではchimera-1.16-linux_x86_64.bin)
Windowsでダウンロードした場合はインストーラーファイルをlinuxの/home/<ユーザー名>などに移動する
chmod +x CHIMERA-INSTALLER.bin
sudo ./CHIMERA-INSTALLER.bin
* CHIMERA-INSTALLERは上記の場合、chimera-1.16-linux_x86_64が入ります
インストール先を聞かれる。 どこでもよいと思うのだが、Youtubeを参考に
/opt/UCSF-Chimera64-1.16
とかにしておきますか。
途中でデスクトップにアイコンを作るか?などと聞かれるが、WSLではデスクトップもクソもないので、Noでよいはず。
さらにSymobolic linkを作るか聞かれる。ここではデフォルトが0(no link)となっているが、作っておいたほうが良さげ。Youtubeに倣って/usr/sbinを選んでおく。
(注)UCSF-Chimeraはすでに次世代のUCSF-ChimeraXが公開されている。ChimeraXがEMAN2とリンクできるかについて検討する必要があるが、一旦ペンディング
EMAN2のインストール
EMANでもGPUを使う箇所があるため、CUDA toolkitがインストールされていない場合は(2)の記事を参照し、インストールしておく
https://qiita.com/bonnginn/items/0fe011afab58ecfdd852
下記からLinux版をダウンロードする。本来、上のファイルで大丈夫なはずだが、回線が貧弱なためか、インストールに失敗したので、"huge"と書かれているファイル(eman2.91_sphire1.4_sparx_huge.linux64.sh)をダウンロードした。
https://cryoem.bcm.edu/cryoem/downloads/view_eman2_version/32
先ほどと同様に
Windowsでダウンロードした場合はインストーラーファイルをlinuxの/home/<ユーザー名>などに移動する
インストールは下記のページに従い
https://blake.bcm.edu/emanwiki/EMAN2/Install/BinaryInstallAnaconda/2.91
bash <path-to-EMAN2-installer>
例: bash eman2.91_sphire1.4_sparx_huge.linux64.sh
インストール後Ubuntuを再起動
試しに
e2version.py
と打って
Emanのバージョン(ここでは2.9.1)が表示されればOK
実際に解析に使う場合には、解析用のフォルダを作製し、そこにdirectoryを変更してから
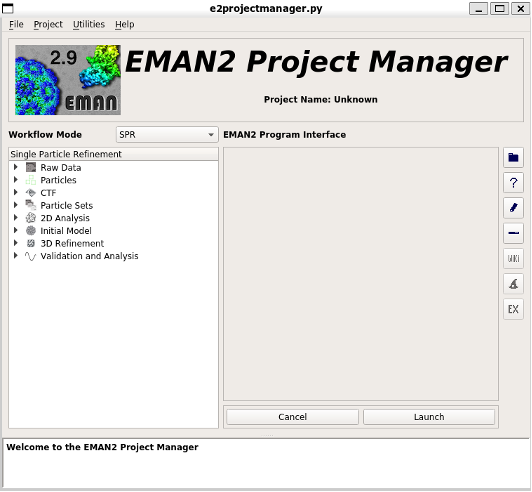
e2projectmanager.py
を実行する