Introduction
COVID-19のデータ(PCR陽性者数など)のデータを簡単にダウンロードして解析できるPythonパッケージ CovsirPhyの紹介記事です。
今回はシナリオ分析(パラメータ比較)についてご説明します。
英語版のドキュメントはCovsirPhy: COVID-19 analysis with phase-dependent SIRs, Kaggle: COVID-19 data with SIR modelをご参照ください。
1. 実行環境
CovsirPhyは下記方法でインストールできます!Python 3.7以上, もしくはGoogle Colaboratoryをご使用ください。
- 安定版:
pip install covsirphy --upgrade - 開発版:
pip install "git+https://github.com/lisphilar/covid19-sir.git#egg=covsirphy"
import covsirphy as cs
cs.__version__
# '2.8.3'
| 実行環境 | |
|---|---|
| OS | Windows Subsystem for Linux / Ubuntu |
| Python | version 3.8.5 |
2. 準備
実データをCOVID-19 Data Hub1よりダウンロードするコード2はこちら:
data_loader = cs.DataLoader("input")
jhu_data = data_loader.jhu()
population_data = data_loader.population()
本記事の表及びグラフでは、下記コード3により2020/10/3時点の日本のデータ(厚生労働省発表の全国の合計値)を用いています。COVID-19 Data Hub1にも日本のデータが含まれますが、厚生労働省発表の全国の合計値とは異なるようです。
→ DataLoader.jhu()内で自動的にCOVID-19 dataset in Japanをダウンロードするようにしました。DataLoader.japan()の呼び出しは不要です。(2020/12/24追記)
# (Optional) 厚生労働省データの取得
# エラーが出る場合は"pip install requests aiohttp"
japan_data = data_loader.japan()
jhu_data.replace(japan_data)
print(japan_data.citation)
実データの確認:
# 解析用クラスのインスタンス生成
snl = cs.Scenario(jhu_data, population_data, country="Japan")
# 実データのグラフ表示
snl.records(filename=None)
S-R trend analysisによるPhaseへの分割3と期間調整4:
# S-R trend analysis
snl.trend(filename=None)
# Separate 0th phase
snl.separate("01Apr2020")
# 一覧表示
snl.summary()
| Type | Start | End | Population | |
|---|---|---|---|---|
| 0th | Past | 06Feb2020 | 31Mar2020 | 126529100 |
| 1st | Past | 01Apr2020 | 21Apr2020 | 126529100 |
| 2nd | Past | 22Apr2020 | 06Jul2020 | 126529100 |
| 3rd | Past | 07Jul2020 | 23Jul2020 | 126529100 |
| 4th | Past | 24Jul2020 | 01Aug2020 | 126529100 |
| 5th | Past | 02Aug2020 | 14Aug2020 | 126529100 |
| 6th | Past | 15Aug2020 | 28Aug2020 | 126529100 |
| 7th | Past | 29Aug2020 | 08Sep2020 | 126529100 |
| 8th | Past | 09Sep2020 | 19Sep2020 | 126529100 |
| 9th | Past | 20Sep2020 | 03Oct2020 | 126529100 |
Parameter estimation with SIR-F model5:
# Parameter estimation
snl.estimate(cs.SIRF)
# 一覧表示(一部)
est_cols = ["Start", "End", "Rt", *cs.SIRF.PARAMETERS, "RMSLE"]
snl.summary(columns=est_cols)
| Start | End | Rt | theta | kappa | rho | sigma | RMSLE | |
|---|---|---|---|---|---|---|---|---|
| 0th | 06Feb2020 | 31Mar2020 | 3.6 | 0.0197967 | 0.000781433 | 0.110404 | 0.0292893 | 0.693445 |
| 1st | 01Apr2020 | 21Apr2020 | 13.53 | 0.00119307 | 0.000954275 | 0.119908 | 0.00789786 | 0.17892 |
| 2nd | 22Apr2020 | 06Jul2020 | 0.37 | 0.090172 | 0.000858737 | 0.0264027 | 0.0636349 | 0.883253 |
| 3rd | 07Jul2020 | 23Jul2020 | 1.93 | 0.000466376 | 8.04707e-05 | 0.133382 | 0.0689537 | 0.0318977 |
| 4th | 24Jul2020 | 01Aug2020 | 1.78 | 7.63782e-05 | 0.000231109 | 0.135991 | 0.0760643 | 0.0193947 |
| 5th | 02Aug2020 | 14Aug2020 | 1.4 | 0.00100709 | 0.000215838 | 0.100929 | 0.0716643 | 0.0797834 |
| 6th | 15Aug2020 | 28Aug2020 | 0.82 | 0.000562179 | 0.000899694 | 0.0783473 | 0.0943161 | 0.021244 |
| 7th | 29Aug2020 | 08Sep2020 | 0.7 | 0.00168729 | 0.00119958 | 0.0616905 | 0.0863032 | 0.0182731 |
| 8th | 09Sep2020 | 19Sep2020 | 0.78 | 0.00293238 | 0.00132534 | 0.0793277 | 0.099966 | 0.0282502 |
| 9th | 20Sep2020 | 03Oct2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 | 0.0274187 |
3. scenarioとは
CovsirphyのScenarioクラスでは、Phase3の設定内容を複数種類登録することができます。「Phase3の設定内容」を"scenario"と呼んでいます。
たとえば次の表のように"Main scenario", "Medicine scenario"を並列して設定し、シナリオ分析を実行することができます。2020/2/6から2020/10/3までの実データが得られている、という前提で日付を記載しています。
| scenario名 | 0th-9th | 10th | 11th |
|---|---|---|---|
| Main | 2/6 - 10/3 | 10/4 - 12/31 | 2021/1/1 - 2020/4/10 |
| Medicine | 2/6 - 10/3 | 10/4 - 12/31 | 2021/1/1 - 2020/4/10 |
各シナリオについて0th-9th phaseのパラメータ推定を行い、10 phaseのパラメータは同一、Medicine scenarioについては10thから11thの間に$\sigma$が2倍になると仮定して患者数のシュミレーションを実行します。このとき2021/1/1-2021/4/10の間の患者数推移を比較すれば、$\sigma$の倍加効果を見積もることができます。
新薬の創出などによって一気に2倍になるとは考えにくいですが、1つのデモンストレーションとしてご参照ください。
4. シナリオの作成と削除
パラメータによる患者数への影響を検討するため、異なるパラメータを設定したシナリオを作成し、患者数のシュミレーションを行って比較します。まずシナリオ自体の作成/初期化/削除方法は次の通りです。
新規作成
Scenario.clear(name="新規シナリオ名", template="コピー元のシナリオ名")メソッドを使用して、設定済のシナリオをコピーして新たなシナリオを作成することができます。Main scenario(デフォルト)をコピー元として"New"シナリオを作成する場合、
# 新規作成
snl.clear(name="New", template="Main")
# 一覧表示
cols = ["Start", "End", "Rt", *cs.SIRF.PARAMETERS]
snl.summary(columns=cols)
(1st - 8th phaseは紙面上省略)
| Start | End | Rt | theta | kappa | rho | sigma | |
|---|---|---|---|---|---|---|---|
| ('Main', '0th') | 06Feb2020 | 31Mar2020 | 3.85 | 0.0185683 | 0.000768779 | 0.112377 | 0.027892 |
| ('Main', '9th') | 20Sep2020 | 03Oct2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
| ('New', '0th') | 06Feb2020 | 31Mar2020 | 3.85 | 0.0185683 | 0.000768779 | 0.112377 | 0.027892 |
| ('New', '9th') | 20Sep2020 | 03Oct2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
削除
削除はScenario.delete(name="シナリオ名")を使用して行います。"New"シナリオを削除し、"Main"シナリオのみ登録された状態に戻します。
# シナリオの削除
snl.delete(name="New")
# 一覧表示
snl.summary(columns=cols)
(1st - 8th phaseは紙面上省略)
| Start | End | Rt | theta | kappa | rho | sigma | |
|---|---|---|---|---|---|---|---|
| 0th | 06Feb2020 | 31Mar2020 | 3.85 | 0.0185683 | 0.000768779 | 0.112377 | 0.027892 |
| 9th | 20Sep2020 | 03Oct2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
5. [追加] 最終日を指定
ここから、phaseを追加する方法についてご説明します。Scenario.add(end_date="31Dec2020")のように最終日を指定して新しいphaseを追加できます。
# Main: 31Dec2020までを10th phaseとして追加
snl.add(end_date="31Dec2020", name="Main")
# 一覧表示
snl.summary(columns=cols, name="Main")
(1st - 8th phaseは紙面上省略)
| Start | End | Rt | theta | kappa | rho | sigma | |
|---|---|---|---|---|---|---|---|
| 0th | 06Feb2020 | 31Mar2020 | 3.61 | 0.0190222 | 0.00099167 | 0.110766 | 0.0290761 |
| 9th | 20Sep2020 | 03Oct2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
| 10th | 04Oct2020 | 31Dec2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
パラメータを特に設定していないため、追加された10th phaseのパラメータは9th phaseと同じ値になっています。
6. [追加] 日数を指定
Scenario.add(days=100)のように日数を指定して新しいphaseを追加できます。
# main: 01Jan2021 (10th最終日の翌日)から100日間を11thとして追加
snl.add(days=100, name="Main").summary(columns=cols)
# 一覧表示
snl.summary(columns=cols, name="Main")
(1st - 8th phaseは紙面上省略)
| Start | End | Rt | theta | kappa | rho | sigma | |
|---|---|---|---|---|---|---|---|
| 0th | 06Feb2020 | 31Mar2020 | 3.61 | 0.0190222 | 0.00099167 | 0.110766 | 0.0290761 |
| 9th | 20Sep2020 | 03Oct2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
| 10th | 04Oct2020 | 31Dec2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
| 11th | 01Jan2021 | 10Apr2021 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
パラメータを特に設定していないため、追加された11th phaseのパラメータは10th phaseと同じ値になっています。
7. [追加] パラメータを指定
Scenario.add(sigma=0.18)のようにパラメータの値を指定して新しいphaseを追加できます。
パラメータ推定値の取得
本記事では$\sigma$の倍加効果を見積もるため、Main scenario 11 phase(last phase)の$\sigma$の推定値をScenario.get()メソッドにより取得します。
sigma_last = snl.get("sigma", phase="last", name="Main")
sigma_med = sigma_last * 2
print(round(sigma_last, 3), round(sigma_med, 3))
# -> 0.09 0.181
Medicineシナリオの設定
Mainシナリオをコピー元としてMedicineシナリオを作成します。このとき将来のphase (10th, 11th phase)は消去されます。その上で10th phase (Mainとパラメータは同一), 11th phase ($\sigma$は2倍)を追加します。
snl.clear(name="Medicine", template="Main")
snl.add(end_date="31Dec2020", name="Medicine")
# Medicine: 01Jan2021 (10th最終日の翌日)から100日間を11thとして追加
snl.add(sigma=sigma_med, days=100, name="Medicine")
# 一覧表示
snl.summary(columns=cols, name="Medicine")
(1st - 8th phaseは紙面上省略)
| Start | End | Rt | theta | kappa | rho | sigma | |
|---|---|---|---|---|---|---|---|
| 0th | 06Feb2020 | 31Mar2020 | 3.85 | 0.0185683 | 0.000768779 | 0.112377 | 0.027892 |
| 9th | 20Sep2020 | 03Oct2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
| 10th | 04Oct2020 | 31Dec2020 | 0.87 | 0.000463924 | 0.000984493 | 0.0793406 | 0.090479 |
| 11th | 01Jan2021 | 10Apr2021 | 0.44 | 0.000463924 | 0.000984493 | 0.0793406 | 0.180958 |
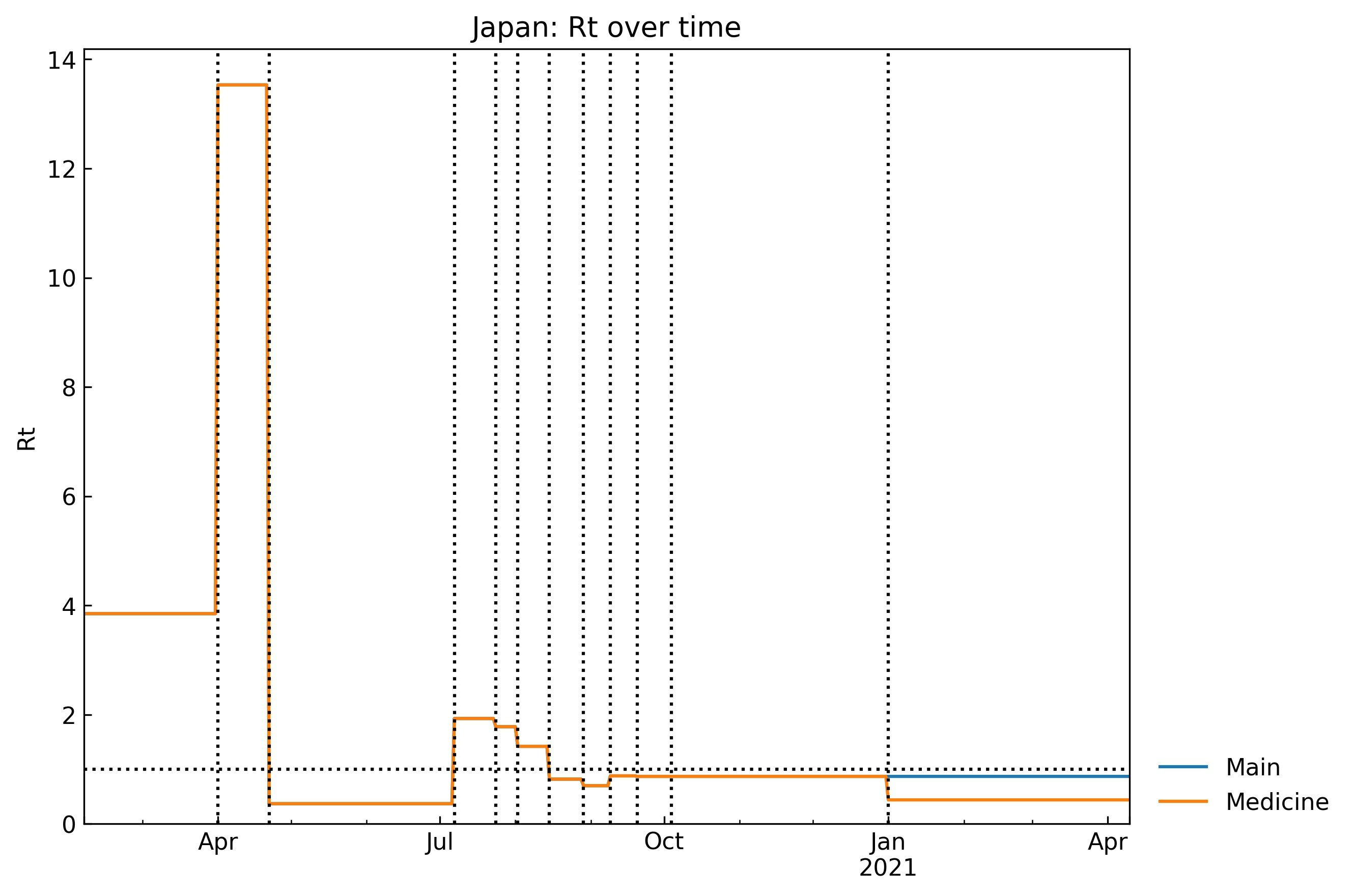
$sigma$が2倍になったため、実効再生産数$Rt$は半減しています。
8. phaseの削除
余談ですが、Scenario.delete(phase=["Phase名"])によってphaseを削除できます。ここでは12th phaseを追加して削除します。
# Phaseの追加
snl.add(days=30, name="Medicine")
# Phaseの削除
snl.delete(phases=["last"], name="Medicine")
9. パラメータ推移の確認
パラメータの推移が予定通り設定されているかどうかをグラフで確認します。Scenario.history(パラメータ名)を使用します。シナリオ名を系列名として使用します。
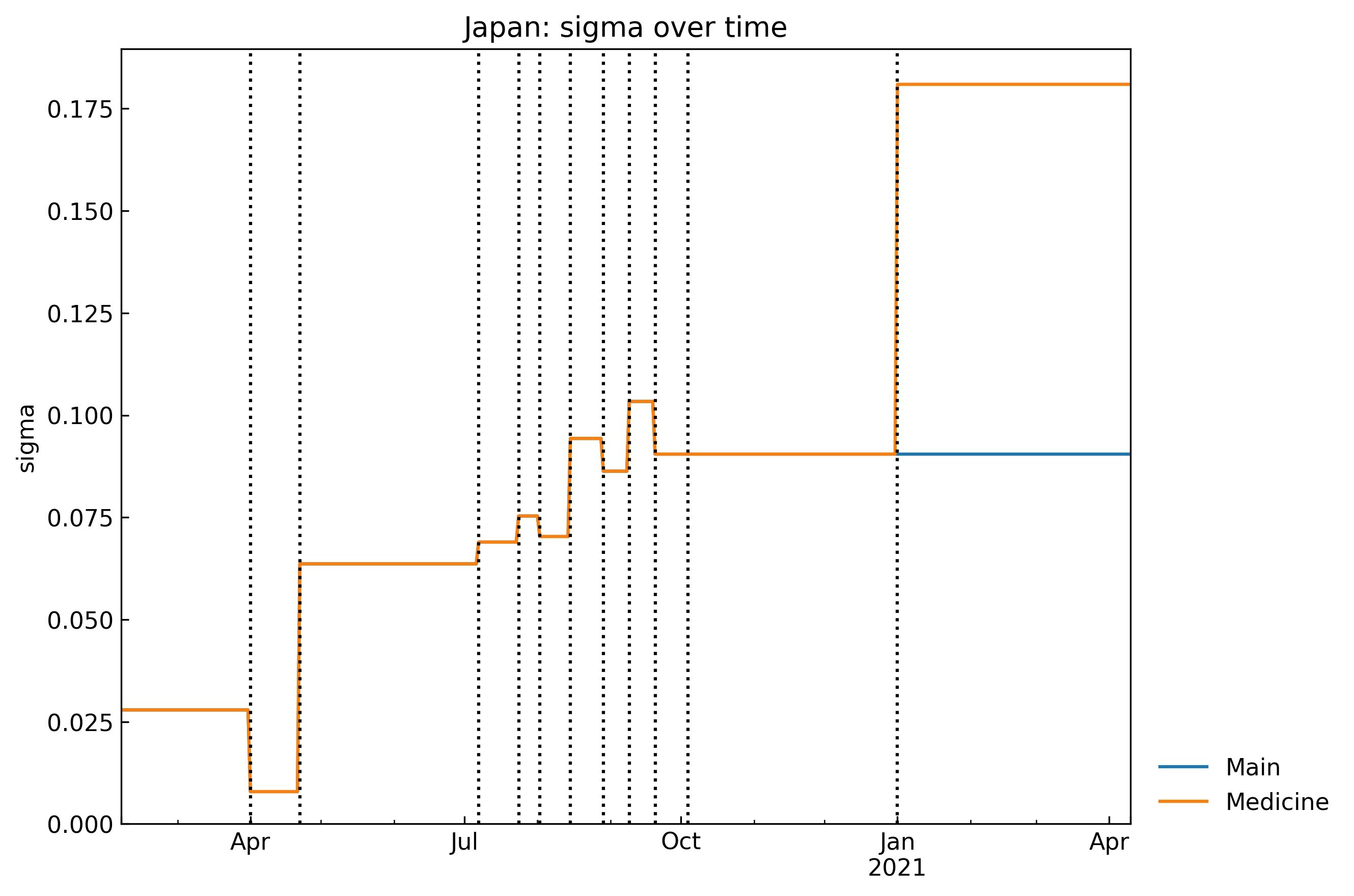
Sigmaの推移
$\sigma$がMedicine scenarioの11th phase (2021/1/1 - 2021/4/1)に倍加しているかどうか:
snl.history("sigma", filename=None)
Rtの推移
実効再生産数$Rt$がMedicine scenarioの11th phase (2021/1/1 - 2021/4/1)に半減しているかどうか:
snl.history("Rt", filename=None)
10. 患者数のシュミレーション
患者数のシュミレーション結果についてもScenario.history(変数名)にて取得できます。
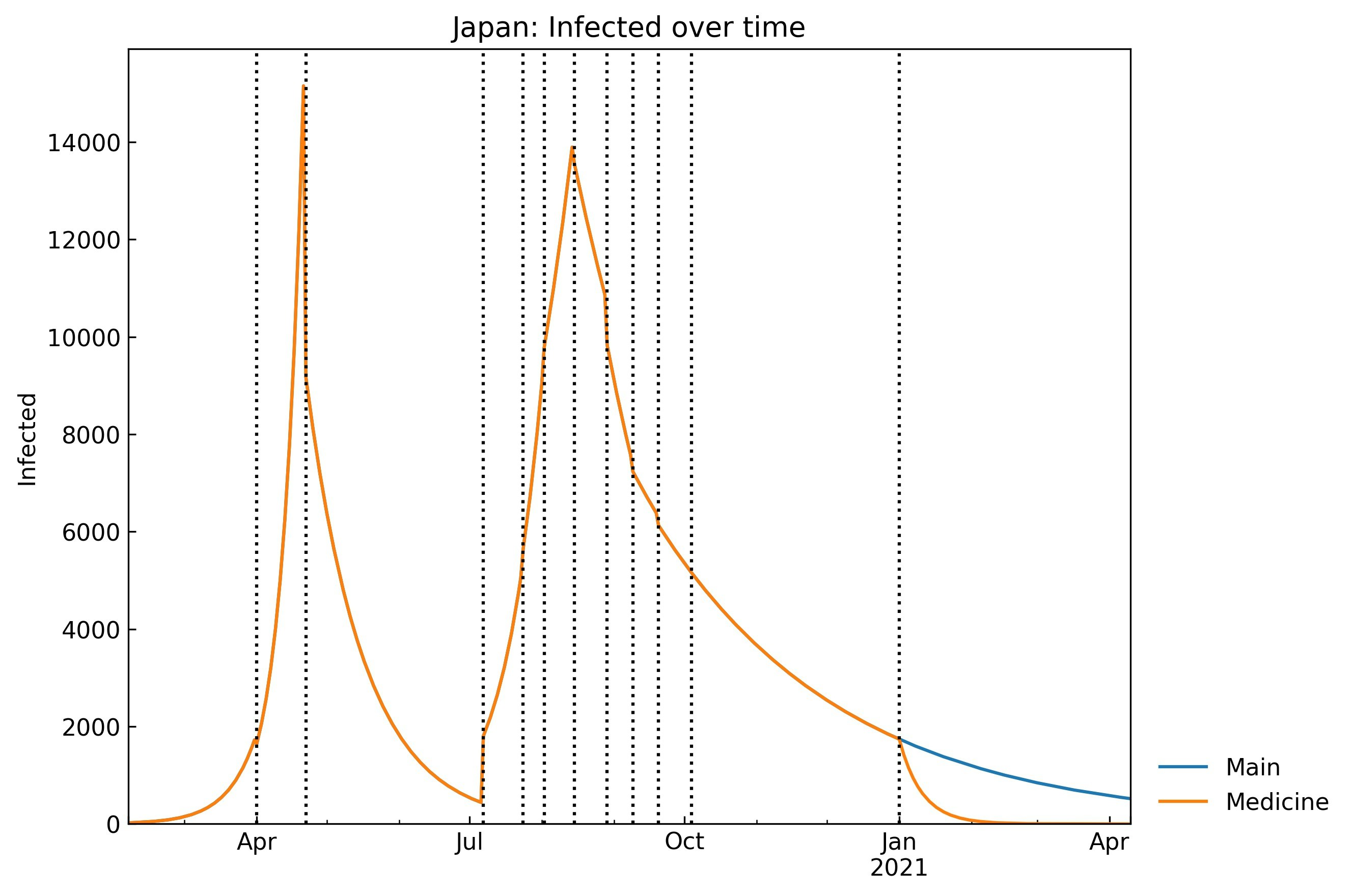
グラフ表示
snl.history("Infected", filename=None)
$\sigma$の倍加により感染者数が急速に減少しています。新薬の創出などによって一気に2倍になるとは考えにくいですが、パラメータの値を現実的な値に変更してシュミレーションを行うことで、今後の感染者数や終息時期を推定することができます。
データフレーム形式
値をデータフレーム形式で取得したい場合はScenario.track()メソッドをご使用ください。
snl.track().tail()
| Scenario | Date | Confirmed | Fatal | Infected | Recovered | Population | Rt | theta | kappa | rho | sigma | 1/alpha2 [day] | 1/gamma [day] | alpha1 [-] | 1/beta [day] | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 425 | Medicine | 2021-04-06 | 108463 | 1884 | 0 | 106579 | 126529100 | 0.44 | 0.000463924 | 0.000984493 | 0.0793406 | 0.180958 | 1015 | 5 | 0 | 12 |
| 426 | Medicine | 2021-04-07 | 108463 | 1884 | 0 | 106579 | 126529100 | 0.44 | 0.000463924 | 0.000984493 | 0.0793406 | 0.180958 | 1015 | 5 | 0 | 12 |
| 427 | Medicine | 2021-04-08 | 108463 | 1884 | 0 | 106579 | 126529100 | 0.44 | 0.000463924 | 0.000984493 | 0.0793406 | 0.180958 | 1015 | 5 | 0 | 12 |
| 428 | Medicine | 2021-04-09 | 108463 | 1884 | 0 | 106579 | 126529100 | 0.44 | 0.000463924 | 0.000984493 | 0.0793406 | 0.180958 | 1015 | 5 | 0 | 12 |
| 429 | Medicine | 2021-04-10 | 108463 | 1884 | 0 | 106579 | 126529100 | 0.44 | 0.000463924 | 0.000984493 | 0.0793406 | 0.180958 | 1015 | 5 | 0 | 12 |
11. 特性値の比較
シナリオの特徴を理解するため、特性値を比較します。ただし感染者数の平均値を比較しても意味がないため、ここでは特性値を下記の通り設定しています。
- 感染者数の最大値とその日付
- 最終日翌日の感染者数
- 最終日翌日の死亡者数
- (追加を検討中)感染者数の積分値
コードと出力結果はこちら:
snl.describe()
| max(Infected) | argmax(Infected) | Infected on 11Apr2021 | Fatal on 11Apr2021 | 11th_Rt | |
|---|---|---|---|---|---|
| Main | 15154 | 21Apr2020 | 512 | 1970 | 0.87 |
| Medicine | 15154 | 21Apr2020 | 0 | 1884 | 0.44 |
シナリオ間で値が異なるPhaseに限り、実効再生産数$Rt$を出力しています。
現在のパラメータが2021/4/10まで続いた場合、4/11時点でも感染者数の予測値が512人程度となるようです...一方で$\sigma$が仮に倍加した場合は感染者数の予測値は0人となっています。
12. あとがき
今回も閲覧ありがとうございました!
特性値の追加など、ご意見をいただけますと幸いです。
次回はワクチンの効果などを評価する方法についてご説明します。
-
Guidotti, E., Ardia, D., (2020), “COVID-19 Data Hub”, Journal of Open Source Software 5(51):2376, doi: 10.21105/joss.02376. ↩ ↩2
-
[CovsirPhy] COVID-19データ解析用Pythonパッケージ: S-R trend analysis ↩ ↩2 ↩3 ↩4
-
[CovsirPhy] COVID-19データ解析用Pythonパッケージ: Parameter estimation ↩