経緯
この記事はPyCon JP 2017 Development Sprintsでの取り組みと、
その後の勉強会(Nagoya.Swift+ 9月度勉強会 - connpass)での取り組みをまとめたものです。
注記
下記の内容は2017年当時のものであり、現在ではよりよい方法があります。下記の内容に従って環境構築を行うのは、次の観点でおすすめできません。
-
pyenvが必要になるケースはかなり限定される一方、環境の構成に与える影響が大きいので導入には慎重になるべき - Docker Container への接続を行った後のデバッグ手法については Remote Development でより容易に実現できる
例えば次に従うことで、Docker Container を開発環境として利用できます。
下記の内容は 2017 年当時の環境を知るための資料として残しますが、現在でもこの手順に従うことを推奨するものではないことは重ねて記しておきます。
目標
普段Jupyterばっかりで他の開発環境を使っていなかったので、VSCodeでPythonやってみます。
次を目標に頑張ります。
-
pyenvで導入したローカルの環境でデバッグする - Dockerで導入した環境でデバッグする
ローカル環境で実行する
環境
環境は次のとおりです。
- OS : macOS
- python : Python3.6
- 利用ツール : Anaconda, pyenv, pyenv-virtualenv
pythonの環境構築
専用のディレクトリを作り、Anacondaの環境を構築します。
環境構築には好き好きあると思いますが、pyenvを使います(やりすぎな気もしているのでそろそろpyenvからvenvに乗り換えたい)。
mkdir ~/Documents/VSCodeHandsOn
cd ~/Documents/VSCodeHandsOn
pyenv install anaconda3-2.5.0
pyenv local anaconda3-2.5.0
参考
VSCodeの環境構築
VSCodeをインストールしたらPython with Visual Studio Codeを参考に進めていけば大丈夫そうです。
僕はPython - Visual Studio Marketplaceを使ってます。
VSCodeの設定
サンプルのために次のコードを作ってみます。
import sys
import numpy as np # Numpyはanacondaを使ったら使いたくなると思うので
print('hello python!')
print('python version:' + str(sys.version_info))
print('numpy version:' + np.version.full_version)
このままPythonコードを走らせると、筆者の環境では「numpyが見つからない、Pythonのバージョンが予期したものと違う」といった不具合が発生しました。
VSCodeが使用するPythonを.vscode/setting.json内で指定することで回避できます。
{
"python.pythonPath": "/Users/[USERNAME]/.pyenv/shims/python"
}
参考
-
uroshika's notes: VisualStudioCode で Python 実行時にモジュールが import できない場合の対処法
-
【Visual Studio Code】v1.12.1 設定のためのSettings.jsonファイルについて - Qiita
デバッグ
Pythonにおけるデバッグ手法にある方法が一通りできるか試しました。
操作方法はVS Code で Python をデバッグする環境構築(+ NumPy, SciPy, Matplotlib を実行する環境構築)に従えば大丈夫です。
この段階で設定ファイルの編集なしでデバッグまで実行できました。
参考
lintツールを変更する
pylintからlintツールをflake8に変更してみました。理由はpylintは厳しすぎたのと(先程のコードだと1行目から注意されます)流行に乗りたかったという2点からです。
flake8のインストール
ターミナルからpipでインストール。
% pip install flake8
lintツールの変更
settings.jsonを編集して、利用するlintツールを変更します。macOSだとCmd+,で設定ファイルが開くので、次の内容を追記すれば大丈夫です。
{
// それぞれのユーザー特有の設定
// "files.autoSave": "afterDelay",
// python関連の設定
"python.pythonPath":"/Users/[USERNAME]/.pyenv/shims/python", // pyenv関連
"python.linting.pylintEnabled": false, // Disable pylint
"python.linting.flake8Enabled": true // Enable flake8
}
参考
Dockerの利用
事前準備
Dockerのインストール
公式サイトのインストール手順が参考になると思います。僕はHomebrewでインストールしました。
$ brew cask install docker # だったと思うけれどだいぶ前のことなので自信がない
参考
Jupyter Notebookのインストール
今回はJupyter Notebook導入済みのイメージを使います。ローカルにインストールする場合はAnaconda推奨のようです。
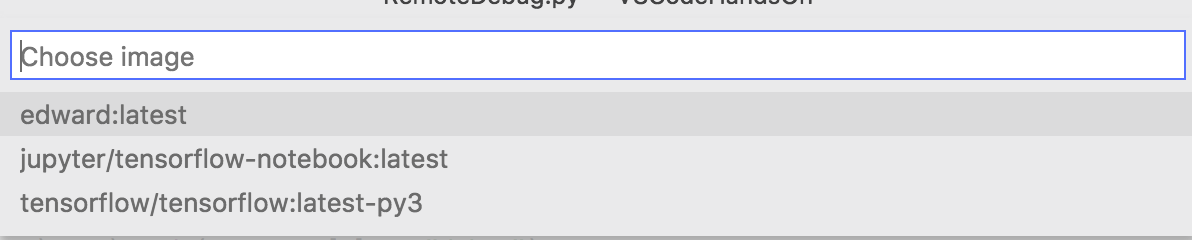
導入済みのイメージの例
前者がTensorflowの実行環境だけではなくて、Scikit LearnとかScipy, Numpy, Jupyter Notebookを含んでいることは事あるごとに広めていこうと思います。Googleの次の動画でもDockerを唐突に薦めてきます。
これ見やすいし、楽しそうに紹介してくれるし、サンプルコードあるしいい感じ! | Hello World - Machine Learning Recipes #1 https://t.co/HQ0RYnBxCO @YouTubeさんから
— 龍一郎 (@K_Ryuichirou) 2017年9月17日
後者でTensorBoardを利用するためには何らかの方法で改めてpip install tensorflowを実行する必要があります。
VSCodeに拡張機能をインストール
DockerとDocker上でJupyter Notebookをどうせ使うので、拡張機能をインストールします。
動作確認
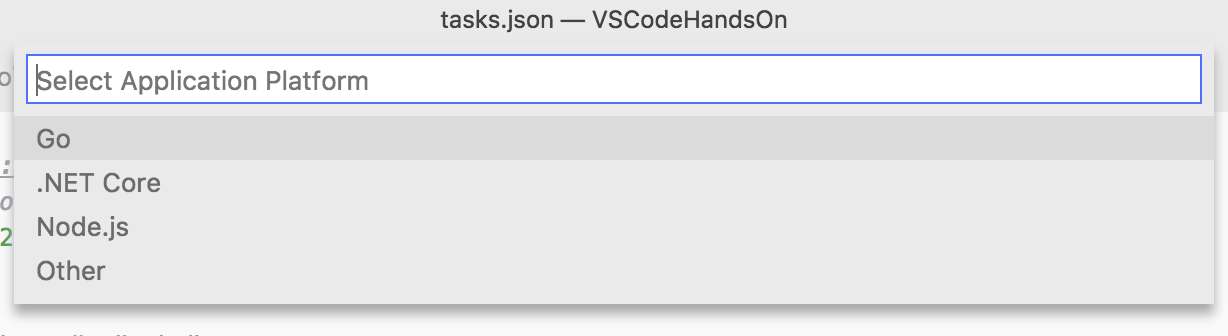
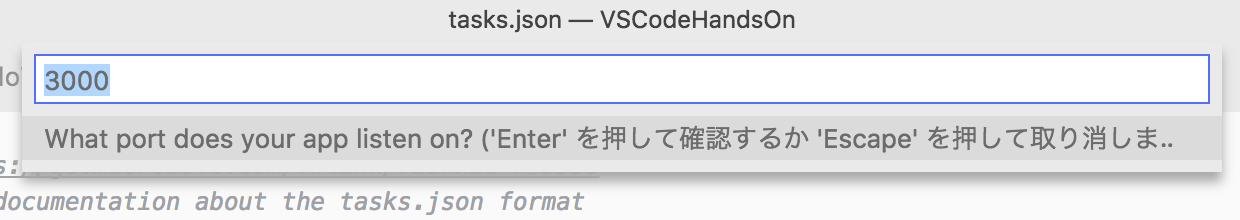
F1キーでコマンドパレットを起動した後に次を入力します。途中まで入力すると補完が効くと思います。
>Docker: Add docker files to workspace
エンターキーを連打します。
Dockerfileが次のような内容で生成されれば大丈夫です。
# golang:onbuild automatically copies the package source,
# fetches the application dependencies, builds the program,
# and configures it to run on startup
FROM golang:onbuild
LABEL Name=vscodehandson Version=0.0.1
EXPOSE 3000
# For more control, you can copy and build manually
# FROM golang:latest
# LABEL Name=vscodehandson Version=0.0.1
# RUN mkdir /app
# ADD . /app/
# WORKDIR /app
# RUN go build -o main .
# EXPOSE 3000
# CMD ["/app/main"]
コンテナの起動と接続
コンテナの起動
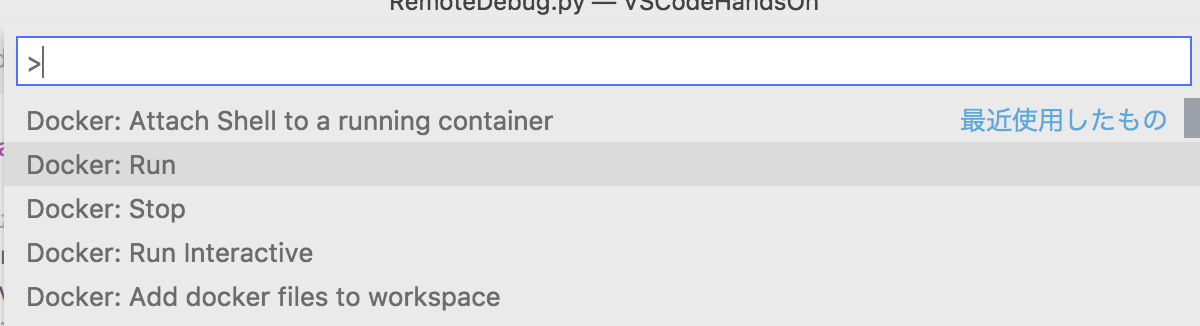
VSCodeから実行できます。F1キーを押してDocker: Runで大丈夫です。
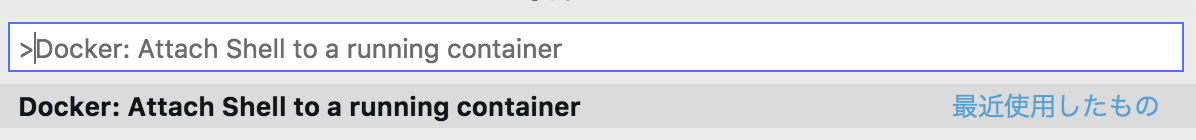
コンテナへの接続
同様にF1キーから>Docker: Attach Shell to a running containerでできます。
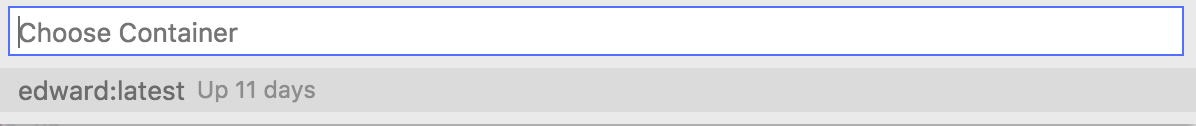
実行すると、現在動いているコンテナが表示されるので、接続したいコンテナを選ぶと接続できます。
おまけ: Kitematic
黒い画面怖い勢というわけでもないのですが、GUIがあれば欲しい勢なのでKitematicを利用しています。紹介記事はこちらが詳しかったです。
[Docker] Kitematicを使ってみよう! - Qiita
KitematicではVOLUMEでマウントするホストのディレクトリを指定できるのですが、dockerfileにVOLUMEが指定されていないイメージだとその機能が使えないようです(2017年9月現在)
次のIssueを見ると2016年から対応が進められているようですが、まだリリースされていないようです。
Tensorflowの公式イメージはVOLUMEコマンドを含んでいませんので、次のようなDockerfileを作っています。
FROM tensorflow/tensorflow:latest-py3
RUN pip --no-cache-dir install \
edward \
ptvsd==3.0.0
RUN mkdir /notebooks/mmt
# TensorBoard
EXPOSE 6006
# Jupyter
EXPOSE 8888
# ptvsd
EXPOSE 3000
WORKDIR "/notebooks"
VOLUME "/notebooks/mmt"
CMD ["/run_jupyter.sh", "--allow-root"]
edwardを入れてみたのは趣味です、またptvsdについては次で解説します。
Docker上のコンテナとのリモートデバッグ
VSCodeはPythonのリモートデバッグに対応しています。ここから設定を見ていきます。
今回は、Docker上のコンテナでPythonのコードを実行し、Dockerホスト(= ローカル環境)のVSCodeからデバッグを行うこととします。
ptvsd
もともとPythonのリモートデバッグにVisual Studioが対応していて、その機能をVSCodeでも使えるようになっている、という状況みたいです。
PythonをVisual Studioで開発するのは、あまり聞き慣れない感じですがChainerの開発を行っているのを見たことがあります。
ptvsd自体はPyPIで配布されています。
ptvsdのインストール
ptvsdはPythonのコードを実行する側にインストールしますので、Dockerfileの中でptvsdのインストールを行う記述を含めます。
また、リモートデバッグではTCP/IPポートで通信するので、Dockerfileなどで外部に公開するポートを指定しておきます。
こんな感じの記述が上のDockerfileにも含まれているのがご確認いただけます。
RUN pip --no-cache-dir install ptvsd==3.0.0
# ptvsd
EXPOSE 3000
なお、現在ではptvsdのバージョンを最新ではなく3.0.0を指定しないと動きません。ptvsd側でversion4.0で対応予定だったと思いますが、すみません、ソースが見つかりませんでした。
ptvsdのセットアップ (Docker側)
ptvsdの使い方はシンプルで、Pythonコード実行時にモジュールを読み込んで実行するだけです。
import ptvsd
ptvsd.enable_attach("my_secret", address = ('0.0.0.0', 3000))
ただし、以降の手順でPythonコードをDocker上に加えて、ローカル環境のVSCodeからも動かす必要がある上、
ローカル環境ではこの行が実行されないようにしなければいけません。
公式ではDocker環境とローカル環境で2つのソースコードを分けて、ローカル環境ではこの2行をコメントアウトするように推奨されていますが、
環境変数を用いてDocker上でのみ実行されるように実行されるようにするのが良いように思います。
今回は、Docker環境での実行時にはコマンドライン引数に"debug"という文字列を与えることで、Docker環境とローカル環境を区別しています。
# usage: $ python RemoteDebug.py debug
import sys
if (len(sys.argv) > 1) and (sys.argv[1] == "debug"):
import ptvsd
print("waiting...")
ptvsd.enable_attach("my_secret", address=('0.0.0.0', 3000))
ptvsd.wait_for_attach()
VSCodeのセットアップ (ローカル側)
Dockerコンテナに接続するため、VSCodeのlaunch.jsonに接続先の情報を書き込みます。多分、ほとんど編集しなくて大丈夫だと思います。
{
"name": "Attach (Remote Debug)",
"type": "python",
"request": "attach",
"localRoot": "${workspaceRoot}",
"remoteRoot": "${workspaceRoot}",
"port": 3000,
"secret": "my_secret",
"host": "localhost"
}
幾つか前提がありますので、補足します。
- DockerコンテナにマウントするフォルダーとPythonスクリプト、jupyter notebookをおくフォルダーは一致させました
- 変更した場合は"localRoot", "remoteRoot"を変更すれば大丈夫だと思います
- 3000番ポートでDockerコンテナとローカル環境との通信ができることは前提とします
- DockerコマンドやKitematicを使って確認できます
- 変更も可能です
デバッグの様子
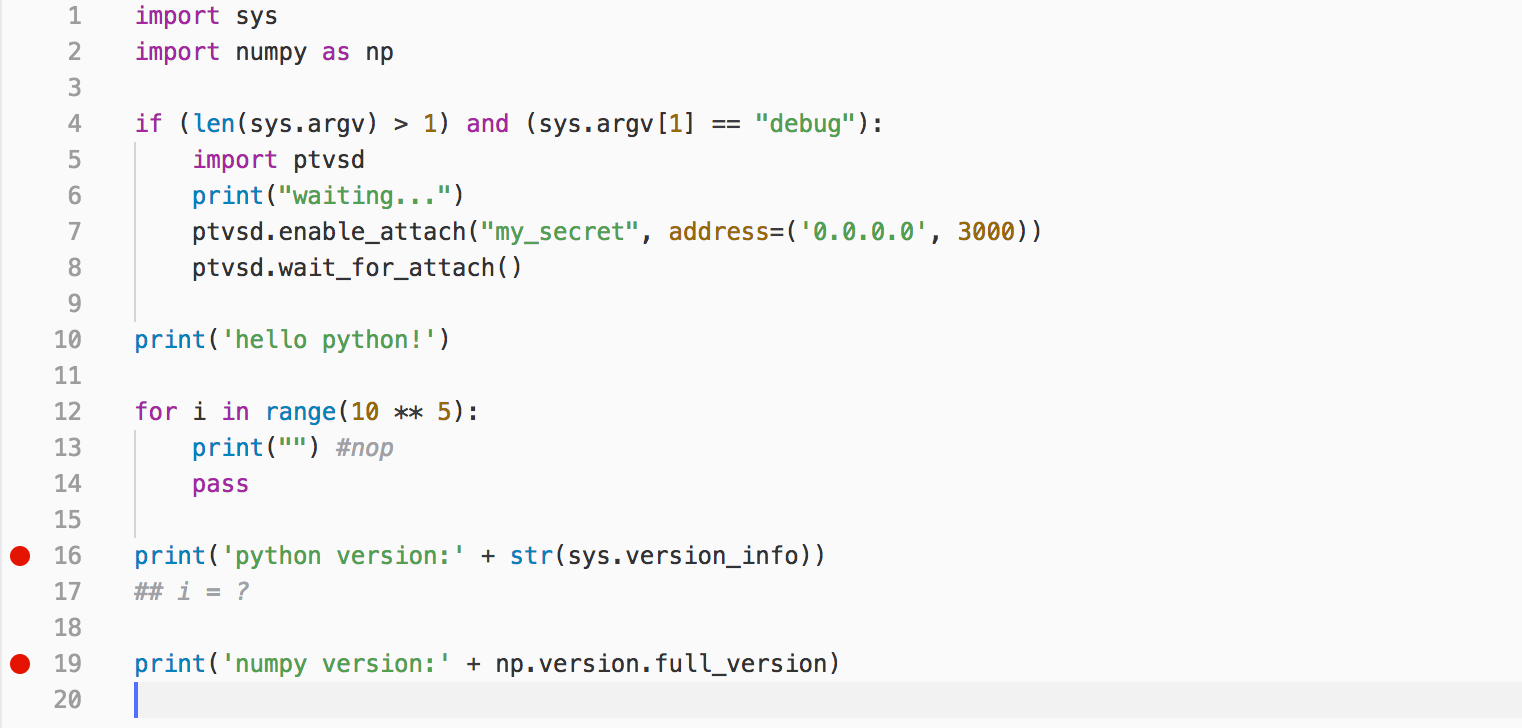
次のコードを使って検証してみます。
import sys
import numpy as np
if (len(sys.argv) > 1) and (sys.argv[1] == "debug"):
import ptvsd
print("waiting...")
ptvsd.enable_attach("my_secret", address=('0.0.0.0', 3000))
ptvsd.wait_for_attach()
print('hello python!')
for i in range(10 ** 5):
print("") #nop
pass
print('python version:' + str(sys.version_info))
## i = ?
print('numpy version:' + np.version.full_version)
ローカル環境での準備(VSCode)
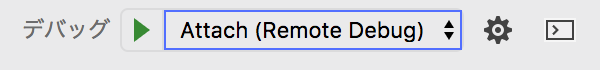
VSCode側でデバッグの構成を "Attach (Remote Debug)" に変更します。
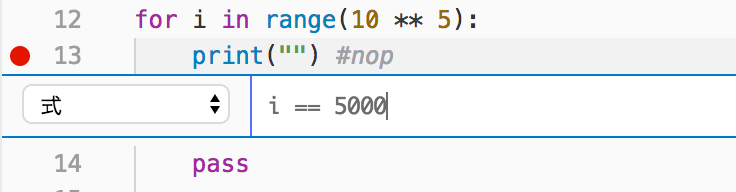
VSCode側でブレークポイントを次のように設けておきます。
また、ブレークポイントを編集し、条件付きのブレークポイントも作成しておきます。
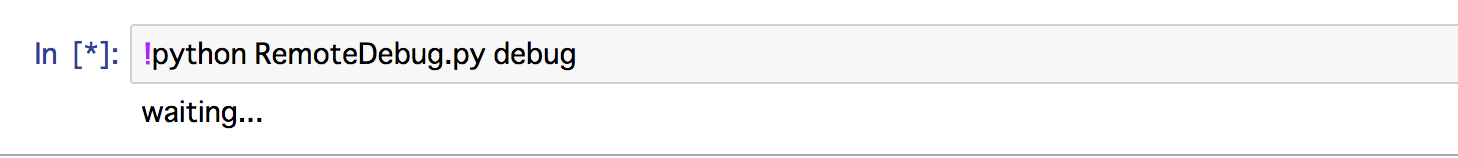
Docker側での準備
何らかの適当な方法で上記のPythonコードを debug 引数付きで実行します。Jupyter Notebookの場合は次をセルに入力し、実行します。
!python RemoteDebug.py debug
接続待ち表示になればOKです。
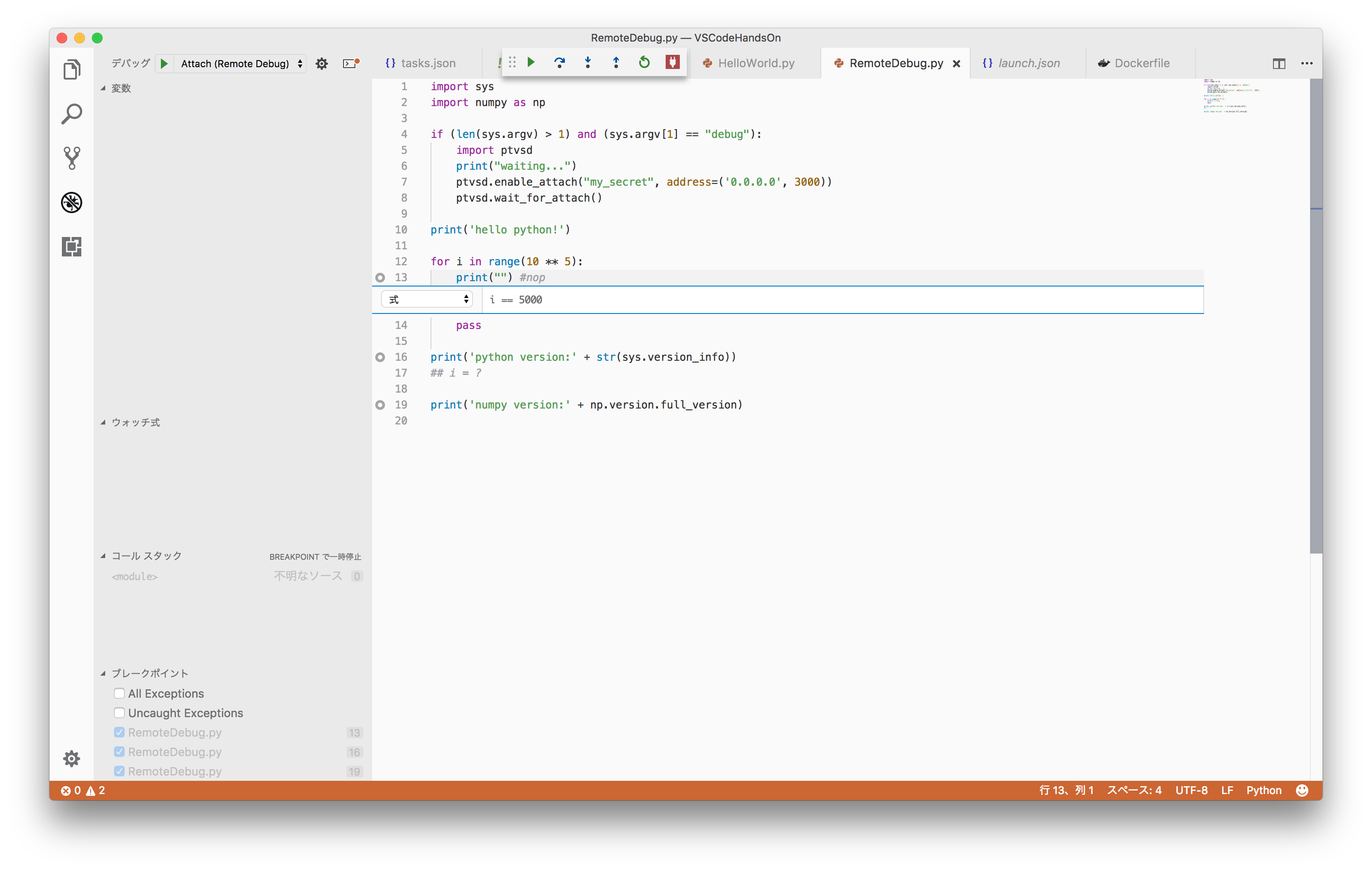
リモートデバッグ
VSCodeからデバッグを実行すると、VSCode側が次のような画面になれば接続できています。(ブレークポイントの編集用ポップアップは表示されていないのが正しいです…。)
Docker側を確認すると次の表示になっているはずです。接続待ちの状態から実行が進み、printが実行されたことがわかります。
VSCode側では変数とその中身が表示されていないため少し焦りますが、「コールスタック」中の<module>をクリックすると表示されます。
このあたりの挙動はちゃんと設定しきれていない気がしますので、要調査ですね。
終わりに
無事VSCodeでDockerの環境に接続してデバッグができました!
これでTensorFlowの環境をDockerで用意してリモートデバッグしていけますね!
あとはTensorFlowのデバッグ用にはtfdbgを使えればいいのですけれど、
明らかにターミナル前提なので諦めてDockerコンテナに乗り込んで実行する方針にします。
上記のプロジェクトをGithubに公開しましたので、お手元でお試しください。