はじめに
ケモインフォマティクスで学ぶPandasに引き続き、リピドミクス(脂質の網羅解析)を題材として、Pythonの代表的なライブラリの一つである「Matplotlib」について解説していきます。
ケモインフォマティクスの実践例を中心に説明していきますので、基本を確認したいという人は以下の記事を読んでからこの記事を読んでみてください。
散布図
Matplotlibは、グラフ描画のためのライブラリです。
データの傾向を可視化したりするのに使えます。
まずは、importでライブラリを読み込みます。
Jupyter Notebookを使っている場合は、%matplotlib inlineと書くことで、ノートブック上でグラフを描画することができます。
ここで、脂肪酸の炭素原子数や二重結合数と物性との関係を解析することを考えましょう。
%matplotlib inline
import matplotlib.pyplot as plt
abbreviations = ['FA 12:0', 'FA 14:0', 'FA 16:0', 'FA 18:0', 'FA 20:0', 'FA 22:0'] # 脂肪酸分子種の略号
Cns = [12, 14, 16, 18, 20, 22] # 脂肪酸の炭素原子数(鎖長)
logPs = [3.99, 4.77, 5.55, 6.33, 7.11, 7.89] # 脂肪酸分子種のlogPowの値
plt.scatter(Cns, logPs) # 散布図の作成
plt.xlabel('Cn') # x軸ラベル
plt.ylabel('logPow') # y軸ラベル
plt.savefig('logP_saturated-fatty-acids.png') # 散布図を画像ファイル(PNGファイル)として保存
plt.show() # 出来上がった散布図を表示
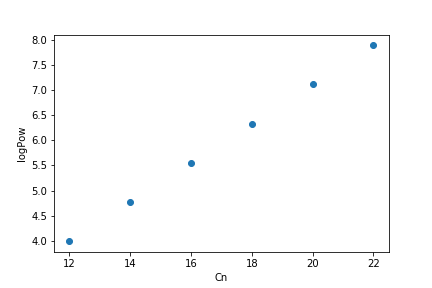
上の例では、飽和脂肪酸(炭素鎖に二重結合がない脂肪酸分子種)について、炭素原子数CnsとlogPowlogPsの関係を図示しています。
logPowというのは、「水オクタノール分配係数」で、化合物の疎水性の大きさを示しています。
今回は、logPowの値は、LIPID MAPSを参照しました。
見て分かるように、炭素原子数が多くなるにつれて、logPowの値も大きくなっていますね。
これは、炭素原子数が増えるにつれて、分子の疎水性が増すことを示しています。
不飽和脂肪酸(炭素鎖に二重結合がある脂肪酸分子種)についても同様に考えてみます。
%matplotlib inline
import matplotlib.pyplot as plt
abbreviations = ['FA 18:1', 'FA 18:2', 'FA 18:3', 'FA 18:4']
Uns = [1, 2, 3, 4] # 脂肪酸の炭素鎖の二重結合数(不飽和度)
logPs = [6.11, 5.88, 5.66, 5.44]
plt.scatter(Uns, logPs)
plt.xlabel('Un')
plt.ylabel('logPow')
plt.savefig('logP_C18-fatty-acids.png')
plt.show()
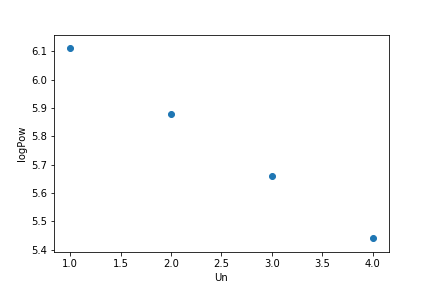
今度は、炭素原子数は同じで、二重結合数(不飽和度)を変えた時に、logPowがどう変わるかを図示しています。
二重結合が増えるにつれて、分子の疎水性が下がることが分かります。
棒グラフ
次にin vitroの実験における細胞中の脂肪酸濃度を図示することを考えます。
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
concs_ctrl = [0.1, 0.05] # コントロールの実験におけるアラキドン酸、ドコサヘキサエン酸の濃度
concs_cmpd_low = [0.05, 0.07] # 化合物添加(低用量)の実験におけるアラキドン酸、ドコサヘキサエン酸の濃度
concs_cmpd_high = [0.01, 0.08] # 化合物添加(高用量)の実験におけるアラキドン酸、ドコサヘキサエン酸の濃度
x = np.arange(len(concs_ctrl)) # 表示する脂肪酸の数
bar_width = 0.3 # 棒グラフの幅
plt.bar(x, concs_ctrl, width=bar_width, align='center') # Controlの条件の棒グラフ
plt.bar(x+bar_width, concs_cmpd_low, width=bar_width, align='center') # 化合物添加時(低用量)の条件の棒グラフ
plt.bar(x+bar_width*2, concs_cmpd_high, width=bar_width, align='center') # 化合物添加時(高用量)の条件の棒グラフ
plt.xticks(x+bar_width, ['AA', 'DHA']) # x軸データ名
plt.ylabel('Concentration (uM)')
plt.legend(('Control', 'Compound X 0.1uM', 'Compound X 1 uM')) # 凡例
plt.savefig('fatty acid concs.png')
plt.show()
ここでは、3種類の実験条件で、アラキドン酸(arachidonic acid: AA)とドコサヘキサエン酸(docosahexaenoic acid: DHA)の細胞中濃度がどう変わるかを示しています。
ちなみに、炭素原子数と二重結合数で表すと、AAは「FA 20:4」、DHAは「FA 22:6」です。
化合物Xを細胞に添加すると、AAの産生が抑制され、DHAの産生量が用量依存的に少しずつ増加しているのが分かりますね。
なお、今回の例ではn=1の実験データとしてグラフを作成しており、エラーバーはつけていません。
まとめ
ここでは、Matplotlibについて、ケモインフォマティクスで使える実践的な知識を中心に解説しました。
もう一度要点をおさらいしておきましょう。
- 2種類のデータの相関を解析する場合には散布図が使え、
matplotlib.pyplot.scatterで描けます。 - 値の大小関係を解析する場合には棒グラフが使え、
matplotlib.pyplot.barで描けます。
続いて、scikit-learnについて以下の記事で解説しています。