はじめに
ケモインフォマティクスで学ぶMatplotlibに引き続き、リピドミクス(脂質の網羅解析)を題材として、Pythonの代表的なライブラリの一つである「Matplotlib」について解説していきます。
ケモインフォマティクスの実践例を中心に説明していきますので、基本を確認したいという人は以下の記事を読んでからこの記事を読んでみてください。
製薬企業研究者がscikit-learnについてまとめてみた
データセットの準備
scikit-learnは、機械学習用のライブラリです。
ここでは、化合物の物性値から、液体クロマトグラフィー(Liquid Chromatography: LC)における保持時間(Retention Time: RT)を、部分最小二乗(partial least squares: PLS)回帰を用いて予測することを考えてみます。
まず、機械学習に用いるデータセットを作成します。
import pandas as pd
params_fatty_acids = ['Heavy atoms', 'Rotatable Bonds', 'van der Waals Molecular Volume', 'logP', 'Molar Refractivity']
lauric = [14, 10, 231.10, 3.99, 59.48]
myristic = [16, 12, 265.70, 4.77, 68.71]
palmitic = [18, 14, 300.30, 5.55, 77.95]
palmitoleic = [18, 13, 297.66, 5.33, 77.85]
stearic = [20, 16, 334.90, 6.33, 87.18]
oleic = [20, 15, 332.26, 6.11, 87.09]
linoleic = [20, 14, 329.62, 5.88, 86.99]
linolenic = [20, 13, 326.98, 5.66, 86.90]
stearidonic = [20, 12, 324.34, 5.44, 86.81]
arachidic = [22, 18, 369.50, 7.11, 96.42]
bishomo_gamma_linolenic = [22, 15, 361.58, 6.44, 96.13]
arachidonic = [22, 14, 358.94, 6.22, 96.04]
eicosapentaenoic = [22, 13, 356.30, 5.99, 95.95]
behenic = [24, 20, 404.10, 7.89, 105.65]
adrenic = [24, 16, 393.54, 7.00, 105.27]
docosapentaenoic = [24, 15, 390.90, 6.77, 105.18]
docosahexaenoic = [24, 14, 388.26, 6.55, 105.09]
df_fatty_acids = pd.DataFrame([lauric, myristic, palmitic, palmitoleic, stearic, oleic, linoleic, linolenic, stearidonic, arachidic, bishomo_gamma_linolenic, arachidonic, eicosapentaenoic, behenic, adrenic, docosapentaenoic, docosahexaenoic], columns=params_fatty_acids)
df_fatty_acids['Experimental Retention Time (min)'] = [4.53, 7.52, 11.02, 10.59, 14.45, 11.86, 9.76, 8.31, 6.71, 17.52, 11.20, 9.96, 8.27, 20.40, 12.75, 11.52, 9.84]
print(df_fatty_acids)
ここでは、説明変数として用いる物性パラメーター名のリストをparams_fatty_acidsとしています。
それぞれの物性値はLIPID MAPSのデータベースに格納されている情報を参照しています。
また、RTは、理化学研究所のPRIMeというウェブサイトで公開されている逆相LCにおけるRTデータを参照しています。
また、実際はpandas.read_csvなどでCSVファイルなどを読み込むことが多いと思います。
さらに、欠損値補完などのデータ前処理が必要になることが多いです。
モデルの構築
次に、予測モデルの構築と、モデルを用いた予測値の算出を行います。
from sklearn.cross_decomposition import PLSRegression
X = df_fatty_acids[params_fatty_acids] # 説明変数
y = df_fatty_acids['Experimental Retention Time (min)'] # 目的変数
pls_rt = PLSRegression()
pls_rt.fit(X, y) # PLS予測モデルを構築
y_pred = pls_rt.predict(X) # 予測値を算出
df_fatty_acids['Predicted Retention Time (min)'] = y_pred
df_fatty_acids['Diff (min)'] = df_fatty_acids['Predicted Retention Time (min)'] - df_fatty_acids['Experimental Retention Time (min)']
df_fatty_acids['Accuracy (%)'] = (df_fatty_acids['Diff (min)'] / df_fatty_acids['Experimental Retention Time (min)']) * 100
print(df_fatty_acids)
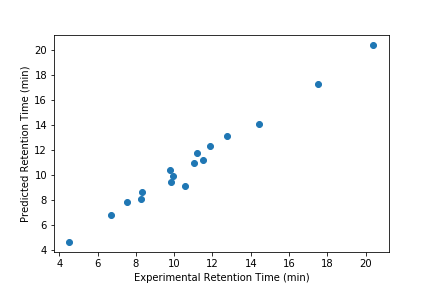
実測値と予測値の関係を図示してみると、以下のようになります。
%matplotlib inline
import matplotlib.pyplot as plt
plt.scatter(y, y_pred)
plt.xlabel('Experimental Retention Time (min)')
plt.ylabel('Predicted Retention Time (min)')
plt.savefig('rts_fatty_acids.png')
plt.show()
今回のデータでは実測値と予測値がよく合っていそうですね。
構築したモデルの当てはまり度合いはr2_scoreで確認することができます。
from sklearn.metrics import r2_score
print(r2_score(y, y_pred))
r2_scoreは、0から1の間の値をとり、1に近いほど実測値と予測値がよく合っていることを示します。
今回のデータでは、r2_scoreが0.98を越える値となっており、かなり当てはまりの良いモデルということになります。
今回は、RTを予測するのに、5種類の物性パラメータを用いましたが、このうちどれが予測に大きく寄与しているのかを見てみます。
print(pls_rt.coef_)
この結果から、今回のデータでは、Rotable Bondsに対する係数(の絶対値)が3.44と最も大きくなっており、この物性値がRTの予測に強く寄与していることが分かります。
モデルを用いた予測
以上で予測モデル構築に用いたデータの予測精度について議論してきましたが、最後に、モデル構築に用いなかったデータがどれほどの精度で予測できるのか見てみます。
lignoceric = [26, 22, 438.70, 8.67, 114.88]
x_lignoceric = pd.DataFrame([lignoceric], columns=params_fatty_acids)
y_pred_lignoceric = pls_rt.predict(x_lignoceric)
y_exp_lignoceric = 22.31 # 実測値
print(y_exp_lignoceric)
print(y_pred_lignoceric)
ここでは、lignoceric acid(FA 24:0)のRTを予測してみました。
予測値と実測値の乖離が1.2分程度となっています。
この差を大きいとみるか小さいとみるかは色々な見方があると思いますが、個人的にはやや予測精度が低いと思います。
理由としては、lignoceric acidが、モデル構築に用いたデータセットに含まれるどの脂肪酸分子種よりも疎水性が高い分子種であり、モデル構築の段階でlignoceric acidの物性に近いデータのfittingが行われていなかったことも関係していると考えられます。
以上のような手順でPLS回帰を行うことができます。
ここでは触れませんでしたが、PLS回帰を行うにあたり、潜在変数の数n_componentsも重要になってきます。
今回はデフォルト値の2を用いましたが、ここを変えることで予測精度も少しずつ変わってきます。
また別の機会に解説したいと思います。
まとめ
ここでは、scikit-learnについて、ケモインフォマティクスで使える実践的な知識を中心に解説しました。
もう一度要点をおさらいしておきましょう。
- scikit-learnを用いることで、機械学習を手軽に行うことができる。
- 機械学習はデータの前処理、モデル構築、予測という流れで行う。
- 構築されたモデルの
r2_scoreや、予測値と実測値の差などを見ながら進めていく。