はじめにお礼
まさか、前回の記事がこんなに伸びてデイリートレンドに載るなんて思いませんでした!
皆様多くのLGTM, ストックありがとうございます!
そして何よりも...

開発者が引用RTするなんて夢にも思いませんでした... ありがとうDaniel Sjoberg、私はこの素晴らしいパッケージを忘れない。
さて、今回は前回の記事の続きとして、実際に**データを整形(pivot_longer + nest) → 図表を同時に作成 (ggplot + gtsummary) → レポート化(Rmarkdown)**という一本化されたプロセスを見ていきたいと思います。
これを通してgtsummaryの実践的な使い方をご紹介します。
完成品はこんな感じで
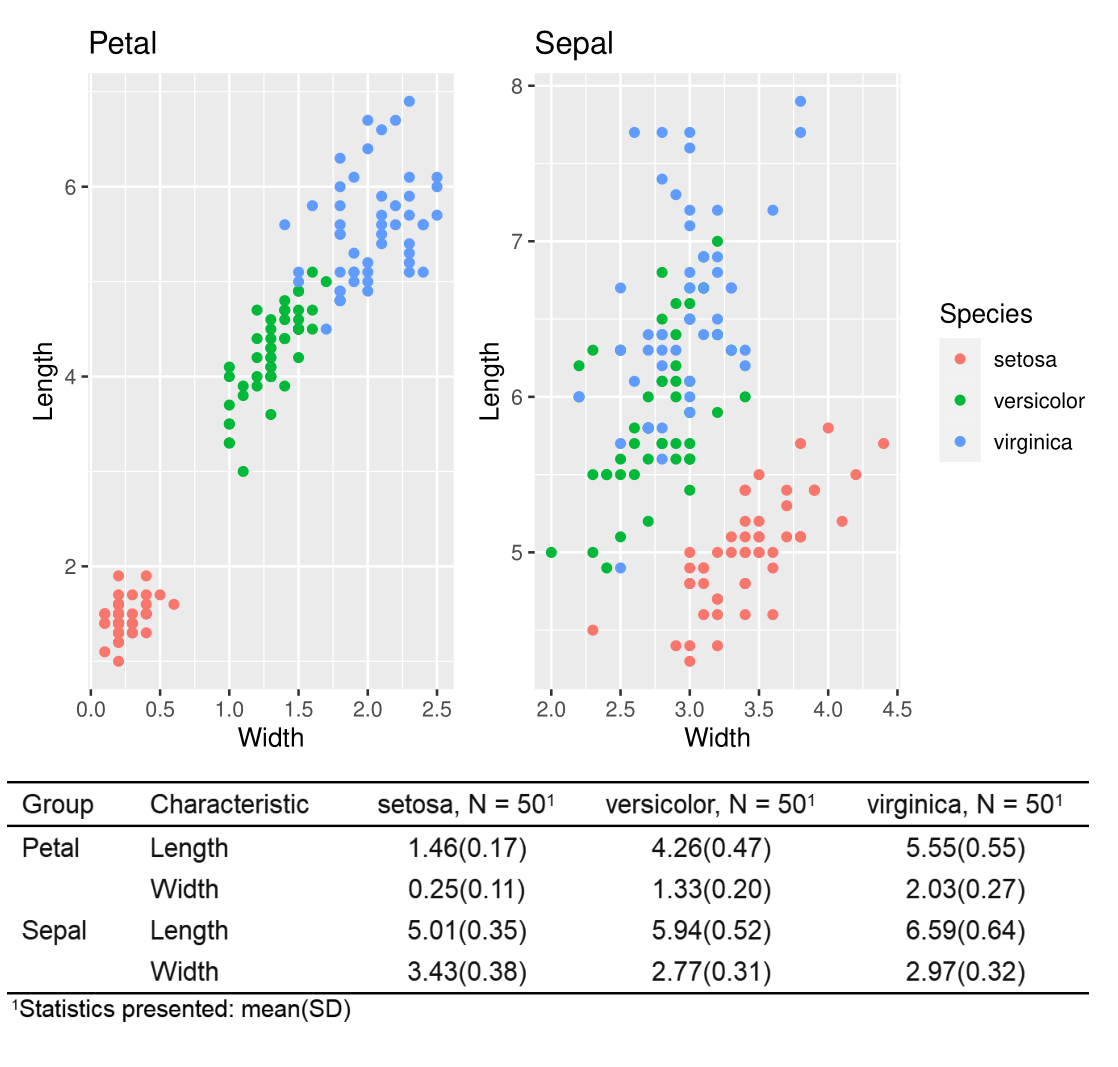
レポートっぽく仕上がっていて、いい感じではないでしょうか?
これを一つのデータから作ります。使うのはみんな大好きiris
注意事項として、Rのバージョンは4.0.2を使用しています。
まずはpivot_longer()
irisのデータを見てみましょう。
library(tidyverse)
iris %>% tibble()
# A tibble: 150 x 5
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
<dbl> <dbl> <dbl> <dbl> <fct>
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
7 4.6 3.4 1.4 0.3 setosa
8 5 3.4 1.5 0.2 setosa
9 4.4 2.9 1.4 0.2 setosa
10 4.9 3.1 1.5 0.1 setosa
# ... with 140 more rows
いつもの見慣れた表が出てきます。これをもう少しTidy dataに変えます。
gather()はもう古いので、pivot_longer()を使いましょう。
iris_longer <-
iris %>%
pivot_longer(
cols = -Species,
names_to = c("key", ".value"),
names_sep = r"(\.)"
)
iris_longer
# A tibble: 300 x 4
Species key Length Width
<fct> <chr> <dbl> <dbl>
1 setosa Sepal 5.1 3.5
2 setosa Petal 1.4 0.2
3 setosa Sepal 4.9 3
4 setosa Petal 1.4 0.2
5 setosa Sepal 4.7 3.2
6 setosa Petal 1.3 0.2
7 setosa Sepal 4.6 3.1
8 setosa Petal 1.5 0.2
9 setosa Sepal 5 3.6
10 setosa Petal 1.4 0.2
# ... with 290 more rows
R 4.0以降ではRAW文字列を使えるようになっています。r"()"で括ることで、エスケープシーケンスを無視します。
また、names_to内で".value"を指定していますが、これはnames_sep で切ったものが直にくるという意味を持った変数のようです。
nest!
nestするにはgroup_nest()!
iris_nest <- iris_longer %>% group_nest(key)
iris_nest
# A tibble: 2 x 2
key data
<chr> <list<tbl_df[,3]>>
1 Petal [150 x 3]
2 Sepal [150 x 3]
これでPetal, Sepalで分かれたデータができました。
ここからがnestの強さです。mutateしてmapしていきましょう。
もう一気に書いたものがこちらです。
library(gtsummary)
iris_nest <-
iris_nest %>%
mutate(
plot = map2(
.x = data, .y = key,
~ ggplot(.x, aes(Width, Length, colour = Species)) +
geom_point() +
theme(legend.position = "none") +
ggtitle(paste0(.y))
),
tbl = map(
.x = data,
~ tbl_summary(
.x,
by = Species,
statistic = list(all_numeric() ~ "{mean}({sd})")
)
)
)
iris_nest
key data plot tbl
<chr> <list<tbl_df[,3]>> <list> <list>
1 Petal [150 x 3] <gg> <tbl_smmr>
2 Sepal [150 x 3] <gg> <tbl_smmr>
ちょっとわかりにくいですが、plotという列とtblという列をmutate関数で作っています。
ここでmap関数を使っているのですがggplotの方ではggtitle()を使ってタイトルを付けたかったため、map2()を使いました。
plotでは各列に対してx軸にWidth, y軸にLengthを取った散布図を作り、tblではデータの要約値をgtsummary()でまとめました。
一部を見てみましょう。
iris_nest$plot[1]
iris_nest$tbl[1]
確かにPetalについてのFigとTableが出力されています。
しかしこのままだとFig、tableが計4種類も作成されてしまい、ごちゃごちゃしてしまいます。
wrap_plots(), tbl_stack(), tbl_merge()
そこで便利なのがpatchwork パッケージがサポートしているwrap_plots()とgtsummaryのtbl_stack()です!
wrap_plots()については@kilometer氏の記事が大いに参考になります。今回は詳細を割愛します。
実際のコードがこちらです。
iris_nest$plot %>% wrap_plots(ncol = 2)+theme(legend.position="right")
工夫として、iris_nest$plot内では凡例をつけないように指定しています(theme(legend.position = "none"))。
wrap_plots()にする際、 +theme(legend.position="right")を付けることでまとめた上で右側に凡例が来るようにできます。
(その他の位置につけることは恐らく無理だと思います...誰か知っていたら教えて下さい)
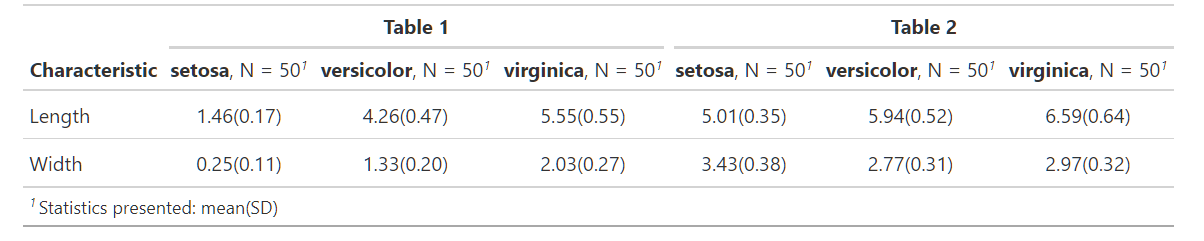
tbl_stack()は2種以上のtbl_summary()を一つにまとめる関数になっており、行方向に結合します。よく似た関数にtbl_merge()がありますが、これは列方向にテーブルを結合させます。
例えば...
iris_nest$tbl %>% tbl_stack()
iris_nest$tbl %>% tbl_merge()
見ていただいてわかるように、tbl_stack()では行数が増え、tbl_merge()では列数が増えています。これを利用して見たものがこちらです。
iris_nest$tbl %>% tbl_stack(group_header = iris_nest$key)
Rmarkdownでpdfに出力
Rmarkdownに関しては@tomotagwork氏の記事など、多くの文献があるので割愛します。
最終的にはこのようなコードでpdfを出力しました。gtsummaryは幅広いtableに変換可能という強みを持っているので、今回はflextableにしてみました。
```{r setup, include=FALSE}
---
knit: (function(inputFile, encoding) {
rmarkdown::render(
inputFile,
encoding = encoding,
output_dir = "C:/Users/",
output_file = paste0(Sys.Date(),".pdf"))
})
output:
pdf_document:
latex_engine: lualatex
documentclass: ltjsarticle
---
```
```{r, include=FALSE}
library(gtsummary)
library(tidyverse)
library(patchwork)
library(flextable)
```
```{r, include=FALSE, echo=FALSE}
theme_gtsummary_journal("jama")
iris_nest <-
iris %>%
pivot_longer(
cols = -Species,
names_to = c("key", ".value"),
names_sep = r"(\.)"
) %>%
group_nest(key) %>%
mutate(
plot = map2(
.x = data, .y = key,
~ ggplot(.x, aes(Width, Length, colour = Species)) +
geom_point() +
theme(legend.position = "none")+
ggtitle(paste0(.y))
),
tbl = map(
.x = data,
~ tbl_summary(
.x,
by = Species,
statistic = list(all_numeric() ~ "{mean}({sd})")
)
)
)
```
```{r, echo=FALSE}
iris_nest$plot %>% wrap_plots(ncol = 2)+theme(legend.position="right")
iris_nest$tbl %>% tbl_stack(group_header = iris_nest$key) %>% as_flex_table()
```
最後に
やはりRの強みの一つとして、レポート作成の際データを整形しながら処理可能であるという点が挙げられます。
かねてより言われているggplotとRmarkdownの親和性の高さに加え、今回gtsummaryも強力な武器として追加されたと思います。
これで美しいレポートづくりがさらに加速し、より良くデータを可視化することも可能ですし、研究者であればそのまま論文に...なんてこともできそうです。
今後も開発の進んでいくパッケージであると思いますので、注視していきましょう!
Enjoy!