概要
こちらは創薬 Advent Calendar 20174日目の記事です。
化合物データベースの構築をより簡便にするためにDocker-composeを作ったよというお話です。
環境
Mac mini (Late 2014) OS: High Sierra (Jupyter Notebookのユーザー側のパソコン)
Docker version 17.09.0-ce, build afdb6d4
Dockerで動いているのでおそらくDockerさえインストールされていたらどの環境でも動くと思います。
前提条件
Django、Docker、Docker-composeの使い方はある程度理解している前提で話を進めます。
(でも結構細かく説明したので初めての方もやってみても面白いかもしれません)
背景
長くなってしまったので、Docker-composeのところだけ読みたい方は読み飛ばして方法へ進んでください。
PostgreSQL, MySQLなどの無償で利用が可能なリレーショナルデータベースはありますが、
まだまだ環境構築が大変、SQL言語がとっつきにくいとなかなか個人で使ってみようというのは
ハードルが高い状況です。
そこでもっと簡単に使えるようにならないかなぁと思って環境をDocker-composeで作ってみました。
1. なぜ化合物データベースが必要なのか
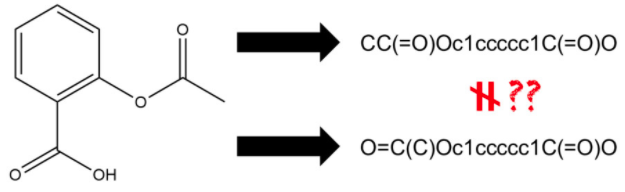
1.1 単純な文字列として保存すると支障が出る
このスライドにもまとめていますが、簡単に言うと化学構造は単純な文字情報では保存できず、
また従来のデータベースでは化学構造に対して行うような検索ができないということです。
- SMILES
どこをスタートとするかで書かれる文字列が異なります。また古いバージョンのSMILESだと立体構造の情報が含まれない場合もあります。(立体構造の情報を含むSMILESをISOMERIC SMILESと呼ぶ場合もあります)
- Canonical SMILES
どこをスタートとするか決めるCanonical SMILESもアプリケーションによって用いているアルゴリズムが異なる可能性があるので様々なアプリケーションで作成したCanonical SMILESを用いる場合一致しない可能性があります。 - InChI
これはSMILESと比べると最近に開発されただけあってかなりいいと思います。もし以下に示す検索の種類で、完全一致検索のみを用いるならこちらを採用し文字列として保存してもいいと思います。
1.2 化学構造ならではの検索をしたい
化学構造は例えばGoogle検索で「創薬」という言葉を検索するような検索方法もある一方で、以下に示すような化学構造だからこそこういう検索をしたいということがあります。
- 完全一致検索
検索にかけた化学構造(以下、検索クエリ)と完全に構造が一致する化学構造を検索ヒットとして抽出する検索方法です。立体異性体も検索ヒットとしてみなすかなどのオプションがある場合があります。 - 部分構造検索
検索クエリを部分構造として含む化学構造を検索ヒットとして抽出する検索方法です。例えばフェノールはベンゼン環を検索クエリとし部分構造検索をした場合、検索ヒットします。 - 類似性検索
例えばfingerprintなどをつかって化学構造同士がどれくらいに似ているかを求め(類似度と呼ばれる指標を計算します)、にている化学構造を検索ヒットとして抽出します。
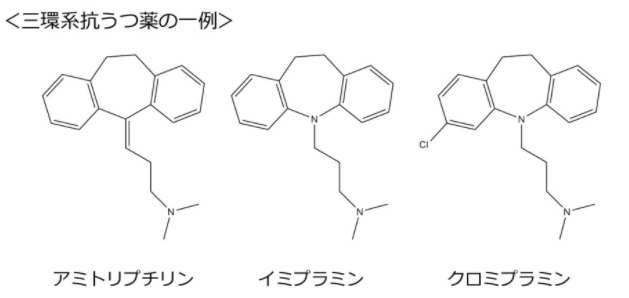
例えば以三環系抗うつ薬のように似ている化学構造は同じ性質を示すことが多いです。そこで創薬では新規の活性を持つ構造が見つかるとその類似構造や共通の部分構造をもつ化合物にも活性があるのではと試験を行うことがあります。このことからも部分構造検索や類似構造検索の機能は重要であるといえます。
以上の検索をする場合は単純な文字列ではなく化学構造データとして保存し、さらにこれらの検索機能を持つデータベースが必要です。
これまでのことから化学構造を扱う化合物データベースがあるとよさそうだなと思ってもらえたら嬉しいです。
1.3 化合物データベースの一例
じゃあ化合物データベースを使うのはいいとして具体的にどういうものがあるのかという話を背景の最後にします。
以下のような有償、無償の化合物データベースやデータベースカートリッジがあります。
| 名前 | 参考サイト | 無償かどうか |
|---|---|---|
| CambridgeSoft | http://www.cambridgesoft.com/ | |
| BIOVIA Isentris | http://ls.ctc-g.co.jp/products/accelrys/accelrys_isentris.html | |
| Pgchem | http://www.pgchem.sk/ | 無償 |
| RDKit database cartridge | http://www.rdkit.org/docs/Cartridge.html | 無償 |
| Bingo | http://lifescience.opensource.epam.com/bingo/index.html | アカデミックフリー? |
データベースカートリッジとは本来データベースにない機能を拡張するためのプラグインみたいなものです。
今回の記事では無償で利用できるようにPostgreSQLにRDKit database cartridgeを組み合わせた化合物データベースを使いたいと思います。
問題点
ただし、まだ以下に示すような問題点があります。
- SQL言語を習得する必要がある。他の言語とちょっと違って独特で大変そう
- 環境構築が大変そう
これらの問題に関して今回は以下のような方法で解決したいと思います。
- SQL言語を習得する必要がある。他の言語とちょっと違って独特で大変そう
O/Rマッパーを使ってPythonでデータベースにアクセスしましょう - 環境構築が大変そう
Docker-composeを使って簡単に環境構築しましょう
1.SQL言語を習得する必要がある。他の言語とちょっと違って独特で大変そう
SQL言語は独特かはまた別の議論としておいといて、新たに学ぶなら学習コストがかかるのは事実です。
そこでO/Rマッパーを使うことで、すでにみなさんが習得しているプログラム言語を使ってデータベースにアクセスし、望む検索などを行えます。
O/Rマッパー: 語弊があるかもしれませんが簡単に言うと一般のプログラミング言語でデータベースにアクセスできることを指します。詳しくはこちらを参照してください。
組み込んでいるWeb Frame Applicationの一例
| 名前 | 用いているプログラム名 |
|---|---|
| Ruby on Rail | Ruby |
| Django | Python |
| Flask | Python |
| Apatch Click | JAVA |
そこで今回はWeb Frame ApplicationであるDjangoのO/Rマッパーの機能の部分のみを利用しちゃおうと思います(もちろんDjango本来の機能である自社内化合物データベースサイトなども作れますよ)。
Djangoは従来のデータベースにのみしか対応していないのでdjango-rdkitという拡張モジュールを使うと化合物データベースにも対応するようになります。
こちらも一緒に導入しちゃいます。
これでSQL言語を学ぶ必要があるという問題は解消されます。
2. 環境構築が大変そう
化合物データベースの構築は大変ですし、リレーショナルデータベースを扱うならSQL言語を学ぶ必要があります。
例えば今回用意した環境構築を一から自分で構築するなら、
- データベースとして従来のPostgreSQLを構築
- RDKit database cartridgeを使ってデータベースを拡張
- SQL言語を使いたくないのでO/Rマッパーとしてdjango-rdkitを用いてDjangoと連携
といったことをしなくてはいけません。
そこでようやくタイトルを回収できますが、今回はその一手間をすべてDocker-composeで行えるものを作ったのでぜひ皆さん使ってみてくださいという話です。
Docker: 仮想環境を構築するツール。Moby Dockerがかわいい。
Docker-compose: 複数の仮想環境を連携しひとまとめにする機能。今回はデータベースとDjangoをひとまとめに連携させている。
Docker-composeを用いることで環境構築が大変とういう問題も解消されます。
方法
まとめると今回は
PostgreSQL + RDKit database cartridge で化合物データベースを構築し
django + django-rdkit でPythonでその化合物データベースにアクセスしたり操作したりできる環境を構築します。
1. Dockerのインストール
Dockerをインストールしましょう。
(DockerはWindows 32bitは対応していないそうです。すいません)
2. リポジトリのクローン
docker-compose-django-rdkitのリポジトリを自身の望むフォルダにクローンしてください。
例えば、ホームディレクトリ直下で良いなら以下のようなコードになります。
cd ~
git clone https://github.com/yamasakih/docker-compose-django-rdkit
その後、docker-compose-django-rdkitというフォルダができていると思うのでその中に移動しましょう。
cd docker-compose-django-rdkit
3. Docker-composeの起動
続けてDocker-composeを起動します。
docker-compose up
はい。これだけでもう
- PostgreSQL + RDKit database cartridgeの化合物データベースが構築され
- django + django-rdkitの仮想環境も構築されています
はやい!今までの無駄な文章はなんだったんだろうか
> $ docker-compose up
Starting dockercomposedjangordkit_db_1 ...
Starting dockercomposedjangordkit_db_1 ... done
Starting dockercomposedjangordkit_web_1 ...
Starting dockercomposedjangordkit_web_1 ... done
Attaching to dockercomposedjangordkit_db_1, dockercomposedjangordkit_web_1
db_1 | LOG: database system was interrupted; last known up at 2017-11-08 00:05:40 UTC
db_1 | LOG: database system was not properly shut down; automatic recovery in progress
db_1 | LOG: invalid record length at 0/18841E0
db_1 | LOG: redo is not required
db_1 | LOG: MultiXact member wraparound protections are now enabled
db_1 | LOG: database system is ready to accept connections
db_1 | LOG: autovacuum launcher started
web_1 | Performing system checks...
web_1 |
web_1 | System check identified no issues (0 silenced).
web_1 | December 04, 2017 - 01:27:34
web_1 | Django version 1.11.7, using settings 'package_root.settings'
web_1 | Starting development server at http://0.0.0.0:8000/
web_1 | Quit the server with CONTROL-C.
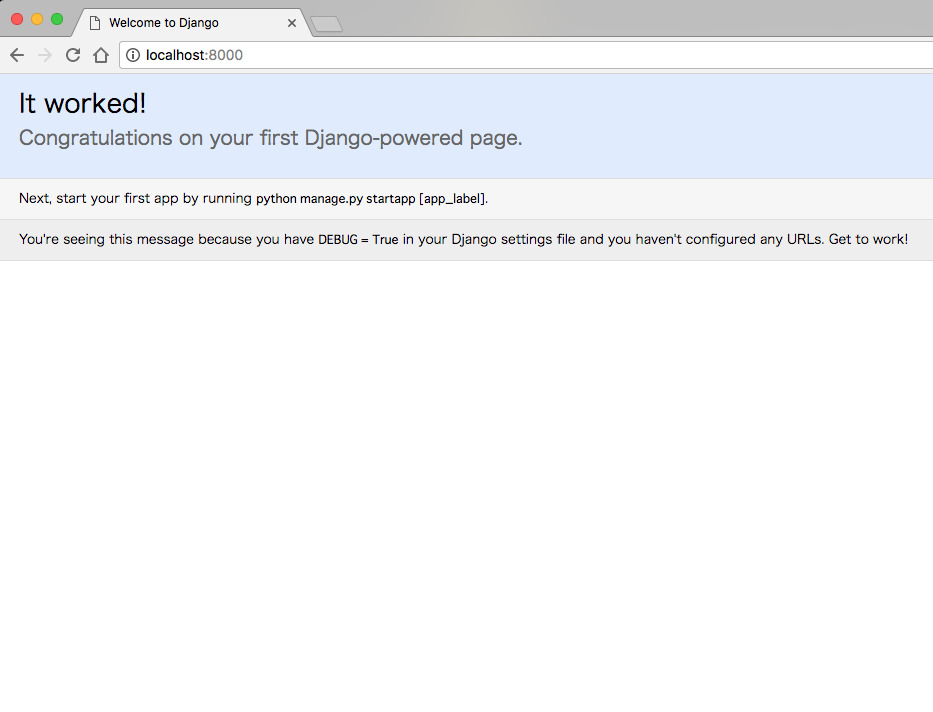
試しにGoogle Chromeなどのブラウザを立ち上げhttp://0.0.0.0:8000あるいはhttp://localhost:8000のいずれかにアクセスすると
仮想サーバーにアクセスすることができ以下の画面が表示されると思います。
たまに最初の1回目だけ正しく表示されない場合があるのでその場合はターミナル上でCtrl + Cで一度仮想サーバーをストップしサイドdocker-compose restartしてからアクセスしてみてください。
(仮想環境dbより先に仮想環境webがbuildされてしまうからでしょうか?)
ちなみに仮想環境を終了する場合にはdocker-compose stopと実行しましょう。
私のパソコンだと終了に少し時間がかかります。
> $ docker-compose stop
Stopping dockercomposedjangordkit_web_1 ... done
Stopping dockercomposedjangordkit_db_1 ... done
4. データベース内にテーブルを作成し化合物を登録してみる
では最後にチュートリアルとして実際に化学構造情報を保存できるテーブルをデータベース内に作成し、化合物を登録して見ましょう。
docker-composeをstopしていた場合はdocker-compose restartで再び起動しておいてください。
> $ docker-compose restart
Restarting dockercomposedjangordkit_web_1 ... done
Restarting dockercomposedjangordkit_db_1 ... done
再起動後はバックエンドで仮想環境が動いている状態になります。
4.1 初期化のmigrateを行う
まずサブコマンドmigrateを行い、Djangoでデフォルトで作られるテーブルを作成したり、化合物データベースの拡張機能を有効にしたりしましょう。
従来の./manage.py migrateなどのコマンドは先頭にdocker-compose run webとつけることで実行できます。
したがって今回の場合以下のように実行します。
> $ docker-compose run web ./manage.py migrate
Starting dockercomposedjangordkit_db_1 ... done
Operations to perform:
Apply all migrations: admin, auth, contenttypes, django_rdkit, sessions
Running migrations:
Applying contenttypes.0001_initial... OK
Applying auth.0001_initial... OK
Applying admin.0001_initial... OK
Applying admin.0002_logentry_remove_auto_add... OK
Applying contenttypes.0002_remove_content_type_name... OK
Applying auth.0002_alter_permission_name_max_length... OK
Applying auth.0003_alter_user_email_max_length... OK
Applying auth.0004_alter_user_username_opts... OK
Applying auth.0005_alter_user_last_login_null... OK
Applying auth.0006_require_contenttypes_0002... OK
Applying auth.0007_alter_validators_add_error_messages... OK
Applying auth.0008_alter_user_username_max_length... OK
Applying django_rdkit.0001_setup... OK
Applying sessions.0001_initial... OK
4.2 tutorial_applicationを作成する
続いて以下のように実行して新しいアプリケーションtutorial_applicationを作成しましょう。
> $ docker-compose run web ./manage.py startapp tutorial_application
Starting dockercomposedjangordkit_db_1 ... done
tutorial_applicationという名前のフォルダがdocker-compose-django-rdkit内に作成されていると思います。
> $ ls
Dockerfile docker-compose.yml package_root/ tutorial_application/
LICENSE manage.py* requirements.txt
続けて./package_root/settings.pyのINSTALLED_APPSの項目にtutorial_applicationを追加しましょう。
tutorial_applicationはstartappで作成したアプリ名に適宜変更してください。
INSTALLED_APPS = [
'django.contrib.admin',
'django.contrib.auth',
'django.contrib.contenttypes',
'django.contrib.sessions',
'django.contrib.messages',
'django.contrib.staticfiles',
'django_rdkit',
'tutorial_application',
]
注:同じ名前のフォルダがあると間違いの元なので、私は間違いがないようにDjangoのsettings.pyなどのファイルができるフォルダをデフォルトの名前docker-compose-django-rdkitからpackage_rootに変更しています。なぜ自動で同じ名前で作られるんだ
4.3 テーブルCompoundを定義する
./tutorial_application/models.pyに以下のように追加しましょう。
from django.db import modelsとデフォルトで書いている場合は削除してください。
from django_rdkit import models
class Compound(models.Model):
name = models.CharField(max_length=256)
molecule = models.MolField()
def __str__(self):
return self.name
これは化合物データベース内に文字列CharFieldを保存するnameと分子構造MolFieldを保存するmoleculeという2つのカラムを含むテーブルを作るという定義になります。
Djangoではテーブルの定義はこのようにクラスを用いることで行えます。`
| 名前 | 化学構造 |
|---|---|
| Benzen | c1ccccc1 |
| Aspirin | CC(=O)Oc1ccccc1C(=O)O |
| Oseltamivir | CCC(CC)OC1C=C(CC(C1NC(=O)C)N)C(=O)OCC |
こんな感じの運用を想定しています。上の化学構造部分にはSMILESキーを書いていますが実際にはSMILESキーでは保存されていません。
4.4 テーブルCompoundを作成する
では実際に定義したテーブルCompoundをデータベース内に作成しましょう。
まず以下のようにサブコマンドmakemigrationsを用いてテーブルの作成の準備をします。
> $ docker-compose run web ./manage.py makemigrations tutorial_application
Starting dockercomposedjangordkit_db_1 ... done
Migrations for 'tutorial_application':
tutorial_application/migrations/0001_initial.py
- Create model Compound
続けて以下のように実行し、テーブルを作成します。
例として以下のように表示されたらテーブルが無事作成されています。
> $ docker-compose run web ./manage.py migrate tutorial_application
Starting dockercomposedjangordkit_db_1 ... done
Operations to perform:
Apply all migrations: tutorial_application
Running migrations:
Applying tutorial_application.0001_initial... OK
4.5 データベース内のテーブルにアクセスしてみる
最後にPythonでデータベース内のテーブルにアクセスし化合物データを登録してみましょう。
化学データベースにアクセスするにはshellサブコマンドを用います。
docker-compose run web ./manage.py shell
上記のように実行するとPythonインタープリターが立ち上がります。この環境下ではデータベースにアクセスができます。
4.5.1 データの登録
from tutorial_application.models import Compound
まずCompoundテーブルにアクセスするためには./tutorial_application/models.pyにあるCompoundクラスをインポートします。
Compound.objects.create(name='benzen', molecule='c1ccccc1')
Compound.objects.createとすることでcreateメソッドを実行することができます。
createメソッドはCompoundテーブルに新規にデータを登録するメソッドです。各カラムname, moleculeの値は引数として与えます。
ちょうどクラスのオブジェクトのコンストラクタと同じだと思ってもらえたらイメージしやすいかなと思います。
これで新しいデータが登録されました。
登録されているデータの数を全て確認するには以下のように実行します。
続けてAspiririnとOseltamivirも登録しましょう。
Compound.objects.create(name='Aspirin', molecule='CC(=O)Oc1ccccc1C(=O)O')
Compound.objects.create(name='Oseltamivir', molecule='CCC(CC)OC1C=C(CC(C1NC(=O)C)N)C(=O)OCC')
4.5.2 全データの取り出し
登録されているデータ全てを取り出すにはCompound.objects.all()を使います。
Compound.objects.all()
取り出した数を知りたいならCompound.objects.count()ですね。
Compound.objects.count()
4.5.3 部分構造検索
部分構造検索にはhassubstructを化学構造を保存しているカラム名(今回はmolecule)の後ろにアンダーバーを2つつけて使います。
さらに検索したい部分構造は適切にわたすためにValue関数でラッパーしSMILESでわたす必要があります。
例えばベンゼン環を部分構造として持つ化合物だけを取り出したいときは以下のように実行します。
from django_rdkit.models import Value
Compound.objects.filter(molecule__hassubstruct=Value('c1ccccc1'))
<QuerySet [<Compound: benzen>, <Compound: Aspirin>]>
ベンゼン環を持つbenzenとAspirinのみが検索ヒットしています。
4.5.4 検索ヒットしたデータの取り扱い
ちなみに検索したい化学構造が複雑でSMILESが書けなくてもRDKitを使うことができるので大丈夫です。
例えばデータベースに保存されているOseltamivirのSMILEは以下のように取り出すことができます。
compound = Compound.objects.get(id=3)
compound
<Compound: Oseltamivir>
検索ヒットが必ず一個となる検索条件の場合はgetメソッドを使うと楽です。今回はid=3で検索しています。
実はDjangoでは自動的にidというカラムもテーブルCompoundに作られて1,2,3,...と割り振られます。
Oseltamivirは3番目に登録したのでid=3です。
さらにテーブルから取り出したデータはclass Compoundのオブジェクトとして扱うことができます。
したがってcompoundはフィールドid, name, moleculeを持っています。
compound.id
3
compound.name
'Oseltamivir'
compound.molecule
<rdkit.Chem.rdchem.Mol object at 0x7f61dc605c38>
しかもcompound.moleculeをみるとこれはRDKitで扱われている化学構造のMolオブジェクトです。
そうなんですデータベースから取り出した後はRDKitのMolオブジェクトとして扱うことができるのです。
試しにRDKitでMolオブジェクトをSMILESに変換するChem.MolToSmiles関数を使ってみましょう。
from rdkit import Chem
Chem.MolToSmiles(compound.molecule)
'CCOC(=O)C1=CC(OC(CC)CC)C(NC(C)=O)C(N)C1'
見事にSMILESを取り出すことができましたね。このようにRDKitを日頃使っている方にとってはすごい扱いやすいデータ構造になっています。
最後にあえてこのSMILESで完全一致検索してみましょう。当然Oseltamivirが検索ヒットします。
完全一致検索はmolecule=Value(検索したいSMILES)としたらできます。
smiles = Chem.MolToSmiles(compound.molecule)
Compound.objects.filter(molecule=Value(smiles))
<QuerySet [<Compound: Oseltamivir>]>
最後に4.5で実行したコードをまとめておきます。
> $ docker-compose run web ./manage.py shell
Starting dockercomposedjangordkit_db_1 ... done
Python 3.6.0 |Continuum Analytics, Inc.| (default, Dec 23 2016, 12:22:00)
[GCC 4.4.7 20120313 (Red Hat 4.4.7-1)] on linux
Type "help", "copyright", "credits" or "license" for more information.
(InteractiveConsole)
>>> from tutorial_application.models import Compound
>>> Compound.objects.create(name='benzen', molecule='c1ccccc1')
<Compound: benzen>
>>> Compound.objects.create(name='Aspirin', molecule='CC(=O)Oc1ccccc1C(=O)O')
<Compound: Aspirin>
>>> Compound.objects.create(name='Oseltamivir', molecule='CCC(CC)OC1C=C(CC(C1NC(=O)C)N)C(=O)OCC')
<Compound: Oseltamivir>
>>> Compound.objects.all()
<QuerySet [<Compound: benzen>, <Compound: Aspirin>, <Compound: Oseltamivir>]>
>>> Compound.objects.count()
3
>>> from django_rdkit.models import Value
>>> Compound.objects.filter(molecule__hassubstruct=Value('c1ccccc1'))
<QuerySet [<Compound: benzen>, <Compound: Aspirin>]>
>>> compound = Compound.objects.get(id=3)
>>> compound
<Compound: Oseltamivir>
>>> compound.id
3
>>> compound.name
'Oseltamivir'
>>> compound.molecule
<rdkit.Chem.rdchem.Mol object at 0x7fcae75917a0>
>>> from rdkit import Chem
>>> Chem.MolToSmiles(compound.molecule)
'CCOC(=O)C1=CC(OC(CC)CC)C(NC(C)=O)C(N)C1'
>>> smiles = Chem.MolToSmiles(compound.molecule)
>>> Compound.objects.filter(molecule=Value(smiles))
<QuerySet [<Compound: Oseltamivir>]>
4.6 その他
チュートリアルを通すことでSQL言語を全く知らない方でもPythonのみでデータベースにアクセスすることが可能だと実感してもらえたと思います。
大量データの登録方法や類似構造検索などその他の機能はこちらを参考にしてみてください。
django-rdkit/tutorial.rst
今後の課題
私が作ったDocker-composeは構築は簡単ですが、弱点の一つとして2.5GBぐらいとすごい容量を使うことがあげられます。
今後はより容量が軽いDockerを作成しそれをベースにしようかなと思います。
あと課題ではないですが無料のPyCharmではDockerのPythonをインタプリターと設定できないのでファイル編集とかがvimとかでしたり
代理になりそうなインタープリターを別途用意する必要があります。
有料のPyCharmではDockerのPythonもインタープリターに設定できるそうです。
今後はDjangoの機能をちゃんと活かして化合物データベースをどう作っていけばなどもGithubのリポジトリで追加していきたいと思っているので
興味のある方はGithubのリポジトリにStarをつけたりWatchしたりむしろ一緒に開発してみましょう。
docker-compose-django-rdkit
最後に
以上でDjango + RDKit database cartridge のDocker-composeで化合物データベースを作った話を終わりにしたいと思います。
現在、同様にDjangoからではなくJupyter Notebookから化合物データベースにアクセスするDocker-composeも作成中です。
そちらの方がより直感的に操作できるかな?と思っています。興味ある方はぜひ気長にお待ちください。
化合物データベースを効果的に使って、よき創薬ライフを過ごしましょう!
参考
Docker Compose — Docker-docs-ja 17.06.Beta ドキュメント
docker-compose コマンドまとめ
docker-composeでmysql & postgreSQL をサクッと起動
The RDKit database cartridge
django-rdkit