概要
『よのなかねかおかおかねかなのよ』
これはR Advent Calendar 2017 5日目の記事になります。
また、先日のJapanR 2017にてLT発表した際に触れていた「データ分析が活かされる組織の5つの要素」のうち、「環境」の面を強化する話でもあります。
Deep Learning界隈ではGPUの枚数が名刺代わりと言われて久しい昨今ですが、Rでも札束を積んでマシンパワーで殴れるクラウド環境を作り使い慣れておくのは、今後を生き抜いていくには必要になるスキルかと思います。
AWSの環境用意や{sparklyr}でS3バケット上のファイルをRで扱うことは以前行いましたので、今回はGoogle Cloud Platform(GCP)を試してみましょう。
中でもデータ分析時に利用しそうなコンポーネントである、Google Cloud Engine(GCE), Google Cloud Storage(GCS), BiqQueryを使います。具体的にはRからGCEのインスタンスを起動・停止・作成したり、GCSにファイルをアップロート・ダウンロードしたり、BiqQueryでクエリを処理させたりします(前半はGCPの設定やGoogle Cloud Platform Console上で操作などを説明しておりますので、ご存知の方は適宜飛ばしてください)。
AWSと同様で無料枠がありますので、個人で練習する分にはこちらの範囲で利用するのがよいでしょう。BiqQueryがクエリ使用料1TBまで無料で使えるのはいいですね(ただし、ストレージ費用に関しては触れていない)。
RStudio Server Proも使えるという富裕層はLauncherで手軽にデプロイできるようです。
- [Getting Started with RStudio Server Pro for GCP]
(https://support.rstudio.com/hc/en-us/articles/115010260627-Getting-Started-with-RStudio-Server-Pro-for-GCP)
また、そもそもGCPで何ができるかわからないという方は、公式サイトや下記の記事や書籍などを読んでおくといいでしょう。
- 公式サイト
- Google Cloud Platform とは
- 【2015年版】AWS ユーザが Google Cloud Platform に15分で入門する!
- プログラマのためのGoogle Cloud Platform入門
事前準備
GCPの利用にはGoogleアカウントとクレジットカードが必要になりますので、あらかじめ調達をお願いします。なお、Googleアカウントを持っていない方は公式ページで開設できます。
これらの準備が終えましたら、次にプロジェクトを作成します(記事中には本来は公開を避ける情報も記載しておりますが、作ったプロジェクトは後ほど削除しており現在はアクセスできません)。
GCPプロジェクトの作成手順
GCPではプロジェクト単位でコンポーネント(GCEやGCSなどのリソース)の利用や課金・ユーザー、API管理などが行えます。このプロジェクトはひとつのGoogleアカウントで複数作成できてプロジェクト別に管理も行えるので、会社のチームや部門・部署、大学の研究室などをくくりにした利用がしやすいかと思います。
作成にはWeb上にスタートガイドがありますので、そちらを参考にして準備します。
- Google Cloud Platform スタートガイド - 公式サイト
- 初心者のためのGCPプロジェクト始め方入門
- Google Cloud Platform のサービスアカウントを作成する(2016年9月版)
以下、メモ代わりに手順を記載していきますが、ご存知の方々は次の節まで飛ばしてください。
Google Cloud Platform Consoleにアクセスしますが、何もプロジェクトを作成していない状態だとこういう感じになっています。

次にGoogle Cloud Platform の無料トライアルに直接アクセスするか、Google Cloud Platform Consoleの画面右上の「無料トライアルに登録」をクリックすると、下記の画面に遷移するので利用規約に同意するを選んで進みます。

すると情報登録を求められますので、「アカウントの種類」を「個人」にして必要事項を記入していきます(「ビジネス」でも違いはなかったような気がしますが)。ここでクレジットカード情報を入力します。無料枠しか使わないと決めていても登録は必須です。ドキドキしますね。
なお、入力時の電話番号は「国際電話番号」ですので、頭を取って続けて入力してください。例えば「090-1234-xxxx」であれば、「90-1234-xxxx」という風にです。

登録をすませるとGoogle Cloud Platform Consoleに遷移し、下記の画面になります。最初と異なりプロジェクトが作成されておりますね(「My First Project」となっており変更可能です)。
また、「プロジェクトID」と「プロジェクト番号」はプロジェクト毎に自動付与されるものになり、プロジェクトIDは後ほど使うことになります。

以上でプロジェクトの作成は終了です。GCP上の様々なコンポーネントが利用できるようになりました。おめでとうございます。
サービスアカウントと鍵ファイルを用意する
個人アカウントに権限を付与するのでもよいですが、今回はサービスアカウントを使ってみましょう。
サービスアカウントを用意すると分析プロジェクトや研究室などで、メンバーの入れ替わりがあっても、権限がなくて処理が実行できないという状況を避けられます(ただし、サービスアカウントには適切な権限設定が重要になります)。
今回は個人プロジェクトですので強い役割を設定してしますが、様々なシステムで利用する際には分けておいた方がいいでしょう。
Google Cloud Platform Consoleの左上にある横線三本のハンバーガーメニュー(由来がよくわからない)をクリックしてメニューが現れたら「IAMと管理」にカーソルを合わせて、さらに現れたメニューの「サービスアカウント」を選びます。
サービスアカウントが作成されていない状態では次の画像のようになります。

この状態では「サービスアカウントの作成」をクリックしましょう。
すると下記のようなウィンドウがポップアップするので、「サービスアカウント名」を入力して「役割」のドロップダウンメニューから設定する権限を選びます。ここでは「BiqQuery 管理者」と「Compute Admin」と「ストレージ管理者」と「サービスアカウントアクター」を選んでいます。「管理者/Admin」はとても強い権限ですので、自分が管理するプロジェクトを他のメンバーが使う場合などは管理者ではなく適宜権限を調整しましょう(このサービスアカウントでインスタンス作成をしなければ「サービスアカウントアクター」の役割は不要です)。
「役割」を設定し終えたら次に「新しい秘密鍵の提供」にチェックを入れ、「キーのタイプ」はJSONを選んでウィンドウ右下の「作成」をクリックします。
サービスアカウントが作成され、対応する鍵ファイルのダウンロードが開始されます。

このファイルもプロジェクトIDとともに利用します。こちらのファイルはGCPの各種コンポーネントの認証・権限に関わりますので、メッセージにある通り安全に保存し、ファイルの公開などはしないようにしましょう(GitHubにソースと一緒にあげるみたいなこと)。
GCEインスタンスを立ち上げる
それではGCEのインスタンスを立ち上げましょう。
GCEのインスタンスにRStudio Serverをインストールしてメモリ4GBのローカルマシンからアクセスすれば自由度が上がりますし、Shinyサーバーにして分析結果を共有するもよいでしょう。
コンソールからインスタンスを立ち上げるまでは、先ほど同じように順々に説明していきます。大丈夫だという方々は次の節まで飛ばしてください。
Google Cloud Platform ConsoleでGCEを操作
先ほどを同じようにGoogle Cloud Platform Consoleのハンバーガーメニューをクリックしてメニューが現れたら「Compute Engine」にカーソルを合わせ、さらに現れたメニューの「VMインスタンス」を選びます。
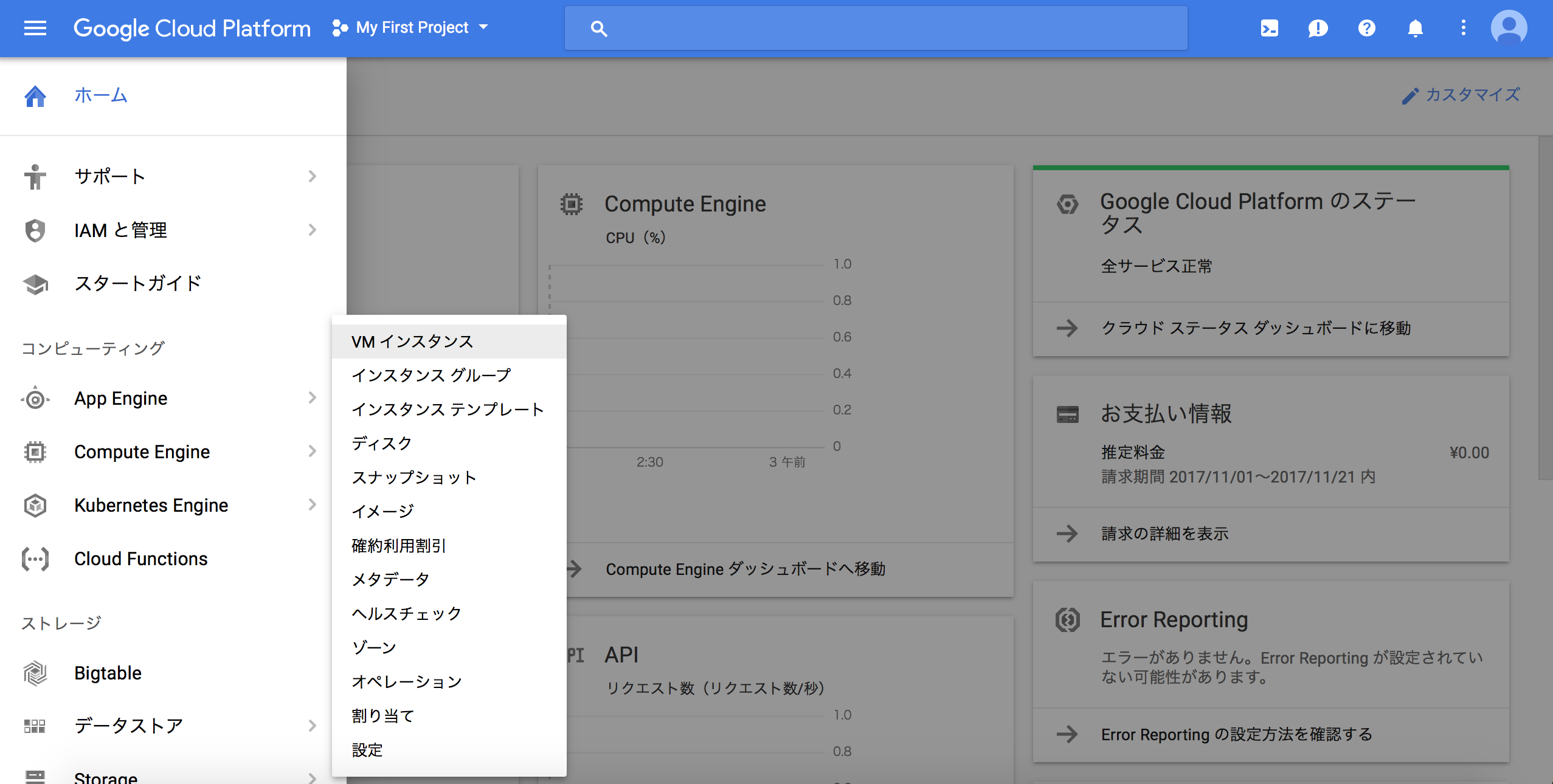
プロジェクト作成後の初回だとしばらく待つ必要がありますが、「作成」を選べるようになったらクリックします。

すると「インスタンスの作成」に切り替わりますので、下記のような感じで選びます。今回選択したマシンタイプはいつまでも無料で使える枠ではない点にご注意ください。
また、「IDとAPIへのアクセス」という項目は権限に影響するところで、先ほど作成したサービスアカウント名を指定します。
その後、ウインドウをさらに下の方にある「作成」のボタンをクリックするとインスタンスが立ち上がり始めます。

「インスタンスの作成」の画面で「作成」をクリックしてインスタンスを立ち上げると「VMインスタンス」に自動的に戻ります。すると先ほどまでは何もなかったですが、作成したインスタンスの起動状態や外部IP(RStudio Serverなどでアクセスするときに用いる)などが確認できます。
インスタンスを作成している状態で「Compute Engine」から「VMインスタンス」に進むとこの画面に来るようになります。
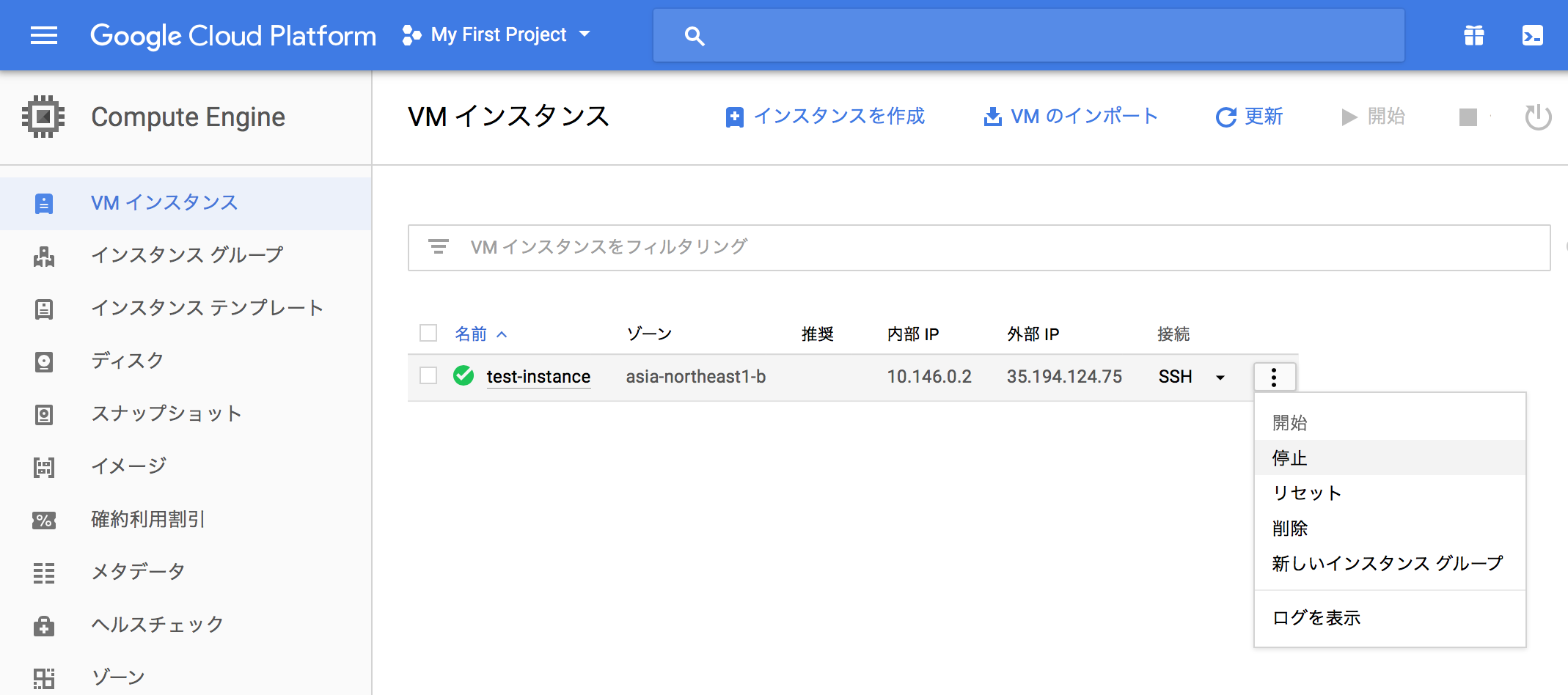
今回は無料枠を利用しているとはいえ、ずっと放置していると枠分を使い切ってしまいお金がかかってしまいます。使わないときはインスタンスは停止しておきましょう。インスタンス名の右端(SSHよりも右)にあるボタンをクリックして表示されるメニューで、「停止」を選びます。
するとインスタンス名の左にあった緑色のチェックが変化し、次のような白い四角になれば停止しています(マウスオーバーするとメッセージが表示されます」)。
逆にインスタンスを起動させたいときは、停止しているインスタンスで先ほど「停止」を選んだところで「起動」の方を選びます。少し待つ必要がありますが、白四角だったアイコンが先ほどと同じく緑丸となって利用できる状態になります。

これでインスタンスの作成と起動、ならび停止ができるようになりました。
他にもディスク容量を増やしたり、接続元のIPを限定したり、一度作ったインスタンスをイメージ化して再利用させたり、様々なことが行えます。
RでGCPを管理する
さて、Rおじさんの方々はRから管理したいと思い始めたころではないでしょうか。
それではGCEインスタンスとサービスアカウントの鍵ファイルが用意できましたので、RをインストールしたローカルマシンからGCPの各コンポーネントを操作してみましょう。
定義・設定部
まずは用意した認証ファイル(サービスアカウントでも個人でも)や必要な設定を定義します。
# 必要なパッケージ名
LOAD_PACKAGE <- c("tidyverse", "googleComputeEngineR", "googleCloudStorageR", "bigQueryR", "bigrquery", "glue")
# 先ほどダウンロードした鍵ファイル
AUTH_JSON_FILE_PATH <- "****.json"
# Google Cloud Platform Consoleで表示されているプロジェクトID
GCP_DEFAULT_PROJECT_ID <- "focal-psyche-186717"
# 各コンポーネントを使うゾーン
GCP_DEFAULT_ZONE <- "asia-northeast1-b"
次に設定した認証情報をライブラリを読み込む前に環境変数に設定します(呼び出した際に確認される)。Webページに遷移して許可を出し、その先に記載のコードをRコンソール上で貼り付けるという手順も取れます。
いくつかの関数で引数にprojectやzoneをしていますが、環境変数で設定していれば省略しても構いません。
Sys.setenv("GCE_AUTH_FILE" = AUTH_JSON_FILE_PATH)
Sys.setenv("GCS_AUTH_FILE" = AUTH_JSON_FILE_PATH)
Sys.setenv("BQ_AUTH_FILE" = AUTH_JSON_FILE_PATH)
Sys.setenv("GCE_DEFAULT_PROJECT_ID" = GCP_DEFAULT_PROJECT_ID)
Sys.setenv("GCE_DEFAULT_ZONE" = GCP_DEFAULT_ZONE)
pacman::p_load(char = LOAD_PACKAGE, install = TRUE, character.only = TRUE)
bigrquery::set_service_token(service_token = AUTH_JSON_FILE_PATH)
これで各種コンポーネントをRから管理する準備が整いました。
RでGoogle Cloud Engineを扱う
まずはGoogle Cloud Platform Console上でインスタンス作成を行ったGCEについて扱います。googleComputeEngineRパッケージの関数を利用して、インスタンスを操作したり作成したりしてみましょう。
GCEインスタンス操作
googleComputeEngineR::gce_list_instances() を実行するとインスタンスのリストを取得できます。前節で作成したインスタンスがあり、停止している(status = TERMINATED)のがわかります。
> gce_instance_list <- googleComputeEngineR::gce_list_instances(
project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
) %>%
print()
==Google Compute Engine Instance List==
name machineType status zone externalIP creationTimestamp
1 test-instance g1-small TERMINATED asia-northeast1-b No external IP 2017-11-23 02:51:02
それではこちらのインスタンスを起動・停止してみましょう。コンソールで処理したときとの同様で、実行して反映されるまで数秒から数十秒を要します。googleComputeEngineR::gce_wait() でJOBが終了するまで数秒スパンで待たせて状況を確認できます。以下では10秒を設定しています。
vm_start_job <- googleComputeEngineR::gce_vm_start(
instance = "test-instance", project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
)
> googleComputeEngineR::gce_wait(operation = vm_start_job, wait = 10)
2017-11-23 21:58:33> Checking operation...PENDING
2017-11-23 21:58:53> Operation complete in 10 secs
==Zone Operation start : DONE
Started: 2017-11-23 04:58:32
Ended: 2017-11-23 04:58:42
Operation complete in 10 secs
# StatusがRUNNINGに変化している
# なお、Google Cloud Platform Console上でも状態が変わります
> googleComputeEngineR::gce_get_instance(
+ instance = "test-instance", project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
+ )
==Google Compute Engine Instance==
Name: test-instance
Created: 2017-11-23 02:51:02
Machine Type: g1-small
Status: RUNNING
Zone: asia-northeast1-b
External IP: 35.200.14.174
Disks:
deviceName type mode boot autoDelete
1 test-instance PERSISTENT READ_WRITE TRUE TRUE
起動時と同じように停止を行います。
vm_stop_job <- googleComputeEngineR::gce_vm_stop(
instance = "test-instance", project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
)
> googleComputeEngineR::gce_wait(operation = vm_stop_job, wait = 10)
2017-11-23 22:16:38> Operation running...
2017-11-23 22:16:48> Operation running...
2017-11-23 22:16:59> Operation running...
2017-11-23 22:17:20> Operation complete in 28 secs
==Zone Operation stop : DONE
Started: 2017-11-23 05:16:37
Ended: 2017-11-23 05:17:05
Operation complete in 28 secs
# StatusがTERMINATEDに変化している
> googleComputeEngineR::gce_get_instance(
+ instance = "test-instance", project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
+ )
==Google Compute Engine Instance==
Name: test-instance
Created: 2017-11-23 02:51:02
Machine Type: g1-small
Status: TERMINATED
Zone: asia-northeast1-b
External IP:
Disks:
deviceName type mode boot autoDelete
1 test-instance PERSISTENT READ_WRITE TRUE TRUE
インスタンスが停止している必要がありますが、マシンタイプを変更してスペックを上げたり下げたりもできます。なお、変更後に起動するとマシンタイプに応じた課金額が発生します。
vm_resize_job <- googleComputeEngineR::gce_set_machinetype(
predefined_type = "n1-standard-1", instance = "test-instance",
project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
)
# 「g1-small」から「n1-standard-1」に変更されている
> googleComputeEngineR::gce_get_instance(
instance = "test-instance", project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
)
==Google Compute Engine Instance==
Name: test-instance
Created: 2017-11-23 02:51:02
Machine Type: n1-standard-1
Status: TERMINATED
Zone: asia-northeast1-b
External IP:
Disks:
deviceName type mode boot autoDelete
1 test-instance PERSISTENT READ_WRITE TRUE TRUE
これらの操作結果は、Google Cloud Platform Consoleでも同じように確認できます。以下のキャプチャーでは「test-instance」のマシンタイプが「n1-standard-1」に変更されているのが見て取れます。

インスタンスを作成する
先ほどはGoogle Cloud Platform ConsoleでGUI操作でスペックを決めて作成しましたが、起動・停止同様に操作できます。インスタンスを作成する関数として、googleComputeEngineR::gce_vm_create()が利用できます。こちらではマシンタイプを指定する方式と、CPU数やメモリなどを自分で入力する方式があります。
# イメージは方式に関わらず設定
> tibble::as_data_frame(
googleComputeEngineR::gce_get_image_family(image_project = "centos-cloud", family = "centos-7")
) %>%
dplyr::select(name, description, status, family, rawDisk)
# A tibble: 2 x 5
name description status family rawDisk
<chr> <chr> <chr> <chr> <list>
1 centos-7-v20171025 CentOS, CentOS, 7, x86_64 built on 2017-10-25 READY centos-7 <chr [1]>
2 centos-7-v20171025 CentOS, CentOS, 7, x86_64 built on 2017-10-25 READY centos-7 <chr [1]>
# マシンタイプを確認
> googleComputeEngineR::gce_list_machinetype(
project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
)$items %>%
dplyr::filter(guestCpus < 3 & memoryMb < 5000) %>%
dplyr::select(
name, description,
guestCpus, memoryMb, imageSpaceGb,
maximumPersistentDisks, maximumPersistentDisksSizeGb
)
name description guestCpus memoryMb imageSpaceGb
1 f1-micro 1 vCPU (shared physical core) and 0.6 GB RAM 1 614 0
2 g1-small 1 vCPU (shared physical core) and 1.7 GB RAM 1 1740 0
3 n1-standard-1 1 vCPU, 3.75 GB RAM 1 3840 10
4 n1-highcpu-2 2 vCPUs, 1.8 GB RAM 2 1843 10
maximumPersistentDisks maximumPersistentDisksSizeGb
1 16 3072
2 16 3072
3 32 65536
4 64 65536
# マシンタイプが「n1-standard-1」でCentOS7のイメージで作成
vm_create_job <- googleComputeEngineR::gce_vm_create(
name = "rtest",
predefined_type = "n1-standard-1",
image_project = "centos-cloud", image_family = "centos-7",
project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
)
> googleComputeEngineR::gce_wait(operation = vm_create_job, wait = 10)
2017-11-23 22:58:26> Checking operation...PENDING
2017-11-23 22:58:47> Operation complete in 9 secs
==Zone Operation insert : DONE
Started: 2017-11-23 05:58:26
Ended: 2017-11-23 05:58:35
Operation complete in 9 secs
# 作成されたインスタンスがインスタンスリストに追加されている
> > googleComputeEngineR::gce_list_instances()
==Google Compute Engine Instance List==
name machineType status zone externalIP creationTimestamp
1 rtest n1-standard-1 RUNNING asia-northeast1-b 35.194.124.75 2017-11-23 05:58:25
2 test-instance g1-small TERMINATED asia-northeast1-b No external IP 2017-11-23 02:51:02
# 作成すると同時に起動するので停止しておく
googleComputeEngineR::gce_wait(
operation = googleComputeEngineR::gce_vm_stop(instance = "rtest"), wait = 10
)
2017-11-23 23:08:49> Operation running...
2017-11-23 23:08:59> Operation running...
2017-11-23 23:09:10> Operation running...
2017-11-23 23:09:30> Operation complete in 26 secs
==Zone Operation stop : DONE
Started: 2017-11-23 06:08:48
Ended: 2017-11-23 06:09:14
Operation complete in 26 secs
# CPU数は最低2CPUからで、メモリは256MBから
vm_sb_create_job <- googleComputeEngineR::gce_vm_create(
name = "rtest-stanby",
cpus = 2, memory = 256 * 8,
image_project = "centos-cloud", image_family = "centos-7",
project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
)
> googleComputeEngineR::gce_wait(operation = vm_sb_create_job, wait = 10)
2017-11-23 23:13:25> Operation running...
2017-11-23 23:13:45> Operation complete in 12 secs
==Zone Operation insert : DONE
Started: 2017-11-23 06:13:19
Ended: 2017-11-23 06:13:31
Operation complete in 12 secs
# machineTypeがcustomで作成されている
> googleComputeEngineR::gce_list_instances(filter = "name eq rtest-stanby")
==Google Compute Engine Instance List==
name machineType status zone externalIP creationTimestamp
1 rtest-stanby custom-2-2048 RUNNING asia-northeast1-b 35.200.14.174 2017-11-23 06:13:19
# 停止
> googleComputeEngineR::gce_wait(
operation = googleComputeEngineR::gce_vm_stop(instance = "rtest-stanby"), wait = 10
)
2017-11-23 23:18:47> Operation running...
2017-11-23 23:18:57> Operation running...
2017-11-23 23:19:08> Operation running...
2017-11-23 23:19:29> Operation complete in 29 secs
==Zone Operation stop : DONE
Started: 2017-11-23 06:18:46
Ended: 2017-11-23 06:19:15
Operation complete in 29 secs
# googleComputeEngineR::gce_vm_createをラップした関数も用意されている
# こちらだとgoogleComputeEngineR::gce_wait()が不要
vm_sb2_create_job <- googleComputeEngineR::gce_vm(
name = "rtest-stanby-2",
cpus = 2, memory = 256 * 8,
image_project = "centos-cloud", image_family = "centos-7",
project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
)
2017-11-24 00:26:03> Creating standard VM
2017-11-24 00:26:05> Checking operation...PENDING
2017-11-24 00:26:09> Operation running...
2017-11-24 00:26:12> Operation running...
2017-11-24 00:26:19> Operation complete in 10 secs
2017-11-24 00:26:21> VM running
> vm_sb2_create_job %>% print()
==Google Compute Engine Instance==
Name: rtest-stanby-2
Created: 2017-11-23 07:26:05
Machine Type: custom-2-2048
Status: RUNNING
Zone: asia-northeast1-b
External IP: 35.194.124.75
Disks:
deviceName type mode boot autoDelete
1 rtest-stanby-2-boot-disk PERSISTENT READ_WRITE TRUE TRUE
これらに加えてgoogleComputeEngineR::gce_vm_template()では、RやRStudio Server, ShinyなどをDockerイメージ(自分が用意したDockerイメージも可)による「テンプレート」を用いたインスタンス作成ができます。下記ではRStudioのテンプレートを指定しています。
rstudio_vm_create_job <- googleComputeEngineR::gce_vm_template(
name = "rstudio", template = "rstudio",
username = "yamano357", password = "yamano357",
predefined_type = "n1-standard-1", disk_size_gb = 20
)
2017-11-24 01:51:10> Checking operation...PENDING
2017-11-24 01:51:30> Operation complete in 8 secs
2017-11-24 01:51:31> External IP for instance rstudio : 35.200.14.174
2017-11-24 01:51:31> ## VM rstudio running at 35.200.14.174
2017-11-24 01:51:31> Wait a few minutes for inital docker container to
download and install before logging in.
外部IP("External IP for ..."に記載がIP)にアクセスすると、RStudio Serverが利用できます。ユーザー名やパスワードなどは、作成した際に設定したものが使えます。

また、ローカルPCでssh-keygenコマンドでSSH認証用の鍵を生成し、googleComputeEngineR::gce_ssh_setup()で設定をしておくとローカルマシンからのSSH接続ができます。
これによって、インスタンス側でコマンド実行した結果を受け取れたり、RStudio Serverにユーザー追加する関数が利用できたりします。
# SSH認証ファイルの設定
rstudio_vm <- googleComputeEngineR::gce_ssh_setup(
instance = rstudio_vm_create_job,
key.pub = "****/.ssh/id_rsa.gcp.pub",
key.private = "****/.ssh/id_rsa.gcp"
)
# 先ほど作成したRStudio Serverのインスタンスにユーザーを追加する
rstudio_vm_adduser <- googleComputeEngineR::gce_rstudio_adduser(
instance = rstudio_vm,
username = "ymn357", password = "ymn357", admin = FALSE,
container = "rstudio"
)
このように追加したアカウントでもRStudio Serverにアクセスできます。

インスタンスの削除
不要になったインスタンスの削除は、googleComputeEngineR::gce_vm_delete()の実行で作成と同様にRから行えます。
googleComputeEngineR::gce_wait(
operation = googleComputeEngineR::gce_vm_delete(
instance = "rtest-stanby-2", project = GCP_DEFAULT_PROJECT_ID, zone = GCP_DEFAULT_ZONE
),
wait = 10
)
2017-11-24 00:26:22> Checking operation...PENDING
2017-11-24 00:26:33> Operation running...
2017-11-24 00:26:43> Operation running...
2017-11-24 00:26:53> Operation running...
2017-11-24 00:27:04> Operation running...
2017-11-24 00:27:14> Operation running...
2017-11-24 00:27:24> Operation running...
2017-11-24 00:27:35> Operation running...
2017-11-24 00:27:45> Operation running...
2017-11-24 00:27:56> Operation running...
2017-11-24 00:28:07> Operation running...
2017-11-24 00:28:17> Operation running...
2017-11-24 00:28:27> Operation running...
2017-11-24 00:28:38> Operation running...
2017-11-24 00:28:48> Operation running...
2017-11-24 00:28:58> Operation running...
2017-11-24 00:29:19> Operation complete in 2.766667 mins
==Zone Operation delete : DONE
Started: 2017-11-23 07:26:21
Ended: 2017-11-23 07:29:07
Operation complete in 2.766667 mins
まとめ(RでGCEを使う)
GCEのインスタンス操作からテンプレート利用でのRStudio Serverの立ち上げなどをRから行いました。
これによりローカルマシンのスペック不足の解消だけでなく、下記のGCSやBiqQueryと組み合わせることで、処理の待ち時間中に別タスクを並行してこなせるようになり、作業効率も格段に上がります。
また、featureパッケージを利用してGCEクラスターで処理を実行させる方法もパッケージ公式ページに記載があり、クラスタ環境を安価に活用できそうですね。
他にもテンプレートにplumberを追加するIssueが立っていたりもします。
RでGoogle Cloud Storageを使う
次にGoogle Cloud StorageをRから扱います。
RでGCSを操作するにはgoogleCloudStorageRパッケージの関数を利用します。バケットを作成したり、GCSにファイルをアップロード・ダウンロードしてみましょう。
GCSバケット操作
バケットがないと始まらないので、まずは作成をしましょう。合わせてバケットのリストを表示して、状況を確認します。
# バケットのリストを表示するが、まだ作成していないためNULLを返す
# すでにプロジェクトで作成している場合は、作成されているバケットが表示されます
> googleCloudStorageR::gcs_list_buckets(projectId = GCP_DEFAULT_PROJECT_ID)
NULL
# バケットを作成する
create_gcs_bucket <- googleCloudStorageR::gcs_create_bucket(
name = "r-sandbox", projectId = GCP_DEFAULT_PROJECT_ID,
location = "asia-northeast1", storageClass = "STANDARD", predefinedAcl = "private"
)
2017-11-24 02:53:00 -- Bucket created successfully:r-sandbox in asia-northeast1
# 再度バケットのリストを表示すると、作成したバケットが表示される
> googleCloudStorageR::gcs_list_buckets(projectId = GCP_DEFAULT_PROJECT_ID)
name storageClass location updated
1 r-sandbox STANDARD ASIA-NORTHEAST1 2017-11-23 17:52:59
GCSでファイル操作
バケットが作成されたので、次にファイルをアップロードしてみましょう。アップロード対象はR上のオブジェクトやファイルが関数では指定できますが、前者の場合はファイルに書き出して処理しています。
# バケット上にはファイルがない
> googleCloudStorageR::gcs_list_objects(bucket = "r-sandbox")
2017-11-24 02:58:10 -- No objects found
0 列 0 行のデータフレーム
# irisデータをGCSにアップロードする
# データフレームを指定すると、write.csv()が適用された形になります
googleCloudStorageR::gcs_upload(file = iris, bucket = "r-sandbox")
# ローカルファイルをアップロードする
readr::write_delim(x = iris, path = "./iris.tsv", append = FALSE)
googleCloudStorageR::gcs_upload(file = "iris.tsv", bucket = "r-sandbox")
# 先ほどと異なりファイルが存在
> googleCloudStorageR::gcs_list_objects(bucket = "r-sandbox") %>%
dplyr::select(id, name)
id name
1 r-sandbox/iris.csv/1511459893643347 iris.csv
2 r-sandbox/iris.tsv/1511461491647409 iris.tsv
Google Cloud Platform Console上で確認すると、下記のようになっています。

次に先ほどアップロードしたファイルをダウンロードしてましょう。ダウンロードする場合は、Rオブジェクトとして代入するか、ファイルとして保存するか、選べます。
# GCSにアップロードしたファイルをオブジェクトとして読み込む
> iris_from_gcs <- googleCloudStorageR::gcs_get_object(object_name = "iris.csv", bucket = "r-sandbox")
Downloaded iris.csv
Parsed with column specification:
cols(
X1 = col_integer(),
Sepal.Length = col_double(),
Sepal.Width = col_double(),
Petal.Length = col_double(),
Petal.Width = col_double(),
Species = col_character()
)
Object parsed to class: tbl_df tbl data.frame
警告メッセージ:
Missing column names filled in: 'X1' [1]
# GCSにアップロードしたファイルを、saveToDiskを指定してローカルにダウンロードする
> googleCloudStorageR::gcs_get_object(object_name = "iris.tsv", bucket = "r-sandbox", saveToDisk = "./iris-from-gcs.tsv")
Saved iris.tsv to ./iris-from-gcs.tsv
[1] TRUE
# ダウンロードしたファイルを元と比較する
> iris %>% tibble::glimpse()
Observations: 150
Variables: 5
$ Sepal.Length <dbl> 5.1, 4.9, 4.7, 4.6, 5.0, 5.4, 4.6, 5.0, 4.4, 4.9, 5.4, 4.8, 4.8, 4.3, 5.8, 5.7...
$ Sepal.Width <dbl> 3.5, 3.0, 3.2, 3.1, 3.6, 3.9, 3.4, 3.4, 2.9, 3.1, 3.7, 3.4, 3.0, 3.0, 4.0, 4.4...
$ Petal.Length <dbl> 1.4, 1.4, 1.3, 1.5, 1.4, 1.7, 1.4, 1.5, 1.4, 1.5, 1.5, 1.6, 1.4, 1.1, 1.2, 1.5...
$ Petal.Width <dbl> 0.2, 0.2, 0.2, 0.2, 0.2, 0.4, 0.3, 0.2, 0.2, 0.1, 0.2, 0.2, 0.1, 0.1, 0.2, 0.4...
$ Species <fctr> setosa, setosa, setosa, setosa, setosa, setosa, setosa, setosa, setosa, setos...
# ファイルを書き出した際に行番号が付与されてそれが列に追加されていたり、Species列がファクターから文字列に型が変わっている
> iris_from_gcs %>% tibble::glimpse()
Observations: 150
Variables: 6
$ X1 <int> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22,...
$ Sepal.Length <dbl> 5.1, 4.9, 4.7, 4.6, 5.0, 5.4, 4.6, 5.0, 4.4, 4.9, 5.4, 4.8, 4.8, 4.3, 5.8, 5.7...
$ Sepal.Width <dbl> 3.5, 3.0, 3.2, 3.1, 3.6, 3.9, 3.4, 3.4, 2.9, 3.1, 3.7, 3.4, 3.0, 3.0, 4.0, 4.4...
$ Petal.Length <dbl> 1.4, 1.4, 1.3, 1.5, 1.4, 1.7, 1.4, 1.5, 1.4, 1.5, 1.5, 1.6, 1.4, 1.1, 1.2, 1.5...
$ Petal.Width <dbl> 0.2, 0.2, 0.2, 0.2, 0.2, 0.4, 0.3, 0.2, 0.2, 0.1, 0.2, 0.2, 0.1, 0.1, 0.2, 0.4...
$ Species <chr> "setosa", "setosa", "setosa", "setosa", "setosa", "setosa", "setosa", "setosa"...
# ファイルの読み込み時に自前で調整する
> readr::read_delim(
file = "./iris-from-gcs.tsv", delim = " ",
col_types = readr::cols(
Species = readr::col_factor(levels = c("setosa", "versicolor", "virginica")),
.default = readr::col_double()
)
) %>% tibble::glimpse()
Observations: 150
Variables: 5
$ Sepal.Length <dbl> 5.1, 4.9, 4.7, 4.6, 5.0, 5.4, 4.6, 5.0, 4.4, 4.9, 5.4, 4.8, 4.8, 4.3, 5.8, 5.7...
$ Sepal.Width <dbl> 3.5, 3.0, 3.2, 3.1, 3.6, 3.9, 3.4, 3.4, 2.9, 3.1, 3.7, 3.4, 3.0, 3.0, 4.0, 4.4...
$ Petal.Length <dbl> 1.4, 1.4, 1.3, 1.5, 1.4, 1.7, 1.4, 1.5, 1.4, 1.5, 1.5, 1.6, 1.4, 1.1, 1.2, 1.5...
$ Petal.Width <dbl> 0.2, 0.2, 0.2, 0.2, 0.2, 0.4, 0.3, 0.2, 0.2, 0.1, 0.2, 0.2, 0.1, 0.1, 0.2, 0.4...
$ Species <fctr> setosa, setosa, setosa, setosa, setosa, setosa, setosa, setosa, setosa, setos...
R上のデータフレームをGCSにアップロードしたり、ダウンロードした結果をRのオブジェクトとして読み込んだりするときに自作関数が指定できます。これによって、先行する処理でミスが発生していたり、別部署管理の元ファイルの仕様変更にも対応できます。
# Species毎に10レコードだけ出力
# ファイル出力にはreadr::write_tsvを用いる
writeSlice10 <- function (input, output) {
return(
input %>%
dplyr::group_by(Species) %>%
dplyr::slice(seq(from = 1, to = 10, by = 1)) %>%
dplyr::ungroup() %>%
readr::write_tsv(x = ., path = output, col_names = TRUE)
)
}
# object_functionに定義した関数を指定してアップロード
googleCloudStorageR::gcs_upload(
file = iris, name = "iris_mod.tsv", bucket = "r-sandbox", object_function = writeSlice10
)
# TSV形式にしたため、アップロードしたファイルをそのまま読み込むとうまくいかない
> googleCloudStorageR::gcs_get_object(object_name = "iris_mod.tsv", bucket = "r-sandbox") %>%
tibble::glimpse()
Downloaded iris_mod.tsv
Object parsed to class: character
chr "Sepal.Length\tSepal.Width\tPetal.Length\tPetal.Width\tSpecies\n5.1\t3.5\t1.4\t0.2\tsetosa\n4.9\t3\t1.4\t0.2\tse"| __truncated__
# 書き込みと同じように読み込み用の関数を定義
# delim引数にタブを指定し、カラムの型も設定する
readTsvIris <- function(object){
return(
readr::read_delim(
file = suppressWarnings(httr::content(object, encoding = "UTF-8")), delim = "\t",
col_types = readr::cols(
Species = readr::col_factor(levels = c("setosa", "versicolor", "virginica")),
.default = readr::col_double()
)
)
)
}
# parseFunctionに読み込み用の関数を指定すると、問題なく読み込めようになる
> googleCloudStorageR::gcs_get_object(
object_name = "iris_mod.tsv", bucket = "r-sandbox", parseFunction = readTsvIris
) %>% tibble::glimpse()
Downloaded iris_mod.tsv
Observations: 30
Variables: 5
$ Sepal.Length <dbl> 5.1, 4.9, 4.7, 4.6, 5.0, 5.4, 4.6, 5.0, 4.4, 4.9, 7.0, 6.4, 6.9, 5.5, 6.5, 5.7...
$ Sepal.Width <dbl> 3.5, 3.0, 3.2, 3.1, 3.6, 3.9, 3.4, 3.4, 2.9, 3.1, 3.2, 3.2, 3.1, 2.3, 2.8, 2.8...
$ Petal.Length <dbl> 1.4, 1.4, 1.3, 1.5, 1.4, 1.7, 1.4, 1.5, 1.4, 1.5, 4.7, 4.5, 4.9, 4.0, 4.6, 4.5...
$ Petal.Width <dbl> 0.2, 0.2, 0.2, 0.2, 0.2, 0.4, 0.3, 0.2, 0.2, 0.1, 1.4, 1.5, 1.5, 1.3, 1.5, 1.3...
$ Species <fctr> setosa, setosa, setosa, setosa, setosa, setosa, setosa, setosa, setosa, setos...
GCS上のファイルを削除する場合は、googleCloudStorageR::gcs_delete_object() を使います。手軽にできてしまうので、必要なファイルを消さないように権限を管理したり、バケット自体を分けたりしておきましょう。
> googleCloudStorageR::gcs_delete_object(object_name = "iris_mod.tsv", bucket = "r-sandbox")
[1] TRUE
まとめ(RでGCSを使う)
GCSのバケット作成から始まり、ファイルのアップロード・ダウンロード、自作関数を指定した操作などを行いました。
これによりローカルマシンやGCEのストレージ領域に捉われず、またバックアップが必要なファイルを退避したり、分析結果出力のやりとりが手軽にできるようになりました。
今回は5MBより小さいファイルをアップロードしていましたが、それ以上のサイズのときはupload_typeを "resumable" にしておきましょうと、のことです。サイズが大きいファイルを扱うときは留意しておきましょう。
また、とても大きいデータを処理したい場合、GCSに対象ファイルを分割して置いておき、GCEインスタンスのスペックを上げたり、複数インスタンスを立ち上げて処理するというアプローチでも対応できますが、下記のBigQueryを利用するのがお手軽でおすすめです。
RでBigQueryを使う
BiqQueryを利用すると、巨大なデータを高速にかつ安価に処理できます。RからBiqQueryを扱うパッケージには次のふたりがあります。ここでは主にbigQueryRパッケージを使います。これにより大規模データを数十秒ほどで処理し、その結果をデータフレームとして受け取ったり、テーブルとして書き込んだりできます。
BigQueryにクエリを発行する
まずはサンプルとして公開されているデータセットに対してクエリを発行してみましょう。bigQueryR::bqr_query()でクエリの結果を受け取れます。ここではGitHubレポジトリ上のアクションデータにクエリを投げて集計しています。
# レコード数を確認
> bigQueryR::bqr_query(
projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "samples",
query = "SELECT COUNT(repository_owner) AS cn FROM `publicdata.samples.github_timeline`",
useLegacySql = FALSE
)
cn
1 5685601
# 指定したプログラミング言語のみに限定してクエリを作成
LANGUAGE <- shQuote(string = "R", type = "csh")
query <- glue::glue(
"
SELECT DISTINCT
repository_owner, repository_name, repository_url
FROM
`publicdata.samples.github_timeline`
WHERE
repository_language = {LANGUAGE}
"
)
github_timeline_r <- bigQueryR::bqr_query(
projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "samples",
query = query,
useLegacySql = FALSE
)
# リポジトリオーナーで集計して降順に並び替え
> github_timeline_r %>%
dplyr::group_by(repository_owner) %>% dplyr::tally() %>%
dplyr::arrange(dplyr::desc(x = n))
# A tibble: 602 x 2
repository_owner n
<chr> <int>
1 ropensci 23
2 hadley 18
3 omegahat 14
4 duncantl 12
5 Sage-Bionetworks 11
6 ggobi 10
7 johnmyleswhite 10
8 jlaake 9
9 cboettig 8
10 rdpeng 8
# ... with 592 more rows
# リポジトリオーナーの頻度の頻度
> github_timeline_r %>%
dplyr::group_by(repository_owner) %>% dplyr::tally() %>%
dplyr::group_by(n) %>% dplyr::tally(wt = n)
# A tibble: 15 x 2
n nn
<int> <int>
1 1 439
2 2 160
3 3 117
4 4 92
5 5 35
6 6 18
7 7 7
8 8 16
9 9 9
10 10 20
11 11 11
12 12 12
13 14 14
14 18 18
15 23 23
WebUIでサンプルデータの詳細を確認でき、下記のようになっています。

BigQueryのテーブル操作
次はサンプルデータではなく自前のテーブルにクエリを投げたいと思います。そのためにはテーブルがないと始まりません。
まずはデータセットを作り、そのデータセットに属するテーブルを作成します。ここでいうデータセットはテーブルを集めたものです。Rで表現するなら、データフレームからなるリストでしょうか。
データセット作成のための関数がbigQueryRパッケージに見当たらなかったため、ここではbigrqueryパッケージのbigrquery::insert_dataset()を使っています。
# データセットが作成されていない状況では、No Datasetsと表示される
> bigQueryR::bqr_list_datasets(projectId = GCP_DEFAULT_PROJECT_ID)
datasetId id projectId
1 **No Datasets** **No Datasets** focal-psyche-186717
# データセット作成
bigrquery::insert_dataset(project = GCP_DEFAULT_PROJECT_ID, dataset = "sandbox")
# データセット作成後に確認すると、作成したデータセットが表示される
> bigQueryR::bqr_list_datasets(projectId = GCP_DEFAULT_PROJECT_ID)
datasetId id projectId
1 sandbox focal-psyche-186717:sandbox focal-psyche-186717
さて、データセットが作成されましたので本命のテーブルを作りましょう。
テーブルはbigQueryR::bqr_create_table()で作れます。作成時にカラム名やデータの型が必要になりますが、ここではtemplate_data引数に元データを指定してデータに応じたテーブルを作成するようにしています。
# テーブル作成前はまだない
> bigQueryR::bqr_list_tables(projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox")
0 列 0 行のデータフレーム
# テーブル作成
> bigQueryR::bqr_create_table(
projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox", tableId = "github_timeline_r",
template_data = github_timeline_r
)
Table created: github_timeline_r
# 確認すると作成されている
> bigQueryR::bqr_list_tables(projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox")
id projectId datasetId tableId
1 focal-psyche-186717:sandbox.github_timeline_r focal-psyche-186717 sandbox github_timeline_r
# 作成したテーブルにクエリを発行しても、データはまだない
> bigQueryR::bqr_query(
projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox",
query = "SELECT COUNT(repository_name) FROM `focal-psyche-186717.sandbox.github_timeline_r`",
useLegacySql = FALSE
)
f0_
1 0
BigQueryテーブルへのデータインサート
テーブルは作成できましたが、まだデータは入っていません。BiqQueryにデータを入れるには、「実行結果を用いる方法」と、「GCS上のファイルを利用する方法」があります。
まずは前者の「実行結果を用いる方法」でデータを入れてみましょう。実行結果を用いる際にはbigQueryR::bqr_query_asynch()を使います。
# queryに発行するクエリを、destinationTableIdに出力先テーブル名を指定します
bq_tb_create_job <- bigQueryR::bqr_query_asynch(
projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox",
query = query,
destinationTableId = "github_timeline_r", useLegacySql = FALSE
)
# JOBが終了するまで待つと作成されている
> bigQueryR::bqr_wait_for_job(job = bq_tb_create_job, wait = 3)
2017-11-26 02:12:04 -- Waiting for job: job_twwJKeGkKZUplx5xsiP-JmUdJCOP - Job timer: 3.002875 secs
2017-11-26 02:12:04 -- Job status: DONE
==Google BigQuery Job==
JobID: job_twwJKeGkKZUplx5xsiP-JmUdJCOP
ProjectID: focal-psyche-186717
Status: DONE
User: gcp-r-69@focal-psyche-186717.iam.gserviceaccount.com
Created: 2017-11-26 02:12:00
Start: 2017-11-26 02:12:01
End: 2017-11-26 02:12:02
## View job configuration via job$configuration
# データを入れたテーブルにクエリを発行すると、結果が返ってくる
> bigQueryR::bqr_query(
projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox",
query = "
SELECT
repository_name
FROM
`focal-psyche-186717.sandbox.github_timeline_r`
WHERE
repository_owner = 'hadley'
",
useLegacySql = FALSE
)
repository_name
1 gtable
2 sfhousing
3 boxplots-paper
4 helpr
5 reshape
6 lubridate
7 evaluate
8 test_that
9 staticdocs
10 stringr
11 plyr
12 httr
13 data-baby-names
14 data-housing-crisis
15 profr
16 scales
17 ggplot2
18 devtools
次は「GCS上のファイルを利用してテーブルを作成する方法」ですが、それ行う前にBiqQueryのテーブルデータをGCS上にコピーしてみましょう。これはbigQueryR::bqr_extract_data()の実行できます(ただし、テーブルフルスキャンになるので大きすぎるテーブルでは注意)。なお、実行にはGCSとBiqQueryの両方に権限が必要になります。
# 出力前の状態
> googleCloudStorageR::gcs_list_objects(bucket = "r-sandbox") %>%
dplyr::select(name)
name
1 iris.csv
2 iris.tsv
# 対象テーブル名と出力先GCSや形式を引数で与える
extract_job <- bigQueryR::bqr_extract_data(
projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox", tableId = "github_timeline_r",
cloudStorageBucket = "r-sandbox", filename = "github_timeline_r",
compression = "GZIP", destinationFormat = "CSV", fieldDelimiter = ",", printHeader = TRUE
)
2017-11-26 02:14:59 -- Extract request successful, use bqr_wait_for_job() to know when it is ready.
> bigQueryR::bqr_wait_for_job(job = extract_job, wait = 3)
2017-11-26 02:15:03 -- Waiting for job: job_m_AVyhAmqrIIO9cQ948HwZsS8v1w - Job timer: 3.002814 secs
2017-11-26 02:15:03 -- Job status: DONE
==Google BigQuery Job==
JobID: job_m_AVyhAmqrIIO9cQ948HwZsS8v1w
ProjectID: focal-psyche-186717
Status: DONE
User: gcp-r-69@focal-psyche-186717.iam.gserviceaccount.com
Created: 2017-11-26 02:14:59
Start: 2017-11-26 02:14:59
End: 2017-11-26 02:15:02
## View job configuration via job$configuration
# 出力後の状態。ファイルが作成されている
googleCloudStorageR::gcs_list_objects(bucket = "r-sandbox") %>%
dplyr::select(name)
name
1 github_timeline_r
2 iris.csv
3 iris.tsv
上記でGCS上に配置されたファイルを用いてテーブルを作成します。
GCS上のファイルを利用する際はbigQueryR::bqr_upload_data()を使います。テーブルへのデータインサートもできますが、テーブルの新規作成もできますのでやってみます。また、bigQueryR::schema_fields()で作成するテーブルのカラム名やデータの型などを設定しています。
# 出力先のテーブル名やデータ元のGCSファイル、テーブルがあった場合の対応などを指定
bq_upload_job <- bigQueryR::bqr_upload_data(
projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox",
tableId = "github_timeline_r_replicate", upload_data = c("gs://r-sandbox/github_timeline_r"),
create = "CREATE_IF_NEEDED", overwrite = FALSE,
schema = bigQueryR::schema_fields(data = github_timeline_r), sourceFormat = "CSV"
)
2017-11-26 02:17:54 -- Uploading from Google Cloud Storage URI
2017-11-26 02:17:56 -- Returning: BigQuery load from Google Cloud Storage Job object: job_U1y3TQkDK0CaCm-oGammuH7yLTkp
> bigQueryR::bqr_wait_for_job(job = bq_upload_job, wait = 3)
2017-11-26 02:18:19 -- Waiting for job: job_U1y3TQkDK0CaCm-oGammuH7yLTkp - Job timer: 3.001228 secs
2017-11-26 02:18:20 -- Job status: DONE
==Google BigQuery Job==
JobID: job_U1y3TQkDK0CaCm-oGammuH7yLTkp
ProjectID: focal-psyche-186717
Status: DONE
User: gcp-r-69@focal-psyche-186717.iam.gserviceaccount.com
Created: 2017-11-26 02:17:55
Start: 2017-11-26 02:17:56
End: 2017-11-26 02:17:58
## View job configuration via job$configuration
# テーブルが作成されている
> bigQueryR::bqr_list_tables(projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox")
id projectId datasetId
1 focal-psyche-186717:sandbox.github_timeline_r focal-psyche-186717 sandbox
2 focal-psyche-186717:sandbox.github_timeline_r_replicate focal-psyche-186717 sandbox
tableId
1 github_timeline_r
2 github_timeline_r_replicate
BigQueryのテーブル削除
テーブル作成と同様に不要になったテーブルの削除も可能です。テーブル削除はbigQueryR::bqr_delete_table()でできます。
> bigQueryR::bqr_delete_table(projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox", tableId = "github_timeline_r_replicate")
[1] TRUE
# 削除確認
> bigQueryR::bqr_list_tables(projectId = GCP_DEFAULT_PROJECT_ID, datasetId = "sandbox")
id projectId datasetId tableId
1 focal-psyche-186717:sandbox.github_timeline_r focal-psyche-186717 sandbox github_timeline_r
>
まとめ(RでBiqQueryを使う)
BigQueryの活用によりメモリ上で処理できない超大規模なデータも扱えるようになるためデータ分析において非常に有用ですが、BiqQueryからRにデータを受け取る際にデータ量に応じた時間とメモリを消費します。
そのため処理結果が大きいままだと「BiqQueryでは数秒で処理された結果をRで受け取るのに数分待つ」といったことも起きてしまいます。Rで受け取るデータは必要な範囲に絞る、別テーブルやGCSに結果を保存してさらに処理する、など工夫が必要でしょう。
また今回はbigQueryRパッケージを取り上げておりますが、bigrqueryパッケージもとても便利です。基本的にほぼ同じ機能を有しており、bigrquery::src_bigquery()でBiqQueryをデータソースとしてdplyrパッケージ同様に処理できますし、DBIパッケージに準じた関数定義は馴染みがある人には使いやすいでしょう。R Markdownのknitr Language Enginesでコネクションオブジェクトを渡すことで、SQL同様にBiqQueryも実行できます。
bq_con <- DBI::dbConnect(
drv = bigrquery::bigquery(), dataset = "publicdata.samples", project = GCP_DEFAULT_PROJECT_ID,
use_legacy_sql = FALSE
)
下記のスクリーンショットのように、上記で定義したBiqQueryコネクションオブジェクトをR Markdownのチャンクオプションで言語エンジンをSQLにした上で設定します。output.varで指定したオブジェクトに結果が保存され、後続のR Markdown上で参照できます。

注意事項として、bigQueryRパッケージの利用時に「Not authenticated with Google BigQuery. 」のエラーが起きる場合ですが、こちらはGCPへの認証スコープで問題が起きております。
適したスコープを設定すればいいのですが、今回のケースでは設定しておりませんのでgoogleCloudStorageRパッケージをbigQueryRパッケージの後に読み込んでBiqQueryに処理させようとすると先のエラーメッセージが出てきます。
プロジェクトの削除
使わなくなったプロジェクトを削除します。プロジェクトの削除はGoogle Cloud Platform Console上の「設定」から行います。
ポップアップウィンドウにあるようにプロジェクト名を入力して、「シャットダウン」をクリックすると削除依頼がされます(クリック時は依頼で削除が実行されるのは30日後ですので、間違って削除してしまってもプロジェクトオーナーは期間内なら取り消せます)。

プロジェクトの削除依頼が出されてプロジェクトがなくなると、次のような画面になります。他にプロジェクトがあるなら「選択」で選べますし、なければ「作成」で新たに作ることもできます。

プロジェクトは手軽に作れますが、割り当て数に限りがありますのでご注意ください。
まとめ
Google Cloud Platform(GCP)のコンポーネントのうち、データ分析に用いることが多そうなGoogle Cloud Engine(GCE), Google Cloud Storage(GCS), BiqQueryをRで操作・利用する方法について説明しました。これによって分析環境が貧弱な状況(例えば、支給されたパソコンのCPUが2Coreとかメモリが4GBとか)に負けず、十分な分析がこなせるようになるでしょう。
今回は個人利用だったため札束で殴れるほどの環境を作っておりませんが、スペックやインスタンス数などを増やしても同じように扱えますので応用はできると思います(ただし、こちらはプロジェクト毎の割り当て量の上限によります)。
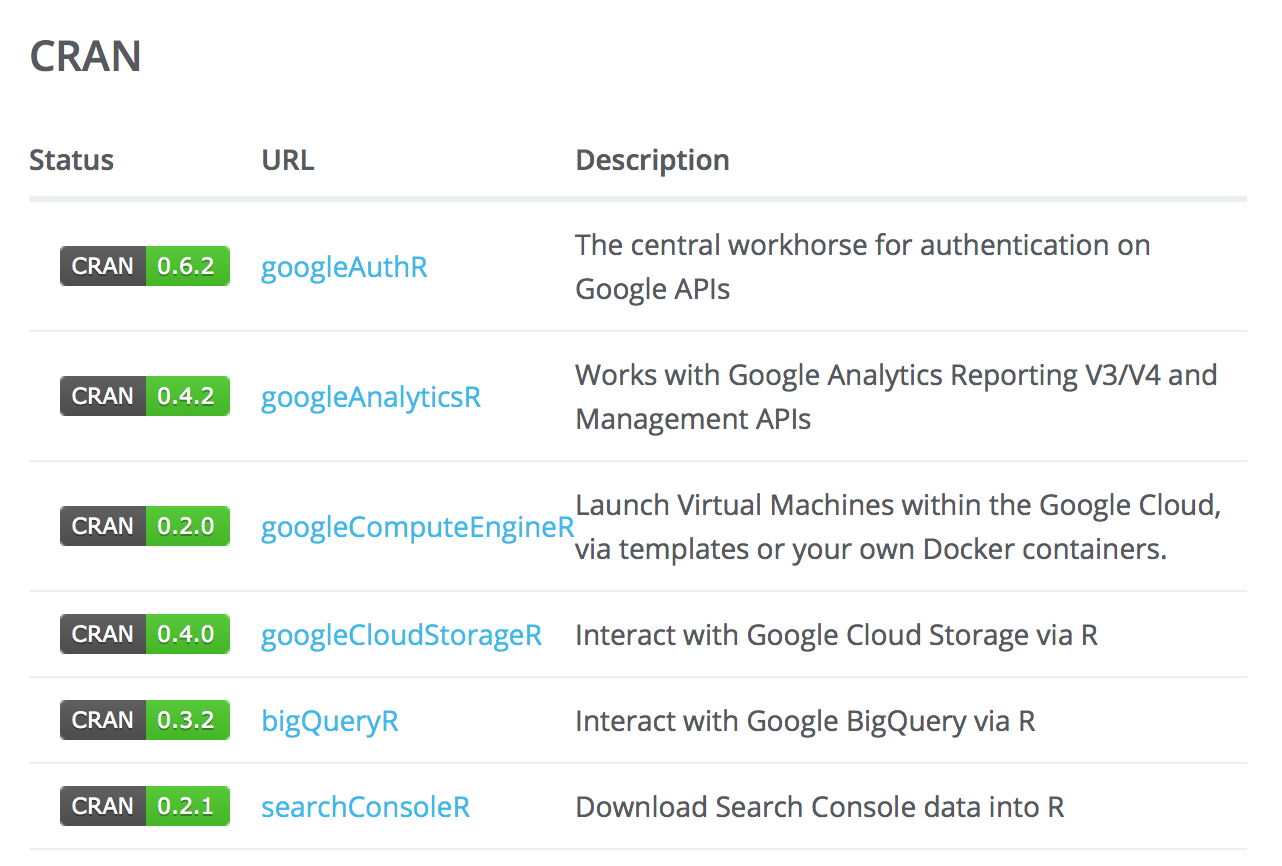
また、GCPの各種コンポーネントはAPIが公開されており、パッケージがないものも自作で処理することも可能です。今回利用したパッケージはすべて同じ作者が作成しており、他にも下記のようなパッケージをCRANに公開しております。
さらにこちらの作成者の方はこれらのパッケージを用いて、RとGCPを活用した分析環境を考えており、今後に期待したいと思います。

参考
実行環境
> devtools::session_info()
Session info -----------------------------------------------------------------------------------------------------------
setting value
version R version 3.4.2 (2017-09-28)
system x86_64, darwin15.6.0
ui RStudio (1.1.383)
language (EN)
collate ja_JP.UTF-8
tz Asia/Tokyo
date 2017-12-03
Packages ---------------------------------------------------------------------------------------------------------------
package * version date source
assertthat 0.2.0 2017-04-11 CRAN (R 3.4.2)
base * 3.4.2 2017-10-20 local
bigQueryR * 0.3.2.9000 2017-11-22 Github (cloudyr/bigQueryR@868f914)
bigrquery * 0.4.1 2017-06-26 CRAN (R 3.4.2)
bindr 0.1 2016-11-13 CRAN (R 3.4.2)
bindrcpp 0.2 2017-06-17 CRAN (R 3.4.2)
broom 0.4.2 2017-02-13 CRAN (R 3.4.2)
cellranger 1.1.0 2016-07-27 CRAN (R 3.4.2)
cli 1.0.0 2017-11-05 CRAN (R 3.4.2)
codetools 0.2-15 2016-10-05 CRAN (R 3.4.2)
colorspace 1.3-2 2016-12-14 CRAN (R 3.4.2)
compiler 3.4.2 2017-10-20 local
crayon 1.3.4 2017-09-16 cran (@1.3.4)
curl 3.0 2017-10-06 CRAN (R 3.4.2)
datasets * 3.4.2 2017-10-20 local
DBI 0.7 2017-06-18 CRAN (R 3.4.2)
devtools 1.13.3 2017-08-02 CRAN (R 3.4.2)
digest 0.6.12 2017-01-27 CRAN (R 3.4.2)
dplyr * 0.7.4 2017-09-28 CRAN (R 3.4.2)
forcats * 0.2.0 2017-01-23 CRAN (R 3.4.2)
foreign 0.8-69 2017-06-22 CRAN (R 3.4.2)
future 1.6.2 2017-10-16 CRAN (R 3.4.2)
ggplot2 * 2.2.1.9000 2017-10-28 Github (tidyverse/ggplot2@ffb40f3)
globals 0.10.3 2017-10-13 CRAN (R 3.4.2)
glue * 1.1.1 2017-06-21 CRAN (R 3.4.2)
googleAuthR 0.6.0 2017-10-20 CRAN (R 3.4.2)
googleCloudStorageR * 0.3.0 2017-05-27 CRAN (R 3.4.2)
googleComputeEngineR * 0.2.0 2017-09-16 CRAN (R 3.4.2)
graphics * 3.4.2 2017-10-20 local
grDevices * 3.4.2 2017-10-20 local
grid 3.4.2 2017-10-20 local
gtable 0.2.0 2016-02-26 CRAN (R 3.4.2)
haven 1.1.0 2017-07-09 CRAN (R 3.4.2)
hms 0.3 2016-11-22 CRAN (R 3.4.2)
httr 1.3.1 2017-08-20 CRAN (R 3.4.2)
jsonlite 1.5 2017-06-01 CRAN (R 3.4.2)
lattice 0.20-35 2017-03-25 CRAN (R 3.4.2)
lazyeval 0.2.0 2016-06-12 CRAN (R 3.4.2)
listenv 0.6.0 2015-12-28 CRAN (R 3.4.2)
lubridate 1.7.1 2017-11-03 CRAN (R 3.4.2)
magrittr 1.5 2014-11-22 CRAN (R 3.4.2)
memoise 1.1.0 2017-04-21 CRAN (R 3.4.2)
methods * 3.4.2 2017-10-20 local
mnormt 1.5-5 2016-10-15 CRAN (R 3.4.2)
modelr 0.1.1 2017-07-24 CRAN (R 3.4.2)
munsell 0.4.3 2016-02-13 CRAN (R 3.4.2)
nlme 3.1-131 2017-02-06 CRAN (R 3.4.2)
openssl 0.9.7 2017-09-06 CRAN (R 3.4.2)
pacman 0.4.6 2017-05-14 CRAN (R 3.4.2)
parallel 3.4.2 2017-10-20 local
pkgconfig 2.0.1 2017-03-21 CRAN (R 3.4.2)
plyr 1.8.4 2016-06-08 CRAN (R 3.4.2)
psych 1.7.8 2017-09-09 CRAN (R 3.4.2)
purrr * 0.2.4 2017-10-18 CRAN (R 3.4.2)
R6 2.2.2 2017-06-17 CRAN (R 3.4.2)
Rcpp 0.12.13 2017-09-28 CRAN (R 3.4.2)
readr * 1.1.1 2017-05-16 CRAN (R 3.4.2)
readxl 1.0.0 2017-04-18 CRAN (R 3.4.2)
reshape2 1.4.2 2016-10-22 CRAN (R 3.4.2)
RevoUtils * 10.0.6 2017-10-17 local
rlang 0.1.4 2017-11-05 CRAN (R 3.4.2)
rstudioapi 0.7 2017-09-07 CRAN (R 3.4.2)
rvest 0.3.2 2016-06-17 CRAN (R 3.4.2)
scales 0.5.0.9000 2017-10-28 Github (hadley/scales@d767915)
stats * 3.4.2 2017-10-20 local
stringi 1.1.5 2017-04-07 CRAN (R 3.4.2)
stringr * 1.2.0 2017-02-18 CRAN (R 3.4.2)
tibble * 1.3.4 2017-08-22 CRAN (R 3.4.2)
tidyr * 0.7.2 2017-10-16 CRAN (R 3.4.2)
tidyverse * 1.2.1 2017-11-14 CRAN (R 3.4.2)
tools 3.4.2 2017-10-20 local
utils * 3.4.2 2017-10-20 local
withr 2.0.0 2017-10-28 Github (jimhester/withr@a43df66)
xml2 1.1.1 2017-01-24 CRAN (R 3.4.2)
yaml 2.1.14 2016-11-12 cran (@2.1.14)