この記事の狙い・目的
機械学習を取り入れたAIシステムの構築は、
①データ分析→ ②データセット作成(前処理)→ ③モデルの構築・適用
というプロセスで行っていきます。
その際「前処理」の段階では、データ分析の考察を踏まえて、精度の高いデータセットが作れるよう様々な対応が必要となります。
このブログでは、「前処理(特徴量エンジニアリング)」の工程について初めから通して解説していきます。
プログラムの実行環境
Python3
MacBook pro(端末)
PyCharm(IDE)
Jupyter Notebook(Chrome)
Google スライド(Chrome)
データ確認
# データ取得
boston_df = pd.read_csv("./boston.csv", sep=',')
# データ確認
boston_df.shape
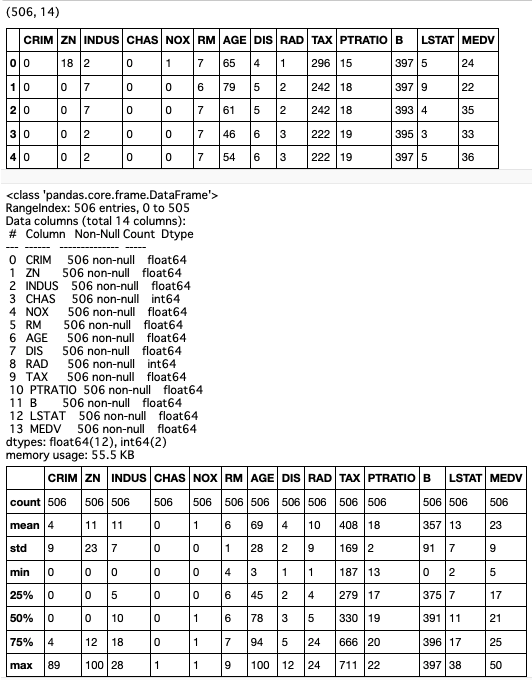
boston_df.head()
boston_df.info()
boston_df.describe()
精度評価
from sklearn.ensemble import RandomForestRegressor as RFR # ランダム・フォレスト(回帰)
from sklearn.model_selection import train_test_split # データ分割
from sklearn.metrics import r2_score, mean_squared_error, mean_absolute_error, mean_squared_log_error # 各評価指標
def learning(model, df):
# データ分割
X = df.drop('MEDV',axis=1)
y = df['MEDV']
X_train,X_test,y_train,y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# 学習、予測
rfr_model = model
rfr_model.fit(X_train, y_train)
y_pred = rfr_model.predict(X_test).round(decimals=1)
# 評価
print('決定係数(R2) = ', r2_score(y_test, y_pred).round(decimals=3))
print('平均絶対誤差(MAE) = ', mean_absolute_error(y_test, y_pred).round(decimals=3))
print('平均二乗誤差(MSE) = ', mean_squared_error(y_test, y_pred).round(decimals=3))
print('対数平均二乗誤差(MSLE) = ', mean_squared_log_error(y_test, y_pred).round(decimals=3))
print('平均二乗平方根誤差(RMSE) = ', np.sqrt(mean_squared_error(y_test, y_pred)).round(decimals=3))
print('対数平方平均二乗誤差(RMSLE) = ', np.sqrt(mean_squared_log_error (y_test, y_pred)).round(decimals=3))
return rfr_model, y_test, y_pred
# 実行
rfr_model, y_test, y_pred = learning(RFR(random_state=42), boston_df)
# 結果
# 決定係数(R2) = 0.892
# 平均絶対誤差(MAE) = 2.04
# 平均二乗誤差(MSE) = 7.902
# 対数平均二乗誤差(MSLE) = 0.02
# 平均二乗平方根誤差(RMSE) = 2.811
# 対数平方平均二乗誤差(RMSLE) = 0.142
非線形モデルでまずまずの評価
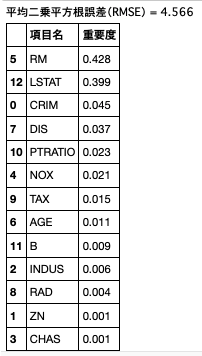
特徴量重要度
ランダム・フォレスト版
def rfr_importance(df):
# データ分割
X = df.drop(columns='MEDV', axis=1)
y = df['MEDV']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=0)
# モデリング
clf_rf = RFR()
clf_rf.fit(X_train, y_train)
y_pred = clf_rf.predict(X_test)
# 評価
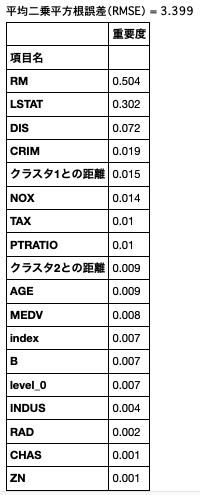
print('平均二乗平方根誤差(RMSE) = {:>.3f}'.format(np.sqrt(mean_squared_error(y_test, y_pred))))
# 重要度
fimp = clf_rf.feature_importances_
# データフレームに変換
imp_df = pd.DataFrame()
imp_df['項目名'] = df.columns[:-1]
imp_df['重要度'] = fimp.round(decimals=3).astype(str)
return imp_df
# 実行
imp_df = rfr_importance(boston_df)
imp_df.sort_values(by='重要度', ascending=False)
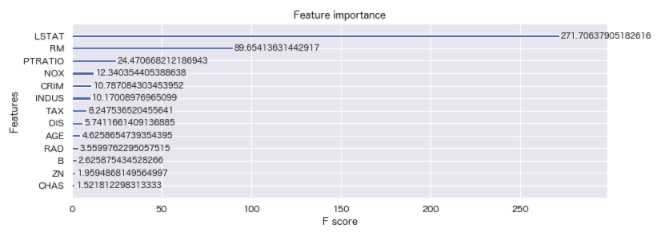
XGBoost版
import xgboost as xgb
def boost_importance(df):
# データ分割
X = df.drop(columns='MEDV', axis=1)
y = df['MEDV']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=0)
# パラメータ
xgb_params = {"objective": "reg:linear", "eta": 0.1, "max_depth": 6, "silent": 1}
num_rounds = 100
# XGBoost用のデータセットの作成
dtrain = xgb.DMatrix(X_train, label=y_train)
# 学習
gbdt = xgb.train(xgb_params, dtrain, num_rounds)
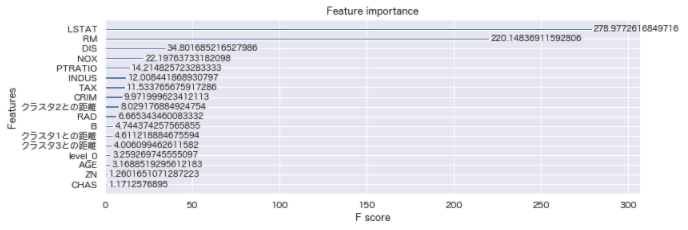
# 重要度
_, ax = plt.subplots(figsize=(12, 4))
# パラメーター:gain 予測精度をどれだけ改善させたることができるできたか(平均値)
xgb.plot_importance(gbdt, ax=ax, importance_type='gain')
# 実行
boost_importance(boston_df)
線形回帰モデル版
from sklearn.linear_model import Ridge, RidgeCV, ElasticNet, LassoCV, LassoLarsCV
from sklearn.model_selection import cross_val_score
def linear_importance(df):
X = df.drop(columns='MEDV', axis=1)
y = df['MEDV']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=0)
def rmse_cv(model):
rmse= np.sqrt(-cross_val_score(model, X_train, y_train, scoring="neg_mean_squared_error", cv = 5))
return(rmse)
# model_ridge = Ridge().fit(X_train, y_train)
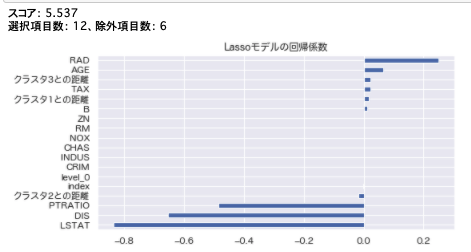
model_lasso = LassoCV().fit(X_train, y_train)
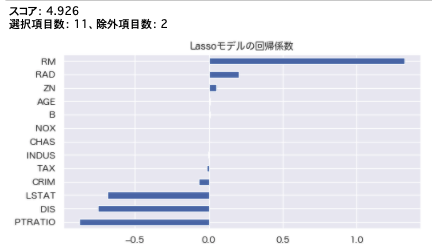
print(f'スコア: {rmse_cv(model_lasso).mean().round(decimals=3)}')
coef = pd.Series(model_lasso.coef_, index = X_train.columns)
print("選択項目数: " + str(sum(coef != 0)) + "、除外項目数: " + str(sum(coef == 0)))
plt.figure(figsize=[8,4])
imp_coef = coef.sort_values()
imp_coef.plot(kind = "barh").grid()
plt.title("Lassoモデルの回帰係数")
# 実行
linear_importance(boston_df)
ランダム・フォレスト + LIME版
import lime
import lime.lime_tabular
def explain_importance(df):
# データ分割
X = df.drop(columns='MEDV', axis=1)
y = df['MEDV']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.3, random_state = 0)
# ランダム・フォレスト(回帰)
model = RFR(max_depth=6, random_state=0, n_estimators=10)
model.fit(X_train, y_train)
# パラメータ
RFR(bootstrap=True, criterion='mse', max_depth=6,
max_features='auto', max_leaf_nodes=None,
min_impurity_decrease=0.0, min_impurity_split=None,
min_samples_leaf=1, min_samples_split=2,
min_weight_fraction_leaf=0.0, n_estimators=10,
n_jobs=None, oob_score=False, random_state=0, verbose=0,
warm_start=False)
# 予測
y_pred = model.predict(X_test)
# 評価
mse = mean_squared_error(y_test, y_pred)**(0.5)
print(f'MSE: {mse}')
# 予測の判断根拠を示す(LIME)
explainer = lime.lime_tabular.LimeTabularExplainer(X_train.values, feature_names=X_train.columns.values.tolist(),
class_names=['MEDV'], verbose=True, mode='regression', random_state=0)
# 解釈結果(予測結果への影響)を表示する
j = 5
exp = explainer.explain_instance(X_test.values[j], model.predict, num_features=6)
exp.show_in_notebook(show_table=True)
# 実行
from IPython.display import Image
explain_importance(boston_df)
plt.savefig("newplot_lime.png")
Image("./newplot_lime.png")
分析通り、RM、LSTATは重要度が高い数値を出している。
CHASは重要度が高いと見ていたが、低い数値となっている。単体では低い数値のため何かしらの変換が必要と思われる。
特徴量エンジニアリング
対応方針
- 正規分布への変換
- MEDV:
予測残差の正規分布性を期待する分析アルゴリズム(線形回帰など)を使用するため、必要と考える。
また、目的変数にマイナス値が既に含まれてしまっているため、Box-Cox変換を用いる。 変換結果にマイナス値を含まれてしまい、線形モデルの評価時にエラーとなるため、Yeo-Johnson変換は行わない。
- MEDV:
- 外れ値
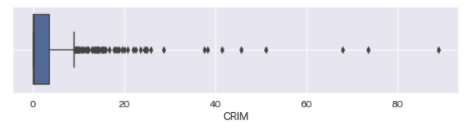
- CRIM:
(大きく外れた)外れ値(80)を含んでいる。犯罪率80%は疑わしい値。線形モデルを使用する(予定)のため、この外れ値を除去する。
- CRIM:
- スケーリング
- NOX, RM, DIS, PTRATIO, LSTAT, MEDV:
(微妙な)外れ値の影響を軽減させたい
線形回帰に対しては有効でないと思われるが、非線形アルゴリズムをアンサンブルで使用する(予定)のため、有効であると考える。
- NOX, RM, DIS, PTRATIO, LSTAT, MEDV:
- 特徴量生成
- クラスタの重心からの距離:
(まだ試してみたことがないため)実験的に効果検証してみる。
- クラスタの重心からの距離:
正規分布への変換
# 変換前の状態
import warnings
warnings.filterwarnings('ignore')
from scipy.stats import norm
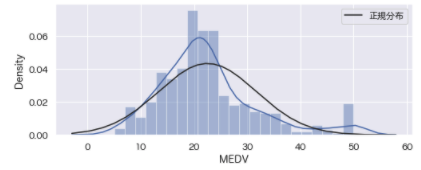
plt.figure(figsize=[8,3])
sns.distplot(boston_df['MEDV'], kde=True, fit=norm, fit_kws={'label': '正規分布'}).grid()
plt.legend();
Yeo-Johnson変換
# Yeo-Johnson変換
from sklearn.preprocessing import PowerTransformer
yeo_johnson_df = boston_df.copy()
sk_yeojohnson = PowerTransformer(method='yeo-johnson') # インスタンス生成
yeojohnson_data = sk_yeojohnson.fit_transform(yeo_johnson_df[['MEDV']]) # 変換
yeo_johnson_df['MEDV'] = yeojohnson_data
import warnings
warnings.filterwarnings('ignore')
from scipy.stats import norm
plt.figure(figsize=[8,3])
sns.distplot(yeo_johnson_df['MEDV'], kde=True, fit=norm, fit_kws={'label': '正規分布'}).grid()
plt.legend()
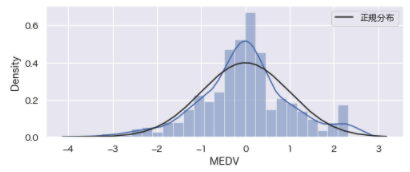
ランダムフォレストには効果があったが、線形モデルには適用できない。評価の過程で目的変数に負値を扱えないため。
Box-Cox変換
# Box-Cox変換
from scipy.special import boxcox1p
boxcox_df = boston_df.copy()
lam=0.15
boxcox_df['MEDV'] = boxcox1p(boxcox_df['MEDV'], lam)
# 描画
plt.figure(figsize=[8,3])
sns.distplot(boxcox_df['MEDV'], kde=True, fit=norm, fit_kws={'label': '正規分布'}).grid()
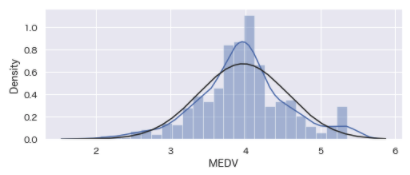
分布の偏りが軽減された。しかし非線形モデルには逆効果だった。
外れ値処理
# 確認
plt.figure(figsize=[10,2])
sns.boxplot(data=boston_df, x='CRIM').grid()
# 外れ値除去
boston_df.shape
boston_df = boston_df[boston_df['CRIM']<40]
boston_df = boston_df.reset_index()
boston_df.shape
# (506, 15)
# (500, 15)
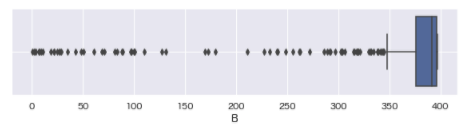
plt.figure(figsize=[10,2])
sns.boxplot(data=boston_df, x='B').grid()
# 外れ値除去
boston_df.shape
boston_df = boston_df[boston_df['B']>10]
boston_df = boston_df.reset_index()
boston_df.shape
# (500, 15)
# (493, 15)
スケーリング
col = ['NOX', 'RM', 'DIS', 'PTRATIO', 'LSTAT', 'MEDV']
boston_df[col].hist(bins=10, figsize=(10,8))
from sklearn.preprocessing import StandardScaler
# 標準化
scaler = StandardScaler()
boston_df['NOX'] = scaler.fit_transform(boston_df[['NOX']])
# 描画
plt.figure(figsize=[8,3])
sns.distplot(boston_df['NOX'], kde=True, fit=norm, fit_kws={'label': '正規分布'}).grid();
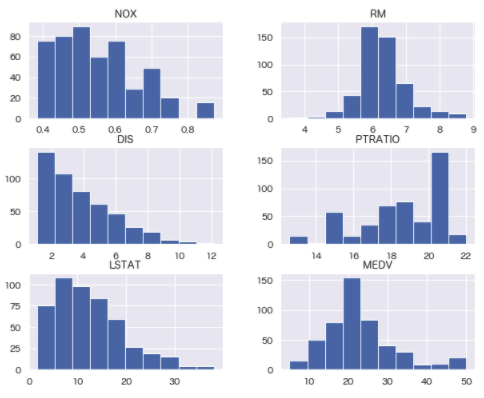
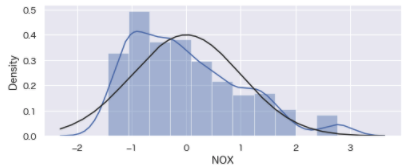
RM, DIS, PTRATIO, LSTAT, MEDVは、標準化しても精度に影響がなかったため、そのままとする。
特徴量生成
クラスタリング、主成分分析
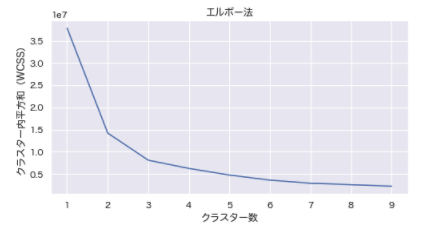
# クラスター数の探索(k-means++)
from sklearn.cluster import KMeans
def elbow(df):
wcss = []
for i in range(1, 10):
kmeans = KMeans(n_clusters = i, init = 'k-means++', max_iter = 300, n_init = 30, random_state = 0)
kmeans.fit(df.iloc[:, :])
wcss.append(kmeans.inertia_)
plt.figure(figsize=[8,4])
plt.plot(range(1, 10), wcss)
plt.title('エルボー法')
plt.xlabel('クラスター数')
plt.ylabel('クラスター内平方和(WCSS)')
plt.grid()
plt.show()
# 実行
elbow(boston_df)
# 主成分分析
from sklearn.decomposition import PCA
# 寄与率
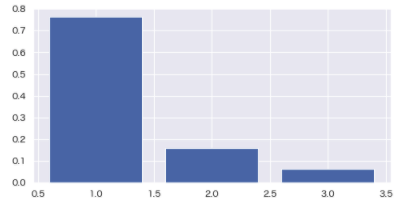
pca = PCA(n_components=3)
pca.fit(boston_df)
plt.figure(figsize=[8,4])
plt.grid()
plt.bar([n for n in range(1, len(pca.explained_variance_ratio_)+1)], pca.explained_variance_ratio_)
# 累積寄与率
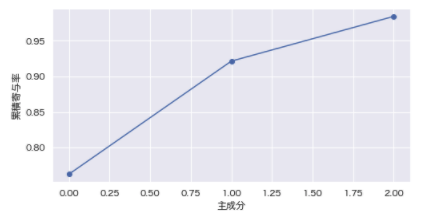
contribution_ratio = pca.explained_variance_ratio_
accumulation_ratio = np.cumsum(contribution_ratio)
cc_ratio = np.hstack([0, accumulation_ratio])
plt.figure(figsize=[8,4])
plt.plot(accumulation_ratio, "-o")
plt.xlabel("主成分")
plt.ylabel("累積寄与率")
plt.grid()
plt.show()
contribution_ratios = pd.DataFrame(pca.explained_variance_ratio_)
contribution_ratios.round(decimals=2).astype('str').head()
print(f"累積寄与率: {contribution_ratios[contribution_ratios.index<5].sum().round(decimals=2).astype('str').values}");
# 結果:累積寄与率: ['0.98']
# クラスタリング(k-means)
from sklearn.preprocessing import StandardScaler
def clustering(df, num):
sc = StandardScaler()
sc.fit_transform(df)
data_norm = sc.transform(df)
cls = KMeans(n_clusters = num)
result = cls.fit(data_norm)
pred = cls.fit_predict(data_norm)
plt.figure(figsize=[8, 4])
sns.scatterplot(x=data_norm[:,0], y=data_norm[:,1], c=result.labels_)
plt.scatter(result.cluster_centers_[:,0], result.cluster_centers_[:,1], s=250, marker='*', c='blue')
plt.grid('darkgray')
plt.show()
# 実行
clustering(boston_df, 3)
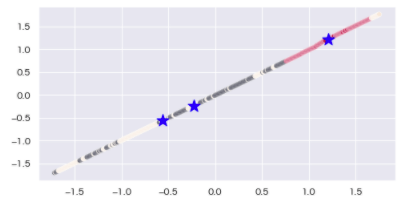
## クラスタリング(k-means)+主成分分析
from sklearn.decomposition import PCA
def cross(df, num):
df_cls = df.copy()
sc = StandardScaler()
clustering_sc = sc.fit_transform(df_cls)
# n_clusters:クラスター数
kmeans = KMeans(n_clusters=num, random_state=42)
clusters = kmeans.fit(clustering_sc)
df_cls['cluster'] = clusters.labels_
x = clustering_sc
# n_components:削減結果の次元数
pca = PCA(n_components=num)
pca.fit(x)
x_pca = pca.transform(x)
pca_df = pd.DataFrame(x_pca)
pca_df['cluster'] = df_cls['cluster']
for i in df_cls['cluster'].unique():
tmp = pca_df.loc[pca_df['cluster'] == i]
plt.scatter(tmp[0], tmp[1])
plt.grid()
plt.show()
# 実行
cross(boston_df, 3)
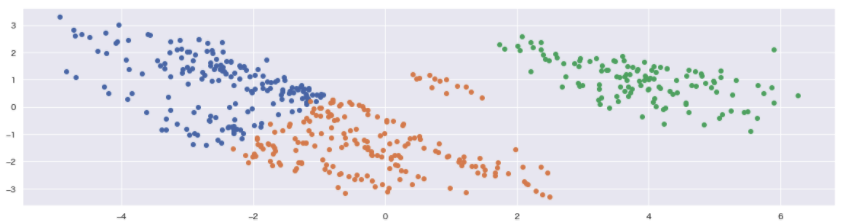
今度はうまく分割することができた。
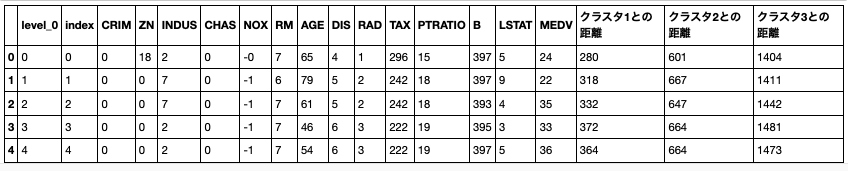
3つのクラスタと、その重心からの距離を算出する。
# 重心からの距離
def get_center_distance(df):
num_cluster=3 # cluster数
clusters = KMeans(n_clusters=num_cluster, random_state = 42)
clusters.fit(df)
centers = clusters.cluster_centers_
columns = df.columns
clust_features = pd.DataFrame(index = df.index)
for i in range(len(centers)):
clust_features['クラスタ' + str(i + 1) + 'との距離'] = (df[columns] - centers[i]).applymap(abs).apply(sum, axis = 1)
return clust_features
# 実行
clust_features = get_center_distance(boston_df)
boston_df[clust_features.columns] = clust_features
boston_df.head()
オーバーサンプリング
# SMOTE
from imblearn.over_sampling import SMOTE
column = 'CHAS'
sm = SMOTE(random_state=42)
X = boston_df.drop(columns=column, axis=1)
y = boston_df[column]
X_sample, Y_sample = sm.fit_resample(X, y)
over_sampling = pd.DataFrame()
over_sampling = X_sample
over_sampling[column] = Y_sample
value_counts = over_sampling[column].value_counts()
df = pd.DataFrame()
df['ラベル'] = value_counts.index
df['件数'] = value_counts.values
ratio=[]
ratio.append((value_counts.values[0] / len(over_sampling[column]) * 100).round(decimals=2).astype('str'))
ratio.append((value_counts.values[1] / len(over_sampling[column]) * 100).round(decimals=2).astype('str'))
df['割合'] = [f'{ratio[0]}%', f'{ratio[1]}%']
print(f"全レコード数:{len(over_sampling[column])}")
df
# 全レコード数:916
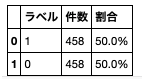
「CHAS」に対するオーバーサンプリングは、ランダムフォレストに対しては効果がある。その際「重心からの距離」がない方が精度が良い。
ただし、線形モデルに対しては、大幅な精度の悪化を招く。一旦、不採用とする。
カウント・エンコーディング
count_map = boston_df['CHAS'].value_counts().to_dict()
count_map
df = boston_df.copy()
df['CHASカウント'] = df['CHAS'].map(count_map)
df[['CHAS', 'CHASカウント']].head()
df = df.drop(columns='CHAS', axis=1)
sns.distplot(df['CHASカウント'])
# 結果:{0: 458, 1: 35}
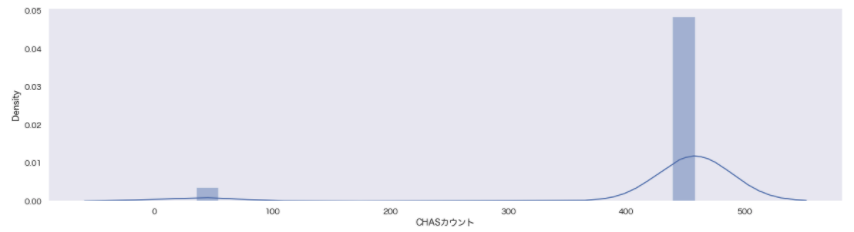
CAHSは住宅価格帯が異なるため、影響があると思ったが、得に影響はなかった。
出現頻度を学習させ、大小を明確にすれば影響が出ると考えたが、むしろ精度が悪化した。
一旦、このカウント・エンコーディングを採用するかは保留とする。
再評価
# 実行
rfr_model, y_test, y_pred = learning(RFR(random_state=42), boston_df)
決定係数(R2) = 0.924
平均絶対誤差(MAE) = 1.761
平均二乗誤差(MSE) = 5.588
対数平均二乗誤差(MSLE) = 0.018
平均二乗平方根誤差(RMSE) = 2.364
対数平方平均二乗誤差(RMSLE) = 0.134
# 線形回帰(重回帰)
from sklearn.linear_model import LinearRegression as LR
rfr_model, y_test, y_pred = learning(LR(), boston_df)
決定係数(R2) = 0.811
平均絶対誤差(MAE) = 2.919
平均二乗誤差(MSE) = 13.928
対数平均二乗誤差(MSLE) = 0.048
平均二乗平方根誤差(RMSE) = 3.732
対数平方平均二乗誤差(RMSLE) = 0.219
# 線形モデル
from sklearn.linear_model import Ridge
rfr_model, y_test, y_pred = learning(Ridge(), boston_df)
決定係数(R2) = 0.81
平均絶対誤差(MAE) = 2.924
平均二乗誤差(MSE) = 13.977
対数平均二乗誤差(MSLE) = 0.048
平均二乗平方根誤差(RMSE) = 3.739
対数平方平均二乗誤差(RMSLE) = 0.219
# 線形モデル
from sklearn.linear_model import Lasso
rfr_model, y_test, y_pred = learning(Lasso(), boston_df)
決定係数(R2) = 0.745
平均絶対誤差(MAE) = 3.172
平均二乗誤差(MSE) = 18.792
対数平均二乗誤差(MSLE) = 0.042
平均二乗平方根誤差(RMSE) = 4.335
対数平方平均二乗誤差(RMSLE) = 0.204
# 線形回帰(重回帰)
from sklearn.linear_model import ElasticNet
rfr_model, y_test, y_pred = learning(ElasticNet(), boston_df)
決定係数(R2) = 0.759
平均絶対誤差(MAE) = 3.112
平均二乗誤差(MSE) = 17.745
対数平均二乗誤差(MSLE) = 0.04
平均二乗平方根誤差(RMSE) = 4.212
対数平方平均二乗誤差(RMSLE) = 0.201
全体的に精度の向上が見られる。
特徴選択
# 実行
imp_df = rfr_importance(boston_df)
imp_df = imp_df.sort_values(by='重要度', ascending=False).reset_index().set_index("項目名")
imp_df = imp_df.drop(columns="index", axis=1)
imp_df
# 実行
boost_importance(boston_df)
# 実行
linear_importance(boston_df)
explain_importance(boston_df)
結果を踏まえて再評価
features = boston_df.drop(columns=['INDUS', 'CRIM', 'level_0', 'index', 'クラスタ3との距離']).columns
# 実行
rfr_model, y_test, y_pred = learning(RFR(random_state=42), boston_df[features])
決定係数(R2) = 0.929
平均絶対誤差(MAE) = 1.712
平均二乗誤差(MSE) = 5.248
対数平均二乗誤差(MSLE) = 0.017
平均二乗平方根誤差(RMSE) = 2.291
対数平方平均二乗誤差(RMSLE) = 0.129
# 線形回帰(重回帰)
from sklearn.linear_model import LinearRegression as LR
rfr_model, y_test, y_pred = learning(LR(), boston_df[features])
決定係数(R2) = 0.827
平均絶対誤差(MAE) = 2.803
平均二乗誤差(MSE) = 12.766
対数平均二乗誤差(MSLE) = 0.036
平均二乗平方根誤差(RMSE) = 3.573
対数平方平均二乗誤差(RMSLE) = 0.189
# 線形モデル
from sklearn.linear_model import Ridge
rfr_model, y_test, y_pred = learning(Ridge(), boston_df[features])
決定係数(R2) = 0.827
平均絶対誤差(MAE) = 2.799
平均二乗誤差(MSE) = 12.736
対数平均二乗誤差(MSLE) = 0.036
平均二乗平方根誤差(RMSE) = 3.569
対数平方平均二乗誤差(RMSLE) = 0.189
# 線形モデル
from sklearn.linear_model import Lasso
rfr_model, y_test, y_pred = learning(Lasso(), boston_df[features])
決定係数(R2) = 0.767
平均絶対誤差(MAE) = 3.124
平均二乗誤差(MSE) = 17.171
対数平均二乗誤差(MSLE) = 0.032
平均二乗平方根誤差(RMSE) = 4.144
対数平方平均二乗誤差(RMSLE) = 0.179
# 線形回帰(重回帰)
from sklearn.linear_model import ElasticNet
rfr_model, y_test, y_pred = learning(ElasticNet(), boston_df[features])
決定係数(R2) = 0.773
平均絶対誤差(MAE) = 3.073
平均二乗誤差(MSE) = 16.698
対数平均二乗誤差(MSLE) = 0.031
平均二乗平方根誤差(RMSE) = 4.086
対数平方平均二乗誤差(RMSLE) = 0.176
新宿の例といい、犯罪率は住宅価格との関係はないのかもしれない。
CHASは「0⇆1」で住宅価格が異なっていたため、影響はあると思われたが、「CHAS」単体では影響があまりなかった。
まとめ
実際に様々な手法を試して見て、効果があったもの、なかったものがあった。特に外れ値はどこまでを外れ値とするのかが判断が付けられず、モデル構築時の評価も加味して決めていきたい。
また過学習かどうかをまだ評価できていないため、モデル構築時(前)に確認することにします。
最後に
他の記事はこちらでまとめています。是非ご参照ください。
解析結果
実装結果:GitHub/boston_regression_preprocessing.ipynb
データセット:Boston House Prices-Advanced Regression Techniques
参考資料