0.はじめに
大阪大学AI & Machine learning Society(AIMS)主催、全国医療AIコンテスト 2020という医療テーブルデータコンペで優勝(1st place, top 6%)したので、解法を投稿します。
今までの記事をご覧いただいている方はご存知かもしれませんが、僕は画像データ専門でやっているので、テーブルデータのコンペはあまり得意ではありません。
間違っている所、合理的でない部分もあるかと思いますがご了承ください。
また、本記事は基本的にはコンペに参加された方に対して書いています。具体的な列名・特徴量について説明はありませんので、そういった部分は読み飛ばして頂いて構いません。(手法的な部分に関しては、ある程度詳しく説明しています!)
同様に、プライベートなコンペであったことから、参加者以外のコンペサイトへのアクセスは出来ませんので、ご了承ください。
1.コンペについて
1-1.コンペ概要
コンペはCOVID-19罹患者の死亡予測に関するテーブルデータコンペです。
与えられた死亡につながると考えられるリスク要因(所在地、肺炎の有無、年齢など)に対してのデータ分析を行い、モデルを作成、提出します。
コンペ期間は2日(9/26 14:00頃 ~ 9/27 12:00)と非常に短期のものとなっており、どれだけ高速に推論と実装、最適化を行えるかが鍵となりました。
また、コンペ参加は学生限定となっていました。
1-2.評価指標
評価指標はROC曲線下面積です。この評価指標に関しては既に下記記事にて説明済みですので、省略します。
メラノーマコンペ-ROC曲線下面積
1-3.データセット
データセットはシンプルに
・train.csv
・test.csv
・sample_submission.csv
の3つから成っています。trainで学習、testを予測、sample_submissionの形式で提出となります。
データ内容については、以下のようなものがありました。
・患者の住所(詳細)
・年齢
・病院の所在
・患者の出身地
・患者が治療を受けた施設
・他のCOVID患者と接触歴があるか
・PCR検査結果
・肺炎
・発症日
・入院日 (受診日)
・挿管
・入院or外来
・慢性腎不全、糖尿病、高血圧、心血管疾患、ぜんそく、その他基礎疾患の有無
test.csvを予測した結果が実際にLeaderBoardに反映されるコンペ(Code Competitionではない)ので、testデータとして与えられる量が多く、Pseudo Labelingなどの手法が有効になると考えられました。
2.EDA
一切する必要がありませんでした。というのも、コンペ運営のAkiyama氏がEDAとbaselineを兼ね備えたnotebookをコンペ開始時に公開しており、殆ど必要な情報は全て揃いました。
特に、LightGBMのfeature_importanceのスコアまで載っており、もう後はモデルを実装するだけ、という状態でした。
非常に分かりやすいデータ分析で、すぐにモデル実装に取り掛かることができました。
3.理論
理論では、今回僕の解法の鍵となった手法に関して説明していきます。
3-1. Target Encoding
target encodingとは、カテゴリ変数において、"そのカテゴリの正解ラベルの平均値"自体を特徴量にする手法です。
正解ラベルそのものを間接的に特徴量として用いるため、リークに注意が必要であり、実装も結構面倒です。
今回この手法が必要になった理由は、477のカテゴリを持つ"place_patient_live2"というカテゴリ変数が存在したからです。通常、NNでカテゴリ変数を用いる場合OneHotEncodingによって処理しますが(GBDTにおいてはカテゴリ変数はそのまま使用できます)、477の列を作成し、そのうち1つだけが意味を持っているとなると、多くの列が無駄な特徴量となってしまうことが分かります(=疎な行列)
そこで、target encodingを用いることによって大幅に列数を減らし(477→1)、効率よく学習を行えるようにする必要がありました。
実際に、OneHotEncodingで学習した場合と、target encodingで学習した場合、僕のモデルでは後者の方が精度が大幅に良かったです。
3-2. Pseudo Labeling
Pseudo Labelingは半教師あり学習手法の1つです。trainingデータのみではなく、testデータも学習することにより、精度の向上を目指します。まず、通常のモデルでtrainingデータを学習、testデータを評価し、予測値を出しますが、この予測値をそのままtestデータのラベルとして使ってしまい、もう一度0からtraining+testデータでの学習を行います。
これによって、trainingの予測結果がある程度正しければ、testデータの分の多様性を獲得することが出来ます。
この手法を用いる場合には、testデータが十分に与えられている必要があり、Code Competitionのようなtestデータが殆ど与えられないコンペにおいては使用できません。また、使用するテストデータの量にも気をつける必要があり、およそtrain:test = 2:1となるような量を用いるのが良いと言われています。
この手法を用いることによって、単体のモデルの精度としては、
- Public LB 0.96696→0.96733
と、大幅な精度の向上を達成しました。
今回は実際には、NNモデルとLGBMモデルのアンサンブルの出力結果をtestデータのラベルとして用いました。
4.検証
4-1.方針
元々、このコンペが始まる前に、テーブルデータであろうということはおおよそ見当はついていたので、EDA→LightGBMによるベースラインの作成→特徴量のWeight抽出→NN実装、の流れで行こう!と考えていたのですが、コンペ開始時点で、運営のAkiyama氏によるEDAとLightGBMによる実装、特徴量のWeightまで公開されていたので、もうあんまりやることがない…という状態でした。(1から実装するのを楽しみにしていたので、コンペ経験者側としてはちょっと残念…)
ということで、LightGBMによる精度はおおよそ分かったので、PytorchによるNNモデルの実装をベースラインとして行いました。
(とりあえずLightGBMのまま、特徴量の作成に取り掛かる、という方針も考えられましたが、Feature Engineeringに関する知識があまり無い為、いきなりNNの作成に取り掛かりました。)
4-2.使用する特徴量の選択と使い方
使用する特徴量は既に公開されているLGBMモデルのものを殆ど全部使いました。
それぞれ適応する特徴量の作成手法が異なるので、ざっくりとベースラインで使用したものを以下に記述します。
・Standardization
標準化。平均0,分散1にスケーリングする。
standard_cols = [
"age",
"entry_-_symptom_date",
"entry_date_count",
"date_symptoms",
"entry_date",
]
・Onehot Encoding
カテゴリ変数の処理。カテゴリの数だけ列を作成し、その列だけ1,他を0とする。
onehot_cols = [
"place_hospital",
"place_patient_birth",
"type_hospital",
"contact_other_covid",
"test_result",
"pneumonia",
"intubed",
"patient_type",
"chronic_renal_failure",
"diabetes",
"icu",
"obesity",
"immunosuppression",
"sex",
"other_disease",
"pregnancy",
"hypertension",
"cardiovascular",
"asthma",
"tobacco",
"copd",
]
・Target Encoding
そのカテゴリ全体の正解ラベルの平均を特徴量として用いる手法
target_E_cols = ["place_patient_live2"]
・日付に関するデータ
まず、日付として与えられるデータ(2020/09/26)を連続な値として考えられるデータ(例:9/26を0とした場合、9/27 : 1, 9/28 : 2)に変更します。その後、最大値を1,最小値を0としたmin_max_encodingを行います。
4-3.ベースライン
name : medcon2020_tachyon_baseline
about : simple NN baseline
model : Liner Model
batch : 32
epoch : 20
criterion : BCEWithLogitsLoss
optimizer : Adam
init_lr : 1e-2
scheduler: CosineAnnealingLR
data : plane
preprocess : OnehotEncoding,Standardization,Target Encoding
train_test_split : StratifiedKFold, k=5
Public LB : 0.96575
4-4.LightGBMモデル
特徴量はAkiyama氏のFeature Engineering結果をそのまま用い、Optunaをかけることによりパラメータチューニングを行いました。
結果、
Akiyama氏のbaseline : Public LB0.96636
Optunaを用いたモデル : Public LB0.96635
と殆ど同じ結果が出たため、このbaselineは相当完成されたモデルであったことが伺えます。
但し、パラメータとしてはある程度異なるモデルなので、アンサンブル効果(多様性によって精度が向上すること)はある程度期待できます。
4-5.アンサンブル提出
4-2のベースラインのパラメータを調整し、ある程度精度が出るようになってから、まずNNモデル1つと、Akiyama氏のbaselineモデルをAverage Ensemblingしたものを提出してみました。
※Public LB
NN単体 : 0.96696
LGBM単体 : 0.96636
Average Ensembling : 0.96738
思ってもみない精度の向上です。NNとGBDTのアルゴリズムが全く異なるため、大幅にスコアが上がったと考えられます。
当時、ブッチギリ1位になりました。
4-6.Pseudo Labeling
2日目(最終日)に、3.理論で説明したPseudo Labelingを実装しました。もっと手っ取り早く精度を上げる手法が存在したのかもしれませんが、僕にはこれしか思い浮かびませんでした…。
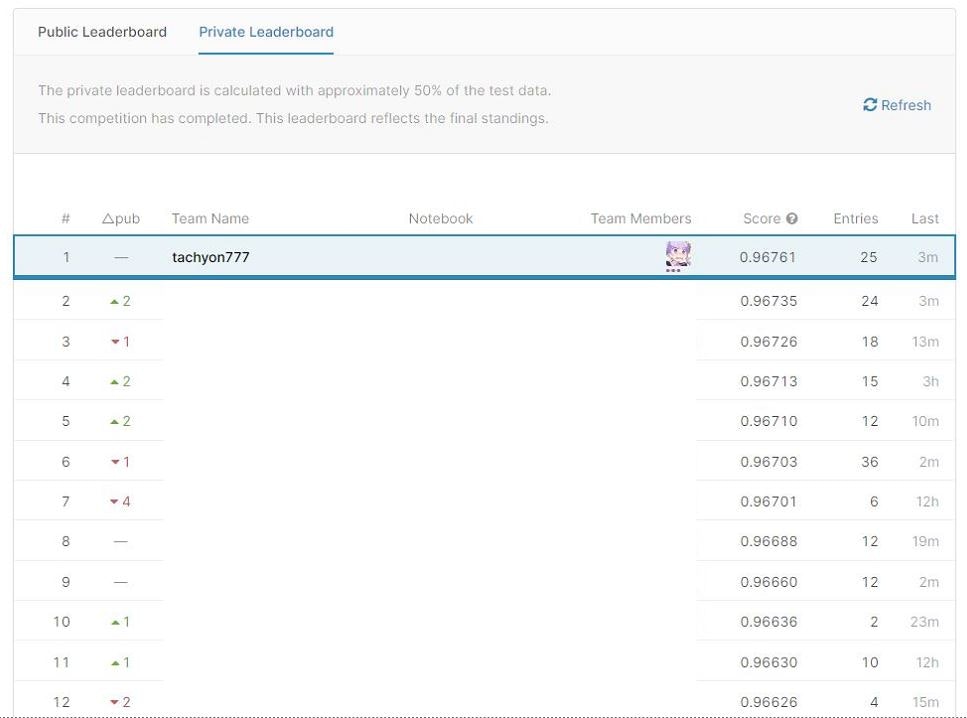
Pseudo Labeling実装前Average Ensembling Score : Private : 0.96753 Public : 0.96761
Pseudo Labeling実装後Average Ensembling Score : Private : 0.96761 Public : 0.96768
結局、この2つを最終submissionに選びました。結果、後者が優勝モデルとなりました。
4-7.Pipeline
PowerPointのスライドで恐縮ですが、最終的に以下のようなパイプラインで完成しました。
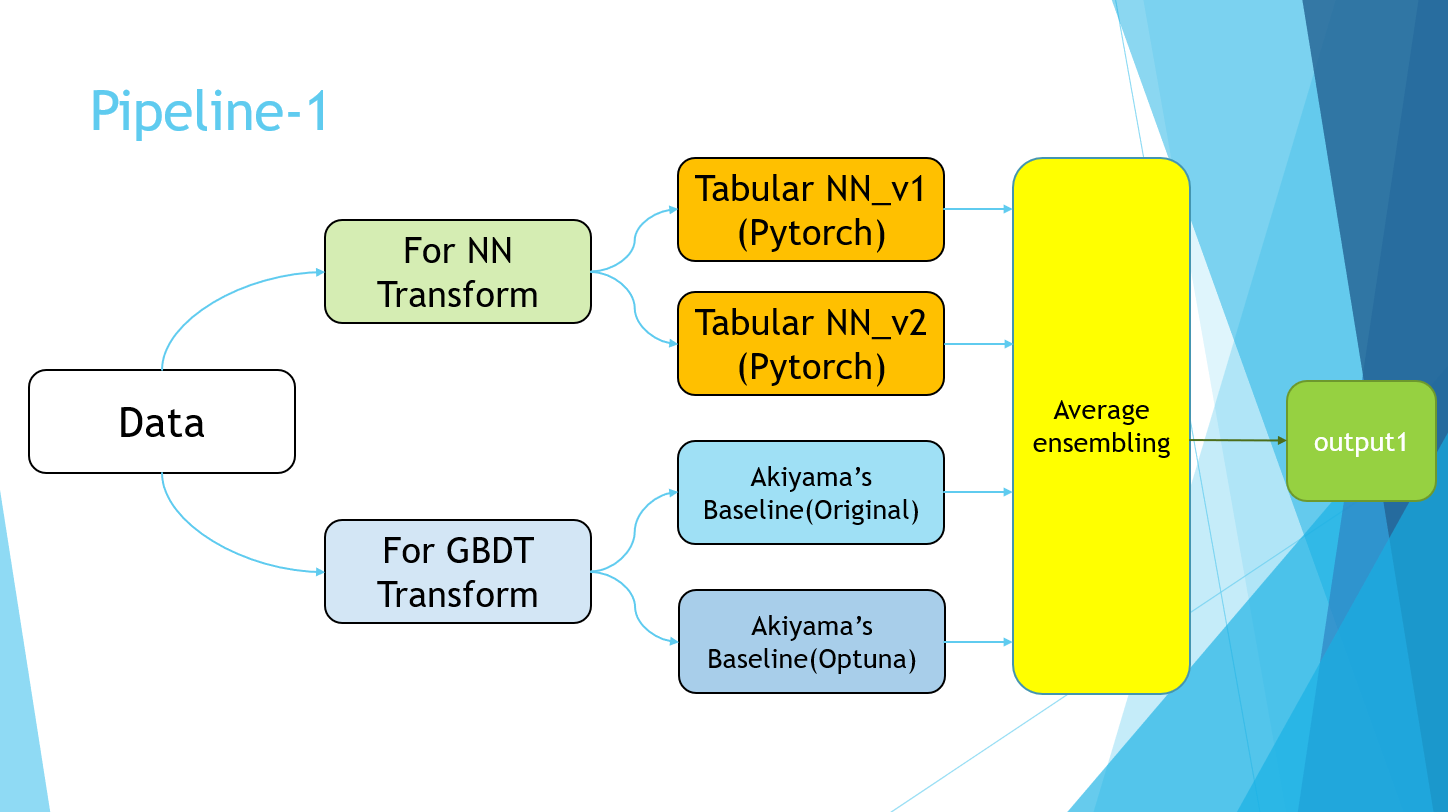
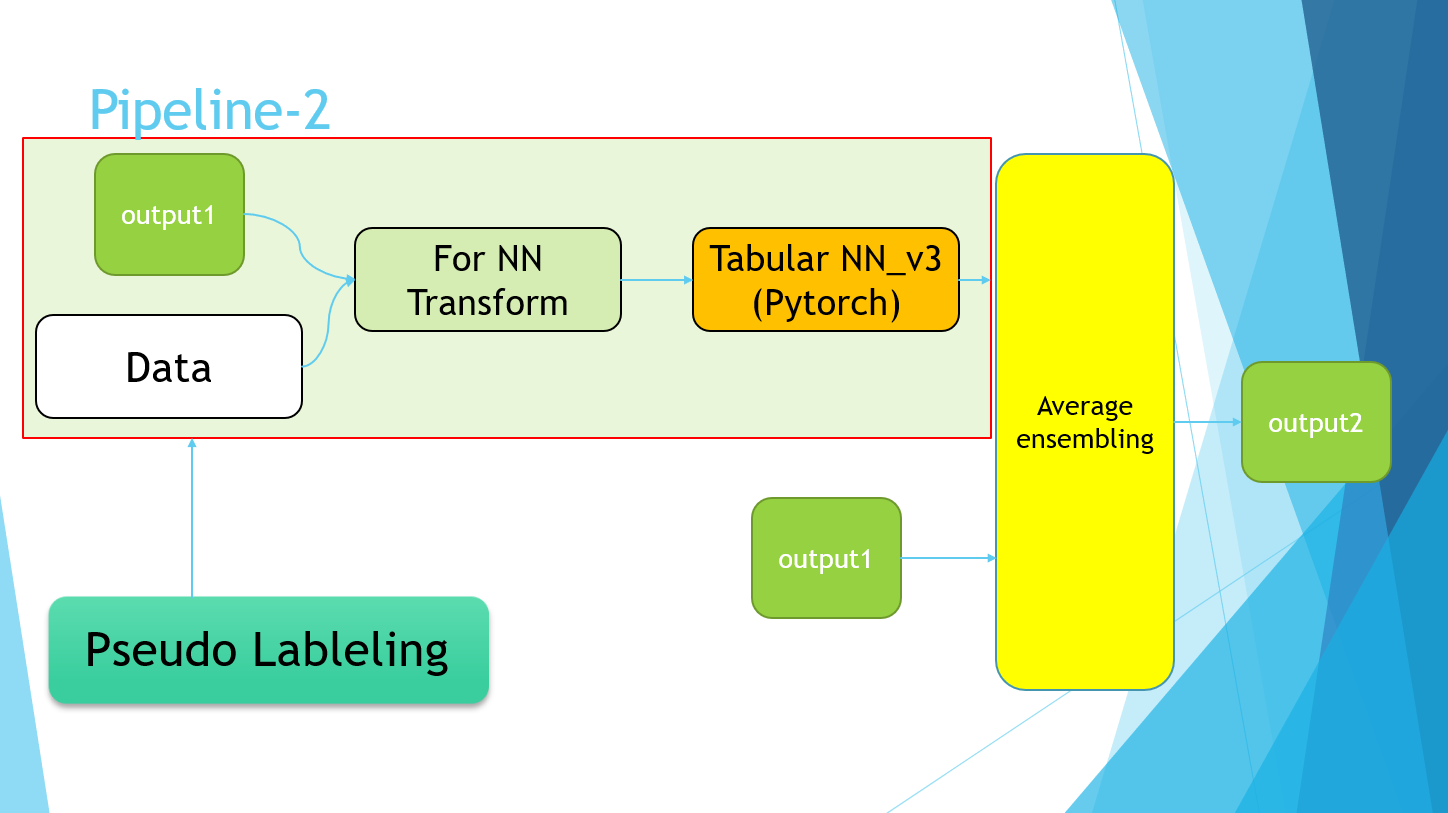
Pipeline-2の部分は、単純にPseudo Labelingの実装を指しています。
最終的に、Pipeline-1の4モデルと、Pipeline-2のPseudo Labelingデータを用いて学習を行ったモデルの平均値を取りました。
5.結果
優勝しました。(今までの人生で、優勝したことなんてあったかな…?)
手法に関しても、自分の中では申し分ないほどやりきったという感じで、達成感があります。
2日目はずっと1位にいて抜かされないか心配だったので、嬉しいと言うよりもホッとした気持ちが大きかったです…
6.考察
今回はプライベートなコンテストというのもあり、他の方の解法を上げるのは控えさせていただきます。
他に試したかったこととしては、上位から
・特徴量の作成
・catboost
・NNモデルのハイパーパラメータ調整
という感じです。
実際にkaggleでこのデータでコンペが開かれたとなれば、僕のスコアも銅メダル以下になるでしょうから、まだまだ改善の余地はあると思われます。
7.終わりに
大阪大学AI & Machine learning Society(AIMS)を始めとした主催者の皆様、一日目の発表者の皆様、コンテスト参加者の皆様、大変お疲れさまでした!
僕の解法が少しでも参考になれば幸いです。
8.参考文献
1.門脇大輔 阪田隆司 保坂桂佑 平松雄司 Kaggleで勝つ データ分析の技術 技術評論社 2019