はじめに
スイスの国立研究所PSIのスピンオフベンチャーDECTRISの1ピクセル1円1とも言われる二次元高分解能X線検出器は、手軽に買えない価格にもかかわらずいまや世界中の大学や放射光実験施設に導入されています。実験室系X線の回折装置や小角散乱装置にも採用されており日常的に使っている方もいるでしょう。

高分解能ということはデータ量が増えることを意味します。ビーム強度が強く測定時間の短い放射光実験では、メガピクセルサイズの32ビット二次元配列が世界中の装置で数秒ごとに生成されています。これらの膨大なデータは、放射光施設自体が仕様策定に関わったHDF5ファイルにバイナリとして保存されています。バイナリデータだけに扱いにはそれなりの道具立てが必須です。数百枚の連続測定データを持ち帰ることがざらにある放射光実験のユーザーには、測定データの多さやファイルサイズに加えてデータそのものの扱いにくさに参っている方もいるでしょう。もちろん、必要なデータを厳選してテキストファイルや扱い慣れたフォーマットに変換し、従来のワークフローに乗せるということも可能です。しかし、PythonとJupyter notebookの組み合わせにより科学データの取り扱い方に新しい潮流が生まれつつある中で、それらを活用して大量のデータを一括で扱うソフトウェア技術を身につけることは、ハードウェアの革命により生まれた新しいサイエンスの可能性を探るのに不可欠な足場とも言えます2。この記事では、X線回折やX線小角散乱という裾野の広い実験手法に起きた超高速な高分解能二次元データ取得というハードウェアによる革命に自力で対応したい方のために、解析の第一歩であるデータの読み取りの方法を述べます。
目的
従来の解析ソフトで扱えるフォーマットに変換するツールをすでに導入しているBLもあると思いますが3、ここでは装置や測定条件に関するメタ情報とカウントデータを、大量データに対する自己流の解析・可視化ワークフローを適用しやすいPythonを使って読み取ることを目的にします4。BLスタッフが用意したデータ処理ソフトウェアを使って扱いやすい形式にすることが多いのか、日本語に限らず英語でも執筆時点ではDECTRISのHDF5ファイル読み取りに関する解説はまだウェブにはないようです。
HDFファイルを扱う際の注意点
DECTRISのデータ(ここではEIGER 4M)を扱うにあたって、知っておくべき点がいくつかあります。詳しくは理研放射光科学研究センターの山下さんのGitHubに書いてありますが、ここでは読み取りに関わるHDF5の圧縮機能と外部リンク機能についてだけ述べます。
圧縮フィルター機能
データ圧縮はHDF5の透過的(transparent)な機能の一つです。透過的というのはユーザーがあまり意識せずとも自動的に機能してくれるという意味です。HDF5関連ドキュメントでは、圧縮機能は透過であることをフィルターに例えて圧縮フィルター(compression filter)とも呼ばれています。DECTRISのHDF5データの圧縮にはbitshuffle+lz4(bslz4)という形式が使われています5。bitshuffleもlz4もHDF5標準の圧縮フィルターではないのでプラグインとしてユーザーが追加する必要があります。Pythonのh5pyパッケージを使ってHDF5を読み取る場合は、ESRFのソフトウェアチームの方が用意したhdf5pluginというパッケージをインストールすれば簡単に両者を導入できます。
外部リンク機能
HDF5の規格には大量の実験データが日々生成される放射光実験施設の提案によるものもあります。外部リンク(External link)機能はその中の一つから発展した機能のようです6。外部リンク機能は、通常のパス表記を使って異なるHDF5ファイルのDatasetにアクセスできる機能です。例えば、連続測定によって大量に得られたデータを扱う際に、全データ共通のメタ情報のみを含むHDF5ファイル(通称マスターファイル)に設定してある各測定結果への外部リンクを使うと、マスターファイルを読み込むだけで別ファイルに保存してある全測定データにシームレスにアクセスできます。I/Oパフォーマンスに関する良し悪しは検証していませんが、コードがかなり簡潔になるのは確かです。
準備
hdf5pluginはpipで、ほかはcondaからインストールできます。
%matplotlib inline
import hdf5plugin # 環境変数のパスを設定をしているので必ずh5pyより先にインポート
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.colors import LogNorm
import h5py
from matplotlib import __version__ as mplv
print('Numpy:', np.__version__)
print('matplotlib:', mplv)
print('h5py:', h5py.__version__)
plt.rcParams['image.origin'] = 'lower' # imshowの原点を左下に
# 明示的に読み込むのはマスターファイルのみ
masterhdf = '/dummy/path/to/data/EIGER_4M_test_master.h5'
この記事ではマスターファイルのあるフォルダには以下のような測定データあるとします。マスターファイルと測定ファイルの名前の共通部分は外部リンクが正しく機能するために必要です。
> ls /dummy/path/to/data
EIGER_4M_test_data_000001.h5
EIGER_4M_test_data_000002.h5
EIGER_4M_test_data_000003.h5
EIGER_4M_test_data_000004.h5
EIGER_4M_test_master.h5
HDFファイルの中身を確認する
この記事に書いてある方法を使って、データ構造とdatasetの中身を確認します。ただし、外部リンクのデータはこの方法では表示されません。
def PrintOnlyDataset(name, obj):
if isinstance(obj, h5py.Dataset):
print(name)
print(obj.value)
with h5py.File(masterhdf,'r') as f:
f.visititems(PrintOnlyDataset)
bslz4フィルターが機能していれば以下のような内容が出力されます。マスターファイルの内容は全測定に共通な検出器情報が主であることがわかります。
entry/instrument/beam/incident_wavelength
0.7208383
entry/instrument/detector/beam_center_x
0.0
entry/instrument/detector/beam_center_y
0.0
entry/instrument/detector/bit_depth_image
32
entry/instrument/detector/bit_depth_readout
12
entry/instrument/detector/count_time
0.48999998
entry/instrument/detector/countrate_correction_applied
1
entry/instrument/detector/description
b'Dectris Eiger 4M'
...
...
データ読み込み後のワークフローで波長や検出器距離などが必要になったときは、ここでパスを確認しておくと必要な時に簡単に取得できます。
測定データとマスクの読み込み
マスターデータのentry/data以下にdata_00****という形で1-2000までの番号が振られた2000個の外部リンクがあらかじめ用意されています。例えば、マスターファイルと同じ場所に4個の連番測定ファイル(000001-000004)が置いてある私の環境で以下のスクリプトを実行してみます。
with h5py.File(masterhdf,'r') as f:
# {i:04}でゼロ埋めした整数4桁というフォーマットを指定
for p in [f'entry/data/data_00{i:04}' for i in [0, 1, 4, 2000, 2001]]:
try:
f[p] # 外部リンク先にファイルが存在するかチェック。
print(p)
except KeyError as e: # ファイルまた外部リンク自体がなければKeyError
print(e)
"Unable to open object (object 'data_000000' doesn't exist)"
entry/data/data_000001
entry/data/data_000004
"Unable to open object (unable to open file: name = '/dummy/path/to/data/EIGER_4M_test_002000.h5', errno = 2, error message = 'No such file or directory', flags = 0, o_flags = 0)"
"Unable to open object (object 'data_002001' doesn't exist)"
五つのprintの結果が意味するところは以下の通りです。
- 外部リンクに
data_000000は存在しない、つまり連番は1始まり - 1番目のデータのマスターファイル上の外部リンクパス(OS上のファイル名は
EIGER_4M_test_data_000001.h5) - 4番目のデータのマスターファイル上の外部リンクパス(OS上のファイル名は
EIGER_4M_test_data_000004.h5) - マスターファイルに外部リンク
data_002000は設定されているが、マスターファイルと同じ場所にEIGER_4M_test_data_002000.h5が存在しない - 外部リンクは1-2000なので
data_002001は存在しない
これはGUIソフトのHDFViewでマスターファイルを開いても確認できます。実際に開いて下までスクロールすれば、実際のファイルが存在しないことを表す"?"がdata_002000まで並んでいることがわかります。
測定データの数はマスターファイルと同じ場所にあるEIGER_4M_test_data_******.h5の数を見ればわかります。ここでは4Mピクセルのデータが500枚格納された測定結果ファイルを二つ指定して500枚の和を読み込みます。可視化で必要なマスクも読み込んでおきます。
imgdatasets = ['entry/data/data_000001', 'entry/data/data_000002'] # 外部リンク
imgs = {} # 読み込み済みデータ格納先
with h5py.File(masterhdf, mode='r') as f:
# 測定ファイル二つからそれぞれ500枚のデータの和をとる
for imgdataset in imgdatasets:
numimg = f[imgdataset].shape[0]
temp = np.zeros_like(mask)
# EIGER 4Mのデータx500が大きすぎるのかnp.sumが異常に遅い
# 通常は遅くてご法度とされるNumpy arrayのforループで代用
for n in tqdm(range(numimg)):
temp += f[imgdataset][n]
imgs[imgdataset] = temp
# マスクデータ
mask = f['entry/instrument/detector/detectorSpecific/pixel_mask'].value
二次元データ表示
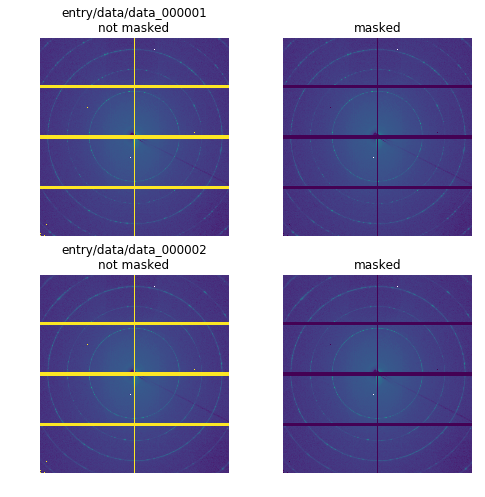
Pythonで測定データを読み込めたらまずやりたいのは可視化です7。ここではもっとも基本的な二次元画像としての可視化をしてみます。
nrow, ncol = len(imgs), 2
fig, axes = plt.subplots(nrow, ncol, figsize=(8, 4*nrow))
vmax = 1e7
for (k, v), ax in zip(imgs.items(), axes):
temp = v.copy()
ax[0].imshow(temp, norm=LogNorm(), vmax=vmax)
ax[0].set_title(f'{k}\nnot masked')
ax[0].axis('off')
# 検出器モジュール間の隙間や異常ピクセルをマスク
# 整数型のarrayにnp.nanは代入できないので弱い強度として1を代入
temp[mask != 0] = 1
ax[1].imshow(temp, norm=LogNorm(), vmax=vmax)
ax[1].set_title('masked')
ax[1].axis('off')
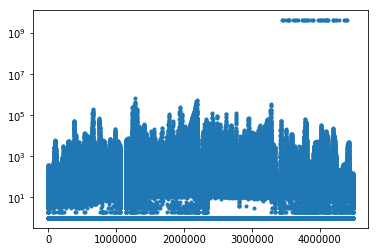
マスク済みのほうを見ると、両データに共通する位置にマスクしきれていない異常ピクセルに見える点がありますが、これは画像の解像度の問題のようで、PDFにして拡大すると何も異常はありません。しかし、全体が妙に暗いのでどこかに周囲に比べて異常に強いピクセルが残ってるようです。マスク済みの最後のtempを一次元プロットして見ると、予想通り一定の強度の異常ピクセルが残っていることがわかるので、この部分を追加でマスク(実際は弱い強度として1を代入)します。
plt.semilogy(temp.ravel(), '.')
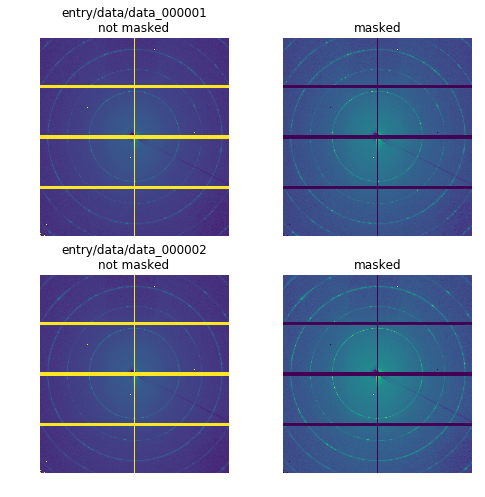
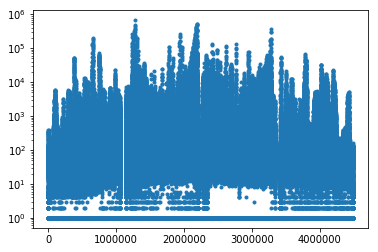
先ほどのマスク部分を以下のようにして再度同じことをしてみます。
temp[mask != 0] = 1
temp[temp > 1e9] = 1
異常ピクセルに$10^0$が代入され、全体の色合いも正しそうな感じになりました。
この先のワークフロー
ここで紹介したデータの読み込みの次はPONIファイルを使った歪み補正やPyFAIによる一次元化、さらには測定やテーマごとの各種解析という具合に進んでいきます。温度などの測定条件を変えた連続実験の場合はpandasのDataFrameを使った測定条件管理が役立つかもしれません。
-
噂で耳にする値段を画素数で割ると、確かにこれくらいのオーダーになります。 ↩
-
ハードウェアとソフトウェアのどちらが足場であるかという議論はここではしません。 ↩
-
競争が激しいタンパク質構造解析関連のBLはやはりこの手の対応が早いようで、例えば名前がおもしろい理研のKAMOやケンブリッジの会社のautoPROCなどがあります。 ↩
-
DECTRIS公式のALBULAというソフトウェア(要ユーザー登録)には測定データの可視化機能に加えてPython APIも用意されていますが、使い方の説明がまだ貧弱です。おそらくこの記事で使っているh5pyのラッパーだとは思いますが、現状では使うにあたって自分でh5pyを使うのと同じ程度の手探りが必要な状況です。むしろラッパーであるがゆえにわかりにくくなっている部分が増えている可能性も高いです。 ↩
-
イギリスの放射光実験施設Diamondの文書にある"VIRTUAL DATA SETS"がsoft link機能として導入され、external linkはその発展形として後のバージョンで導入されたそうです。 ↩
-
ちなみに、可視化だけであればALBULAでも可能です。画面サンプルはEIGERのマニュアルにあります。試しに使って見ましたが、EIGER 4Mの連続測定データ数百回分のアニメーション再生が非常に軽快でした。ただ、測定中にその場でデータ確認する分には非常に有用だと思いますが、まだできることが少なくシンプルな可視化機能しかありません。また、漏れなくアップデートを通知するためかユーザー登録も必須です。 ↩