一覧表の一歩先へ
時分割測定、温度や磁場依存性測定、あるいは合成条件の異なる複数試料の比較測定などを行った後は、試料や測定条件をリストにまとめることが多いでしょう。リストを作るといえばエクセルですね。装置の測定リストCSV出力機能やまさかの手入力&コピペ、あるいはフリーソフトを駆使するなど入力経路は色々あると思いますが、最終的にはエクセル等の表計算ソフトで測定リストを管理してる人が多いのではないでしょうか。紙へ出力したりスクロールでことが済む数のデータを眺める目的ならばエクセルはちょうどいい道具です。しかし、___測定リストをエクセルで管理すると「測定パラメータをある順番で並べ、セルの色やフォントを変えて視認性を向上させて整然と表示する」というただの一覧表以上の使い方はなかなか実現しづらい___と思います1。ここではpandasという統計や機械学習でよく使われるパッケージのDataFrameというオブジェクトを使った、ただの一覧表から一歩進んだ測定条件の管理や解析への活用アイディアを紹介します。
パッケージ
Python 3.6 + Jupyter notebookを使った環境で以下のパッケージをimportしています。
import glob # ファイルパス取得に使う
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import h5py
DataFrameの作成
HDFファイルから測定パラメータを読み込む方法と、すでになんらかの方法で測定パラメータをリスト化したCSVファイルから読み込む方法を書きます。どちらの場合も一覧表以上の使い方をするための肝は__生データへのパスの列を作ること__です。
HDFファイルから測定パラメータを読み込む
DataFrameの作成方法は[マニュアル] (https://pandas.pydata.org/pandas-docs/stable/dsintro.html#dataframe)にいくつか書いてあります。ここでは[From dict of ndarrays / lists] (https://pandas.pydata.org/pandas-docs/stable/dsintro.html#from-dict-of-ndarrays-lists)という方法を採用しています。`datasets`はDataFrame作成時の列の名前とHDFファイルから読み込みたい測定パラメータをペアにしたdictionaryです。`header`は列の名と測定パラメータをペアにしたdictionaryで、これをDataFrameに変換します。
# glob.globは必ずしもファイル名順にならない仕様なのでsortedを使う
hdfpaths = sorted(glob.glob('hdf/*.hdf'))
# 大量の測定データがある場合はDataFrameを作成した後にHDF形式で保存し、次回以降はそれを読み込むとストレスなく作業を続けられる。
if os.path.exists('df.h5'):
df = pd.read_hdf('df.h5', 'df')
else:
# datasetsのkeyはDataFrameの列の名前、valueはその列に代入する測定パラメータのHDFファイル内のパス
# 自分のデータに合わせて変更する
datasets = {'lambda':'entry1/SANS/Dornier-VS/lambda',
'time':'entry1/SANS/detector/counting_time',
'SD':'entry1/SANS/detector/x_position',
'mon1':'entry1/SANS/monitor1/counts',
'comment':'entry1/comment',
# 'mf':'entry1/sample/magnetic_field',
'temperature':'entry1/sample/temperature',
'sample_name':'entry1/sample/name'}
header = {'path':hdfpaths} # このdictionaryのkeyに列名、valueに測定パラメータのリストを追加していく。
for hdfpath in hdfpaths:
with h5py.File(hdfpath,'r') as f:
for colname, dataset in datasets.items():
if not colname in header : # headerのkeyにcolnameがない場合はcolnameを新規作成
try: # 文字列はバイトなのでutf-8へ。 なぜかlist()ではなく[]しか使えない
header[colname] = [f[dataset][()][0].decode('utf-8')]
except AttributeError: # other data types
header[colname] = list(f[dataset][()])
else : # colnameがある場合はそのvalueであるlistにappendメソッドで値を追加
try:
header[colname].append(f[dataset][()][0].decode('utf-8'))
except AttributeError:
header[colname].append(f[dataset][()][0])
df = pd.DataFrame(header)
# 測定データが多い場合はDataFrameをHDFファイルとして保存しておくとよい。
# df.to_hdf('df.h5', 'df')
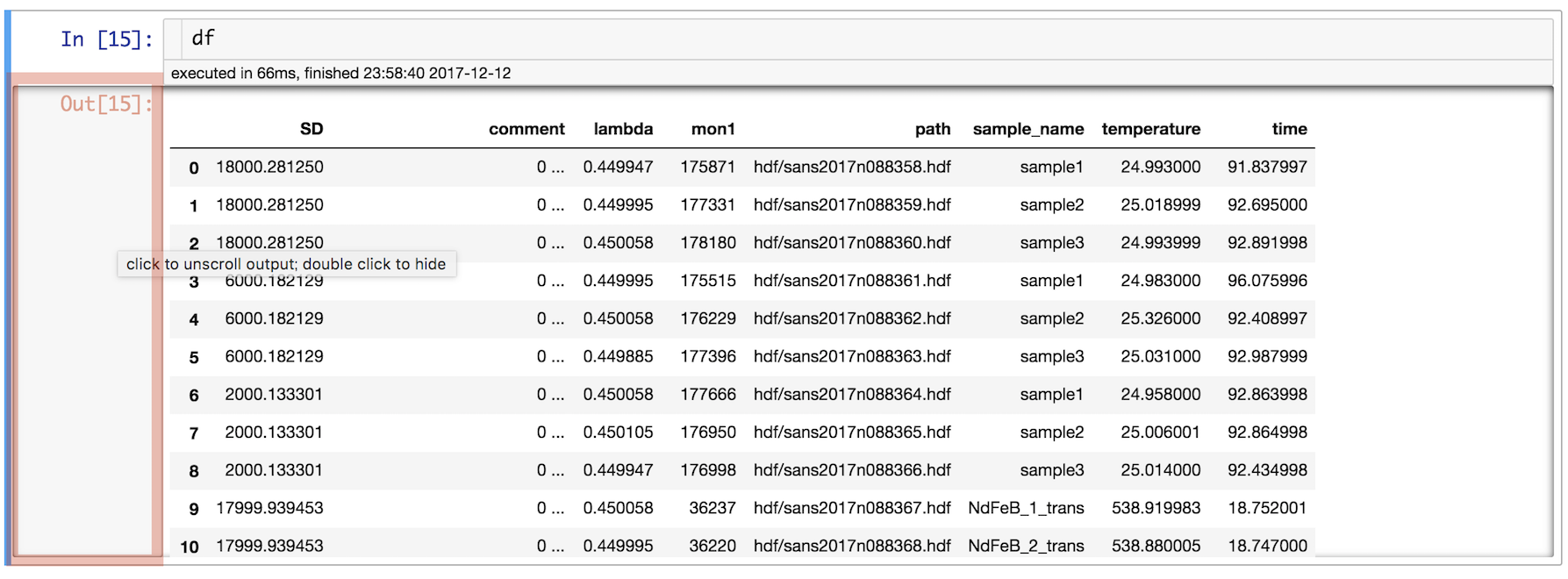
上のコードを実行すると、hdfフォルダにある全HDFファイルから読み取った測定パラメータを下のようなDataFrameにできます。12行分しかスクリーンショットを取っていませんが、数十行あります。

大量のHDFファイルから欲しい情報を抽出してDataFrameにまとめる部分に関しては個別の記事を書きました。詳細は以下の記事を見てください。
Pythonを使いHDFファイルの階層構造を把握してデータを読み込む - Qiita
他の方法で用意した測定パラメータリストをDataFrameとして読み込む
例えば、以下のような測定パラメータのCSVファイルをなんらかの方法で出力できたとします。
SD,comment,lambda,mon1,path,sample_name,temperature,time
18000.281250,0 ...,0.449947,175871,hdf/sans2017n088358.hdf,sample1,24.993000,91.837997
18000.281250,0 ...,0.449995,177331,hdf/sans2017n088359.hdf,sample2,25.018999,92.695000
18000.281250,0 ...,0.450058,178180,hdf/sans2017n088360.hdf,sample3,24.993999,92.891998
...
...
pd.read_csvを使えばオプションなしで先のDataFrameと全く同じものができます。
df = pd.read_csv('test.csv')
delimiter, usecols, dtypeなどのオプションが使えます。 詳しくは[マニュアル] (https://pandas.pydata.org/pandas-docs/stable/generated/pandas.read_csv.html)を参照してください。
HDFファイルにない項目をDataFrameの列に追加する
磁場の代わりに記録されていた電磁石の電流値を磁場に変換したり、二次元検出器全体の合計カウント数を計算したり、測定パラメータとしては記録されていない数字を新しい列として追加したいことはよくあります。マニュアルの[Column selection, addition, deletion] (https://pandas.pydata.org/pandas-docs/stable/dsintro.html#column-selection-addition-deletion)にある通り、dictionaryと同様の方法で既存の列に代入するかのようにしれっと列を追加することができます。
DataFrameにある数字だけから新しい列の値を計算できる場合
これはエクセルでも簡単にできますね。
# 例:HDFファイルに記録されていた電流値を磁場に変換
df['mag'] = df['current']*magcurcoef # dfにmag列が新しく追加される
生データから計算した何らかの値を追加する場合
測定データそのものから求めた値を測定条件と一緒にリストにできると、異常の発見や解析に役立ちます。forループでpandas.DataFrame.iterrowsを使って各行にアクセスしてHDFファイルを読み込んでいるのが肝です。これは少なくともエクセル単体ではできないと思います。HDFファイルの読み込みには[matplotlibを使って二次元検出器の二次元強度マップを作る] (https://qiita.com/skotaro/items/d1c02681fef4783838e3)にもでてきた関数を再利用します。
# 例:二次元検出器全体の合計カウント数を求める
def ReadDataFromHDF(hdfpath): # 以前の記事で定義した関数を再利用
with h5py.File(hdfpath,'r') as f:
data = f['entry1/SANS/detector/counts'][()].astype('float')
return data
total = []
# iterrows()はDataFrameの各行をイテレートする際に使う。iはここでは使わないインデックス番号。
for i, row in df.iterrows():
data = ReadDataFromHDF(row.path) # row.path: rowのpath列の値を返す row['path']と同じ
total.append(data.sum()) # sum(): numpy arrayの全要素の和を返す
df['total intensity'] = total
一覧表から一歩進んだDataFrameの活用方法
すでにお気づきかもしれませんが、このエントリでいう「一歩」は、要は___測定リストからある測定条件に該当する測定データを抽出してからグラフ作成や解析処理などアレコレする___ことです。DataFrameを使うとエクセルよりも多様なアレコレがはるかに簡単にできます。
まずは一覧表として
Jupyter notebookではDataFrameオブジェクトの名前(ここまでの例ではdf)のみをセルで実行すれば、DataFrameの内容が表示されます。pandasの表示オプションの設定によりますが、おそらくたいていの場合は途中の行も列も省略されているでしょう。最大表示数は以下で確認できます。
pd.get_option('max_rows')
pd.get_option('max_columns')
この二つのパラメータをset_optionでNoneに設定すれば全ての行と列を表示します。
pd.set_option('max_rows', None) # 行が多いDataFrameだと表示に時間がかかるようになるかもしれない
pd.set_option('max_columns', None)
以下の図で赤い線で囲った領域をクリックかダブルクリックすると出力セル内部でのスクロールが可能になったり、出力セルごと隠すことができます。
ある条件に該当する測定を探す
表計算ソフト一般で言うところのフィルタです。当然エクセルでもできます。pandasのマニュアルではindexingやslicing、selectingなどと呼ばれています。pandasマニュアルの[Indexing and Selecting Data] (https://pandas.pydata.org/pandas-docs/stable/indexing.html)に詳しく書いてありますが、ここでは表計算ソフトのフィルタのように使える[Boolean indexing] (https://pandas.pydata.org/pandas-docs/stable/indexing.html#boolean-indexing)を使います。
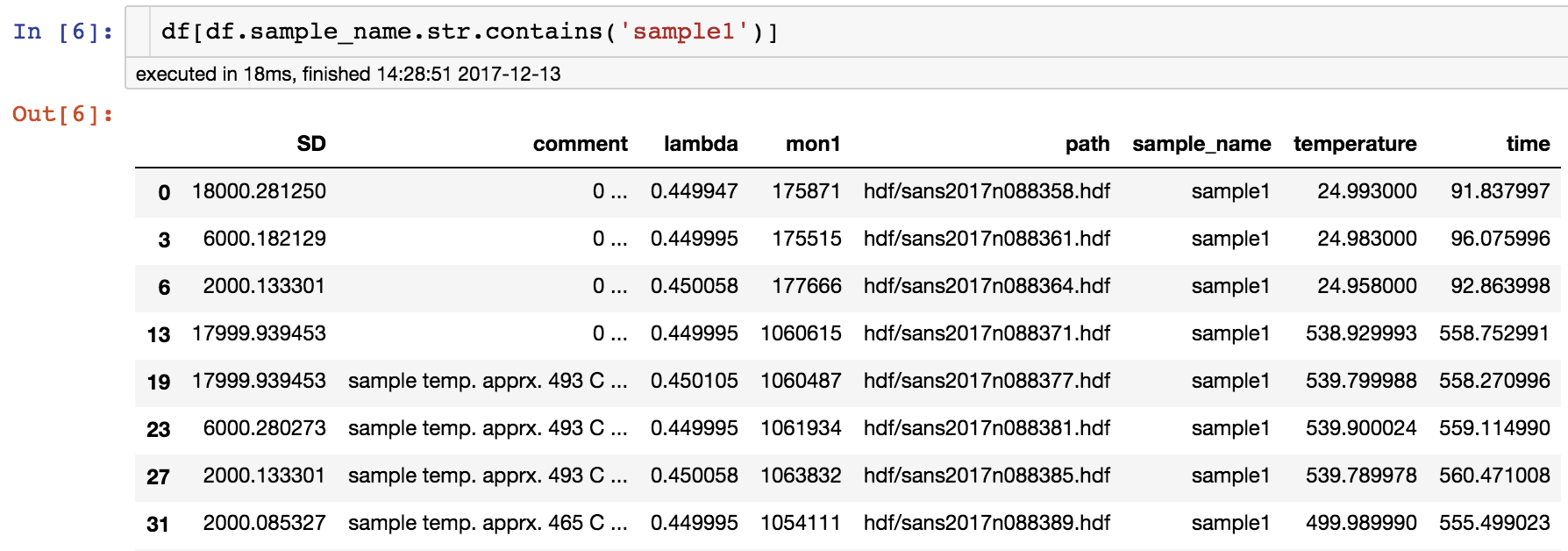
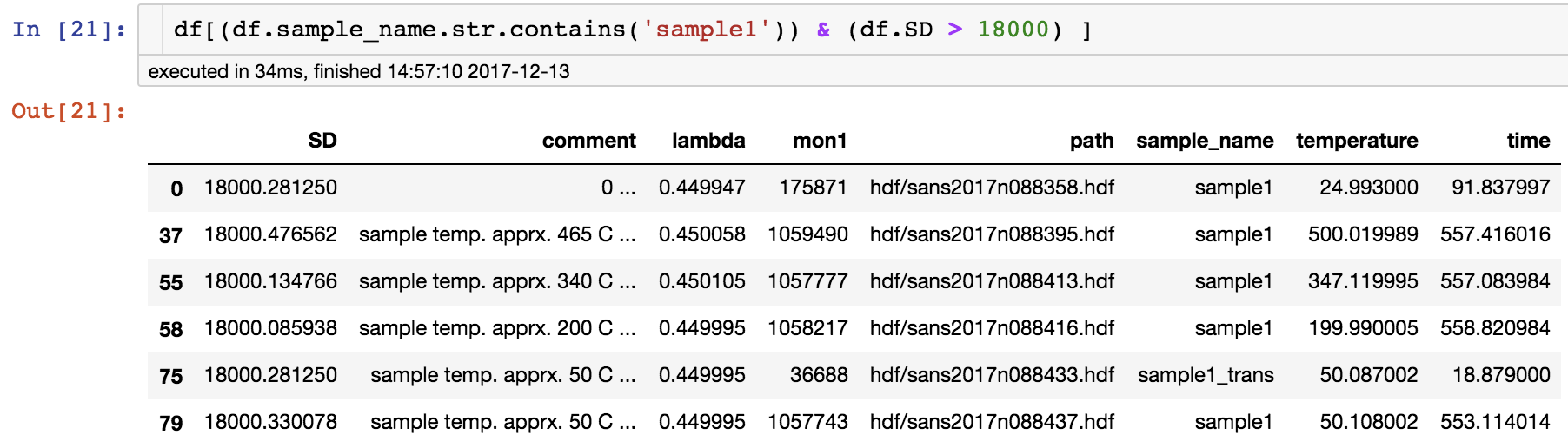
# sample_nameにsample1を含む行を抽出
df[df.sample_name.str.contains('sample1')] # 完全一致にしたい場合はdf.sample_name == 'sample1'
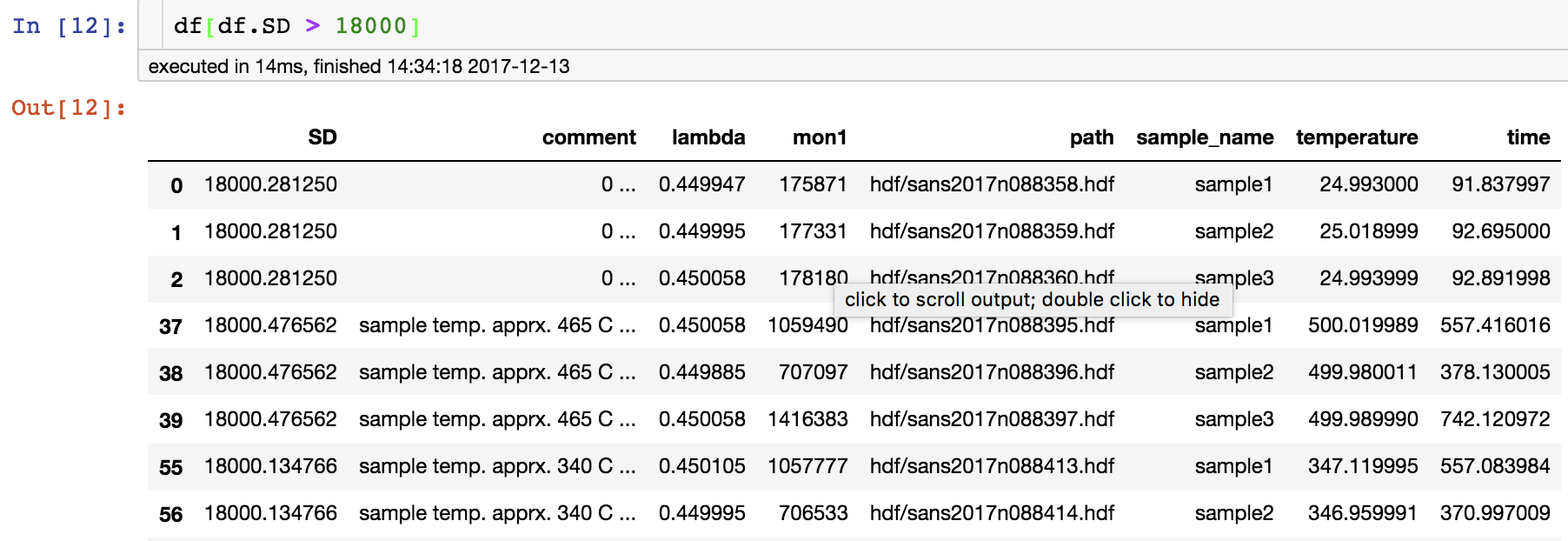
# SD(検出器距離)が18000より大きい行を抽出
df[df.SD > 18000]
# 上記二つの条件に合致する行を抽出
df[(df.sample_name.str.contains('sample1')) & (df.SD > 18000)] # (条件1)&(条件2)のように丸括弧で囲う
Boolean indexingの名前の通り、df[フィルタ条件]のフィルタ条件部分はboolean vectorになっています。フィルタ条件部分のみをセルで実行するとこのboolean vectorの中身を見ることができます。例えばdf.sample_name.str.contains('sample1')は以下のようなboolean vectorを返します。
0 True
1 False
2 False
3 True
4 False
5 False
6 True
7 False
8 False
9 True
...
...
よく組み合わせる条件があればlambda式(1行で簡易的な関数を書けるPythonの機能)を定義するのが便利です。例を紹介する前に、SD列の値に無視できる揺らぎがあるので整数値に丸めましょう。
df.SD = np.around(df.SD, decimals=-1).astype(int)
これで先に例示したdf.SD > 18000ではなくdf.SD == 18000という等号を使った表現で絞込みができます。以下は上で示した二つの条件を組み合わせたフィルタの例と同じ結果を返します。
# 試料名と検出器距離で検索できるconditions(name, SD)という関数を定義
conditions = lambda name, SD : (df.sample_name.str.contains(name)) & (df.SD == SD)
df[conditions('sample1', 18000)]
エクセルにもフィルタ機能はありますが、DataFrameだと絞り込んだ後のDataFrameに対して簡単にアレコレできます。以下では試料名で絞り込みをした後のDataFrameの行数をprintしています。
names = ['sample1', 'sample2', 'sample3']
for name in names:
# 抽出した行数をprint
print('{} rows for {}'.format(len(df[conditions(name, 100)]), name))

このように測定データを分類してアレコレするのにさらに便利なグループ化機能は後述します。
測定パラメータの変化をプロットする
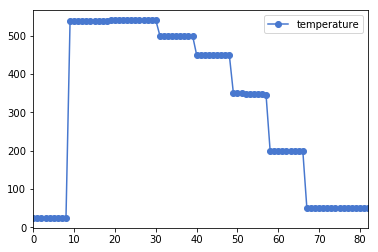
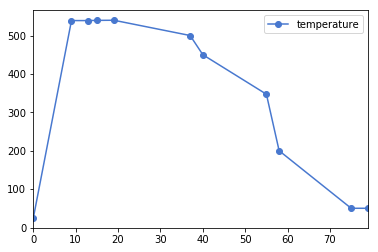
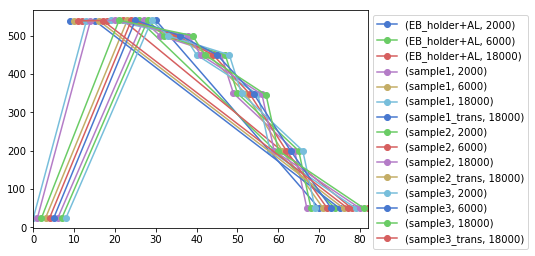
測定温度を測定順にプロットして温度履歴を確認したり、少しずつ変えてるはずのモーターが予期せぬ値になっていないかなどの確認に使えます。ある列の数字をプロットするだけなら当然エクセルでもできるので、この一歩は大したことないかもしれません。ただ、プロット対象が増えてくると一括アレコレできるDataFrameの優位性が一気に上がり大きな一歩になります。
df.plot(y='temperature', marker='o') # 横軸xはデフォルトではインデックス
df[conditions('sample1', 18000)].plot(y='temperature', marker='o') # 横軸xのインデックスは絞り込む前のインデックス
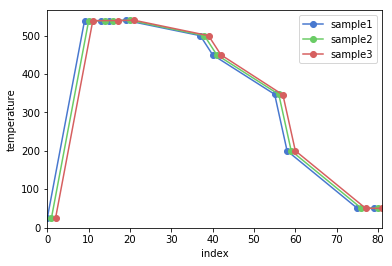
# 複数の試料について絞り込み、それぞれの絞り込み結果の温度変化を同じAxesオブジェクトにプロット
ax = plt.gca()
names = ['sample1', 'sample2', 'sample3']
for name in names:
df[conditions(name, 18000)].plot(y='temperature', marker='o', ax=ax, label=name)
# ax=axがないとnameの数だけグラフが作成されてしまいます。
ax.legend()
ax.set(xlabel='index', ylabel='temperature')
ある条件に該当する測定に対して生データを取得してアレコレする
生データを取得できると、一括可視化と解析処理の組み合わせによってアレコレの可能性が一気に広がります。これはエクセル単体ではやりづらい部分だと思います。以下の例では、ここで公開しても問題のないバックグラウンド測定データを使ってひとまず二次元強度マップを作ってみます。
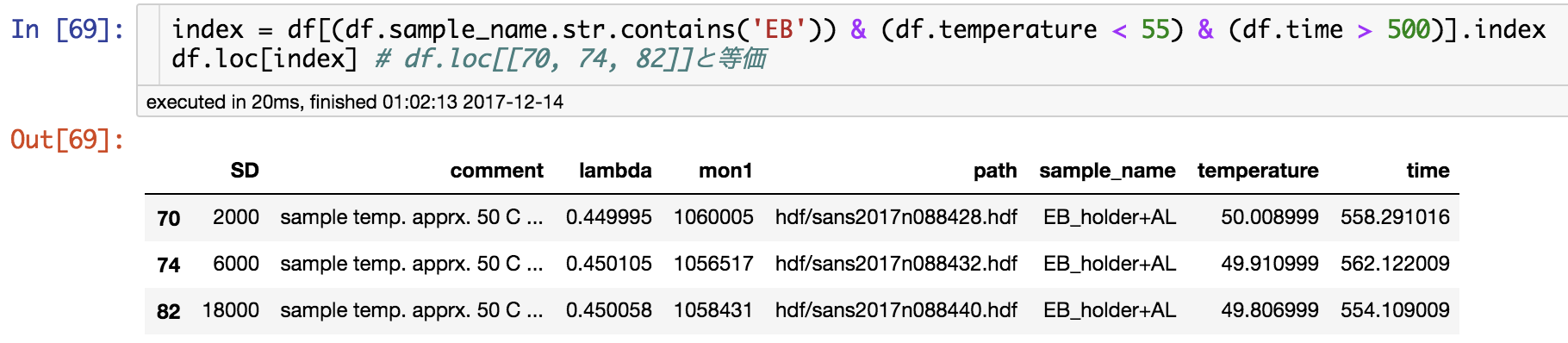
# バックグラウンドデータの抽出
df[(df.sample_name.str.contains('EB')) & (df.temperature < 55) & (df.time > 500)]

DataFrame.locを使ってindexを直接指定しても抽出できます。
# indexを使った抽出
index = df[(df.sample_name.str.contains('EB')) & (df.temperature < 55) & (df.time > 500)].index
df.loc[index] # df.loc[[70, 74, 82]]と等価
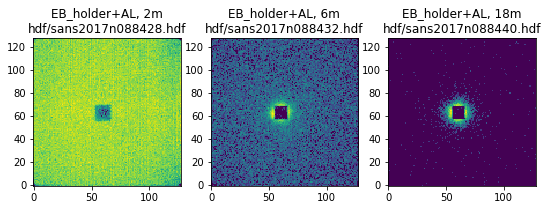
三つのバックグラウンド測定データをDataFrame.iterrowsを使ってforループでイテレートし、二次元強度マップを作ります。path, SD, sample_nameを使っています。
fig, axes = plt.subplots(1, 3, figsize=(9, 3))
for ax, (i, row) in zip(axes, df.loc[[70, 74, 82]].iterrows()):
data = SafeLog10(ReadDataFromHDF(row.path), nansub=0)
ax.set_title('{}, {:.0f}m\n{}'.format(row.sample_name, row.SD*0.001, row.path))
ax.imshow(data, origin='lower')
# このほかの好きな処理もできる
二次元強度マップの代わりにフィッティングや統計処理をして最後に各測定に対するいろいろな数値を一つまたは複数の図にプロットすることももちろん可能です。例えば、温度依存性のあるピークが観測された時にガウス関数でフィッティングした時のピーク位置等のパラメータの温度依存性を一気にプロットできます。
測定条件に基づいてグループ分けをしてアレコレする
DataFrame.groupbyを使うと測定条件によるグループ分けができます。「大量の実験データの測定条件管理と解析」という目的に対するDataFrameの1番の御利益はこれです。例えば、試料ごとに分けてソートし直して表示するだけならエクセルでもできますが、これもやはり図の一括作成などアレコレの部分でDataFrameの有用性が際立ちます。私の周辺分野でありがたみが想像できそうな例としては以下のようなものが挙げられます。
- 温度依存性測定結果を温度を凡例のラベルとしてプロットして測定試料の個数分の図を作る
- 複数試料の温度依存性データをフィッティングして、パラメータの温度依存性を試料間で比較できる図を作る
グループ分けの基準にしたいcolumn名をリストにしてgroupbyに渡すとgroupbyオブジェクトが作成され、それをforループでイテレートしてグループ化されたDataFrameにアクセスするという手順になります。
# グループ分けの基準にした値(key)とグループ分けされたDataFrame(group)の行数をprint
grouped = df.groupby(['sample_name', 'SD'])
for key, group in grouped:
# グループ分けの基準が二つあるのでkeyは要素2個のtuple
print('{}:{} rows'.format(key, len(group)))

# DataFrameの各行にはアクセスせずにtemperature列の数字を各keyをラベルにしてプロット
# 特に意味のないグラフ
ax = plt.gca()
for key, group in grouped:
group.plot(y='temperature', marker='o', ax=ax, label=key)
ax.legend(bbox_to_anchor=(1, 1), loc='upper left')
# グループ化されたDataFrame(group)の各行にアクセスして、
# ある条件に合うグループの生データへのパスをprint
for (sample, SD), group in grouped: # タプルの要素2個を別々に代入
if sample in ('sample1', 'sample2', 'sample3') and SD == 6000:
print(sample+', '+'SD='+str(SD))
for i, row in group.iterrows():
# ここでアレコレできる
print(row.path)

紙への印刷・PDF化はやっぱりエクセル
上述した通り、測定リストからうまいこと必要な部分を切り出して生データに対してアレコレできるのがDataFrameを使った測定条件管理の強みであり、それを私は「一覧表の一歩先」と表現してるのですが、一覧表のけっこう大事な機能である___紙への出力あるいはPDF化にはDataFrameは全く向いていません___2。こればかりは、プリントアウトできる行数ならばDataFrame丸ごと、行が多すぎるなら細切れにしてCSVファイル等に一旦出力してからエクセル等表計算ソフトを使って体裁を整えて出力するしかないと思います。
-
ただ、私はほとんどエクセルを使わないので、実はうまいことやるとエクセルでもできることをできないと思い込んでいる可能性は高いです。 ↩
-
[このstackoverflowの回答] (https://stackoverflow.com/questions/33155776/best-way-to-export-pandas-df-into-a-pdf-file-using-python)によるとできないこともないようですが、明らかに面倒なので私は試していません。 ↩