はじめに
本記事は, こちらの論文の解説です.
原題: Improving Chemical Autoencoder Latent Space and Molecular De-novo Generation Diversity with Heteroencoders
著者: E. J. Bjerrum and B. Sattarov
論文誌: Biomolecules
発表日: 2018年6月15日(第1版)
arXiv: https://arxiv.org/abs/1806.09300
ケモインフォでなるべく新しい論文を選びました.
簡潔さ・伝わりやすさを重視して一部省略・加筆していますので, ご興味を持たれた方は論文を参照してください.
概要
分子の性質を表す特徴ベクトルとして様々なfingerprintがあり, 近年では深層学習を応用したfingerprint設計に注目が集まっています.
本研究では, 分子の文字表現であるSMILESをautoencoderで学習させる手法を発展させて, 同じ分子を表す複数のSMILES間の変換を行うChemical Heteroencoderを提案しました.
これにより得られたfingerprintは, 5通りの活性予測や潜在空間上でのPCAにより, 分子の物理化学的特性と生体活性の両方を捉えており, かつSMILESの記号的特徴から独立していることを示しました.
このfingerprintは様々な活性予測で汎用に使うことができ, また, 新薬の生成モデルにも応用できることが期待されます.
背景
ケモインフォマティクスに馴染みの無い方が多いと思うので, がんばって解説します!
分子の表現方法
分子は, 構造式や**SMILES (simplified molecular-input line-entry system)**などによって表現されます.
構造式は説明不要と思いますが, このような図のことです(安息香酸の例).
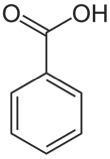
SMILESはこれを文字で書き下したもので, 安息香酸なら「c1ccccc1C(=O)O」と表せます.
構造式は人の目にわかりやすく, SMILESは解釈性を損なわない範囲で計算機上でも扱いやすい形式にしたものと言えます.
しかし, 機械学習などへの応用を考えた場合は分子を固定長のベクトルで表現できることが望ましく, fingerprintと呼ばれる分子の特徴ベクトルが多数考案されてきました.
Fingerprint
ここでは, fingerprintを3種類に分類して紹介します.
人の手で設計したもの
部分構造に注目して1024次元程度の二値ベクトルに直すアプローチで, 比較的歴史が長いです.
わかりやすいものとしてMACCS Keysが, よく使われるものとして**ECFP (extended circular fingerprint)**などがあります.
離散かつ疎なので精度が出しにくいのが問題です.
(イメージ図. Molecular fingerprint similarity search in virtual screeningより)
グラフ畳込みを利用したもの
分子をグラフとして扱うアプローチで, 深層学習とともに使われることが多いです. ECFPの一部の処理をニューラルネットで置き換えた**NFP (neural fingerprint)**や, **GGNN (gated graph neural network)**などがあります.
連続値をとります.
autoencoder系
SMILESをautoencoderで学習させることが多いです.
機械翻訳のアルゴリズムを応用したseq2seq fingerprintなどがあります.
連続で密な特徴ベクトルになります.
本研究では, こちらのアプローチで性能改善を目指します.
問題点
RNNベースのautoencoderで得られたfingerprint(潜在表現)をPCA(primal component analysis; 主成分分析)により2次元でプロットすると, このようになります.
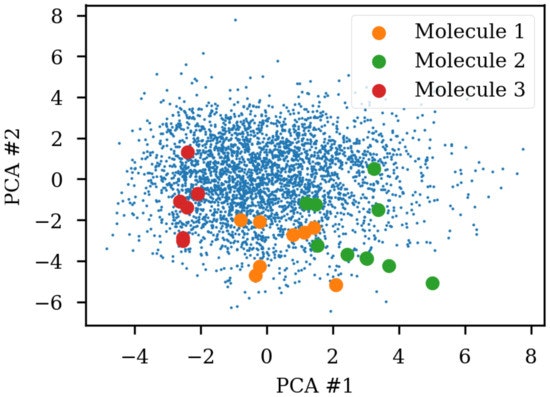
これを見ると, 同じ分子を表す複数のSMILESが別のところにプロットされており, よい表現にはなっていない(SMILESの記号的特徴に依存している)ことがわかります.
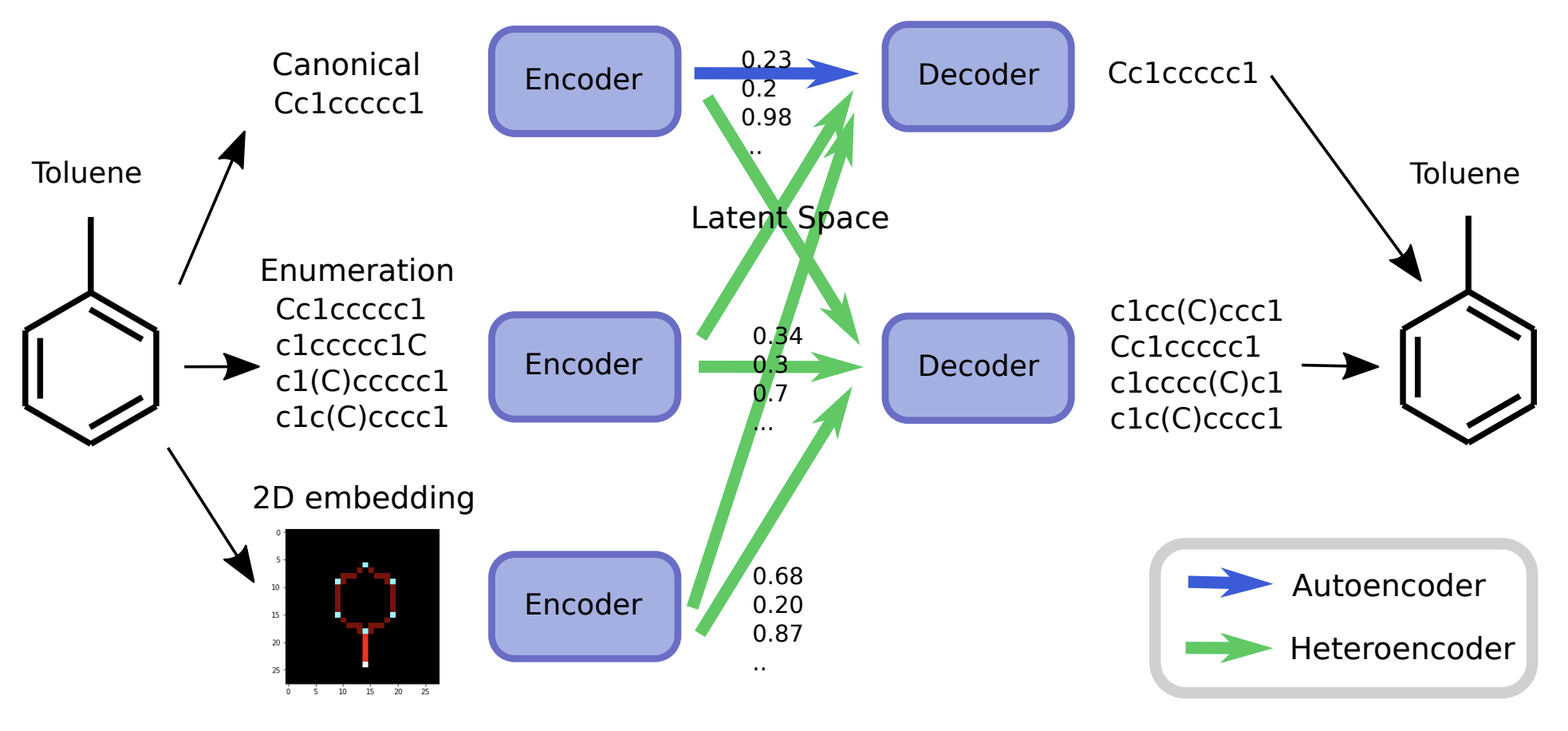
そこで, 本研究では, 分子の様々な表現に対してencode/decodeを行う「heteroencoder」を提案し, SMILESのみから良いfingerprintを獲得できることを示します.
そして, そのfingerprintを活性予測の性能の観点から比較します.
手法
こちらも論文より丁寧に説明します.
SMILESについて
SMILESは構造式を文字列で書き下したもので,
- 水素原子は省略する
- 環構造は切り開いて両端に同じ番号を振る
- 芳香環は小文字を使う
- 二重結合は「=」, 三重結合は「#」で表す
- 分岐構造は()でくくる
などのルールに従います.
SMILESは1つの分子に対して複数の表現を取り得ることに注意してください.
例えば, 先ほど「c1ccccc1C(=O)O」とした安息香酸ですが, ヒドロキシ基から辿れば「OC(=O)c1ccccc1」のようにも書けます.
本研究では, 分子と1対1対応になるようにした**正準化SMILES(Canonical)と, 1つの分子に対して複数の表現を取りうる列挙SMILES(Enumerated)**を用います.
列挙SMILESは, 正準化SMILESをランダムに変換することで得られます.
Autoencoderについて
autoencoderは教師なし学習の一種で, 入力の特徴量抽出に使うことができます.

入力$x$を一度低次元の$y$にエンコードし, それから$y$を高次元の$x'$にデコードします. そして, $x'$が$x$になるべく近くなるように学習していきます.
学習が終わると, 入力とほぼ同じものを出力できるようになります. これがautoencoder(自己符号化器)と呼ばれる所以です.
入力と同じものを出力して何が嬉しいのかというと, 一度低次元の潜在ベクトル$y$に$x$を圧縮して$x$を復元できたということは, $y$は$x$の情報を十分に保持していると考えられるからです.
情報をなるべく落とさずに密なベクトルが得られたので, これを特徴量として使うことができます.
seq2seqについて
autoencoderのエンコーダとデコーダのネットワークをLSTMなどで作ると, 自然言語のような系列入力を与えることができます. これを**seq2seq (sequence-to-sequence)**と呼びます.

seq2seqは日本語-英語のような対訳のデータが大量にあれば(つまり教師あり学習), 入力$x$を日本語, 出力$x'$を対応する英語として, 機械翻訳を行うことができます.
このとき, 学習済みのseq2seqの潜在ベクトル(Hidden)には, 言語という記号表現に依存しない「意味」が詰め込まれていると考えられます.
この研究のモチベーションを一言で言うと, 「これをSMILESでやって『意味』を取り出そう」ということになります.
*seq2seqは機械翻訳だけでなく, 対話AIなどにも使われます.
Metricsについて
あとで出てくる評価基準です.
Fingerprint類似度
出力されたSMILESがどれくらい答えに近いかを, ECFPの観点から評価します.
2つのSMILESについて, ECFPのユークリッド距離を計算します.
Alignmentスコア
出力されたSMILESがどれくらい答えに近いかをS, MILESレベルで評価します.
適当に「-」を挿入した後で, 文字が一致していたら+1, 異なっていたら-1, 「-」なら-0.5, 「-」の延長なら-0.05とします.

全体像
入力として正準化SMILES, 列挙SMILES, 画像(構造式)の3種類, 出力として正準化SMILES, 列挙SMILESの2種類を考えます.
この6通りの変換を, 大量のSMILESデータでそれぞれ学習させた後, 得られたエンコーダをfingerprint抽出器として利用します.
そのfingerprintで分子の様々な特徴を予測することで, fingerprintの性能を評価します.
実験1
まず, 入出力の形式を6通りから絞るための予備実験をしました.
(水素原子を除いて)8個以下の原子からなる分子を集めたGDB-8というデータセットからSMILESを収集し, 正準化SMILES, 列挙SMILES, 画像の3通りの入力をそれぞれLSTM, LSTM, CNNでエンコードしました.
出力は正準化SMILESと列挙SMILESの2通りで, ともにLSTMでデコードしました.
これらのheteroencoderを, 損失関数をSMILES間の交差エントロピーとして学習させました.
潜在ベクトルは64次元としました(既存のfingeprintは1024次元や2048次元が一般的です).
学習結果はこのようになりました.
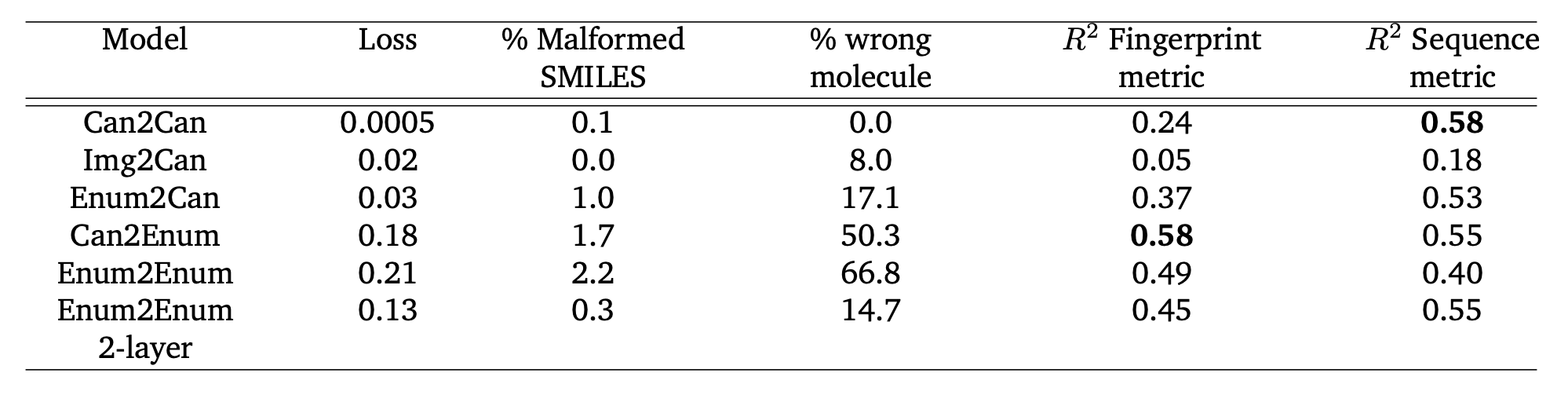
R2 Fingerprint Metricという列は, SMILES間の潜在空間でのユークリッド距離の対数に$-1$をかけたものを説明変数としたときの, Fingerprint類似度の決定係数です.
R2 Sequence Metricという列は, 同じ説明変数によるAlignmentスコアの決定係数です.
Can2EnumやEnum2Enumは答えが不定なので, Wrong moleculeの割合が高くなるのは当然ですが, LSTMを2層にしたら改善できました.
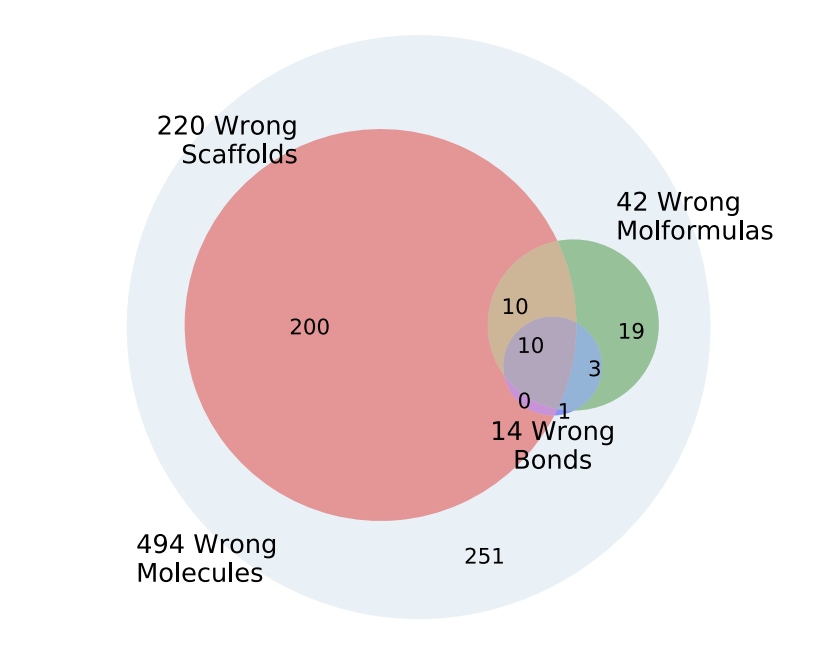
Can2Enumの誤答例を分類するとこのようになりました.
なお, Img2Canはmetricsの値が低く, 使えなさそうなので次の実験では外しました.
また, 学習後の潜在空間をPCAで次元削減してプロットすると次のようになりました.
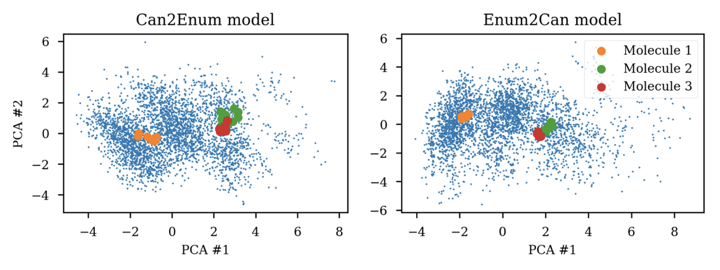
同じ分子に対するSMILESが潜在空間上で近くに集まっていることがわかります.
プロットはありませんでしたが, Enum2EnumでもEnum2Canと同様の良い空間が得られたそうです.
実験2
実験1の結果を踏まえて, 4通りのheteroencoder(Can2Can, Can2Enum, Enum2Can, Enum2Enum)を, ChEMBLというより大きなデータセットから得られたSMILESで事前学習させました.
実験1では原子数が(水素原子を除いて)8個以下という制約がありましたが, 今度はSMILESが100文字以下という制約で40万種類の分子を学習に使いました.
潜在ベクトルの次元数は256に増やしました.
次に, それぞれのfingerprint及び比較対象のECFPを使って5つのデータセットで活性予測を行いました.
| 名前 | 内容 | 件数 |
|---|---|---|
| IGC50 | テトラヒメナの成長阻害量 | 1434 |
| LD50 | ラットに対する致死量 | 5931 |
| BCF | 生物濃縮係数 | 541 |
| Solubility | 水溶性 | 1297 |
| MP | 融点 | 7509 |
データセットは物理的な特性やタンパク質に対する活性などに偏らないように選びました.
モデルはニューラルネットを使い, ハイパーパラメータはすべてのfingerprintに対して統一しました.
図に, 予測結果のR2とRMSEを示します.
Can2CanでもほとんどのセットでECFPを上回りましたが, 他のheteroencoder, 特にEnum2Enumではさらなる性能改善を確認できました.
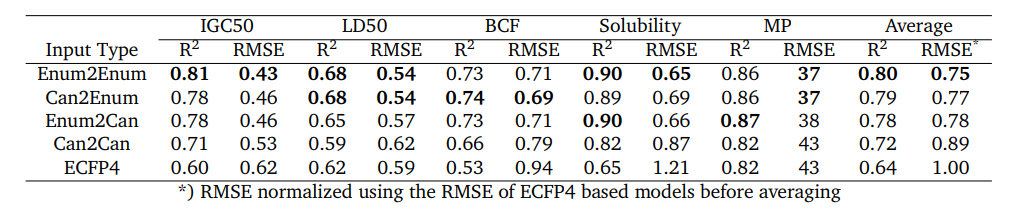
一方で, デコーダに列挙SMILESを使うと異なるSMILESを出力することが多く($69.9%$), 損失関数の値が下がりにくくなりました.
まとめ
本研究では, autoencoderの入出力に同じ分子を表す複数のSMILESを用いるheteroencoderを提案しました.
heteroencoderはSMILESのセットさえあれば学習させることができ, 大量のラベルデータを必要としません.
そして, 学習後に得られる潜在表現は分子のfingerprintとして利用できることを, 潜在空間上でのPCAや5通りの活性予測により実証しました.
本研究で得られた潜在空間は分子の物理化学的特性と生体活性の両方を捉えており, かつSMILESの記号的特徴から独立しています.
この空間上で探索して得られるfingerprintをデコードすることで, heteroencoderを新規薬剤の発見にも応用できると考えられます.
ひとこと感想
NLPのケモインフォへの応用が熱いですね.
ただし, ベースラインとしてECFPのみを用いるのはこのご時世では良くないと思います.
タンパク質への結合なども扱えるようにしたらより創薬っぽさが出てくるのですが, まだ難しいのでしょうか...