はじめに
Claude Code や OpenAI Codex CLI をはじめとした AI コーディングエージェントが広く使われるようになってきました。
これらは基本的にクラウド上のモデルを利用しますが、 週の利用上限に到達してしまった、機密保持やコストの観点からローカル完結させたい、といった場面もあるかと思います。
そこで今回は、ローカル AI コーディングエージェントを opencode + Ollama + gpt-oss の組み合わせで構築し、試してみました。
検証環境
- GPU: NVIDIA RTX 6000 Ada Generation * 4
- VRAM容量 50GB * 4
- Ubuntu: 22.04
- opencode: 1.0.134
- Ollama: 0.13.0
- local model: gpt-oss:20b
- uv: 0.5.31
ツール概要
opencode
opencode はターミナル内で動作するオープンソースのAIコーディングエージェントです。
機能的には Claude Code とよく似ていますが、特定のモデルプロバイダーに依存せず多くのモデルと接続可能です。
TUI(ターミナルユーザーインターフェース) にも注力しているとのことで、軽量で見やすく、操作体験が良好でした。
Ollama
Ollama はローカル環境で LLM(大規模言語モデル)を手軽に実行できるオープンソースのランタイムです。
opencode も Ollama に対応しているため、モデルと接続するインターフェースとして利用します。
コマンド数回だけでローカル LLM 実行環境が整うのが便利でした。
gpt-oss
gpt-oss は OpenAI が公開しているLLMです。
AI コーディングエージェントは AI (LLM) がシェルコマンドなどのツールを使うことで実現されるため、
ツール使用(tool calling, function calling)に対応したモデルを使う必要があります。
gpt-oss は公式にこれらに対応しているとのことので、今回は比較的手軽な 20B モデルで試してみました。
検証は十分なVRAMを備えたPCで行いましたが、 gpt-oss-20b なら 16GB 、 gpt-oss-120b なら 80GB あれば実行可能です。
設定手順
お試し用のサンプルプロジェクトディレクトリを用意し、以下の手順を実行します。
パス中には日本語を含めないようにしてください。
$ mkdir </path/to/project>/sample_project
$ cd </path/to/project>/sample_project
1. Ollama + gpt-oss のインストール
以下のコマンドで Ollama + gpt-oss:20b をインストールします。
$ curl -fsSL https://ollama.com/install.sh | sh
$ ollama pull gpt-oss:20b
2. Ollama + gpt-oss のコンテキストサイズ設定
Ollama のドキュメント にあるように、 Ollama のデフォルトのコンテキストサイズは 4096 トークンです。
4096 トークンではすぐに上限に達してしまい opencode のエージェントが上手く実行できない問題があったため、コンテキストサイズを拡張します。
拡張の設定は環境変数 OLLAMA_CONTEXT_LENGTH を設定する方法と、 Ollama のモデル毎に設定する方法があります。
ここでは、後者について説明します。
まず、プロジェクトディレクトリに以下のテキストファイル Modelfile_gpt-oss-20b-128k を作成します。
FROM gpt-oss:20b
PARAMETER num_ctx 131072
これを使用し、 Ollama create コマンドでコンテキストサイズを128kに拡張したモデルを作成します。
$ ollama create gpt-oss:20b-128k -f ./Modelfile_gpt-oss-20b-128k
ollama show コマンドで num_ctx が 131072 になっていることを確認し、
$ ollama show gpt-oss:20b-128k
Model
architecture gptoss
parameters 20.9B
context length 131072
embedding length 2880
quantization MXFP4
Capabilities
completion
tools
thinking
Parameters
num_ctx 131072
temperature 1
License
Apache License
Version 2.0, January 2004
...
ollama run による実行確認の後、 ollama ps で出力される CONTEXT が 131072 になっていれば完了です。
$ ollama run gpt-oss:20b-128k
>>> こんにちは
Thinking...
The user says "こんにちは" which is Japanese greeting: "Hello". They might be greeting us. We should respond in Japanese, maybe polite. We can ask how can we help.
...done thinking.
こんにちは!何かお手伝いできることはありますか?お気軽にどうぞ。
>>> /bye
$ ollama ps
NAME ID SIZE PROCESSOR CONTEXT UNTIL
gpt-oss:20b-128k 771f083c726f 16 GB 100% GPU 131072 4 minutes from now
3. opencode のインストール
以下のコマンドで opencode をインストールします。
$ curl -fsSL https://opencode.ai/install | bash
4. opencode のローカルモデル接続設定
プロジェクトディレクトリに以下の設定ファイル opencode.json を作成します。
これにより、コンテキストサイズを拡張した gpt-oss:20b-128k を opencode で呼び出せるようになります。
{
"$schema": "https://opencode.ai/config.json",
"provider": {
"ollama": {
"npm": "@ai-sdk/openai-compatible",
"name": "Ollama (local)",
"options": {
"baseURL": "http://localhost:11434/v1",
},
"models": {
"gpt-oss:20b-128k": {
"name": "gpt-oss:20b-128k",
}
}
}
}
}
設定ファイルを保存し、opencode を起動します。(opencode コマンドが有効でない場合は再ログインを)。
$ opencode .
TUIが起動したら、
/models
を実行し、 gpt-oss:20b-128k を選択します。 以上で準備完了です。
TUIを終了する場合は
/exit
で元のターミナルに戻れます。
実行例
テーブルデータの分析を行うサンプルコードと、それを動かす python 仮想環境が構築できるか、具体的に試してみました。
まっさらな作業ディレクトリで試すため、以下のように新規のディレクトリを作成して opencode を起動しなおします。
$ mkdir </path/to/project>/sample_project
$ cd </path/to/project>/sample_project
$ opencode .
1ターン目
opencode の TUI から「irisデータセットのEDAを行う eda.ipynb を実装してください。」を入力すると、以下のような出力が得られました。
※パスなど一部テキストをマスク、省略しています。
Thinking: とタグ付けされた出力テキストを見るとユーザ入力を解釈し、実装内容を検討する様子が確認できます。
ツールを利用しながら情報収集と実装内容の検討を行い、 eda.ipynb ファイルの生成まで完了できました。
折り畳み領域:実行例1ターン目
irisデータセットのEDAを行う eda.ipynb を実装してください。
Thinking: The user asks: "irisデータセットのEDAを行う eda.ipynb を実装してください。" Japanese: "Implement eda.ipynb to perform EDA on the iris dataset."
So need to create a Jupyter notebook eda.ipynb with EDA code using iris dataset. Likely using pandas, seaborn, matplotlib. Provide code. Since it's a code file, need to write it. The environment is a git repo. The target is to create or edit eda.ipynb. We need to create new file eda.ipynb.
We should read current repo to see if eda.ipynb exists. Use read? It's presumably not. We just create it. But guidelines: use write tool but must read before writing. Since new file, no need to read.
Better to output a notebook JSON. Use minimal Jupyter notebook structure. Need code cells: import libs, load iris, basic info, summary stats, visualizations: pairplot, correlation heatmap, distribution histograms, maybe boxplots. Also maybe label encoding, etc.
Also maybe using sklearn.datasets.load_iris. Use seaborn for pairplot.
We just create notebook. Provide minimal but functional. Use plain JSON. Let's construct.
Notebook cells:
1. Title cell (markdown): "Iris Dataset EDA"
2. Imports
3. Load dataset into DataFrame
4. Show first rows
5. Describe
6. Value counts of species
7. Pairplot
8. Correlation heatmap
9. Histograms of features
10. Boxplots of features per species
11. Maybe scatter matrix with colors.
Add code accordingly.
After writing, tests? None. But we just create file. Let's create using write tool.
We need absolute path: *****/sample_project/gpt-oss-20b/eda.ipynb? Based on repo root, it's current working directory. So path: eda.ipynb.
We'll write file with proper JSON. Use triple-quoted string.
Let's draft JSON.
Use nbformat v4.
Cells:
{
"cell_type":"markdown",
"metadata":{},
"source":"# Iris Dataset EDA\nA basic exploratory data analysis for the classic Iris dataset."
}
Cell 2:
import statements.
import pandas as pd
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
sns.set(style='whitegrid')
%matplotlib inline
<省略:ノートブックのコード生成>
Thinking: We have created eda.ipynb file. Need to check if any tests or other requirements? The user just asked: "irisデータセットのEDAを行う eda.ipynb を実装してください。". We wrote a notebook with EDA code. Might need to import some packages installed? We'll assume env has pandas, matplotlib, seaborn, sklearn.
I want to ensure we didn't mis-format JSON. Write tool expects string content. We passed JSON representation of notebook. That should create file correctly.
Now, maybe we need to ensure the file is valid JSON. Use triple quotes? The write tool expects string. We gave JSON string using backslashes to escape. Should be fine.
Check for any syntax errors in notebook code. For magics, we used %matplotlib inline. In a .ipynb code cell, we should write that. Good.
Check for missing imports: We used seaborn, matplotlib, sklearn. Might need to import seaborn as sns, matplotlib.pyplot as plt. Already included.
We used df = pd.DataFrame(data=iris.data, columns=iris.feature_names) - this uses feature names with a trailing newline maybe includes spaces: "sepal length (cm)". That is fine.
We map target: df'species' = df'target'.map({i: name for i, name in enumerate(iris.target_names)}). That maps label to string.
We didn't use 'species' column for EDA. Use pairplot with hue etc uses species. Good.
One potential issue: In pairplot, we may need to import seaborn earlier. done.
One issue: 'sns.heatmap' may require that the matrix be a DataFrame. We used corr = dfiris.feature_names.corr(), which is a DataFrame. Good.
We create hist plots for each feature. Good.
Boxplots.
All fine.
Potential missing: after map, we didn't set species to string? Yes.
Check for stray indentation or extra metadata.
Also we didn't set 'metadata' inside cells. They had {} but no necessary. Might be fine.
Maybe we should include a kernel metadata to specify language. But not necessary.
To ensure this is valid .ipynb, we might need "cells" as array. Good.
Now, we need to ensure the .ipynb file exists. Already wrote.
We might need to check if any tests import this file. Likely nothing.
Now finish.
The eda.ipynb notebook has been created in the specified directory and contains a complete, runnable EDA workflow for the Iris dataset. You can open it with Jupyter or any compatible notebook viewer to explore the data visualizations and statistics.
2ターン目
eda.ipynb が作成できたので、実行環境も作ってもらいました。
「現在のディレクトリに、 eda.ipynb を動かすのに必要な python 仮想環境を uv を使って構築してください。」を入力すると、 以下の出力とともに仮想環境が構築されました。
エージェントとしての自律的な動きが分かりやすいのは以下の部分です。
最初は uv init を存在しないオプションを付けて実行してエラーが出ていますが、エラーメッセージを踏まえて修正、再度実行しています。
# Initialize uv project and install dependencies for Iris EDA
$ uv init -y && uv add pandas numpy seaborn matplotlib scikit-learn ipykernel notebook
error: unexpected argument '-y' found
tip: to pass '-y' as a value, use '-- -y'
Usage: uv init [OPTIONS] [PATH]
For more information, try '--help'.
Thinking: We cannot pass -y directly. Use uv init without -y, then uv add. Let's just run uv init (creates pyproject.toml with default). Then uv add packages. We'll run sequentially.
# Initialize uv project
$ uv init
Initialized project `gpt-oss-20b`
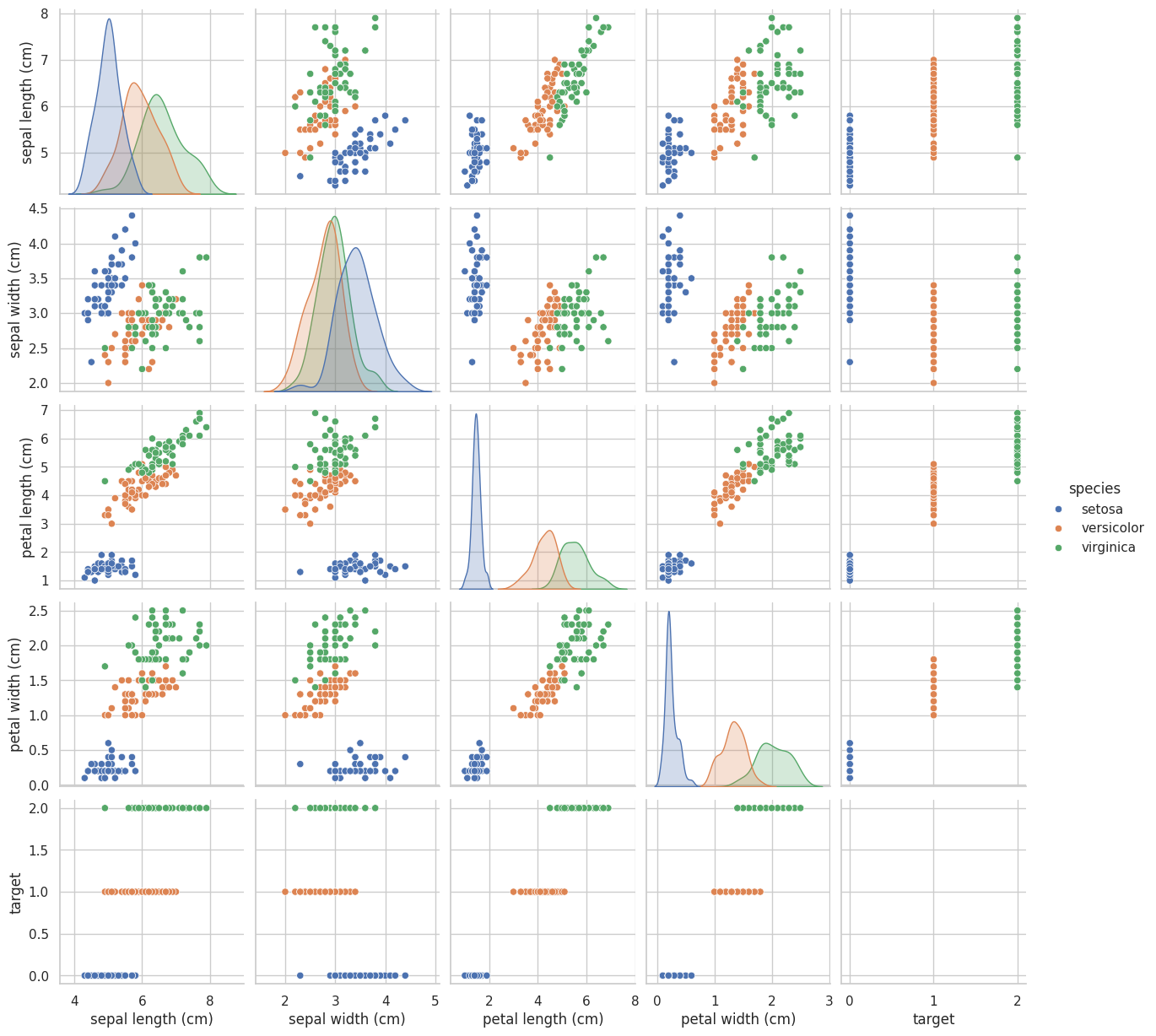
eda.ipynb を実行するとペアプロットなどの可視化結果が出力されました。
折り畳み領域:実行例2ターン目
現在のディレクトリに、 eda.ipynb を動かすのに必要な python 仮想環境を uv を使って構築してください。
Thinking: We need to create a python virtual environment using uv, presumably a project env or global? They want to setup environment to run the eda.ipynb. Use uv to create a virtual environment and install dependencies: pandas, numpy, seaborn, matplotlib, scikit-learn. Also maybe ipykernel. Provide instructions? The user asks: "現在のディレクトリに、 eda.ipynb を動かすのに必要な python 仮想環境を uv を使って構築してください。". So we should create a uv project. Steps: run uv init -y to create pyproject.toml. Add dependencies. Also maybe create a uv.lock. Also create a virtual environment in .venv. Use uv's local env? We might just use uv venv . maybe or uv sync. But must do within repository.
We must execute commands via function. Use type bash. Provide description. We'll need to check that uv is installed. It likely is.
We need to call multiple commands: maybe uv init -y, then uv add pandas numpy seaborn matplotlib scikit-learn ipykernel. That will create pyproject.toml and .venv in the repo. Then maybe instruct user to run uv sync --frozen. We'll do all as single command? We can chain: uv init -y && uv add .... Use semicolons or &&. Provide description.
Then we need to ensure the environment is recognized.
Better to run them individually? But we can combine.
We'll do:
uv init -y
uv add pandas numpy seaborn matplotlib scikit-learn ipykernel
Then to create the environment: uv venv . maybe. But uv automatically installs into local env if not set? The uv add will create .uv or similar? I believe uv add will add dependencies to pyproject and also install them into local env. If environment not exist, uv will create it.
Thus we just run commands.
We'll use bash with description "Initialize uv project & add dependencies".
Note: no newline allowed; but we can use semicolons.
We'll use uv init -y && uv add pandas numpy seaborn matplotlib scikit-learn ipykernel.
Add also jupyter maybe? Not necessary, but to run notebook, you need jupyter. But user may run notebook via Jupyter, requiring jupyter. So install jupyter. So dependencies: pandas, numpy, seaborn, matplotlib, scikit-learn, ipykernel, notebook. Add notebook.
Thus command: uv init -y && uv add pandas numpy seaborn matplotlib scikit-learn ipykernel notebook.
Let's do it.
# Initialize uv project and install dependencies for Iris EDA
$ uv init -y && uv add pandas numpy seaborn matplotlib scikit-learn ipykernel notebook
error: unexpected argument '-y' found
tip: to pass '-y' as a value, use '-- -y'
Usage: uv init [OPTIONS] [PATH]
For more information, try '--help'.
Thinking: We cannot pass -y directly. Use uv init without -y, then uv add. Let's just run uv init (creates pyproject.toml with default). Then uv add packages. We'll run sequentially.
# Initialize uv project
$ uv init
Initialized project `gpt-oss-20b`
# Install dependencies in uv project
$ uv add pandas numpy seaborn matplotlib scikit-learn ipykernel notebook
Using CPython 3.12.9
Creating virtual environment at: .venv
Resolved 111 packages in 215ms
Downloading scikit-learn (9.0MiB)
Downloading scipy (34.0MiB)
Downloaded scikit-learn
Downloaded scipy
Prepared 5 packages in 768ms
Installed 107 packages in 109ms
+ anyio==4.12.0
<省略:インストールパッケージ一覧>
The virtual environment has been created at .venv and all required packages for running eda.ipynb are installed. You can now activate the environment, e.g.:
source ./.venv/bin/activate
jupyter notebook eda.ipynb
If you prefer, the environment is already set up for running notebooks directly from the uv executable. No further action needed.
分かったこと
上記の構成でローカル環境でもAIエージェントとしての自律的な振る舞いを確認できました。
仮に途中で失敗してもある程度自動で軌道修正できるのはメリットだと思います。
ただし、繰り返し使ってみると誤りを含んだままのアウトプットが提示されるケースもありました。
例:
- 出力されたノートブックファイル の json フォーマットが崩れており VSCode で開けない
- ノートブックのコードにバグがあり実行時エラーが出る
今回は構築と定性的な動作確認までとしましたが、
今後は定量的な安定性の検証や、gpt-oss:120bとの比較、
既存プロジェクトに導入する想定での実験もやってみたいと思います。
おわりに
最後まで読んでいただき、ありがとうございました!
本記事がローカルAIエージェントのお試しの参考になりましたら幸いです。