はじめに
本稿はpandasユーザーに向けて、複数のCSVファイルからデータを読み込んで1つのDataFrameを作る高速な方法を書きました。
最初にfor文で1ファイルづつ読み込む一般的な方法を確認した後に、高速な方法を紹介しています。
環境
- Google Colaboratory (無償版CPU)
- pandas ver 1.5.3
対象CSVファイルの仕様
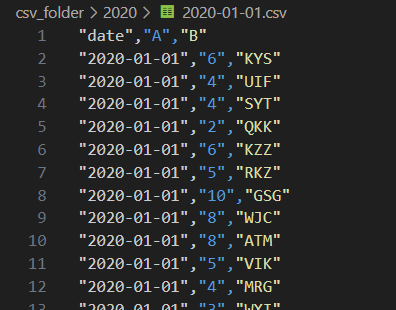
- 1つのCSVファイルに50,000レコード x 3列のデータが入ってます。
- 3列は「日付」「数値」「文字列」の型に対応したデータが入っています。
- date列: 「YYYY-MM-DD」の書式でファイル名と同じ日付の文字列が入ってます。
- A列: 0~9のランダムな数値が入っています。
- B列: A-Zのランダムな3文字の文字列が入ってます。
- 「csv_folder」フォルダの直下に「2020」「2021」のように「年」のフォルダがあり、「年」フォルダの中にCSVファイルが入っています。
- 全部で2,000ファイルあります。 2,000ファイル x 50,000レコード = 計1億レコードのデータになります。
- CSVファイルの名前は「YYYY-MM-DD.csv」の書式になっています。
csv_folder/
├─ 2020/
│ 2020-01-01.csv
│ ...
│ 2020-12-31.csv
├─ 2021/
│ 2021-01-01.csv
│ ...
│ 2021-12-31.csv
├─ 2022/
│ 2022-01-01.csv
│ ...
...
1. 一般的な複数のCSVファイルを読み込む方法
for文でCSVファイルの場所を取得して、 pd.read_csv() で1ファイルづつDataFrameに読み込んで、最後に pd.concat() で1つのDataFrameにまとめています。
import pathlib
import pandas as pd
_list = []
# 1)「csv_folder」フォルダ配下の全てのCSVファイルのパスを日付順に1個づつfor文で取得する
for file in sorted(pathlib.Path("./csv_folder").glob("**/*.csv")):
# 2) .read_csv()でCSVファイルから読み込んだDataFrameを_listへ入れる
_list.append(pd.read_csv(file))
# 3) _listへ入れた全てのDataFrameを1つのDataFrameにまとめる
df = pd.concat(_list, ignore_index=True)
df.shape
# (100000000, 3)
最終的に 1億レコード x 3列(date列, A列, B列) のDataFrameが出来上がりました。
ここが今回のゴール地点です。
2. 高速版
それでは高速な方法を紹介します。それは「pyarrow」というライブラリを使って読み込みます。
pyarrow
このpyarrowを用いると、モダンなCPUやGPUで効率的にデータ分析処理を行える「Apache Arrow フォーマット」でCSVファイルを読み込んで、pandasへDataFrameを渡すことができます。
pandasの生みの親である「Wes McKinney」が2015年に立ち上げた「Apache Arrow」プロジェクトから提供されていて、pandasと深く統合されているライブラリです。
インストール方法
pipコマンドでインストールできます。本稿執筆時点の最新版はver 14.0.2です。
pip install pyarrow
使い方
pyarrowでは、複数CSVファイルの読み込みをこのように書くことができます。
import pyarrow.dataset as ds
# 1)「csv_folder」フォルダ配下の全てのCSVファイルを読み込む
table = ds.dataset("./csv_folder", format="csv").to_table()
# 2) 読み込んだデータをpandasのDataFrameへ変換する
df_pa = table.to_pandas()
df_pa.shape
# (100000000, 3)
最上位フォルダ「csv_folder」の場所を渡すだけで、全てのサブフォルダのCSVファイルを読み込んで1つにまとめてくれます。for文でループしたりpd.concat()する必要はありません。
データを読み込んだらpandasのDataFrameへ変換して完了です。
2つの方法の速度比較
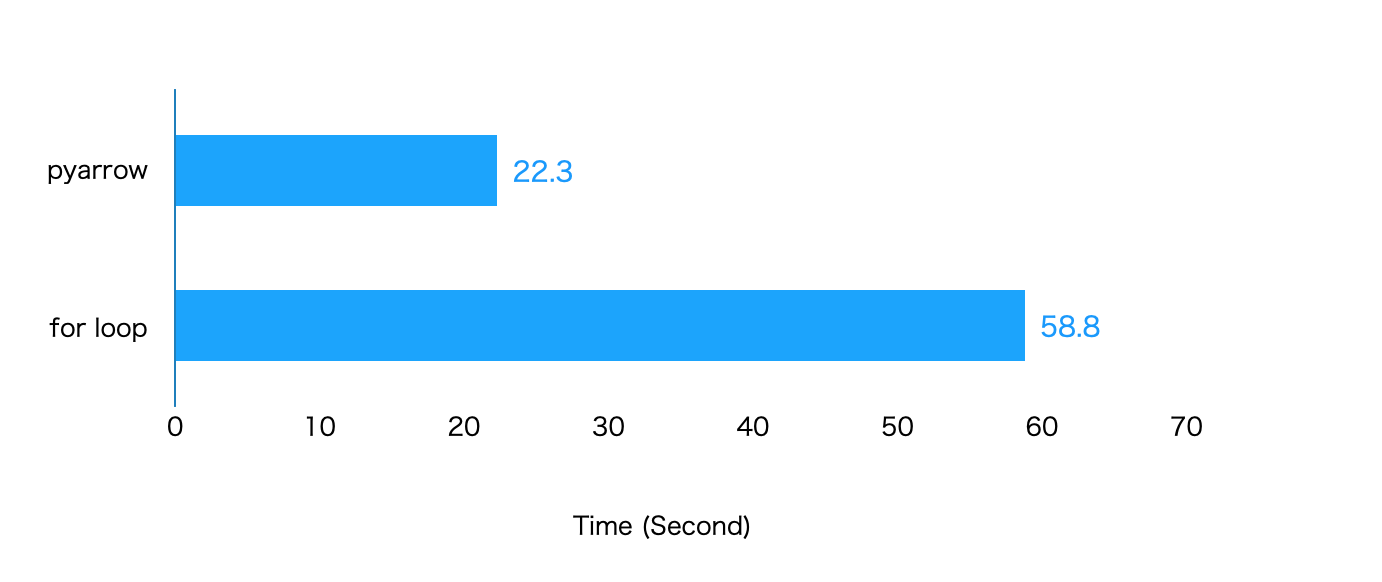
「for文を使った方法」と「pyarrowを使った方法」の速度を比較してみましょう。
timeitで同じ処理を7回繰り返した結果の平均値になります。
2.6倍ほどpyarrowの方が高速に処理していることが確認できます。
もう少しpyarrowの世界に触れてみる
それでは読み込んだ2つのDataFrameの内容が一致してるか確認してみましょう
# 2つのDataFrameの内容が一致しているか確認する
df.equals(df_pa)
# False
!?
DataFrameの内容が不一致であることを示すFalseが出力されました。一体どういうことでしょう。
実は、あえてこのような結果になるようにサンプルのCSVファイルを用意しました。この原因を今から説明しながら、もう少しpyarrowを上手に使うためのポイントを紹介しようと思います。
まずFalseになってしまったのは、2つのDataFrameのdate列の「要素の型」が異なっているためです。
# for loop で作ったDataFrameのdate列を調べてみる
df["date"].dtype.name # => 'object' # dtypeはobject
df["date"].iat[0] # => '2020-01-01'
type(df["date"].iat[0]) # => str # 要素の型はstr型
# pyarrowから受け取ったDataFrameのdate列を調べてみる
df_pa["date"].dtype.name # => 'object' # dtypeはobject
df_pa["date"].iat[0] # => datetime.date(2020, 1, 1)
type(df_pa["date"].iat[0]) # => datetime.date # 要素の型はdatetime.date型
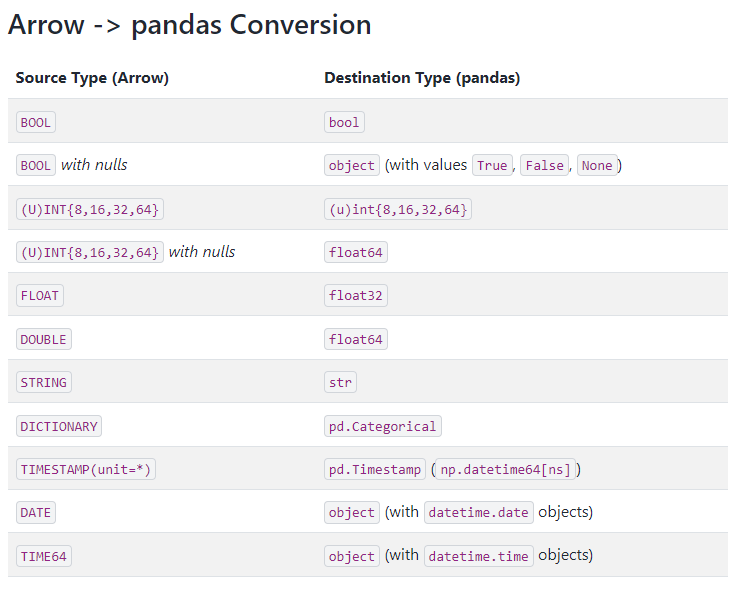
これはfor文の中でpd.read_csv()がdate列を文字列として読み込んだ事に対して、pyarrowはpa.date32型という日付を表現する型で読み込んだ事に起因しています。
pyarrowはpandasのDataFrameへデータを変換する際に、pyarrowとpandasの型の変換ルールに基づいて変換しますが、pa.date32型はdatetime.date型に変換する決まりになっています。
これにより df_pa["date"] の要素はdatetime.date型になっていました。
そのためdate列の要素をstr型へ変換してあげると
# date列の要素をstr型へ変換してからequals()へ渡す
df.equals( df_pa.assign(date=df_pa["date"].astype("str")) )
# True
きちんと一致します。
またpyarrowがCSVファイルを読み込む時に、「date列を文字列型で読み込んで!」と指定することもできます。
import pyarrow as pa
import pyarrow.dataset as ds
df_pa = (
ds
.dataset(
"./csv_folder", format="csv",
schema=pa.schema([
('date', pa.string()), # date列をpa.string型で読み込むように
# pyarrowへ指定している
('A', pa.int64()),
('B', pa.string())
])
)
.to_table()
.to_pandas() # date列はpa.string型なので変換ルールに基づいてstr型へ変換される
)
# 2つのDataFrameの内容が一致しているか確認する
df.equals(df_pa)
# True
さらには、そもそも文字列型ではなくpandasのTimestamp型(Numpyのdateteme64型)で受け取りたい場合もあるでしょう。その場合は
import pyarrow as pa
import pyarrow.dataset as ds
df_pa = (
ds
.dataset(
"./csv_folder", format="csv",
schema=pa.schema([
('date', pa.timestamp('ns')), # date列をpa.timestamp型で読み込むように
# pyarrowへ指定している
('A', pa.int64()),
('B', pa.string())
])
)
.to_table()
.to_pandas() # date列はpa.timestamp型なので変換ルールに基づいて
# pd.Timestamp型(np.datetime64[ns]型)へ変換される
)
# date列のdtypeを確認する
df_pa["date"].dtype.name
# 'datetime64[ns]'
のように書くことができます。
そして、先ほど.to_pandas()によってpa.date32型からdatetime.date型へ変換された場合でも .to_pandas(date_as_object=False)と書いてあげると
import pyarrow.dataset as ds
df_pa = (
ds
.dataset("./csv_folder", format="csv")
.to_table()
.to_pandas(date_as_object=False) # pa.date32型をdatetime.date型ではなく、
# Timestamp型(Numpyのdateteme64型)へ変換してくれる
)
# date列のdtypeを確認する
df_pa["date"].dtype.name
# 'datetime64[ns]'
datetime.date型ではなく、pd.Timestamp型(Numpyのdateteme64型)で受け取ることができます。
pandas側でdtypeを確認したり、変換したりする
pyarrowがCSVファイルを読み込む時や .to_pandas() でpandasのDataFrameへ変換する時に型の制御が出来ることをお伝えしました。
受け取ったDataFrameのdtypeを確認したり、変換する方法も見てみましょう。
# 'pandas.api.types.'の記述を省略するためにimportしてます
from pandas.api.types import (
is_string_dtype, is_integer_dtype,
is_object_dtype, is_datetime64_dtype,
infer_dtype
)
####################
# dtypeを確認する方法
# 全ての列のdtypeを確認する
df_pa.dtypes # or df_pa.info()
# dtypeの数を集計する
df_pa.dtypes.value_counts()
# string dtypeか確認する
is_string_dtype(df_pa["B"])
# -> bool
# integer dtypeか確認する
is_integer_dtype(df_pa["A"])
# -> bool
# object dtypeか確認する
is_object_dtype(df_pa["date"])
# -> bool
# dateteme64 dtypeか確認する
is_datetime64_dtype(df_pa["date"])
# -> bool
# 要素がdateteme.date型か確認する
infer_dtype(df_pa["date"]) == "date"
# -> bool
####################
# dtypeを変換する方法
# str型の要素を持つobject dtypeを string dtype へ変換する
df_pa["date"].astype('string')
# -> pd.Series
# str型の要素を持つobject dtypeをpd.Timestamp型(Numpyのdateteme64型)へ変換する
pd.to_datetime(df_pa["date"], format="%Y-%m-%d")
# -> pd.Series
# datetime.date型の要素を持つobject dtypeをpd.Timestamp型(Numpyのdateteme64型)へ変換する
pd.to_datetime(df_pa["date"])
# -> pd.Series
##########################################
# dtypeを条件にDataFrameから列を抽出する方法
# object dtypeの列を抽出する
df_pa.select_dtypes('object')
# -> pd.DataFrame
# int型やfloat型などの数値のdtypeの列を抽出する
df_pa.select_dtypes('number')
# -> pd.DataFrame
# dateteme64 dtype の列を抽出する
df_pa.select_dtypes('datetime')
# -> pd.DataFrame
# DataFrameから dateteme64 dtype を"除いた"列を抽出する
df_pa.select_dtypes(exclude='datetime')
# -> pd.DataFrame
dtypeに関連した操作の例をいくつか挙げてみました。きちんと確認、変換をしておけば、その後のDataFrameの操作も安心です。
3. 他ライブラリと速度比較
ここでは他ライブラリのPolars(本稿執筆時点の最新版 ver 0.20.2)と速度比較してみます。
Polars公式サイトに掲載されている、複数CSVファイルを読み込んで1つのDataFrameにまとめる書き方を採用しています。
import polars as pl
# 1)「csv_folder」フォルダ配下の全てのCSVファイルを読み込む
polars_df = pl.read_csv("./csv_folder/**/*.csv")
# 2) 読み込んだデータをpandasのDataFrameへ変換する
df_pl = polars_df.to_pandas()
df_pl.shape
# (100000000, 3)
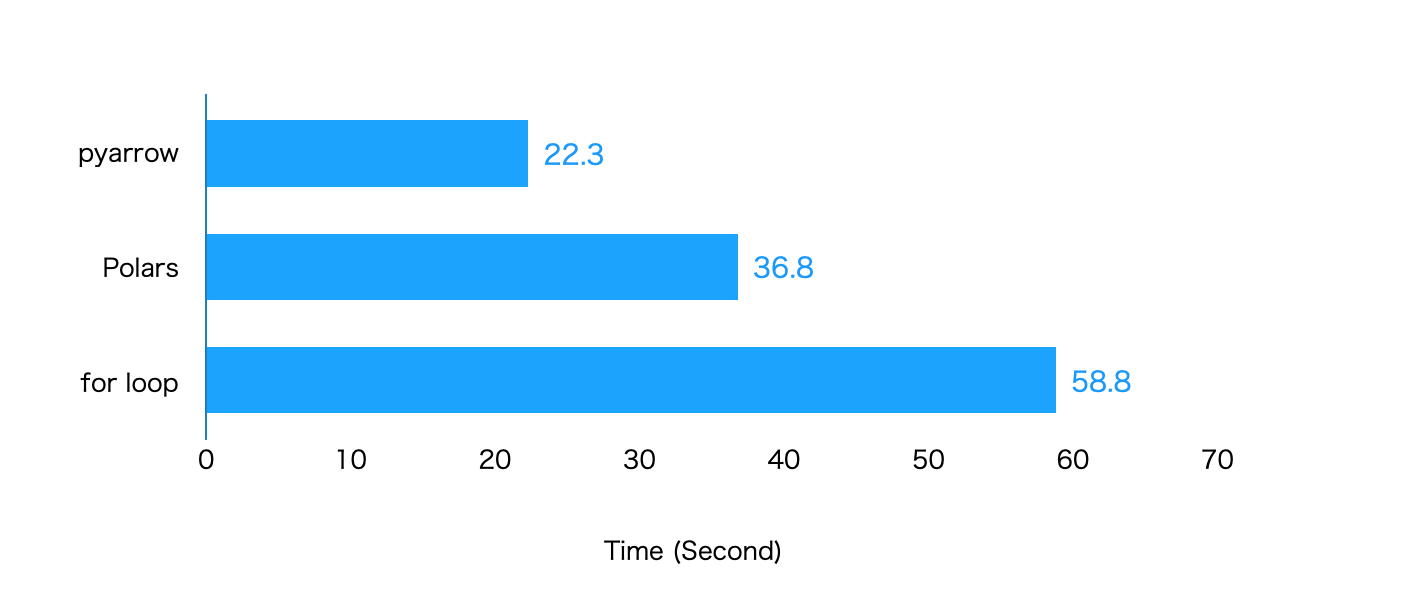
計測結果を先ほどのグラフに追加して並べてみます。
pyarrowがPolarsよりも高速に読み込んでいる事が確認できます。
おわりに
本稿では、モダンなCPUやGPUで効率的にデータ分析処理を行える「Apache Arrow フォーマット」のPythonライブラリ「pyarrow」を用いて、大量のCSVファイルを高速に読み込む方法を紹介しました。今回紹介した内容はpyarrowが出来ることのごく一部に過ぎません。
条件にマッチしたレコードだけ読み込んだり、ローカルのファイルシステムだけでなく「AWS S3」や「Google Cloud Storage (GCS) 」といったクラウドのファイルシステムの操作も可能です。
そして、本稿を読まれた方は、今この瞬間から複数CSVファイルの読み込み操作に関して「for文を使った方法」と「pyarrowを使った方法」の二刀流の使い手になっていることでしょう。もし今後、大量データを扱う機会に遭遇した際は、pyarrowで読み込んでpandasに渡す方法を試してみてください。