Rではheatmap(x)と打つだけで、x, y各軸に対してクラスタリングされたデンドログラム付きヒートマップが描画でき、遺伝子の発現量が・・・など日々呟いているbioinformaticiansにはお馴染みの機能なのですが、Python+matplotlib環境で同じことをやろうとした際、あまり情報が無かったので、試行錯誤してみました。
備忘録ついでに公開しときます。
データの準備
まずは、適当なデータとして、細胞組織ごとの遺伝子発現量の増減を表したようなデータを作ります。
ここでの組織のチョイスも、遺伝子のチョイスも、全て適当です。
後でクラスタリングしたときにそれっぽく見えるように、乱数で生成したデータを若干小細工してpandas.DataFrameオブジェクトにします。
# !/usr/bin/env python3
genes = [
'HIST1H4H', 'SPRN', 'DNLZ', 'PYCARD', 'PRDM11', 'DKKL1', 'CYBRD1', 'DMAP1',
'MT1E', 'PDGFRL', 'SERTM1', 'PIFO', 'FAM109A', 'ST5', 'ABCA7', 'FAM160A1',
'SAMD15', 'NUAK1', 'GLTP', 'HIST3H2A', 'SCN5A', 'PNPLA5', 'SFRP5', 'CCBE1',
'PTCD1', 'RFTN1', 'SYTL2', 'FAM65B', 'NFKBIZ', 'RHOG', 'KIF3A', 'DYRK1B',
'NXPH2', 'APLN', 'ZNF526', 'NRIP3', 'KCNMA1', 'MTSS1', 'ZNF566', 'TNC',
'GPX2', 'AQP3', 'TSACC', 'SNX15', 'TRIM22', 'THAP6', 'GRIP1', 'DLGAP3',
]
tissues = [
'brain', 'kidney', 'lung', 'heart',
'liver', 'pancreas', 'testis', 'placenta',
]
def draw_heatmap(a):
print(a.head())
def _main():
from random import choice
import numpy as np
from pandas import DataFrame
v = np.random.random([4, len(tissues)]) * 1.2 - 0.6
w = np.random.random([3, len(genes)]) * 0.8 - 0.4
v = np.vstack([choice(v) for x in genes])
w = np.vstack([choice(w) for x in tissues]).T
a = DataFrame(v + w, index=genes, columns=tissues)
draw_heatmap(a)
if __name__ == '__main__':
_main()
brain kidney lung heart liver pancreas \
HIST1H4H -0.085630 0.074054 0.058026 -0.142751 -0.767515 -0.348885
SPRN -0.424203 0.251821 -0.012052 -0.037645 -0.000477 0.714727
DNLZ 0.372402 0.532086 0.097971 -0.102806 -0.727570 0.109148
PYCARD -0.561378 0.114647 0.706732 0.681139 0.718306 0.577552
PRDM11 -0.698969 -0.022945 0.240133 0.214540 0.251708 0.439960
testis placenta
HIST1H4H 0.324709 -0.319531
SPRN -0.815679 -0.010529
DNLZ 0.402956 -0.241284
PYCARD -0.728135 0.077015
PRDM11 -0.773637 0.031513
これ以降、draw_heatmap(a)関数のみをいじっていきます。
手っ取り早く表示する場合
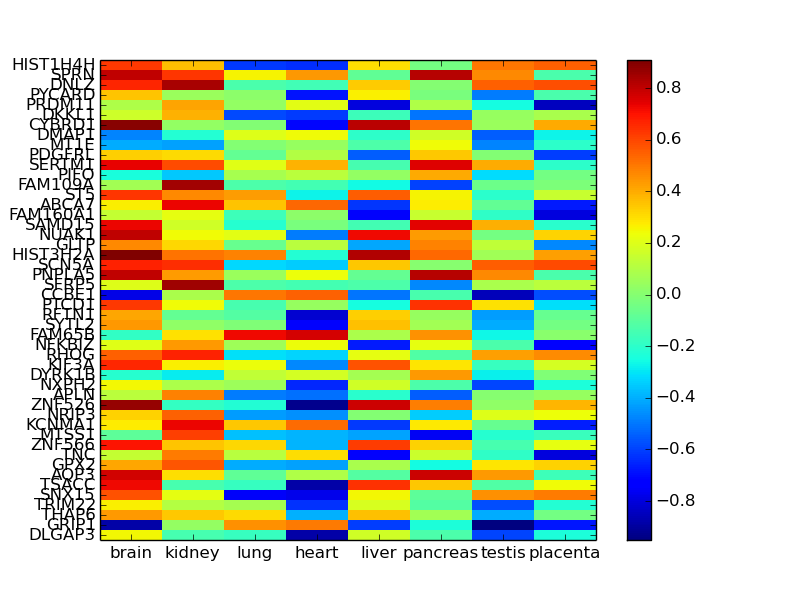
資料作成などではなく、とりあえずデータだけ確認したいときは、次のようにmatplotlibのimshow()にデータを突っ込めば表示してくれます。
def draw_heatmap(a):
from matplotlib import pyplot as plt
plt.imshow(a, aspect='auto', interpolation='none')
plt.colorbar()
plt.xticks(range(a.shape[1]), a.columns)
plt.yticks(range(a.shape[0]), a.index)
plt.show()
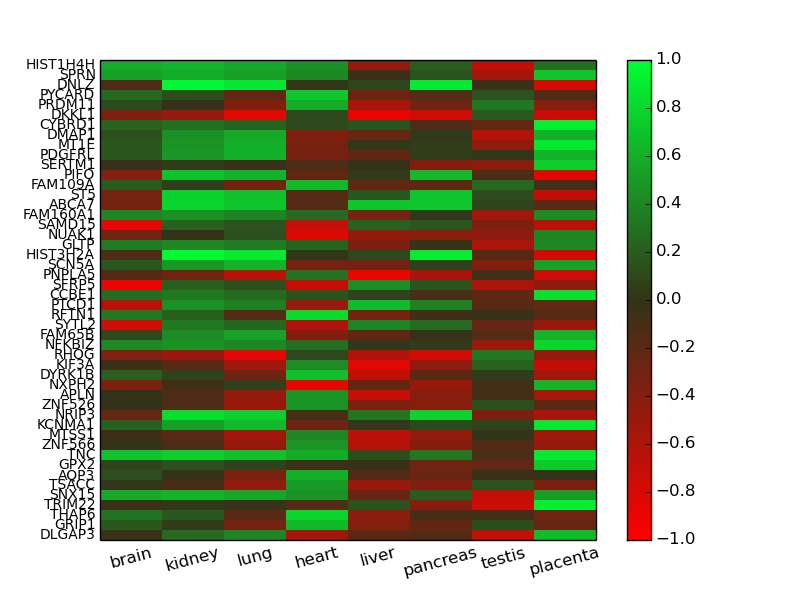
見栄えを良くする
上の例に対して、少し見栄えを良くしていきます。
- 目盛りは不要なので消します。
- ラベルが見にくいので、サイズや位置を調整します。
- 二色法マイクロアレイの名残か、Down regulationが赤、Neutralが黒、Up regulationが緑・・・という色分けが比較的多いようなので、これに倣います。
- スケールが正負で対称となっていないので、上限と下限を指定します。
from matplotlib.colors import LinearSegmentedColormap
microarray_cmap = LinearSegmentedColormap('microarray', {
'red': [(0.0, 1.0, 1.0), (0.5, 0.2, 0.2), (1.0, 0.0, 0.0)],
'green': [(0.0, 0.0, 0.0), (0.5, 0.2, 0.2), (1.0, 1.0, 1.0)],
'blue': [(0.0, 0.0, 0.0), (0.5, 0.2, 0.2), (1.0, 0.0, 0.0)],
})
def draw_heatmap(a, cmap=microarray_cmap):
from matplotlib import pyplot as plt
plt.imshow(a, aspect='auto', interpolation='none',
cmap=cmap, vmin=-1.0, vmax=1.0)
plt.colorbar()
plt.xticks(range(a.shape[1]), a.columns, rotation=15)
plt.yticks(range(a.shape[0]), a.index, size='small')
plt.gca().get_xaxis().set_ticks_position('none')
plt.gca().get_yaxis().set_ticks_position('none')
plt.show()
赤→黒→緑、と変わるColormapはmatplotlibのプリセットに無いので、LinearSegmentedColormapで自前で定義しています。
0を完全な黒とすると、0近傍の差が明度が低すぎて分かりにくいので、敢えて中間は濃い灰色にしています。
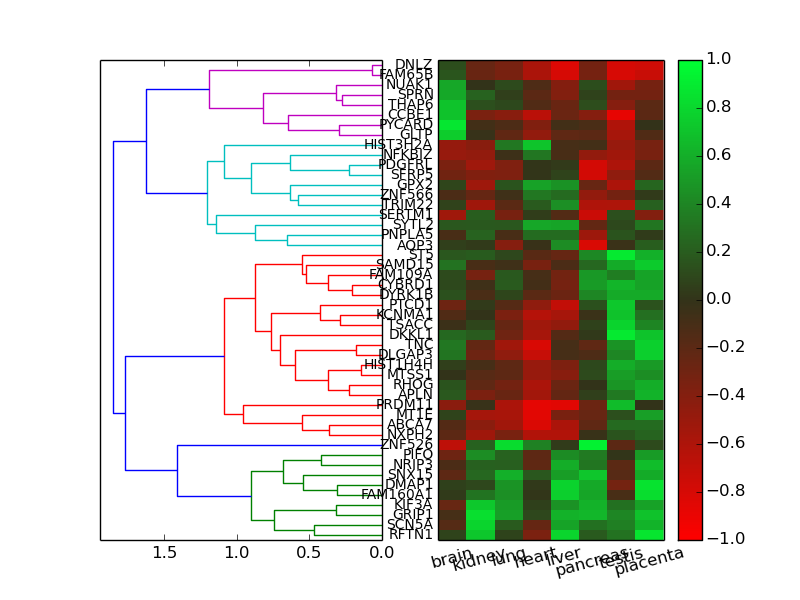
クラスタリングをする
似た発現パターンを持つ遺伝子をクラスタリングすることで、見やすくします。
def draw_heatmap(a, cmap=microarray_cmap):
from matplotlib import pyplot as plt
from scipy.spatial.distance import pdist
from scipy.cluster.hierarchy import linkage, dendrogram
metric = 'euclidean'
method = 'average'
plt.subplot(1, 2, 1)
ylinkage =linkage(pdist(a, metric=metric), method=method, metric=metric)
ydendro = dendrogram(ylinkage, orientation='right', no_labels=True,
distance_sort='descending')
a = a.ix[[a.index[i] for i in ydendro['leaves']]]
plt.subplot(1, 2, 2)
plt.imshow(a, aspect='auto', interpolation='none',
cmap=cmap, vmin=-1.0, vmax=1.0)
plt.colorbar()
plt.xticks(range(a.shape[1]), a.columns, rotation=15)
plt.yticks(range(a.shape[0]), a.index, size='small')
plt.gca().xaxis.set_ticks_position('none')
plt.gca().yaxis.set_ticks_position('none')
plt.gca().invert_yaxis()
plt.show()
Scipyの機能を用いて、階層クラスタリングを行い、デンドログラムと並べて描画します。
- 距離の定義は、euclideanの他にも様々なものを使用できる。詳しくはScipyのドキュメント参照。
- 階層クラスタリング方法も、averageの他にも様々なものを使用できる。詳しくはScipyのドキュメント参照。
- デンドログラム描画後、葉の順番に従いDataFrameの行を入れ替える。
- クラスタリングはしたいけれど、デンドログラムは要らない場合、
dendrogram(..., no_plot=True)を指定すれば良い。 - y軸に対し、
imshow()は左上を原点とするのに対し、dendrogram()は左下を原点とするので、invert_yaxis()を使って軸の方向を入れ替える。
見栄えを良くする
上の例の見栄えを良くします。
- デンドログラムとヒートマップをつなげて、y軸ラベルは右側に移動します。
- デンドログラムの枠は要らないので消します。
- デンドログラムの色も要らないので、黒のみにします。
- デンドログラムの幅を、もうすこし詰めます。
def draw_heatmap(a, cmap=microarray_cmap):
from matplotlib import pyplot as plt
from mpl_toolkits.axes_grid1 import make_axes_locatable
from scipy.spatial.distance import pdist
from scipy.cluster.hierarchy import linkage, dendrogram
metric = 'euclidean'
method = 'average'
main_axes = plt.gca()
divider = make_axes_locatable(main_axes)
plt.sca(divider.append_axes("left", 1.0, pad=0))
ylinkage = linkage(pdist(a, metric=metric), method=method, metric=metric)
ydendro = dendrogram(ylinkage, orientation='right', no_labels=True,
distance_sort='descending',
link_color_func=lambda x: 'black')
plt.gca().set_axis_off()
a = a.ix[[a.index[i] for i in ydendro['leaves']]]
plt.sca(main_axes)
plt.imshow(a, aspect='auto', interpolation='none',
cmap=cmap, vmin=-1.0, vmax=1.0)
plt.colorbar(pad=0.15)
plt.gca().yaxis.tick_right()
plt.xticks(range(a.shape[1]), a.columns, rotation=15, size='small')
plt.yticks(range(a.shape[0]), a.index, size='small')
plt.gca().xaxis.set_ticks_position('none')
plt.gca().yaxis.set_ticks_position('none')
plt.gca().invert_yaxis()
plt.show()
-
subplot()の代わりに、make_axes_locatable()を使っています。 - デンドログラムの枠を
set_axis_off()で非表示にし、link_color_funcで線を黒のみにしています。 -
yaxis.tick_right()で、y軸ラベルを右側に持ってきます。このままではカラーバーと重なるので、colorbar(pad=...)として間隔を広げています。
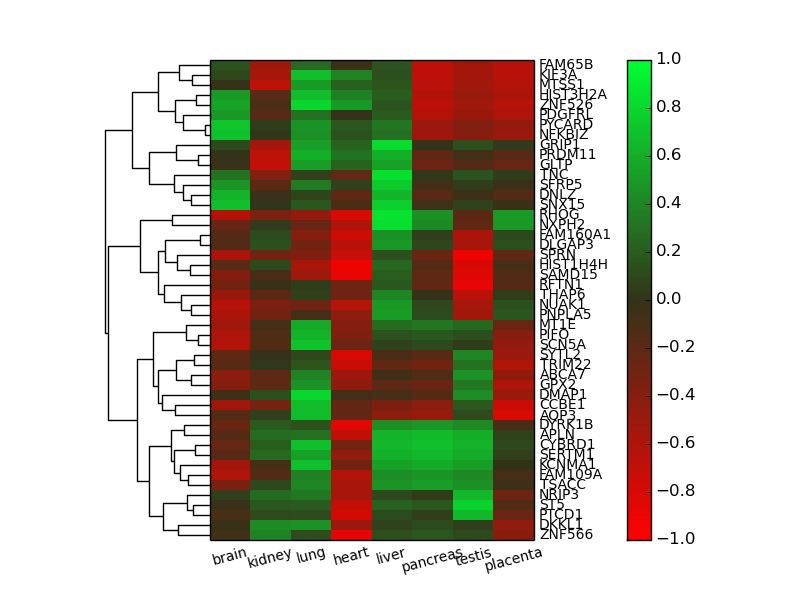
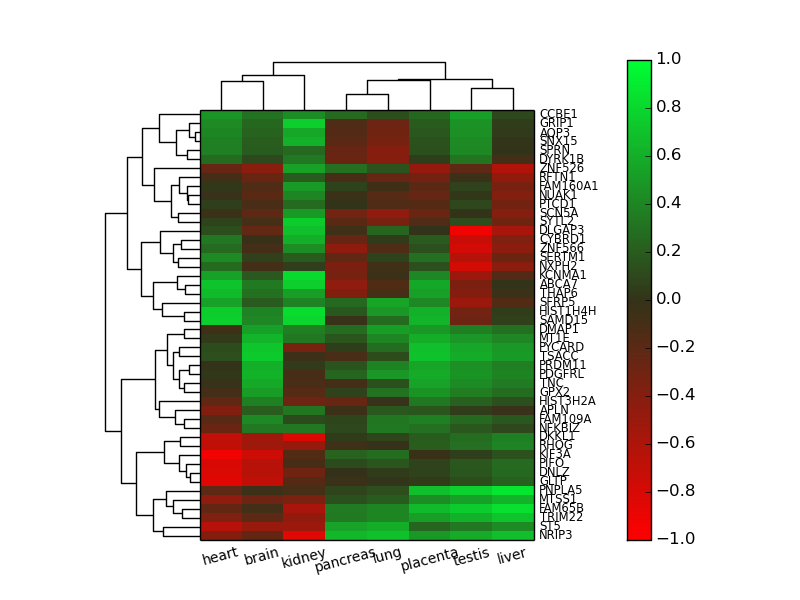
x軸もクラスタリングする。
時系列データなどならば不要ですが、組織ごとのデータなどではx軸もクラスタリングした方がわかりやすい場合もあります。やることは、基本的に今までと同じです。
def draw_heatmap(a, cmap=microarray_cmap):
from matplotlib import pyplot as plt
from mpl_toolkits.axes_grid1 import make_axes_locatable
from scipy.spatial.distance import pdist
from scipy.cluster.hierarchy import linkage, dendrogram
metric = 'euclidean'
method = 'average'
main_axes = plt.gca()
divider = make_axes_locatable(main_axes)
xdendro_axes = divider.append_axes("top", 0.5, pad=0)
ydendro_axes = divider.append_axes("left", 1.0, pad=0)
plt.sca(xdendro_axes)
xlinkage = linkage(pdist(a.T, metric=metric), method=method, metric=metric)
xdendro = dendrogram(xlinkage, orientation='top', no_labels=True,
distance_sort='descending',
link_color_func=lambda x: 'black')
plt.gca().set_axis_off()
a = a[[a.columns[i] for i in xdendro['leaves']]]
plt.sca(ydendro_axes)
ylinkage = linkage(pdist(a, metric=metric), method=method,
metric=metric)
ydendro = dendrogram(ylinkage, orientation='right', no_labels=True,
distance_sort='descending',
link_color_func=lambda x: 'black')
plt.gca().set_axis_off()
a = a.ix[[a.index[i] for i in ydendro['leaves']]]
plt.sca(main_axes)
plt.imshow(a, aspect='auto', interpolation='none',
cmap=cmap, vmin=-1.0, vmax=1.0)
plt.colorbar(pad=0.15)
plt.gca().yaxis.tick_right()
plt.xticks(range(a.shape[1]), a.columns, rotation=15, size='small')
plt.yticks(range(a.shape[0]), a.index, size='x-small')
plt.gca().xaxis.set_ticks_position('none')
plt.gca().yaxis.set_ticks_position('none')
plt.gca().invert_yaxis()
plt.show()
あとはsavefig('hoge.eps')などして論文に貼り付ければおしまい。
ところで、dendrogram(orientation=...)の意味が、横向きと縦向きで異なっているような気がするのですが・・・Scipyのバグ?