この記事はMATLABで機械学習を実装する方法について説明します。
前置き
機械学習をやると言ったら、殆どの人はPythonですよね。でもRやJuliaなど他の言語は少ないながら使っている人がある程度います。MATLABも機械学習の実装に向いている言語の一つです。
私も今までPythonだけ使っていたのですが、今の仕事でMATLABを使うことになったので、勉強し始めたばかりです。意外と興味深い言語です。
Pythonと違って、MATLABでは外部ライブラリを必要とせずに公式で機械学習用のパッケージを持っているので、インストールなどの面倒なことなしで簡単にすぐ実装できて便利です。公式なので安定として安心で使えますし。
それにPythonのライブラリーは色々すぎどれを使うか悩むこともありますね。MATLABでは機械学習機能が公式のものだから、使い方は統一しています。
MATLABは有料なので、所有者であるMathWorks会社も色んな機能を充実させるように頑張って工夫しているはずでしょうね。
MATLABの機械学習に関する関数を使ってみたら確かに意外とよくできています。
ということでここではMATLABで機械学習をやってみたい人のための入門となります。
なお、これはあくまでMATLABの入門で、機械学習の入門ではないので注意。既にPythonなどで機械学習を実装した経験を持つ人を前提として書いているつもりなので、機械学習の基本に関する説明は殆ど省いています。
MATLABのもっと基本の内容は前の記事で書いたので、配列の扱い方に関してはまずこの記事を読むのをおすすめします。
また、MATLABで機械学習をやるにはまずStatistics and Machine Learning Toolboxというパッケージをインストールする必要があります。
更に深層学習をやりたい場合は特にDeep Learning Toolboxが必要となります。
今回の記事では深層学習のことまで説明しませんが、深層学習の実装に関しては続編として次の記事に書いてあります。
既存のデータセットを使う
機械学習の実装を試すためにはまず何らかのデータセットが必要ですね。自分で準備してもいいのですが、今回はMATLABの既存のデータを使います。
Statistics and Machine Learning Toolboxパッケージの中には機械学習でよく使われるデータセットも含まれているので、勉強など使えます。
どんなデータセットがあるか公式サイトのこの頁に書いてあります。load関数で呼び出せます。
今回は「fisheriris」という有名なアヤメの花のデータセットを使います。
データを読み込んだら特徴量はmeasという変数に、ラベルはspeciesという関数に入れられます。
load fisheriris
size(meas)
size(species)
ans =
150 4
ans =
150 1
データ数は150で、特徴量は4つ:がく片の長さ、がく片の幅、花びらの長さ、花びらの幅。
一部だけのデータを表示してみます。
meas(1:5,:)
species(1:30:end)
ans =
5.1000 3.5000 1.4000 0.2000
4.9000 3.0000 1.4000 0.2000
4.7000 3.2000 1.3000 0.2000
4.6000 3.1000 1.5000 0.2000
5.0000 3.6000 1.4000 0.2000
ans =
5×1 の cell 配列
{'setosa' }
{'setosa' }
{'versicolor'}
{'versicolor'}
{'virginica' }
ラベルのデータはcell配列になっているので、扱いにちょっと面倒なところもあるかも。
cell配列についてはこの記事に纏めてあるので、まず読んでおいたら楽です。
教師あり学習の予測モデルを作る
MATLABでは色んな機械学習モデルが準備してあるので、これを使って簡単に実装できます。
よく使われる分類モデルを作る関数はここに纏めておきます。
| 学習関数名 | 分類モデルクラス名 | 方法 |
|---|---|---|
| fitclinear | ClassificationLinear | 線形 |
| fitcknn | ClassificationKNN | k最近傍 |
| fitcnb | ClassificationNaiveBayes | 単純ベイズ |
| fitctree | ClassificationTree | 決定木 |
| fitcsvm | ClassificationSVM | サポートベクターマシン |
| fitcnet | ClassificationNeuralNetwork | 全結合のフィードフォワードニューラルネットワーク |
| fitcensemble | ClassificationEnsemble | アンサンブル学習 |
その他に、回帰するためのモデルは大体関数名が分類学習関数名を「c」から取って「r」に入れ替えるだけです。例えば決定木回帰はfitrtree関数を使います。ここでは主に分類モデルで例を挙げますが、回帰モデルも同様に使えます。
普段使う時にこれらの学習関数に特徴量とラベルのデータを入れて、そうしたらモデルが作られて学習が始まって、学習済みのモデルが返り値とされます。そしてそのモデルを使って予測できます。
まずは例として決定木で例を挙げます。アヤメの花のデータを読み込んで準備しておいたらfitctree関数ですぐ学習を始められます。
load fisheriris
model = fitctree(meas,species)
model =
ClassificationTree
ResponseName: 'Y'
CategoricalPredictors: []
ClassNames: {'setosa' 'versicolor' 'virginica'}
ScoreTransform: 'none'
NumObservations: 150
fitctreeの学習で返されたのはClassificationTreeという決定木モデルクラスのオブジェクトです。
そしてこの学習済みのモデルにはpredictというメソッドを持っています。これを使って予測して結果を得られます。ここで学習データをそのまま使ってみます。
species_yosoku = model.predict(meas);
結果と実際のラベルを比べてみます。ただしここでラベルは文字列で、MATLABでは文字列を数値みたいに==で比較することができません。ちょっとややこしいですが、strcmpi関数を使う必要があります。
比較して性格率を正確度を計算してみます。
seikakudo = mean(strcmpi(species_yosoku,species))
seikakudo =
0.9800
かなり高い正確度です。ただしこれは学習データだから当然ですね。訓練データとテストデータを分ける方法については後で説明します。
他のモデルも使ってみましょう。例えばk近傍法。学習はfitcknn関数を使います。
load fisheriris
model = fitcknn(meas,species);
species_yosoku = model.predict(meas);
seikakudo = mean(strcmpi(species_yosoku,species))
seikakudo =
1
100%?こんなにいい結果?
いいえ、実はあまり不思議なことではありません。k近傍法では最近傍の数が1なら学習データ自身を予測させたら100%となるのは当然。これは常識ですね。
fitcknnをそのまま使ったら既定値で最近傍の数が1になってしまいます。これは非常に過学習に陥りやすいのです。
確認するために先程学習した後のモデルの詳細を見てみましょう。
disp(model)
ClassificationKNN
ResponseName: 'Y'
CategoricalPredictors: []
ClassNames: {'setosa' 'versicolor' 'virginica'}
ScoreTransform: 'none'
NumObservations: 150
Distance: 'euclidean'
NumNeighbors: 1
ここでやはりNumNeighbors、つまり最近傍の数プロパティが1になっていますね。
モデルを調整する
学習関数をそのまま使ったら既定値が使われて、それは望ましい結果になるとは限らないのです。例えばk近傍法は普段もっと最近傍の数をもっと多くした方がいいはずです。
既定値とは違う値にしたいパラメータを学習関数のキーワードとして入れることで設定することができます。
例えば上述と同じk近傍法で、最近傍の数を3にしてみます。
load fisheriris
model = fitcknn(meas,species,NumNeighbors=3);
species_yosoku = model.predict(meas);
seikakudo = mean(strcmpi(species_yosoku,species))
seikakudo =
0.9600
結果は違って、正確度が下がりましたね。でもこれで過学習を避けらるでしょう。
その他にも色々調整できます。例えば標準化をしたい場合はStandardize=trueを書いたらいいです(既定ではfalse)。今回のアヤメの花のデータは標準化する必要がないが、使う必要がある場合も多いでしょう。
混同行列
混同行列はconfusionmat関数で作れます。
例えば単純ベイズを使って予測して結果を比較してみます。
load fisheriris
model = fitcnb(meas,species);
species_yosoku = model.predict(meas);
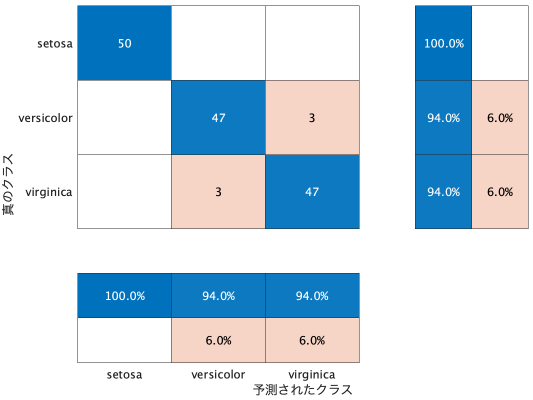
confusionmat(species,species_yosoku)
50 0 0
0 47 3
0 3 47
又はconfusionchartを使って図を書くこともできます。
confusionchart(species,species_yosoku)
わかりやすくて綺麗にできますね。
これでsetosaがちゃんと分類できるが、versicolorとvirginicaは混同しやすいとわかります。
キーワードを入れることで表示の設定ができます。例えば横と下にパーセントに纏める結果も表示したいならこう書きます。
confusionchart(species,species_yosoku, ...
ColumnSummary="column-normalized", ...
RowSummary="row-normalized")
データを分割する
モデルの学習は普段訓練データとテストデータに分ける場合が多いです。Pythonならtrain_test_splitみたいな便利な関数があってすぐ分割できますが、残念ながらMATLABはこんな関数がないのです。
ないなら自分で作るしかない。簡単に作れますし。
function [x_train,x_test,y_train,y_test] = train_test_split(x,y,test_size)
n_data = length(x);
n_train = round(n_data*(1-test_size));
i_rand = randperm(n_data);
i_train = i_rand(1:n_train);
i_test = i_rand(n_train+1:end);
x_train = x(i_train,:);
x_test = x(i_test,:);
y_train = y(i_train);
y_test = y(i_test);
end
そしてこうやって作っておいた関数を使ってデータを分けて訓練データで学習してテストデータで検証します。
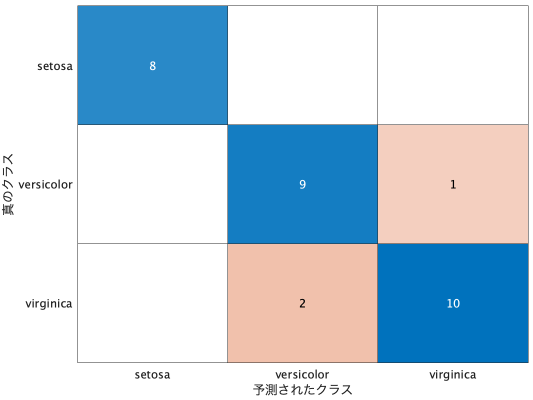
load fisheriris
[meas_train,meas_test,species_train,species_test] = train_test_split(meas,species,0.2);
model = fitcnb(meas_train,species_train);
species_yosoku = model.predict(meas_test);
figure
confusionchart(species_test,species_yosoku)
スコアと事後確率
predictメソッドを使う時に実は返り値が予測値だけでなく、もし2つの変数で返り値を受けたら2つ目の変数に予測のスコアというものが入ります。
そのスコアの解釈は学習モデルによって違うのですが、基本的に予測値を判断する基準となる数値です。例えばfitcnb(単純ベイズ)の場合は「事後確率」、つまり各ラベルに分類する確率です。
ちょっとその数値を見てみます。
load fisheriris
[meas_train,meas_test,species_train,species_test] = train_test_split(meas,species,0.2);
model = fitcnb(meas_train,species_train);
[species_yosoku,sukoa] = model.predict(meas_test);
disp(sukoa)
0.0000 0.0000 1.0000
0.0000 0.0073 0.9927
0.0000 1.0000 0.0000
1.0000 0.0000 0.0000
0.0000 0.0000 1.0000
1.0000 0.0000 0.0000
1.0000 0.0000 0.0000
0.0000 0.9845 0.0155
0.0000 0.0000 1.0000
0.0000 0.0417 0.9583
0.0000 1.0000 0.0000
0.0000 0.0000 1.0000
0.0000 0.9978 0.0022
0.0000 0.0000 1.0000
1.0000 0.0000 0.0000
0.0000 0.9998 0.0002
1.0000 0.0000 0.0000
0.0000 0.0000 1.0000
0.0000 0.0503 0.9497
0.0000 0.9997 0.0003
1.0000 0.0000 0.0000
0.0000 0.0081 0.9919
0.0000 0.0000 1.0000
1.0000 0.0000 0.0000
1.0000 0.0000 0.0000
0.0000 0.9997 0.0003
0.0000 0.9996 0.0004
0.0000 0.9835 0.0165
0.0000 0.0067 0.9933
0.0000 0.9895 0.0105
このように3つのラベルのスコアが並びます。予測値だけでなく、この確率の数値が必要とする場合もあるでしょう。
k-fold交差検証
MATLABでは交差検証を簡単に実装できます。
ちなみに、交差検証について私は初めてのqiita記事でこんな絵を作成して説明したことがあります。
学習関数を使う時に分割したい数をKFoldキーワードに入れたら自動的にランダムで分けて交差検証を行うことになります。
そしてこの方法で学習したモデルは普段とは違うクラスになります。予測したい時にpredictメソッドではなく、kfoldPredictを使います。
load fisheriris
model = fitcnb(meas,species,KFold=5)
species_yosoku = model.kfoldPredict;
kondou = confusionmat(species_yosoku,species)
model =
ClassificationPartitionedModel
CrossValidatedModel: 'NaiveBayes'
PredictorNames: {'x1' 'x2' 'x3' 'x4'}
ResponseName: 'Y'
NumObservations: 150
KFold: 5
Partition: [1×1 cvpartition]
ClassNames: {'setosa' 'versicolor' 'virginica'}
ScoreTransform: 'none'
kondou =
50 0 0
0 47 3
0 3 47
テーブルのデータを使う
学習関数には、特徴量とラベルをそれぞれ入れる以外に、データが纏まっているテーブルオブジェクトを入れて使うこともできます。
自分でデータを準備する時などにreadtableなどの関数でテーブルとして読み込むことが多いでしょう。その場合でもtableとしてそのまま使えるので便利です。
例として今回はこのようにアヤメの花のデータセットをテーブルに入れておきます。
load fisheriris
ayame = table( ...
meas(:,1), ...
meas(:,2), ...
meas(:,3), ...
meas(:,4), ...
species, ...
VariableNames=["がく片の長さ" "がく片の幅" "花びらの長さ" "花びらの幅" "種類"])
ayame =
150×5 table
がく片の長さ がく片の幅 花びらの長さ 花びらの幅 種類
__________ _________ __________ _________ _____________
5.1 3.5 1.4 0.2 {'setosa' }
4.9 3 1.4 0.2 {'setosa' }
4.7 3.2 1.3 0.2 {'setosa' }
4.6 3.1 1.5 0.2 {'setosa' }
5 3.6 1.4 0.2 {'setosa' }
: : : : :
6.7 3 5.2 2.3 {'virginica'}
6.3 2.5 5 1.9 {'virginica'}
6.5 3 5.2 2 {'virginica'}
6.2 3.4 5.4 2.3 {'virginica'}
5.9 3 5.1 1.8 {'virginica'}
このように学習に使うテーブルの準備が整いました。
学習関数に使う時そのテーブルオブジェクトと、ラベルが入っている列の名前を入れます。
model = fitcknn(ayame,"種類",NumNeighbors=3)
model =
ClassificationKNN
PredictorNames: {'がく片の長さ' 'がく片の幅' '花びらの長さ' '花びらの幅'}
ResponseName: '種類'
CategoricalPredictors: []
ClassNames: {'setosa' 'versicolor' 'virginica'}
ScoreTransform: 'none'
NumObservations: 150
Distance: 'euclidean'
NumNeighbors: 3
こうやって特徴量とラベルを別々として入力する時と同じように学習が行われてモデルが作られますね。更にPredictorNamesとResponseNameのプロパティにテーブルの列の名前が付いています。
そしてpredictを使う時にテーブルオブジェクトを入れることもできます。
model = fitcknn(ayame,"種類",NumNeighbors=10)
species_yosoku = model.predict(ayame);
confusionchart(species_yosoku,ayame.("種類"));
2次元への可視化
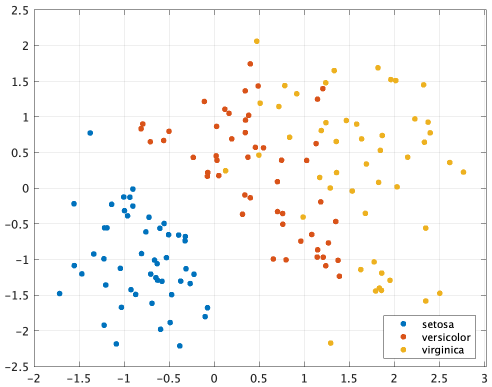
多次元のデータを2次元に圧縮して可視化する方法としてt-SNE(T分布型確率的近傍埋め込み法)というアルゴリズムは有名ですね。MATLABではtsne関数を使って簡単に実装できます。
アヤメの花のデータに試して、2次元になったら散布図を描きます。
load fisheriris
meas2d = tsne(meas);
figure
gscatter(meas2d(:,1),meas2d(:,2),species)
grid
既定値以外に、色んなキーワードで違う設定をして違う結果ができます。
meas2d = tsne(meas, ...
Algorithm="exact", ...
Perplexity=100, ...
Standardize=true, ...
Distance="mahalanobis");
どうしてもversicolorとvirginicaが絡み合って分別なんて難しいですね。
2次元の他に、NumDimensionsを設定して3次元などにすることもできます。
meas3d = tsne(meas,NumDimensions=3);
クラスタリング
上述は主に教師あり学習ですが、教師なし学習も試してみます。一番よく使われるのはk平均法というクラスタリング手法です。これをkmeans関数で簡単に実装できます。
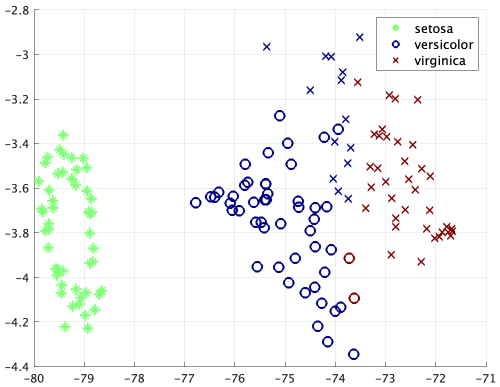
アヤメの花のデータをラベル指定なしで学習させて3つのクラスターに分けて番号を付けてみます。そして結果を表示するためにtsneを使って2次元でプロットします。
load fisheriris
ic = kmeans(meas,3);
meas2d = tsne(meas,Perplexity=70);
figure
hold on
ii = strcmpi(species,"setosa");
scatter(meas2d(ii,1),meas2d(ii,2),90,ic(ii),"*",LineWidth=2)
ii = strcmpi(species,"versicolor");
scatter(meas2d(ii,1),meas2d(ii,2),90,ic(ii),"o",LineWidth=2)
ii = strcmpi(species,"virginica");
scatter(meas2d(ii,1),meas2d(ii,2),90,ic(ii),"x",LineWidth=2)
legend(unique(species),FontSize=12)
colormap(gca,"jet")
grid
ここで色は予測結果を示します。藍色の✕と赤色の◯は完全に分別できないところです。versicolorとvirginicaの領域は重ね合っているから教師あり学習でなければ分別するのは無理ですね。
簡単なニューラルネットワーク
MATLABにおいてニューラルネットワークは主にDeep Learning Toolboxを使うのですが、ただの簡単な全結合のフィードフォワードニューラルネットワークを使いたいならStatistics and Machine Learning Toolboxだけで実装できます。分類モデルならfitcnetを、回帰モデルならfitrnetを使います。使い方も簡単で、特に設定せずに既定値で実装できます。
ではアヤメの花のデータセットで試しましょう。使い方は殆ど上述のモデルと同じ。
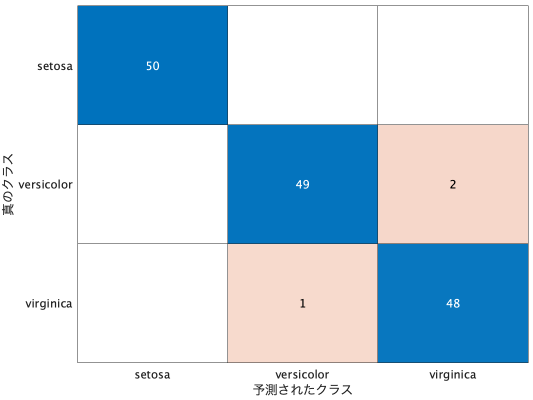
load fisheriris
model = fitcnet(meas,species)
species_yosoku = model.predict(meas);
kondou = confusionmat(species,species_yosoku)
model =
ClassificationNeuralNetwork
ResponseName: 'Y'
CategoricalPredictors: []
ClassNames: {'setosa' 'versicolor' 'virginica'}
ScoreTransform: 'none'
NumObservations: 150
LayerSizes: 10
Activations: 'relu'
OutputLayerActivation: 'softmax'
Solver: 'LBFGS'
ConvergenceInfo: [1×1 struct]
TrainingHistory: [170×7 table]
kondou =
50 0 0
0 49 1
0 1 49
ニューラルネットワークはたとえ一番簡単な全結合のフィードフォワードニューラルネットワークでも設定する必要があるハイパーパラメータが沢山ありますね。でもfitcnet関数は既定値があって特に考えずにそのまま使ってもいいです。
ここでモデルのプロパティを見るとLayerSizesのところは10になっています。これは隠れ層の出力サイズです。既定では一つだけで出力サイズは10になっていますが、LayerSizes=[16 32 64]など3層に変えることができます。その他にもActivations(活性化関数)など色々設定できます。ただしやはり機能が限られているので、本格的なニューラルネットワークを作るのにあまり向いていないと思います。
このように全結合層を重ねただけの簡単なニューラルネットワークならこれで作れますが、もっと複雑な構造もしくは学習方法など細かく設定したいならやはりDeep Learning Toolboxを使う必要があるでしょう。
学習結果はConvergenceInfoにstructとして保存されています。
model.ConvergenceInfo
フィールドをもつ struct:
Iterations: 170
TrainingLoss: 0.0132
Gradient: 1.5561e-07
Step: 0.0016
Time: 0.0182
ValidationLoss: NaN
ValidationChecks: NaN
ConvergenceCriterion: '勾配の相対許容誤差に達しました。'
History: [170×7 table]
学習の歴史はTrainingHistoryに保存してあります。
model.TrainingHistory
170×7 table
Iteration TrainingLoss Gradient Step Time ValidationLoss ValidationChecks
_________ ____________ __________ _________ __________ ______________ ________________
1 0.36355 0.074598 0.045987 0.00030425 NaN NaN
2 0.35479 0.065764 0.084421 0.00020304 NaN NaN
3 0.3366 0.24084 0.25967 0.00018733 NaN NaN
4 0.33351 0.12719 0.10369 0.00032108 NaN NaN
5 0.31654 0.12996 0.10265 0.00032075 NaN NaN
: : : : : : :
166 0.013221 2.4135e-05 0.0053553 6.0917e-05 NaN NaN
167 0.013221 2.1091e-05 0.0031286 6.3458e-05 NaN NaN
168 0.013221 3.6114e-06 0.0021568 0.00032692 NaN NaN
169 0.013221 3.5813e-06 0.0064717 6.0167e-05 NaN NaN
170 0.013221 1.5561e-07 0.0015599 0.0010566 NaN NaN
ValidationLossとValidationChecksがNaNになっているのは今回検証データを入れていないからです。検証データがあればValidationDataキーワードに入れることで学習の時に検証も行われます。
学習の時の損失の数値はここにあるので、これを取ってクラフを描いてみます。
figure
semilogy( ...
model.TrainingHistory.Iteration, ...
model.TrainingHistory.TrainingLoss, ...
"m",LineWidth=2)
xlabel("反復")
ylabel("損失")
grid
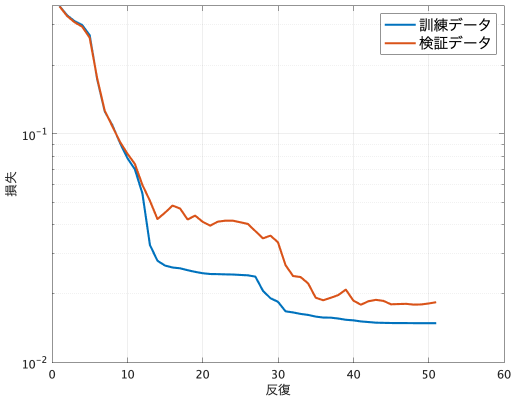
検証データも準備して学習に使う場合も実装してみましょう。隠れ層も4層入れてみます。そして最後に訓練データと検証データの損失を一緒にプロットします。
load fisheriris
[meas_train,meas_test,species_train,species_test] = train_test_split(meas,species,0.2);
model = fitcnet(meas_train,species_train, ...
LayerSizes=[32 64 32 16], ...
ValidationData={meas_test,species_test}, ...
ValidationPatience=10);
figure
semilogy( ...
model.TrainingHistory.Iteration, ...
model.TrainingHistory.TrainingLoss, ...
model.TrainingHistory.Iteration, ...
model.TrainingHistory.ValidationLoss, ...
LineWidth=2)
xlabel("反復")
ylabel("損失")
legend("訓練データ","検証データ",fontsize=14);
grid
参考&もっと読む
- MATLABの機械学習で脳波を識別 その1
- MATLABの機械学習で脳波を識別 その2
- 言語処理100本ノックで MATLAB 入門!第6章: 機械学習 50-54
- 言語処理100本ノックで MATLAB 入門!第6章: 機械学習 55-59
- 5.6.2.1 ディープラーニング:畳み込みニューラルネットによる分類
- 5.6.6 ディープラーニング:点群データのサポート
- MathWorks機械学習・深層学習セミナーの感想
終わりに
以上機械学習によく使われる機能を色々紹介しましたが、その他にも沢山あります。
MATLABの機械学習パッケージは機能が整っているので、Pythonで機械学習をやっていた人はMATLABでやることになっても色々同じようにできるはずです。ただし色々違うところもあるので、ある程度勉強する時間が必要でしょう。