内容
NVIDIA RAPIDSが提供するGPU DataFrameライブラリ cuDFを使ってみました。
Pandasと似たような操作でデータの前処理/加工をGPUで行うことができるみたいです。
CPUで処理するよりも早いとの事で気になったので入れて試してみました。
実行環境
- OS
- Windows 10 Home 64bit
- WSL2 Ubuntu 20.04
- Docker 20.10.10
- CPU
- Intel Core i7-7820X
- Memory
- DDR4-3200MHz 96GB
- GPU
- NVIDIA GeForce RTX3060Ti
cuDFについて
- pipでは提供されていない
- Python対応バージョンは3.8 or 3.9
- Dockerコンテナでの提供もされている
前提条件確認
導入するための前提条件をチェック

GPU
- Pascalコア採用以降のGPUかつcompute capabilityページでの数字が6.0+以降の製品
- Pascalコア採用は一般向けビデオカードでいうとGTX1xxxシリーズ以降が対応
- GT1030はPascalコアだがcompute capabilityに未記載
- 自分が使用しているRTX3060Tiは対応
- Pascalコア採用は一般向けビデオカードでいうとGTX1xxxシリーズ以降が対応
OS
- Ubuntu 18.04/20.04 or CentOS 7/8 かつ gcc/++ 9.0以降
- 後半の要件は
gcc -vでバージョンチェック
- 後半の要件は
- 自分の環境では
gcc -v結果9.3.0
Docker
- Docker CE(コミュニティ エディション) v19.03
- CEはエディションの違い。無料版。
-
docker -vでバージョンチェック
- 自分の環境では
docker -v結果20.10.10
CUDA & NVIDIA Drivers
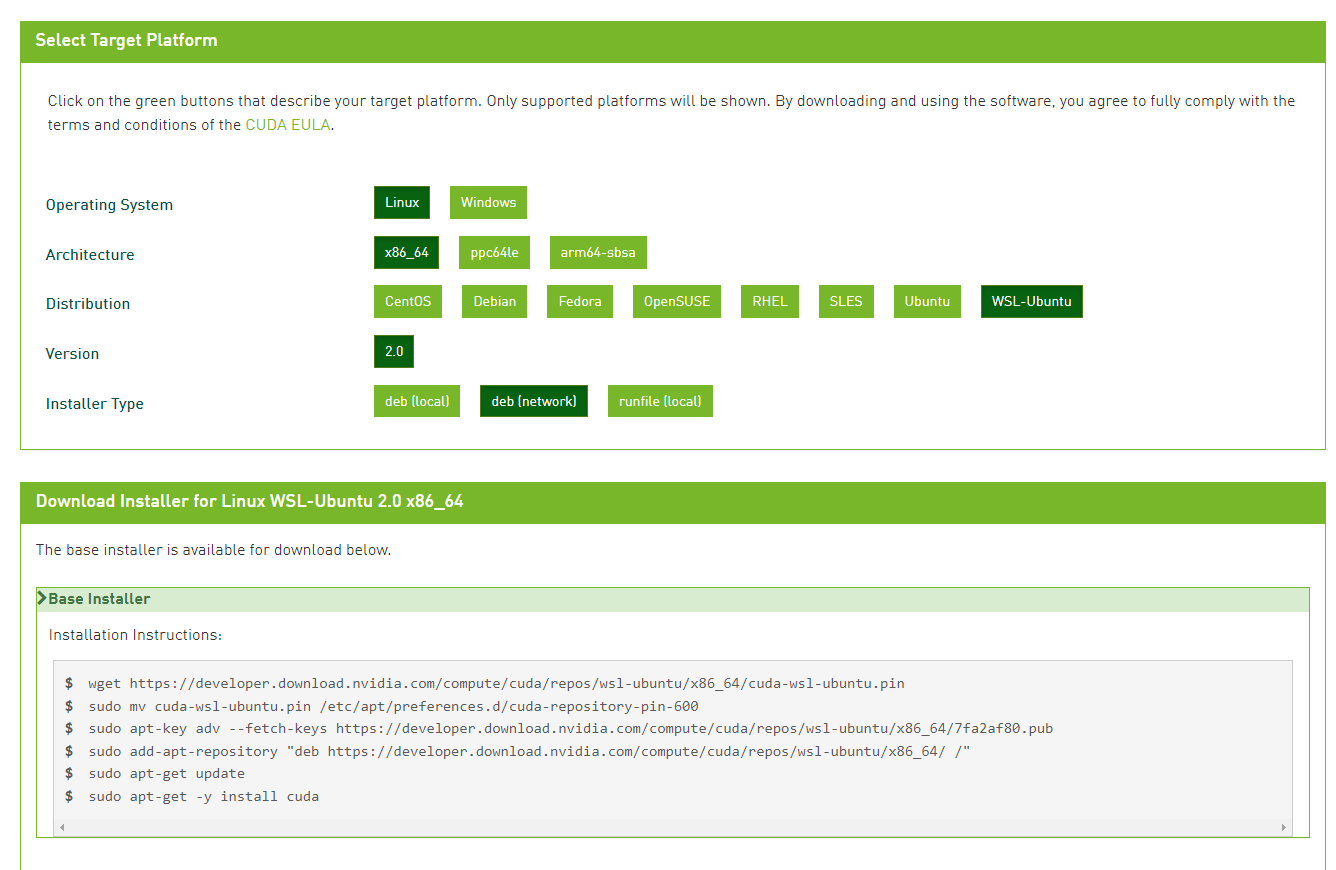
- 上記画像の4種類のドライババージョンとCUDA Toolkitのバージョンが必要
- 自分の環境はNVIDIA Driver 511.79&CUDA Toolkit未インストール
-
Dockerの項目にnvidia-container-toolkitが必要とあるが、そのページでは
Note that you do not need to install the CUDA Toolkit on the host system, but the NVIDIA driver needs to be installedとある。CUDA Toolkitは入れなくてもいい? - 一応以下のように項目を選択して入れた
-
nvidia-smiで確認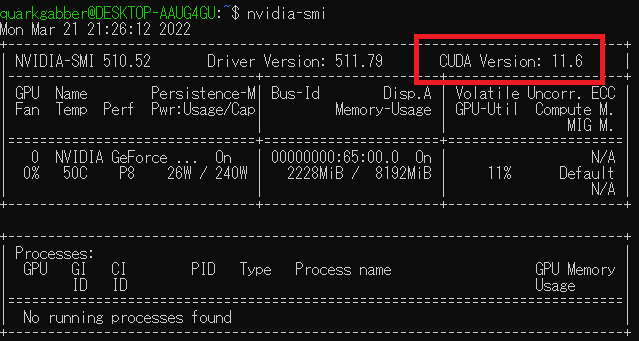
-
Dockerの項目にnvidia-container-toolkitが必要とあるが、そのページでは
導入
RAPIDS RELEASE SELECTORなるもので環境を選んで入れられるようなので以下のように選択した
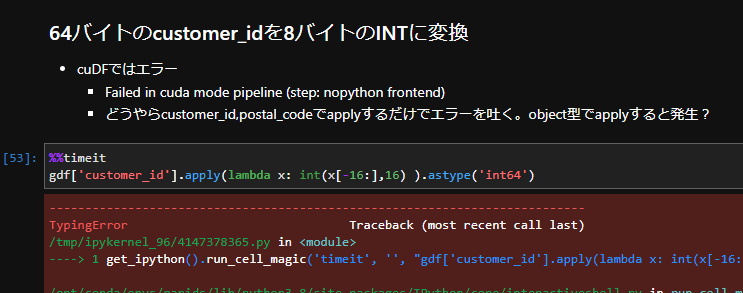
ボリュームマウントしたいのでdocker runのコマンドを以下に変更
docker run -v /mnt:/rapids/notebooks/host --gpus all --rm -it -p 8888:8888 -p 8787:8787 -p 8786:8786 \
rapidsai/rapidsai-core:22.02-cuda11.5-runtime-ubuntu20.04-py3.8
/rapids/notebooks/hostへbindすることにより、hostフォルダからボリュームマウントしたフォルダ群が見れるようになる
WSL2で/mntとしてボリュームマウントするとWindows側のフォルダ群が見れる
処理速度比較
cuDFドキュメントを見ながら、Pandasとの処理速度の違いを比較してみました。
ノートブックはGistにアップロードしています。
対象データ
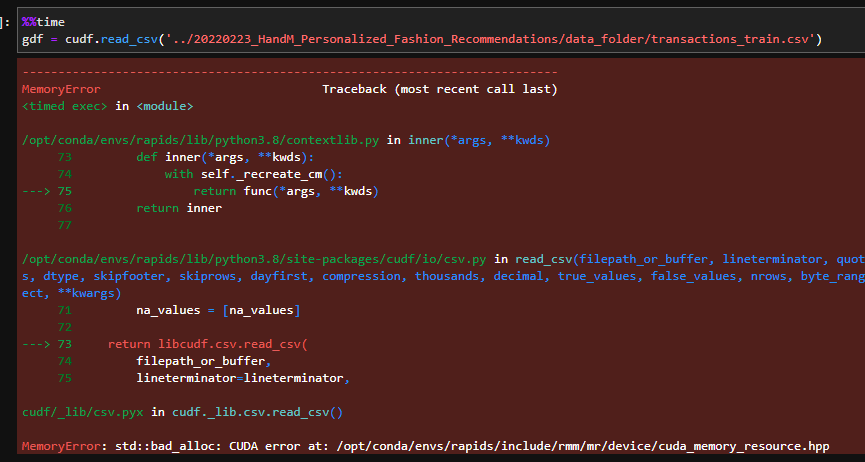
対象データはKaggle H&M Personalized Fashion Recommendationsのcustomers.csv(207MB)とtransactions_train.csvをcustomer_id毎に集計したデータを用いました。
余談ですが、transactions_train.csv(3.49GB)をそのまま用いた場合、cuDF.read_csv時にエラーを吐いてしまいました。
RAPIDS 概要資料の8ページに
必要なメモリサイズ
作業メモリとしてデータセットサイズの2-3倍
とありましたので、おそらくVRAMが足りなかったのかと思います。
結果
Notebookの%%timeitを使って処理に掛かった時間を見ています。
| 関数 | cuDF処理速度 | Pandas処理速度 | 処理速度倍率 |
|---|---|---|---|
| describe | 76.2ms | 203ms | 2.66倍 |
| apply | 140ms | 385ms | 2.75倍 |
| merge | 46.6ms | 1.63s | 34.98倍 |
| fillna | 50.7ms | 544ms | 10.73倍 |
| sort_values | 85.6ms | 2.07s | 24.18倍 |
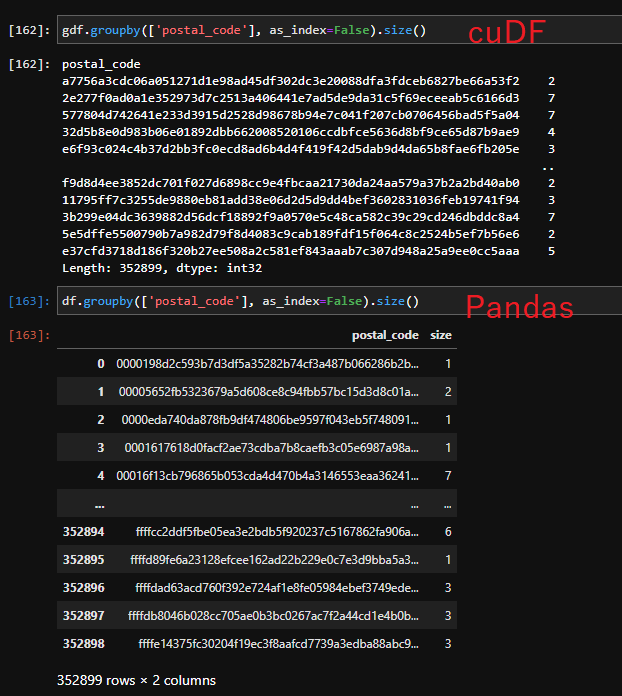
| groupby | 16.9ms | 1.21s | 71.60倍 |
| drop_duplicates | 120ms | 550ms | 4.58倍 |
| round | 1.41ms | 1.57ms | 1.11倍 |
試した関数については、すべてcuDFのほうが速いという結果になりました!
groupbyやmergeはびっくりするくらい速かったです。
多少気になったところ
- pickleファイルの読み書き関数が無い
-
pickle.dump(),pickle.load()を使えばいい
-
- groupby後の結果がDataFrameにならない
- object型に対してcuDF.applyができない?
感想
- 速くてとてもいい!けどPandasと思って使うとちょいちょい躓く部分があるかも
- VRAM量が無いと大きなデータについては抑々読み込めない
- colaboratoryで使えるT4,P100で使うのが良いかも
- 買える方ならVRAM 12GB以上のビデオカード
- 実行環境もDockerで提供されてて楽に使える
- GPUで機械学習アルゴリズムが使えるcuMLも良さそう