総研大 統計科学コースの平元です。アドベントカレンダーを書くことになったのでデータ同化普及のための記事を書きます。
粒子フィルタを実装するときのプログラミング言語 粒子数ドカ盛り編
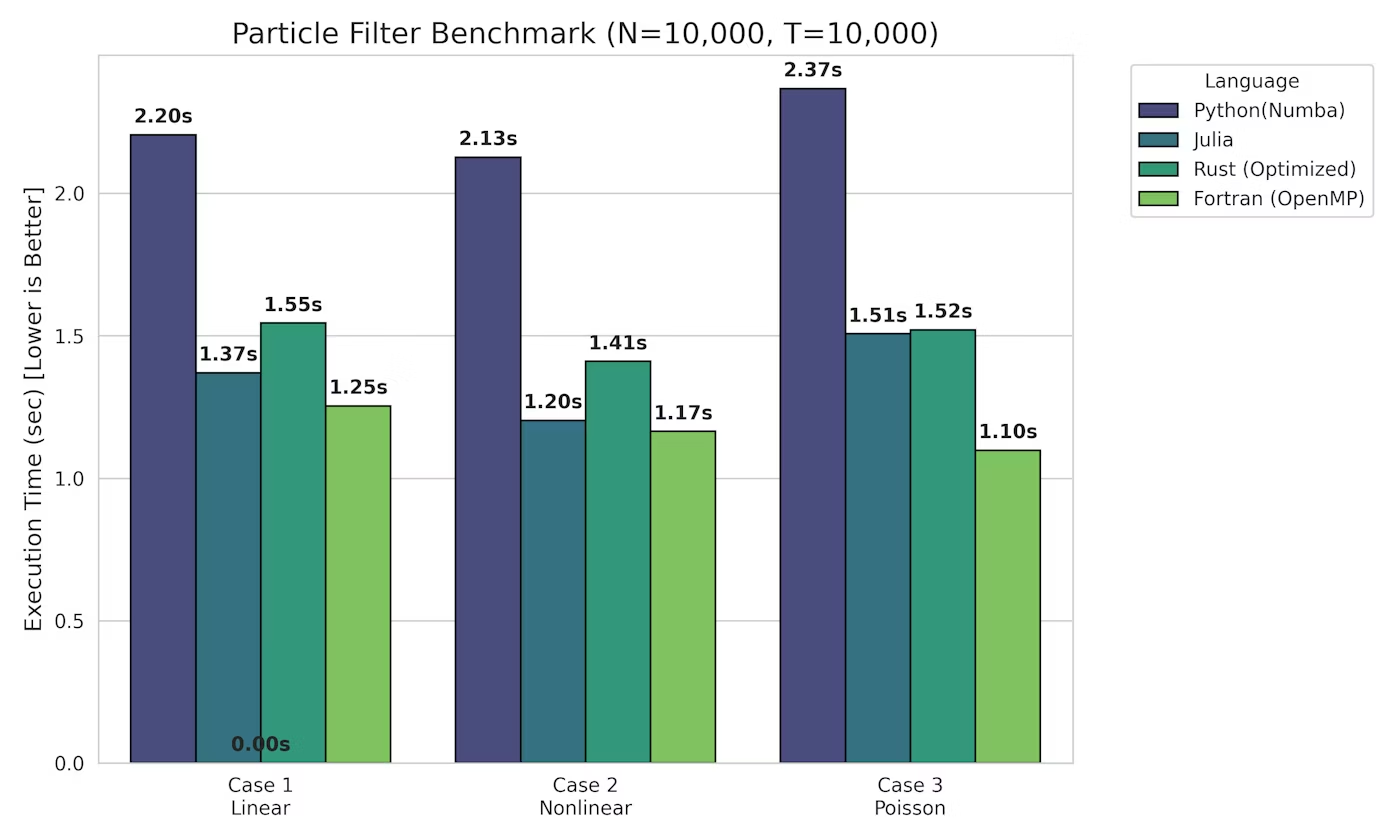
前回の記事では粒子フィルタを Python / Rust / Julia / Fortran の 4 言語で実装し速度を比較しました。
前回の記事でも書きましたが、 この結果は小規模な試験だからこその結果である可能性が高く、(特にPythonが得意な)並列化計算はむしろ遅くなりがちな試験内容でした。今回は補足として、粒子数を(個人宅のPCで実行可能な範囲で)増やした結果について検証します。
今回、粒子数を $N = 1{,}000{,}000$ に増やし、
Fortran / Python / Julia の 3 言語で Case 1 と Case 2 を比較しました(Rust は高速化に失敗したので今回は除外)。
モデル・データは前回の記事のCase1/2をそのまま用いて推定を実行した結果がこちらになります。

だいたいのイメージとしては、
Fortran:約 8 分
Python:約 8〜9 分
Julia:約 9〜11 分
といったところです(もちろんマシンやスレッド数によって前後します)。
前回との違いとして、Fortran が最速なのは変わらないが、Python と Fortran の差がかなり縮まっているという点が挙げられます。Julia は今回の実装だと Python よりやや遅いくらいとなっています(この辺は作者の技量・得意分野もあると思います)。
小規模編と合わせて考えると、どのケースでも Fortran が最速というのは変わりません。しかしながら、開発の容易さと速度のコストパフォーマンスではJuliaが相当に優秀であり、大規模化すればPythonもFortran並の速度を達成できるということがわかっただけでも今回は満足です。また、この辺りになってくると言語間の差というよりもアルゴリズム側の工夫も思います。
使用したコードは以下にあります。
https://github.com/nhiramoto10363/Qiita_PF_largeparticles