知っていると便利かもしれない小技から,どーでもいい小技まで.ググったサンプルをコピペして完了!系大学院生が読むと,何か新しい発見があるかもしれません.使わなさ(当社比)を4段階(☆☆☆,★☆☆,★★☆,★★★)で表記しますので,ああ確かに知らなくていいなって思ってください.新しい技を仕入れ次第,更新します.
多次元データへのアクセス
次元を落とさない
使わなさ:☆☆☆(そこそこ役に立つ)
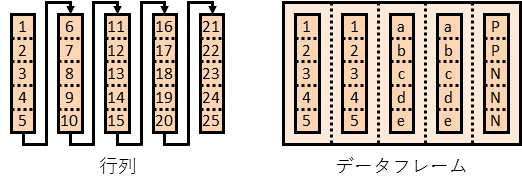
デフォルト設定では,行列や配列の一部分にアクセスした場合,その戻り値はできる限り次元を落としたものになります(次元を落とすことを「ドロップする」と言うことにします).データフレームでも同様です.
> x <- array(1:1000, c(10, 10, 10))
> class(x[1, 1, 1]) # 3次元配列 → 要素数1のベクトル(スカラ)
[1] "integer"
> class(x[1, 1, ]) # 3次元配列 → ベクトル
[1] "integer"
> class(x[1, , ]) # 3次元配列 → 行列
[1] "matrix"
引数 drop に FALSE を指定することで,次元を維持できます.
> class(x[1, 1, 1, drop=F])
[1] "array"
> class(x[1, 1, , drop=F])
[1] "array"
> class(x[1, , , drop=F])
[1] "array"
これは,行列から条件を満たす部分を取り出して処理したい場合に役立ちます.
index <- which(x[ , 1] != 0);
x.sub <- x[index, -1, drop=F];
apply(x.sub, 1, sum);
この例では index の要素数が1になる可能性があり,その場合 x[index, -1] は行列ではなくベクトルになります.しかし,関数 apply はベクトルに対して適用できないので,このままではエラーになります.そこで drop=F を指定し,常に行列として値を返すよう設定します.
データフレームの列をカンタンに取り出す
使わなさ:★☆☆
データフレームから特定の列を取り出す場合,以下のようなコマンドを使うことが多いのではないでしょうか.
data(iris)
iris[ , "Species"]
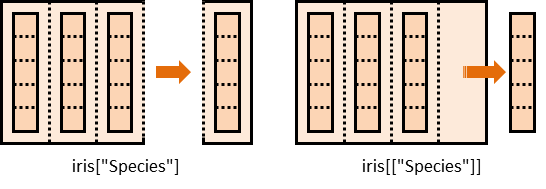
これでもいいのですが,データフレームの場合は単純に列名を指定するだけで列を取り出せます.
iris["Species"]
これは,データフレームが列ごとにベクトルを作り,ベクトルのリストとしてデータフレーム全体を管理しているためです.一方で,行列は行列全体を一本のベクトルとして管理しているので,10列目を取り出そうとして x[10] と書いても表全体の10番目の値が得られるだけですので注意してください.
ちなみに,iris[ , "Species"] はドロップされてデータフレームからベクトルになっています.一方で,iris["Species"] はデータフレームを返しますので,厳密には iris[ , "Species", drop=F] でアクセスした結果と同じになります.
データフレームの列をベクトルとして取り出す
使わなさ:★☆☆
データフレームはリストと同じように扱うことができるので [[]] で列をベクトルとして取り出せます.
data(iris)
class(iris["Species"]) # data.frame
class(iris[["Species"]]) # factor
指定列以外の列を取り出す
使わなさ:☆☆☆(みんな知ってる)
負の値を用いることで指定列以外の列を取り出すことができます
iris[ , -1]
番号ではなく列名で同じことをやりたいのですが,列名の場合は番号のときのような方法は用意されていないようです.比較的短いコードで書ける方法として,僕は以下のような方法を使っています.
data(iris)
# このコマンドは *エラー* となる
iris.sub <- iris[-"Sepal.Length"]
# 取り除きたい列がひとつの場合
index <- colnames(iris) != "Sepal.Length"
iris.sub <- iris[index]
# 取り除きたい列が複数ある場合
index <- setdiff(colnames(iris), c("Sepal.Length", "Sepal.Width"))
iris.sub <- iris[index]
正規表現で列を取り出す
使わなさ:☆☆☆(そこそこ役に立つ)
R にも grep が用意されているので,これで必要な列の番号を取り出します.次の例では,列名に Width を含む列のみを取り出します.複雑なパターンを書きたい場合は Perl 形式の正規表現を使えたほうが便利なので,常に perl=T を指定しています.
data(iris)
ls(iris)
# "Petal.Length" "Petal.Width" "Sepal.Length" "Sepal.Width" "Species"
index <- grep("Width", colnames(iris), perl=T) # 2, 4
iris[index]
ファイル読み込み
クリップボードから読み込む
使わなさ:☆☆☆(そこそこ役に立つ)
Excel などからクリップボードにコピーしたテーブルは,ファイル名に clipboard と指定するだけで読み込めます.
read.table("clipboard") # for Windows
read.table(pipe("pbpaste")) # for Mac
ヘッダーをそのまま読み込む
使わなさ:★☆☆
ヘッダーに変数名として使えない文字が含まれている場合,read.table は自動的に使える文字からなる文字列に変換します.check.names に FALSE を指定することで,自動変換を防ぎます.
read.table(file="input.txt", header=T, check.names=FALSE)
詳しくは hoxo_m さんの「read.table() でヘッダーをそのまま読み込みたい」をご覧ください.ヘッダーの自動変換の例や,自動変換をオフにするデメリットが説明されています.
重複した列を取り除く
使わなさ:☆☆☆(そこそこ使える)
次の表のような,重複した列を含むデータを読み込みたい場合があります("a" という名前の列が2回登場しています).例えば,複数のテーブルを列名を確認せずに結合した場合などに起こります.
| a | b | c | a | d |
|---|---|---|---|---|
| 1 | 4 | 7 | 1 | x |
| 2 | 5 | 8 | 2 | y |
| 3 | 6 | 9 | 3 | z |
重複した列はいらないので削除します.上の表をドラッグ&コピーして,以下のコマンドを実行します.
# check.names=F を指定することで,2回目の a が a.1 に変換されることを防ぐ
x <- read.table("clipboard", header=T, check.names=F)
# 重複のない列名の一覧を取得し,必要な列のみ取り出す
x.unique <- x[unique(colnames(x))]
連番のファイルを読み込む
使わなさ:★☆☆
R でもsprintf が使えるので,これでパスを生成してファイルを読み込みます.
for(i in 1:10)
{
path <- sprintf("input_%02d.txt", i) # input_01.txt, input_02.txt ...
read.table(file=path)
}
paste を使うよりも複雑な文字列を生成できるので,計算の途中経過を表示するときにも使えます.
高速化
rbind,cbind が遅い
使わなさ:★☆☆
ベクトルを返す関数をさまざまな条件で実行して,その結果をひとつの表にまとめる,という処理を行う場合があると思います.for などループを用いる場合,rbind で関数の戻り値を結合して表を作っているのではないでしょうか.
func <- function(x) { rep(x, 10) } # ベクトルを返す関数
x <- NULL
for(i in 1:10)
{
x <- rbind(x, func(i))
}
これは表が大きくなると急激に重くなります.おそらく結合するたびにメモリ確保と要素の全コピーが発生するのでしょう.以下のように表をあらかじめ初期化して,そこに代入するようにすると速くなります.
func <- function(x) { rep(x, 10) } # ベクトルを返す関数
x <- matrix(0, 10, 10)
for(i in 1:10)
{
x[i, ] <- func(i)
}
もちろん apply 系を使えるのであれば,それに越したことはありません.
func <- function(x) { rep(x, 10) } # ベクトルを返す関数
x <- sapply(1:10, func)
演算子
演算子のヘルプを検索する
使わなさ:★★☆
バッククォートで演算子を括り,ヘルプを検索します.
?`*`
配列の特定要素にアクセスする演算子 [] は [ だけで検索します.配列の特定要素を置き換える演算子は [<- で検索します.
?`[` # x[i]
?`[<-` # x[i] <- value
データフレームの特定要素にアクセスする演算子は別に定義されており,[.data.frame で検索します.データフレームの特定要素を置き換える演算子も同様で,[<-.data.frame で検索します.
?`[.data.frame` # x[i, j]
?`[<-.data.frame` # x[i, j] <- value
演算子 [] の [i, j, drop=F] の例のように,他言語の感覚では取りえないような値を取る演算子が存在するので,演算子のヘルプの検索方法を知っておくと役に立つ日が来るかもしれません.
関数として演算子を呼び出す
使わなさ:★★★
バッククォートで演算子を括り,演算子に渡す値を引数として指定します.単項演算子の場合は一つ,二項演算子の場合は二つ,引数に値を指定します.
`-`(10) # -10
`-`(10, 2) # 10 - 2 = 8
属性の置換関数を自作する
使わなさ:★★★
R では「関数の戻り値に代入を行う」という(ように見える)意味わからんコマンドを打つことがあります.
x <- 1:10
x.names <- sprintf("A%d", 1:10)
# 関数の戻り値に代入?
names(x) <- x.names
これはシンタックスシュガーです.内部的には以下のような処理が行われています.
# names(x) <- x.names と等価
`*tmp*` <- x
x <- `names<-`(`*tmp*`, value=x.names)
rm(`*tmp*`)
【2014/10/31 追記】
この コメントで kohske さんが教えてくださいました.以下のように書きます.colnames<- みたいな関数って自作できるんでしょうか?知っている方いたら教えてください.
`f<-` = function(object, value) {
paste0(object, value)
}
a = "hoge"
f(a) <- "orz" # f(a) = "orz" でもOK
a # hogeorz
関数名の後に <- を張り付けた関数を定義するだけです.最後の引数の名前は必ず value にします.
参考:R Language Definition > 3.4.4 Subset assignment
謎のエラーメッセージを読み解く
使わなさ:★★☆
データフレームの定義されていない列にアクセスしようとすると,次のようなエラーメッセージが表示されます.入力したコマンドとメッセージ中のコマンドが異なっていて謎ですね.
> data(iris)
> ls(iris)
[1] "Petal.Length" "Petal.Width" "Sepal.Length"
[4] "Sepal.Width" "Species"
> iris["sepal.length"]
Error in `[.data.frame`(iris, "sepal.length") :
undefined columns selected
これは演算子 [] が内部的には関数 [.data.frame として扱われているから起こる現象でしょう.演算子を関数として呼び出せることを知っていれば,この類の謎メッセージは読み解けます.
演算子を上書きする
使わなさ:★★★
バッククォートで演算子を括り,関数オブジェクトを代入します.
2 * 4 # 8
`*` <- function(x, y) { x / y }
2 * 4 # 0.5
オーバーロードではなくオーバーライドです.嫌がらせにどうぞ.
その他
列名チルダドット?
使わなさ:☆☆☆
SVM で分類器を作る方法をググると,次のようなコードが出てきます.
ksvm(Species~., data=iris)
初めて見る人からすると Species~. ってなんだよ(´・ω・`)という感じです.これは fomula というオブジェクトで,目的変数と説明変数の対応関係を定義するためのものです.目的変数 ~ 説明変数1 + 説明変数2 + 説明変数3 という風に書きます.
ただし説明変数の数が多いと,いちいちプラスで結合するの大変です.特に SVM の場合,説明変数の数(特徴ベクトルの次元)が数千とか数万とかになりますので,手動で列挙するのは現実的ではありません.そこで「目的変数として指定した変数以外を,すべて説明変数とする」という略記方法が用意されており,それが . です.
オブジェクトを保存する
使わなさ:☆☆☆(そこそこ使える)
特定のオブジェクトのみをファイルに保存できます.
data(iris)
model <- ksvm(Species~., data=iris)
save(model, file="svm")
オブジェクトを復元するときは load を使います.
load(file="svm")
機械学習のモデルは計算に時間がかかる場合もあるので,コードや結果に加えてモデルも保存しておくと再実験の際に役立つかもしれません.
【2014/10/31 追記】
hoxo_m さんから「saveRDS の方が便利」という情報をコメントでいただきました.
data(iris)
model <- ksvm(Species~., data=iris)
saveRDS(model, file="svm")
model.read <- readRDS(file="svm")
save load が保存前の名前で環境中へ強制的にリストアしてしまうのに対し,saveRDS readRDS は好きな名前を付けることができます.確かにこちらの方が使い勝手良いです.
気が向いたら調べる
以下,完全に自分用メモです.参考になりそうなページご存知でしたら教えてください(^q^)
- 変数のスコープ(レキシカルスコープ,ダイナミックスコープ)
- 環境とは?
- 代入演算子の違い(=,<-,->,<<-,->>)
- クロージャ
- カリー化
- デバッグ機能
- 型判定関数の違い(class,typeof,mode)
- クラスの種類(S3,S4)
- Generic function