はじめに
今回は、複数(主に2つ)のdata.frameを結合する方法についてまとめます。
実際に分析する際に、1つのdata.frameだけで完結することはあんまりなく、マスタデータの結合など複数のdata.frameを組み合わせて使う必要がありますが、またまたdplyrを使えば簡単にできます。
結合と言っても、結構色んな方法があるので、分かりやすいようにチートシートからイメージ図を拝借してきました。
1つのdata.frameを操作する場合は、↓の記事を参照してください。
dplyrを使いこなす!基礎編
dplyrを使いこなす!Window関数編
JOINの種類
JOINの種類は大きく分けて以下の3つあります。
| 種類 | 説明 |
|---|---|
| Mutating joins | マッチした行の列を、元のdata.frameに追加します。 |
| Filtering joins | マッチしたか否かで、元のdata.frameをフィルタリングします。 |
| Set operations | 2つのdata.frameを組み合わせます。 |
それでは、順番に見ていきましょう。
Mutating Joins
Mutating Joinsには、以下の4つの種類があります。
SQLで書くと、下表のようなイメージです。
inner join、left join等はSQLで使うことも多いのでイメージしやすいと思います。
| dplyr | SQLイメージ |
|---|---|
| inner_join | SELECT * FROM x JOIN y ON x.a = y.a |
| left_join | SELECT * FROM x LEFT JOIN y ON x.a = y.a |
| right_join | SELECT * FROM x RIGHT JOIN y ON x.a = y.a |
| full_join | SELECT * FROM x FULL JOIN y ON x.a = y.a |
dplyr0.5.0では、non-equi joinのサポートが予定されているようです。
結合の条件が=だけではなく、>、<等が使えるようになります。(たぶん
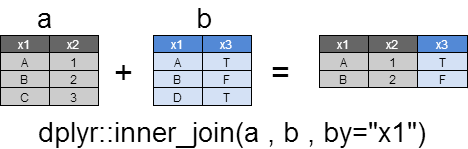
inner_join
inner_joinは、xとyのbyで指定した列がマッチした行のみを返却します。
もし、xとyの間で複数マッチする場合は、すべての組み合わせが返却されます。
> inner_join
function (x, y, by = NULL, copy = FALSE, ...)
a <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c(1,2,3)
)
b <- dplyr::data_frame(
x1=c("A","B","D") ,
x3=c(TRUE , FALSE , TRUE)
)
dplyr::inner_join(a , b ,by="x1")
x1 x2 x3
1 A 1 TRUE
2 B 2 FALSE
オプション
Mutating Joinsのオプションは、すべて同じなのでここでまとめて解説します。
といっても、2種類しかなく、copyは特殊ケースの場合のみなので、byの使い方が理解できれば十分です。
| オプション | 説明 |
|---|---|
| by | JOINのキーに指定する変数。 NULLの場合、Natural JOINつまり、同じ名前の列を使用してJOINする。 異なる変数でJOINしたい場合、by = c("a" = "b") のように指定する。 |
| copy | データソースをDBとし、かつxとyが異なるデータソースである場合、yをxのデータソースにコピーするか否か。 データ量が多い場合は、重い操作になるので、基本は同じデータソースを使用する。 |
a <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c(1,1,1),
x3=c(1,2,3)
)
b <- dplyr::data_frame(
x1=c("A","B","D") ,
x2=c(2,1,3),
x4=c(TRUE , FALSE , TRUE)
)
dplyr::inner_join(a , b ,by=c("x1","x2"))
a <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c(1,2,3)
)
b <- dplyr::data_frame(
x2=c("A","B","D") ,
x3=c(TRUE , FALSE , TRUE)
)
dplyr::left_join(a , b ,by=c("x1" = "x2"))
a <- dplyr::data_frame(
a1=c("A","B","C") ,
a2=c(1,1,1),
a3=c(1,2,3)
)
b <- dplyr::data_frame(
b1=c("A","B","D") ,
b2=c(2,1,3),
b3=c(TRUE , FALSE , TRUE)
)
dplyr::inner_join(a , b ,by=c("a1" ="b1" , "a2" = "b2"))
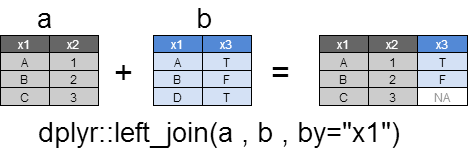
left_join
すべてのxの行を返却し、yとマッチする行はyの列も返却されます。
yとマッチしない行のyの列は、NAとなります。
xとyの間で複数マッチする場合は、すべての組み合わせが返却されます。
> left_join
function (x, y, by = NULL, copy = FALSE, ...)
a <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c(1,2,3)
)
b <- dplyr::data_frame(
x1=c("A","B","D") ,
x3=c(TRUE , FALSE , TRUE)
)
dplyr::left_join(a , b ,by="x1")
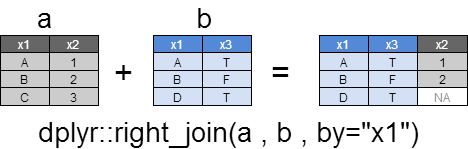
right_join
left_joinのxとyが逆になったイメージですが、個人的にはleft_joinを使うためほとんど使用しません。
すべてのyの行を返却し、xとマッチする列はxの列も返却されます。
xとマッチしない行のxの列は、NAとなります。
xとyの間で複数マッチする場合は、すべての組み合わせが返却されます。
> right_join
function (x, y, by = NULL, copy = FALSE, ...)
a <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c(1,2,3)
)
b <- dplyr::data_frame(
x1=c("A","B","D") ,
x3=c(TRUE , FALSE , TRUE)
)
dplyr::right_join(a , b ,by="x1")
x1 x2 x3
1 A 1 TRUE
2 B 2 FALSE
3 D NA TRUE
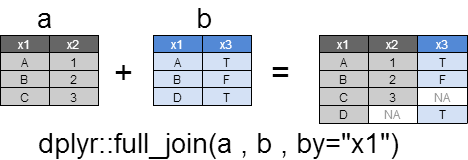
full_join
xとyのすべての行と列を返却します。
マッチしない箇所は、NAとなります。
> full_join
function (x, y, by = NULL, copy = FALSE, ...)
a <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c(1,2,3)
)
b <- dplyr::data_frame(
x1=c("A","B","D") ,
x3=c(TRUE , FALSE , TRUE)
)
dplyr::full_join(a , b ,by="x1")
x1 x2 x3
1 A 1 TRUE
2 B 2 FALSE
3 C 3 NA
4 D NA TRUE
Filtering Joins
xの行のフィルタリングに、yとマッチしたか否かを使用します。
| dplyr | SQLイメージ |
|---|---|
| semi_join | SELECT * FROM x WHERE EXISTS (SELECT 1 FROM y WHERE x.a = y.a) |
| anti_join | SELECT * FROM x WHERE NOT EXISTS (SELECT 1 FROM y WHERE x.a = y.a) |
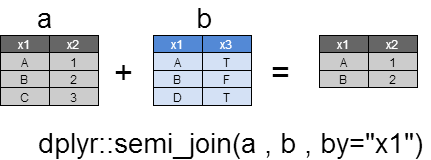
semi_join
yとマッチしたすべてのxの行を返却します。
返却される列は、すべてxの列のみでyの列は返却されません(inner_joinと違うところです。)
> semi_join
function (x, y, by = NULL, copy = FALSE, ...)
a <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c(1,2,3)
)
b <- dplyr::data_frame(
x1=c("A","B","D") ,
x3=c(TRUE , FALSE , TRUE)
)
dplyr::semi_join(a , b ,by="x1")
x1 x2
1 A 1
2 B 2
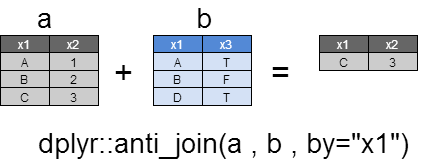
anti_join
yとマッチしなかったすべてのxの行を返却します。
返却される列は、すべてxの列のみでyの列は返却されません。
> anti_join
function (x, y, by = NULL, copy = FALSE, ...)
a <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c(1,2,3)
)
b <- dplyr::data_frame(
x1=c("A","B","D") ,
x3=c(TRUE , FALSE , TRUE)
)
dplyr::anti_join(a , b ,by="x1")
x1 x2
1 C 3
Set Operations
xとyは、同じ列から構成されるdata.frameである必要があります。
| dplyr | SQLイメージ |
|---|---|
| intersect | SELECT * FROM x INTERSECT SELECT * FROM y |
| union | SELECT * FROM x UNION SELECT * FROM y |
| setdiff | SELECT * FROM x EXCEPT SELECT * FROM y |
intersect
xとyの両方にある列が返却されます。
> intersect
function (x, y, ...)
y <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c( 1 , 2 , 3 )
)
z <- dplyr::data_frame(
x1=c("B","C","D"),
x2=c( 2 , 3 , 4)
)
dplyr::intersect(y , z)
x1 x2
1 B 2
2 C 3
union
xとyの重複がないユニークな行が返却されます。
> union
function (x, y, ...)
y <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c( 1 , 2 , 3 )
)
z <- dplyr::data_frame(
x1=c("B","C","D"),
x2=c( 2 , 3 , 4)
)
dplyr::union(y , z)
x1 x2
(chr) (dbl)
1 B 2
2 A 1
3 C 3
4 D 4
setdiff
yにはない、xの行が返却されます。
> setdiff
function (x, y, ...)
y <- dplyr::data_frame(
x1=c("A","B","C") ,
x2=c( 1 , 2 , 3 )
)
z <- dplyr::data_frame(
x1=c("B","C","D"),
x2=c( 2 , 3 , 4)
)
dplyr::setdiff(y , z)
x1 x2
1 A 1
まとめ
JOIN編は以上となります。
SQLに詳しい人は、SQLでできるJOINはほぼdplyrでもできることが分かったと思います。
inner_join、left_join、semi_join、anti_join辺りが使えれば、実務にはほぼ困らないのではないでしょうか。
dplyrの機能としては、DBとの接続周りを除けば、ざっくり解説できたと思うのでtidyrの解説に移りたいと思います。
dplyrとtidyrを使いこなせると、仕事が捗ること間違いなしです!