Binderでxvfbが簡単に使えることに気がついた
昨日は「ヘッドありアプリを含むワークフローでxvfbを使うのがむずい」とxvfbをディスる記事を書いておいて速攻で手のひら返しになります。
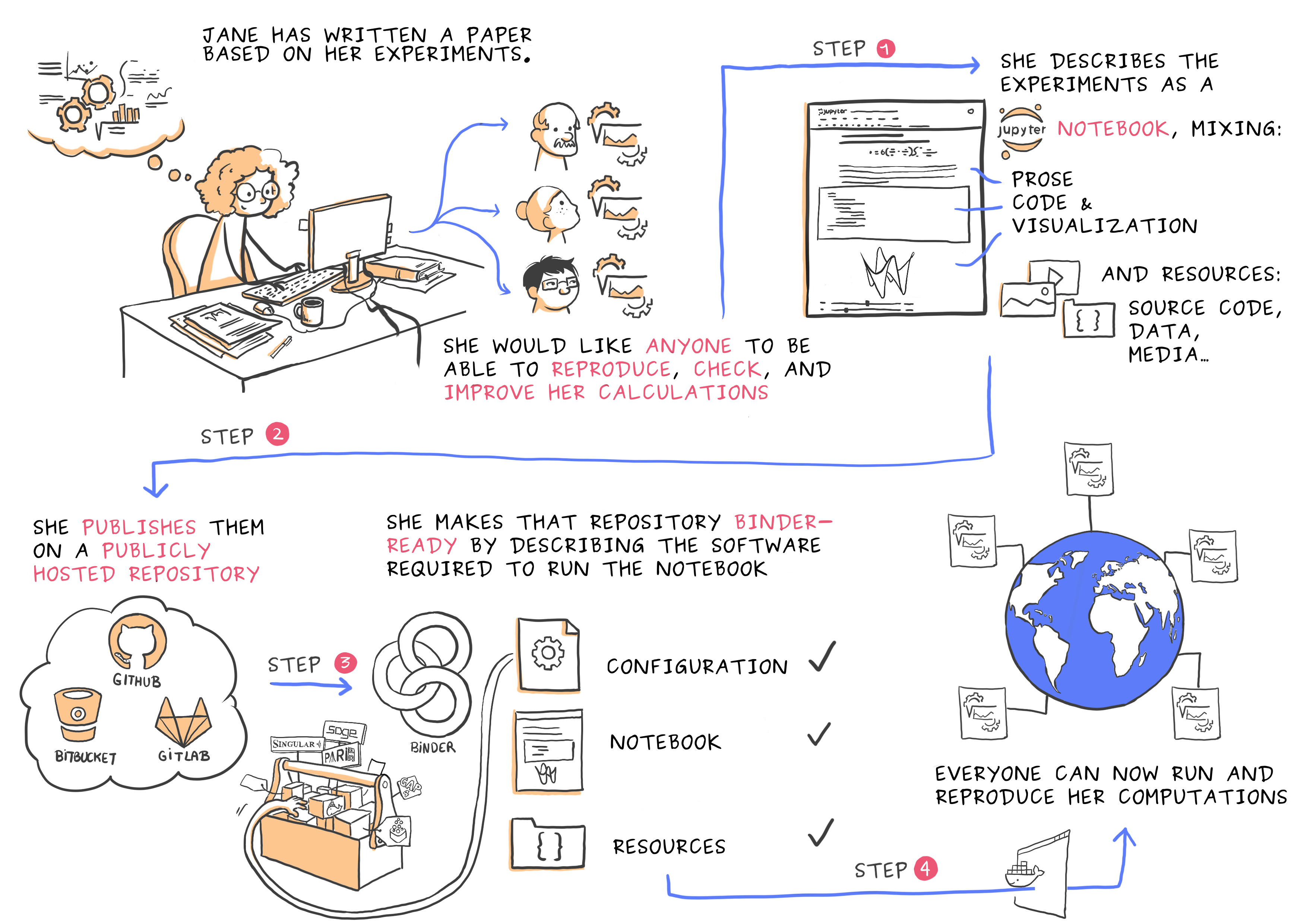
Binderという
- (GitHubなり何なりの)ノートブックリポジトリ(≒ワークフローリポジトリ)を
- 無償で誰もが利用可能なR, Python, Julia のお試しノートブックサーバ(JupyterだけでなくRStudioも利用可能)として公開する
サービスがあります。
(最近はshiny, bokehのダッシュボードアプリのお試し公開用の場としても有用そうです。)
Figure credit: Juliette Taka, Logilab and the OpenDreamKit project
ひさしぶりにBinderをいじってみると
- 簡単に設定ファイル書けるやん(Dockerfile要らんやん)
- Rも3.6が使えるやん(ちょい前は使えなかった,BioconductorはRのVersionが新しくないと使えない)
- xvfbもインタラクティブにシェルから実行できるやん
- CIでxvfbを使おうとするのに比べたら全然楽やん
となったのでBinder環境でxvfbを使う方法を紹介します。
X410+Dockerに対するRCy3のAuthorの反応
昨日のX410+Docker記事のRCy3の使い方を「どうすかね?」とRCy3のAuthorに聞いてみると
「うーん,何か違うんだよねえ。何つうの? "リンクを踏むだけで使える"?,"学生教育用"? 的な環境をイメージしてるんだけど...」
という反応で
「まあそうだわな」
となり,やはりBinderだわな,となってBinderでxvfbを使おうとするに至った次第です。
(そしてそれをアドベントカレンダーとして使いまわすに至った次第です。)
Binderでxvfbを使うには
まず当然ですがxvfbのDebianパッケージをaptでインストールする必要があります。
最近のBinderではほとんどの場合でDockerfileを書く必要が無く,設定するのが非常に簡単になっています。
下記のような環境構築用設定ファイルをリポジトリのトップかリポジトリのトップ/binderフォルダー
下に配置し,https://mybinder.org/ でリポジトリに対するビルドをかければ利用可能になる,という具合です。
- apt.txt : aptでinstallするパッケージ名を列挙します。
- environment.yml : condaでinstallするパッケージ名をYAMLで書きます。
- requirements.txt : pipでinstallするパッケージ名を列挙します。
- install.R : RパッケージをinstallするRスクリプトを書きます。
- runtime.txt : 使用するRのバージョン情報などを書きます。
- postBuild : Binder環境のビルド後に実行するシェルスクリプト
他にも設定ファイルはありますが,ほとんどの場合 上記で事足りると思います。
xvfbをインストールしたいのでapt.txtは下記になります。
ここではCytoscapeをxvfbといっしょに使うためにopenjdk-8-jdkもインストールします。
xvfb
openjdk-8-jdk
その他のファイルも加えるとこのリンク のような感じになります。
すると 
RStudioに行き着いたらTerminalタブで下記のコマンドを実行します。
xvfb-run bash ~/cytoscape-unix-3.7.2/cytoscape.sh
これでCytoscapeがバックグラウンドで立ち上がります。
Cytoscapeの画面を見ることはできませんがCytoscapeはREST APIを持っているのでRからワークフローを実行しスクリーンショットを保存することができます。
簡単な例として,RStudioのConsoleタブで下記のようなワークフローを試してもらえます。
- STRINGというデータベース用の追加機能(App)をインストールし
- タンパク質名で相互作用ネットワークを取得し
- 画像を保存
するって感じです。
installApp('stringApp')
string.cmd = 'string protein query query="SORCS2" species="Homo sapiens"'
commandsRun(string.cmd)
exportImage('string','PDF')
まとめると
- Binder簡単,有用,すばらしい
- Binderでxvfb簡単に使えました(使うの難しい,は思いこみだったかも)
- こんな感じでヘッドレス版が無いアプリを含むワークフローをBinder環境で実行できそう
という感じです。
以上。