ヘッドレス版が無いデスクトップアプリがワークフローに必要な場合
ワークフローの実行環境の構築はDockerで実現するのが主流かと思います。
またそういった実行環境はヘッドレスのアプリで構成されることがほとんどかと思います。
ですがヘッドレス版が無いデスクトップアプリがワークフローに必要な場合もあります。
そういったアプリは画面の出し先が無いと起動に失敗し,ワークフローを実行することができません。
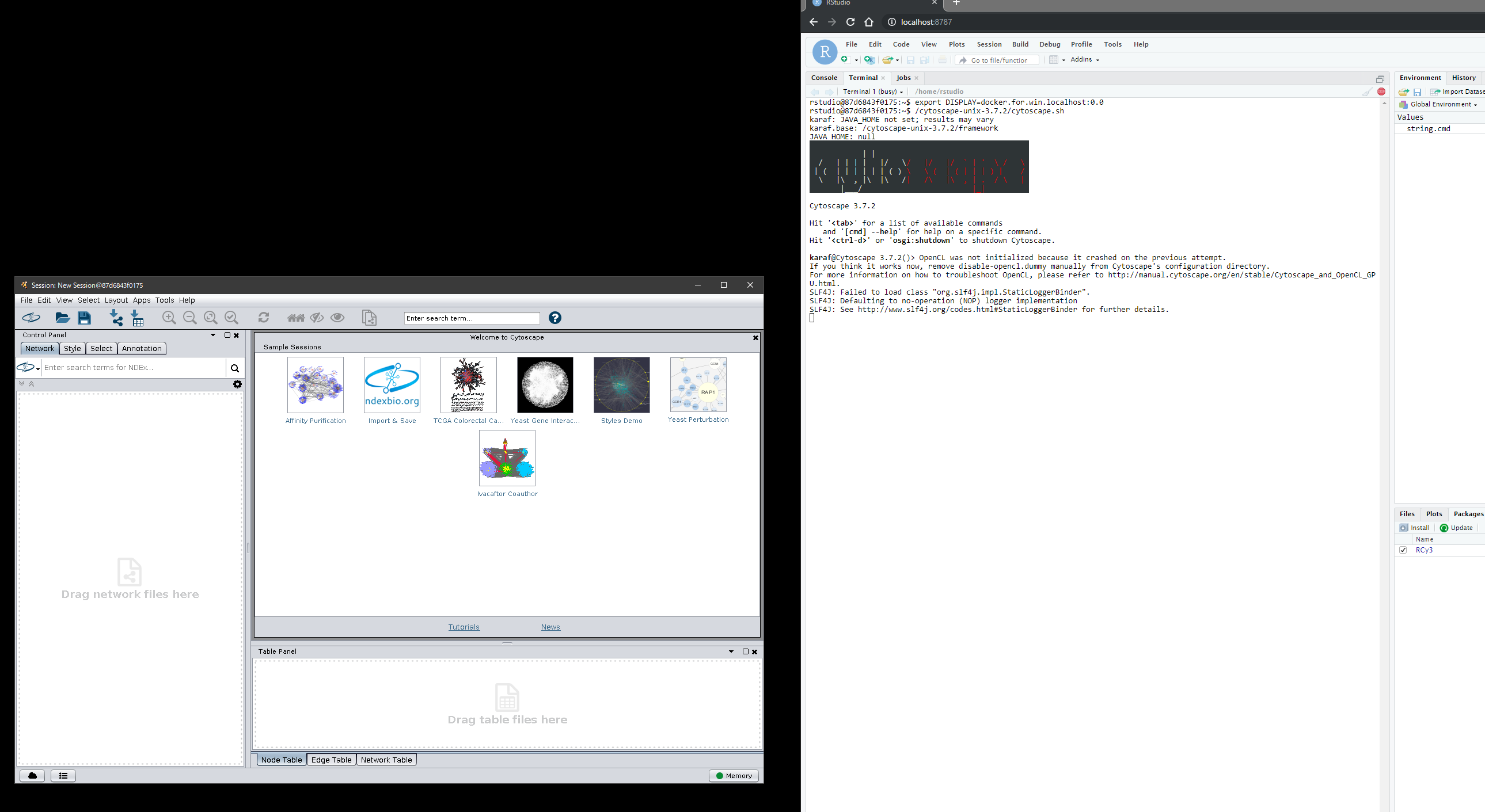
このようなデスクトップアプリの具体的な例としてCytoscape Desktopがあります。このアプリは起動時にREST serverにもなりGUI操作を行わずともPythonやRからコントロールできるのですが,前述の問題があるためDockerコンテナ中に押し込むことが難しいと思われていました。
(https://github.com/cytoscape/docker-cytoscape-desktop のようにseleniumベースのイメージでxvfbとVNC serverを使う手でがんばっていた時があったんですがやはり構成が複雑で,また利用する側としても操作が難しくプロジェクトとして行き詰まっていました。)
X410がよかった
そこでX410というWindows10のためのX Serverを使ってX窓を飛ばしてみると今までの苦労が嘘のようにシンプルなDockerfileで済んだので「みんなX410(買って)使おう」というのがこの記事の趣旨です。
ちなみにそのDockerfileは https://github.com/kozo2/rcy3-docker/blob/master/Dockerfile になります。
https://github.com/cytoscape/docker-cytoscape-desktop/blob/master/Dockerfile と比べるとかなりシンプルです。
X410をDockerといっしょに使ってみよう
例として
CytoscapeとそのRESTサービスをRから利用するためのpackage RCy3, そしてRStudioと全部コミコミのDocker image(前述のDockerfile由来)をX410と組み合わせて使う方法を示します。
下記の順に実行します。
- X410を起動
- Docker for Windowsを起動
- コマンドプロンプトなどで
docker run -e PASSWORD=hoge -p 8787:8787 kozo2/rcy3-dockerを実行 - ウェブブラウザで
localhost:8787を開く - ユーザー名
rstudioパスワードhogeでRStudio Serverにログイン - RStudioのTerminalタブに移動
- Terminalタブで
export DISPLAY=docker.for.win.localhost:0.0を実行 - Terminalタブで
/cytoscape-unix-3.7.2/cytoscape.shを実行
するとWindowsデスクトップにLinux版のCytoscapeの窓が現れるかと思います。
あとはRStudioでR Markdownに書いたRCy3を活用するワークフローを実行してめでたしめでたしという感じです。
この先は?
- Mac でも使えるようにするには?
- リモートのDocker実行環境に対して同じことをやるには?
- ワークフローのテストのためにCIでも動かすようにするには?
などいろいろこの先やりたいと思われることもあるかと思うんですが
とりあえずローカルのX410とWindowsで済むことであればこれでいいんではないかと,という感じです。
ここではネットワーク可視化ソフトを取り上げたんですが
バイオイメージインフォマティクスの分野でもX窓飛ばしが必要になるワークフローもあるんじゃないかなーと思っています。
なのでもしX410がお役に立ちそうであればぜひご購入ください。
以上。