はじめに
こんにちは。この記事はバイオインフォマティクス Advent Calendar 2020 の記事です。
本当は、今日までにRuby-htslibの開発を進めて、公開したかったのですが、あまり進んでないので、もうあきらめて、背景的なことをタラタラと書いていこうと思います。
この記事は未完成ですが見切りをつけて公開します。ruby-htslibの開発が進んだら更新しようと思います。
続編
Ruby-htslibの開発はその後も継続しており、2022年05月に新たなQiita記事を書きました。
Rubyに対するこだわり
R、Python, Perl, Julia, C++..世の中には星の数ほど素晴らしい言語があります。しかし私はRuby言語が好きです。私がたまたま最初に書けるようになったのはRuby言語だったからです。そこで、あたかもカルガモのヒナが最初に見たプログラミング言語を親だと思いこむように、Rubyが母国語になりました。(嘘です。Ruby言語の前にObjective-C言語とかに挑戦したことがありますが諦めました。Objective-C言語に比較すると、Rubyは単純で美しく見えました)
そんなRuby言語の特徴は、RやJulia等と比較すると、ほとんどあらゆるものがオブジェクトで構成され、関数のかわりにメソッドを使うことです。この世界では「データ」と「手続き」がひとまとまりの「オブジェクト」として存在しています。これは一般的にデータ解析では不利な性質だと思います。一方で「何かを道具を作りたい」という人を力強くサポートしてくれます。私も気がつくと、Rubyでツールを作ろうとしてしまいます。
Rubyでも次世代シーケンサーのデータを扱いたい
さて、Ruby言語はデータ解析にはあまり向いていないかもしれないと言いましたが、中には根強いファンもいて、BioRubyという生命情報用のライブラリもります。
- BioRuby: http://bioruby.org/
- リポジトリ: https://github.com/bioruby/bioruby
- ドキュメント: http://bioruby.org/rdoc/
けれども、今日の、何やらすごい研究のデータは、次世代シーケンサーの出力するデータを利用していることが多いです。次世代シーケンサーとは、DNAやRNAの配列を高速に読み取ることができる装置です。そういった次世代シーケンサーが出力するファイル形式、つまりSAM, BAM, VCF, BCF を扱うのはBioRubyでは難しいらしい、ということが私にもわかってきました。
誰か素敵なライブラリを作っていないのかな? と思って探しました。ひとつありました。Bioruby-samtoolsです。
- リポジトリ: https://github.com/helios/bioruby-samtools
- 論文: Bio-samtools: Ruby bindings for SAMtools, a library for accessing BAM files containing high-throughput sequence alignments
けれどもこのプロジェクトはSamtoolsをコマンドラインから呼び出すツールでして、細かくファイルを操作できるツールとはちょっと違う感じでした。(このプロジェクトは以前FFIを利用してSamtoolsのバインディングとして構成されていましたが、SamtoolsからHTSlibが分離された時にOpen3を使用するようになったそうです。Open3とは、プログラムを実行し、そのプロセスの標準入力・標準出力・標準エラー出力にパイプをつなぐライブラリです。つまり外部のコマンドを実行しているのであり、htslibのバインディングではありません。APIが安定したらFFIを利用してHTSlibのバインディングを作成して切り替えていく方針だったそうですが、開発者の方の余裕がなくて結局そのままになってしまったそうです。)
一方で、Python、Rといった言語では次世代シーケンサーのファイルを利用できるライブラリが存在します。そして結果として解析用のtoolやパイプラインも多く開発されています。
普通であれば、目的に応じてツールを選択するということで、柔軟に言語を切り替えて利用するべきです。私も頭ではわかっているのですが、なかなかそのような合理的な方法をとることができません。モグラのように、なんとかしてRuby言語でもこれらのファイルを扱う方法はないだろうかと考えはじめました。
SAM/BAMというフォーマット
SAM ファイルや BAM ファイルといった形式は、リードのアラインメントを表したファイルです。次世代シーケンサーはFASTQというリードのファイルを生成します。このファイルを、bwaやSTARといったアライナーでリファレンスゲノムにマッピングした結果のファイルがSAM/BAMファイルです。
これは少し奇妙なファイル形式だと思います。私たちの身の回りには、SQLiteなどもっと一般的なデータベースがあります。タブ区切りテキストや、MS Excelといったファイル形式もあります。しかし生命情報の世界ではそれらのファイルではなく、SAM/BAMファイルが好んで利用されているようです。
@HD VN:1.6 SO:coordinate
@SQ SN:ref LN:45
r001 99 ref 7 30 8M2I4M1D3M = 37 39 TTAGATAAAGGATACTG *
r002 0 ref 9 30 3S6M1P1I4M * 0 0 AAAAGATAAGGATA *
r003 0 ref 9 30 5S6M * 0 0 GCCTAAGCTAA * SA:Z:ref,29,-,6H5M,17,0;
r004 0 ref 16 30 6M14N5M * 0 0 ATAGCTTCAGC *
r003 2064 ref 29 17 6H5M * 0 0 TAGGC * SA:Z:ref,9,+,5S6M,30,1;
r001 147 ref 37 30 9M = 7 -39 CAGCGGCAT * NM:i:1
↑ 仕様書(hts-spec)に載っているSAMファイルの例。見慣れないヘッダー、フラグ、タグがついている。フラグは、リードがマッピングされているかいないかといった非常に重要な情報が含まれている。
- SAM/BAMに関連するリンク
フラグは2進数で表記されており、パット見ても何を意味しているのか人間にはわかりません。samtools flag コマンドを使うと、フラグの意味を表示してくれます。
samtools flag 2064
# 0x810 2064 REVERSE,SUPPLEMENTARY
↑ samtoolsでフラグの意味を表示したところ
| Integer | Binary | Description |
|---|---|---|
| 1 | 000000000001 | ペアリードがある |
| 2 | 000000000010 | 両方マップされている |
| 4 | 000000000100 | read1 はマップされていない |
| 8 | 000000001000 | read2(相方) はマップされていない |
| 16 | 000000010000 | read1 は逆相補鎖にマップされている |
| 32 | 000000100000 | read2(相方) は逆相補鎖にマップされてる |
| 64 | 000001000000 | read1 である |
| 128 | 000010000000 | read2 である |
| 256 | 000100000000 | 複数個所にマップされている |
| 512 | 001000000000 | マッピングQVが低い |
| 1024 | 010000000000 | PCR or optical duplicate |
| 2048 | 100000000000 | supplementary alignment (e.g. aligner specific, could be a portion of a split read or a tied region) |
↑ フラグの表
BAMファイルはSAMファイルの圧縮形式で、gzipと互換性のあるフォーマットだそうです。BAMファイルは圧縮されても検索しやすくなるように工夫されているようです。CRAM というよく似たファイル形式もあります。こちらはBAMよりも圧縮率が高いファイル形式のようです。これら特殊な形式のファイルを、Samtoolsというコマンドラインツールを使っていろいろ操作していくのです。
実際のところ、この初心者に対する嫌がらせのようにも思えるファイル形式が使われている理由は、歴史的な経緯と、人類の技術的限界によるところが大きいのだと思います。RNA-SeqのBAMファイルは小さいものでも数GBあったりしますし、全ゲノムシーケンスなら数十GBにおよびます。これだけでも十分に巨大ですが、SQLiteやTSVにするとその数倍以上のサイズになるでしょう。そのような巨大なファイルをエクセルで検索することは当然難しくなります。解析するのも大変でしょうし、保存するスペースを確保するのも大変でしょう。サイズの問題、そして可能な限り高速に検索するために、特殊なフォーマットが開発されたということなのでしょう。(興味がある方は下のエントリーをお読みください)
-
Forum: Why does bioinformatics use a special format like a BAM file instead of a database like SQLite?
- 煽り気味の質問にも親切に答えてくれるBiostarsのみなさん
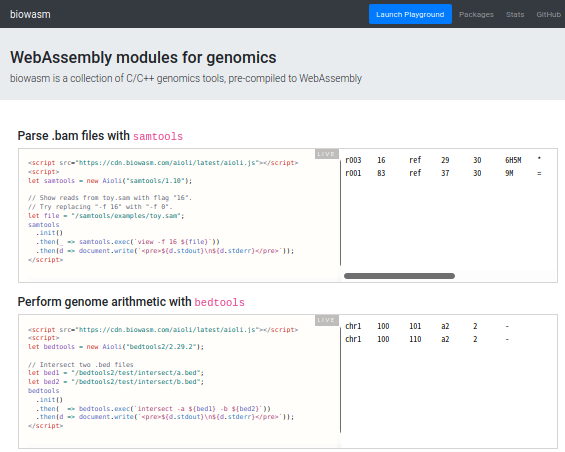
しかしながら、コンピュータの性能は上がり続けています。いつまでも同じ状況が続くとは限りません。10年後、20年後には、現在の配列情報の処理は、スマートフォンで十分であるという時代が来るかも知れません。たとえばbiowasmとかいうプロジェクトがあります。これは、なんとWebAssemblyを使うことでブラウザ上でsamtools bedtools bowtie seqkit 等を動かしてしまうというようなものだそうです。
↑ ブラウザ上でゲノミクスのツールを動かす野心的なプロジェクトbiowasm
現状ではこのようなツールは役に立たないかも知れません。世間的には巨額な予算を獲得して、ワークフロー言語を使ってクラウド環境やスーパーコンピューターなど多数のPCを操作して、数の暴力により効率的に大量に検体を処理して新事実を発見しないとなかなか評価の対象にならないのかもしれません。しかしそんな勇ましいことを言っても、そんなことができるのは現実的には地球上でもごく少数の人だけでしょう。ですから、そのように物事をとらえてしまうと、どんどんと暗い気持ちになってしまうのではないでしょうかね。(少なくとも私は暗い気持ちになります)
ですから、そういった世知辛さとは遠いところにあるツールに触れたり眺めたりしている時間こそ、心あたたまる大切な時間だという気がいたします。
SAM/BAMのアイコンが見当たらない話
話がとびましたが、さらに話がとびます。
SAMファイルやBAMファイルのアイコンというものが見つからないことが気になっています。バイオインフォ用アイコンセットというものがないか結構探しました。コマンドライン上の操作に強い人はそんなの気にならないのかもしれませんが、個人的には気持ちよく作業をするためにはファイル・アイコンの存在はとても大事なことだと考えています。
もしもバイオインフォ用の素晴らしいアイコンセットを知っている方はコメント欄で教えてほしいなと思います。多分世界のどこかにはそういうものを作っている人がいるけれども、見つかってないだけだと思います。まあ自分でデザインして、身銭を切ってデザイナーの方に仕上げてもらい、それを著作権フリーで公開するという最終手段もありますが。
SAM/BAMの仕様書を読む
- SAM/BAM仕様書: https://github.com/samtools/hts-specs
は、Githubのリポジトリとして公開されています。日本語の情報があまりないので、詳しい内容を知りたい時は原典にあたる必要があります。しかし、PDFファイルであるため、Google翻訳がなかなか効きません。なので、PDFから文字起こしをして、Qiitaに記事として投稿しました。
↑のサイトをGoogle翻訳することによって、ある程度日本語でSAM/BAMファイルの仕様を把握することができます。
VCF形式とBCF形式
VCF/BCFは突然変異の形式です。こちらはまだ表面的にも理解できているとは言えないので、今後の勉強が必要です。まずはSAM/BAM形式のように仕様書を機械翻訳して全体の概要をつかもうと考えています。
Samtoolsの内部ではHTSlibが動いている
さて、このSamtoolsはC言語で作られていて、Samtoolsの内部ではhtslibというライブラリが、いわばエンジンのように動いていることがわかりました。HTSLibは、もともとSamtoolsの一部だったものをHTSLibとして切り分けたようです。そして、Python言語やJava言語など、いろいろな言語でhtslibに対するバインディングが作成されていることがわかりました。PythonやRのような言語でも、SAM/BAM/VCF/BCFを直接扱うようなツールは、htslibを間接的に利用していることを知りました。
↑ 各種言語におけるhtslibバインディング
面白いところはRustやNimといった、新しい静的型付き言語において熱心にバインディングが開発されていることです。これは、速度に対する需要が大きいのだろうなと思います。ゲノムのデータは膨大なので、Pythonのようなスクリプト言語よりも、RustやNim、Goといった言語を使って高速に処理することが求められているのでしょう。
上から5番目のJulia言語のバインディングは日本の方が開発されているようです。中をみてみますと、C言語のヘッダーファイルをパースしてバインディングをある程度自動生成しているようでした。
 図:HTSLib.jl には独自のバインディング[自動生成スクリプト](https://github.com/bicycle1885/HTSLib.jl/blob/master/scripts/translate_exported_functions.jl)が付属している
図:HTSLib.jl には独自のバインディング[自動生成スクリプト](https://github.com/bicycle1885/HTSLib.jl/blob/master/scripts/translate_exported_functions.jl)が付属している
そこで私は考えました。Ruby言語も htslibのバインディングを作成すれば、ある程度は次世代シーケンサーのファイルが扱えるようになるのではないかと。
こういう時、PythonやR、Juliaなど流行の言語であれば、世界の素晴らしい方が作ったバインディングを探してどれを使えばいいか悩むのかも知れませんが、Rubyでは解決策は一つしかないのであれこれ悩む必要はありません。
自分で作るか、作らないかです。
HTSLibの論文が出たようです
また話がずれますが、偶然にも、HTSLibの論文が12月16日にアップロードされました。これを受けて、いろいろな言語でhtslibのバインディングの作成が活性化するかも知れません。

↑ 論文中の図

↑ Nim言語のバインディングhts-nimの作者が「便利そうダナー」とコメントしているツイッター
C言語のRubyのバインディングを作るということ
では、どうやってRubyのバインディングを作っていけばいいのだろうか、ということになります。方法は大きく分けて2通りあります。ネイティブ拡張を利用する方法と、FFIを利用する方法です。ネイティブ拡張を使う方法は、C言語をゴリゴリ書いていく必要があります。私はC言語を扱えませんのでこの方法を採用することはできません。もうひとつはFFIを利用する方法は簡単で、あまりC言語に詳しくなくても、Rubyバインディングを作ることができます。
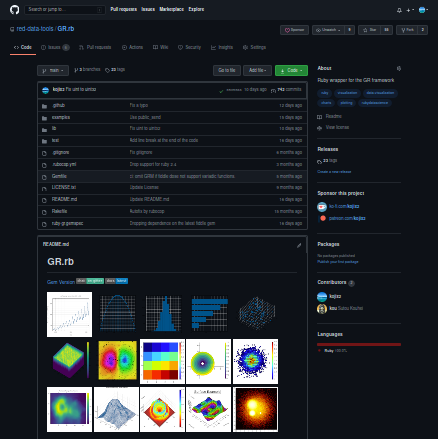
私は1年前から、GR.rbというRubyでグラフを描出するツールを作っています。
↑ Rubyでもきれいなグラフや図が描出できるGR.rb
Julia言語では事実上のデファクトスタンダードとなっているGRというグラフ描出ライブラリのバインディングです。FFIはFiddleというRuby標準ライブラリを使っています。Rubyバインディングを作るのは、配線をつなぎ合わせてプラモデルを作るようなもので、そこまで高度な技術がなくてもできてしまいます。
先行している素晴らしいバインディングのコードを観察してパターンを学び、同じように作れば、ほとんどの場合うまく動作するものが作れます。このプロッティングツールも、Rubyでグラフを描出できるライブラリが足りないため、自分で作り始めたものです。
RubyコミュニティではAndrew Kaneさんという方が、2年ほど前から精力的にC言語の機械学習のツールのバインディングを作成されており、RubyでC言語のバインディングをFFIを利用して作ろうとする方は、Ankaneさんのリポジトリを観察すると、とても参考になると思います。
Rubyのhtslibバインディングを作る意味
次に、htslibのRubyバインディングを作る意義は何かという話になります。
開発する個人的なメリットとしては、ruby-htslibを作っているうちに、SAMやVCFのファイル仕様を読む必要が出てくるため、自然にフォーマットに対する知識がいろいろ身につくのではないかという気がします。また、これから先、次世代シーケンサーを超えて、さまざまな新しいモダリティが出現する時がかならず来ると思います。その場合も、いろいろなフォーマットが出現し、それを処理する基盤となるツールやライブラリが作成されて、各種言語からのバインディングが作られるという人間の営みは大きく変わりません。共通の課題に対していろいろな人々が頑張っている場を学ぶことには大きな意義がある気がします。
つぎに、Rubyよりももっとデータ解析に適した言語がいろいろ存在する中で、Ruby言語でバインディングを作る社会的な意味を考えます。これはやってみないとわかりません。RubyはActiveRecordに代表されるように、データをオブジェクトに変換して扱うという分野では使いやすさに定評があります。ですから、作り続けていくうちにHTSLibのRubyバインディングの良さのようなものが現れるとしたら、一つそういった方面があると思います。もちろんRuby言語を利用した解析ツールや、解析パイプラインのようなものが作れればそれに越したことはないですが、これはなかなか実現が難しいのではないかと考えています。いずれにせよ、プログラミング言語の世界でも多様性は大事であると考えます。
基本的な方針
さて、ここから先は、技術力がない人が、どうすればruby-htslibを作れるだろうか?という現実的な話になります。
最初のポイントはFFIのライブラリ選びです。Ruby言語では、Ruby-FFIと、Fiddleの2種類のFFIバインディング作成のためのライブラリがあります。どちらも歴史あるライブラリです。Ruby-FFIはより高機能なサードパーティのライブラリです。FiddleはRubyの標準ライブラリですが機能は簡易的で普及度はあまり高くありません。
- Ruby-FFI: https://github.com/ffi/ffi
- Fiddle: https://github.com/ruby/fiddle
今回Ruby-FFIを採用することにしました。Ruby-FFIは多くのプロジェクトで採用されており、Ruby配列とCポインタの相互変換、ネストされた構造体などを扱うのが得意です。Ruby-FFIのあまりよろしくない点としては、Ruby標準ライブラリではないため追加のライブラリのインストールが必要になることと、将来的にRubyの構成が大きく変わった時に変化についていけないリスクがあること、日本人開発者がいないため、日本語で質問しにくいという点があります。
↑まずはRuby FFIの全体像を得るために、Google翻訳を使って FFI wikiの全訳を作成しました。
さて、低レベルのバインディングでRuby-FFIを利用しても作るのが大変だというところは2点ほど思いつきます。
一つはRuby-FFIがC言語の bit field に対応していないところです。FFIを bit field に対応させましたというGemもあるのですが、公開日が古く、そのまま使うわけにはいかないと思っています。
もう一つ難しい点は、メモリ管理です。
Rubyではガーベッジコレクションが参照されなくなったオブジェクトを自動的に削除します。そのタイミングでFFIもメモリを開放するようです。しかし、何かの理由でうまく動作せず、開放するべきメモリが開放されなかったり、逆にGCが勝手にメモリを開放してしまうような場面が出現するかも知れません。そうなったとき、その問題を発見したり、デバッグしたりするのは私には大変難しいだろうなと考えています。
そういったものに当たったときには、人の助けを借りるしかないと思います。
自動的にCのヘッダーファイルをパースしてバインディングを生成できないのか?
FFIのバインディングを作る場合、通常はC言語で書かれたヘッダーファイルの関数を、正規表現などを用いて置換を繰り返して、RubyのFFIモジュールに書き換えるのではないかと思います。けれども自分で一つ一つ関数をメソッドに書き換えるケースも悪くはありません。一つ一つのメソッドを手書きして実装するのは、時間と手間をかければ、誰でもやることができます。単調な作業に見えますが、実は大きなメリットがあります。それはライブラリのAPIを写経することで、ツールの全体像を深く理解できるようになることです。
けれども人間が手作業でバインディングを作成する場合、どうしても書き損じやタイポが生じます。HTSLibは現在でも開発が続いており、今後も関数は追加されたり、変更されたりします。HTSLibのバージョンが上がれば、Rubyバインディングも微修正を加えなければなりません。私は手作業は好きですし大事だと思っていますが、それでけでなく、何らかの自動化も考えていきたいところです。
一つの方針は、Julia言語のケースのように自分で変換器を作成することです。この場合、正規表現による置換を利用したり、Ruby言語で実装されたC言語のパーサを使って、バインディングを自動生成するスクリプトを作成すれば良いでしょう。もう一つは、RustのHTSlibバインディングのように、すでにある誰かが作った変換器を利用することも考えられます。
Ruby-FFIには自動的にバインディングを生成するプロジェクトがありましたが開発が事実上中断しています。最終更新日が古いのでそのまま使うことはできないでしょう。
また、次のようなプロジェクトもあるようです。こちらはうまく動作するようですので、利用できないかどうか検討しています。
nmコマンド
関連する方法として、共有ライブラリのバイナリファイルを直接観察するという考え方があります。
nm htslib.so
とすると、共有ライブラリのバイナリファイルから直接関数の名前を見ることができます。
実装できる関数をなるべくカバーするという目的では、このようなバイナリを直接見る手段もどこかで役に立つかも知れません。
Fat Gemの問題 共有ライブラリをパッケージに含めるべきかどうか?
共有ライブラリをパッケージ含めるメリットは、バージョンの調節が容易になることです。しかしGemのサイズが大きくなりますし、ユーザーによってはGemの中にバイナリファイルが含まれていることは好まず、自分で用意したhtslibを使いたいと考えるはずです。また、クロスプラットフォームにすることを考えた時、gemをプラットフォームごとに分割しない場合は、Windows, Mac, Linuxの3種類のバイナリを梱包しておく必要があります。(詳しくないのでもっとよい方法があるかも)
共有ライブラリを探す/指定する方法には、環境変数でパスを指定する方法のほか、Pkg-configを利用して、ファイルを見つける方法などがあります。htslibもpkg-configに対応しているっぽいです。しかしAnacondaなどでhtslibを入れた場合などの動作確認が必要と思われます。
高レベルのAPI
ffiを使ったバインディングは、2種類のAPIを作成します。
低レベルのAPIは、FFIモジュールを作成して、特異メソッドとして用意します。FFIモジュールの特異メソッドを呼び出すと、htslibの関数が呼び出されます。
高レベルのAPIでは、よりRubyらしいAPIを提供します。Rubyらしさとは何か、というのは難しい問題です。NGS関連ファイルを無理なくオブジェクト指向で扱えることが目標になると思います。まだしっかり理解できていないのでhts-pythonを移植して、それを改良することを考えています。
bamファイルを検索するような使い方では、ActiveRecordsのAPIが参考になるかもしれません。私はActiveRecordの使い方は詳しくないので、いろいろ勉強する必要があります。
高レベルのAPIを考えるためには他の言語のツールとかをよく使ってみてhtslib全体を理解する必要がありそうです。私の実力はまだまだで、ここからかなり勉強と経験が必要な気がするので、年単位で実現していけばなあと思っています。
現在の進捗状況
今の段階では、低レベルのAPIを主に手書きで作り終えたところです。
次のようなサンプルで、bamファイルを読み込むことができます。
require 'htslib'
bam_path = File.expand_path('../assets/poo.sort.bam', __dir__)
htf = HTS::FFI.hts_open(bam_path, 'r')
idx = HTS::FFI.sam_index_load(htf, bam_path)
hdr = HTS::FFI.sam_hdr_read(htf)
b = HTS::FFI.bam_init1
nuc = { 1 => 'A', 2 => 'C', 4 => 'G', 8 => 'T', 15 => 'N' }
cig = {
0 => :BAM_CMATCH,
1 => :BAM_CINS,
2 => :BAM_CDEL,
3 => :BAM_CREF_SKIP,
4 => :BAM_CSOFT_CLIP,
5 => :BAM_CHARD_CLIP,
6 => :BAM_CPAD,
7 => :BAM_CEQUAL,
8 => :BAM_CDIFF,
9 => :BAM_CBACK
}
10.times do
HTS::FFI.sam_read1(htf, hdr, b)
p b[:core].members.zip(b[:core].values)
p name: b[:data].read_string,
flag: b[:core][:flag],
pos: b[:core][:pos] + 1,
mpos: b[:core][:mpos] + 1,
mqual: b[:core][:qual],
seq: HTS::FFI.bam_get_seq(b).read_bytes(b[:core][:l_qseq] / 2).unpack1('B*')
.each_char.each_slice(4).map { |i| nuc[i.join.to_i(2)] }.join,
cigar: HTS::FFI.bam_get_cigar(b).read_array_of_uint32(b[:core][:n_cigar])
.map { |i| s = format('%32d', i.to_s(2)); [s[0..27].to_i(2), cig[s[28..-1].to_i(2)]] },
qu
ご覧のように、低レベルのAPIだけ使って無理やり表示しているのでごちゃついています。
まだまだですね…。先は長いです。これを実行すると次のように表示されます。
[[:pos, 3289], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 2], [:flag, 133], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3289], [:isize, 0]]
{:name=>"poo_3290_3833_2:0:0_2:0:0_119", :flag=>133, :pos=>3290, :mpos=>3290, :mqual=>0, :seq=>"GGGGCAGCTTGTTCGAAGCGTGACCCCCAAGACGTCGTCCTGACGAGCACAAACTCCCATTGAGAGTGGC", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
[[:pos, 3292], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 3], [:flag, 133], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3292], [:isize, 0]]
{:name=>"poo_3293_3822_1:0:0_0:0:0_76", :flag=>133, :pos=>3293, :mpos=>3293, :mqual=>0, :seq=>"TTCGATGCGGGACCCCCAAGACGTCGTCCTGACGAGCACAAACTCCCATTGAGAGTGGCACATGATTTCC", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
[[:pos, 3293], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 2], [:flag, 69], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3293], [:isize, 0]]
{:name=>"poo_3294_3861_2:0:0_2:0:0_2d7", :flag=>69, :pos=>3294, :mpos=>3294, :mqual=>0, :seq=>"TGGGGACCGTGTGACTATCAGAAGGGTGGGGTCAGCTTGTTCGATGCGGGACCCCCAAGACGTCGTCCTG", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
[[:pos, 3298], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 2], [:flag, 69], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3298], [:isize, 0]]
{:name=>"poo_3299_3808_0:0:0_4:0:0_2e0", :flag=>69, :pos=>3299, :mpos=>3299, :mqual=>0, :seq=>"CCCAAGACGTCGACCTGAGGAGCACAAACTCCCAATGAGAGTGGCACATGATTTGCCCAACCATACCATT", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
[[:pos, 3303], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 2], [:flag, 133], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3303], [:isize, 0]]
{:name=>"poo_3304_3813_0:0:0_2:0:0_2f5", :flag=>133, :pos=>3304, :mpos=>3304, :mqual=>0, :seq=>"GGACCCCCAAGACGTCGTCCTGACGCGCACAAACTCCCATTGAGAGTGGCACATTATTTCCCCAACCATA", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
[[:pos, 3311], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 3], [:flag, 133], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3311], [:isize, 0]]
{:name=>"poo_3312_3825_0:0:0_1:0:0_6f", :flag=>133, :pos=>3312, :mpos=>3312, :mqual=>0, :seq=>"TTGTTCGATGCGGGACCCCCAATACGTCGTCCTGACGAGCACAAACTCCCATTGAGAGTGGCACATGATT", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
[[:pos, 3317], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 2], [:flag, 133], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3317], [:isize, 0]]
{:name=>"poo_3318_3847_3:0:0_1:0:0_142", :flag=>133, :pos=>3318, :mpos=>3318, :mqual=>0, :seq=>"CTATCAGAAGGGTGGGGGCAGCTTGTTCGATGCGGGACCCCCAAGACGACGTCCTGACGAGCACAAACTC", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
[[:pos, 3319], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 2], [:flag, 69], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3319], [:isize, 0]]
{:name=>"poo_3320_3822_3:0:0_6:0:0_333", :flag=>69, :pos=>3320, :mpos=>3320, :mqual=>0, :seq=>"TTCGATGCGGGACCCCCAAGACGTCGTGCTGACGAGCACAACCTCGCAATGAGAGTGGCACAAGATTTGC", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
[[:pos, 3321], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 2], [:flag, 69], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3321], [:isize, 0]]
{:name=>"poo_3322_3847_1:0:0_0:0:0_2cb", :flag=>69, :pos=>3322, :mpos=>3322, :mqual=>0, :seq=>"CTATCAGAAGGGTGGGGGCAGCTTGTTCGATGCGGGACCCCCAAGACGTCGTCCTGACGAGCACAAACTC", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
[[:pos, 3327], [:tid, 0], [:bin, 4681], [:qual, 0], [:l_extranul, 2], [:flag, 69], [:l_qname, 32], [:n_cigar, 0], [:l_qseq, 70], [:mtid, 0], [:mpos, 3327], [:isize, 0]]
{:name=>"poo_3328_3840_1:0:0_2:0:0_211", :flag=>69, :pos=>3328, :mpos=>3328, :mqual=>0, :seq=>"AAGGGTGGGGGCAGCTTGTTCGATGCGGGACCCCCAAGACGTCGTCCTGACGAGCACCAACTCCGATTGA", :cigar=>[], :qual=>"2222222222222222222222222222222222222222222222222222222222222222222222"}
pooというのは、wgsimを利用して作られた適当なbamファイルです。
Rubyアソシエーション開発助成
このプロジェクトは2020年度のRubyアソシエーション開発助成に選ばれました。
この記事は以上です(未完)