はじめに
謎の生物 プー それは、太古から存在するかもしれない 謎の生物です。
wgsimとはなんじゃらほい?
wgsimとはバイオインフォ界の巨星 Heng Liさんが作ったNGSのシミュレーターです。
2011年のソフトですが問題なく動きます。
インストール
Biocondaを導入している場合は
conda install wgsim
〜 謎の生物プー 〜
謎の生物プーは、まったくの気まぐれから生み出されました。
神は生物を創造します。(進化など言語道断です。)
あなたは神なので、ちょっとしたウイルスと同じぐらいの配列、5000塩基の生物を創造しましょう。この生物の名前を「プー」と名付けました。
プーのリファレンス配列は、ランダムで生成しています。(筆者は絶滅危惧種のRuby派なのですが、ほかのプログラミング言語でも同じことができるでしょう。)
genome = Array.new(5000){ %w[A C T G].sample }
puts '>poo chr1'
genome.each_slice(80) do |l|
puts l.join('')
end
このスクリプトを実行すると、こんな感じで配列が出力されると思います。
これが謎の生物プーのリファレンスゲノムだッ!!!
>poo chr1
AAAGCGCTGGATCATGCTTTAAGGCTTAGTTTTATGGCCCTAAACACGGAGACTGAAGCCGCTTGAGTCCGGTTTCAGAG
AGATTTGGTTTCACCCACAACTGCCAGCATATAATGGACCGTCGGCCGTACACGTTATGAGCCTGGCAGGGGTTACTACG
CGCCACGGGCTAGAGACCTAGTGGCGAAACGTGAAGGGCAATTATGGAATCCCGTACGCGCAATAAATGTCTACGCATTC
CCGGGGGGTGTTGTAGTCCCACGGCCCCGGACCTGCATTGGTCCAAATTCTCTTCGGTGCATATTAGCTTCATCGGAACC
GGCCTTTCTAATTCAGAACTGTTCGGTCATACACTATCTTCCAACACGCCGTGCACGAGTTGCACGGAGATCTAGTTGTT
ACTCGACGCGCGTACGACCGAGGCGGGGCTTAACACGCGAGTAGAAGCCCTGGGTTATCCACCACTTAATTTTGTAAAAA
GCGCTTTCAGTACTACACGGCTTGACCGTAGCTCTGTCGGACTTGTGACCCCTTCCGGCGAACATTAATTGTACCCCTTC
TTCTGACGGATTGGTTAGTAGTCCAAGCAGTCTTCACCCCAGGAGCACTTCTTCTTGCCGCGAGGCCATTCAGTCCAGTT
CGTGATGGCGGGGGTACCGATGCAAAAGCTCGTCTTTCTTGAGGCATCCACAGCCAGAGGCGGGGTATTTACGAGATTAA
GTTCTATCATTGCACTAGACAAAGCAAGTACAGCTCGGCAAAAAGAAACTCGTGGGCGCGTCTTGACCTGCAGCTAATAC
TGACTAGCATCCGTAGATATTACCGATCGTGCCGCACAAGGGAAATCTCGAGGCTGACAACGTCTTTGCCAAGGCAGTGG
CCGTGTCTTCTCGCCAGGGGTAAAGGTGCCGCTGGACTAGCGCTAACTTAGTGTCGTAATATCTCCATATGGACGATTTC
CCCGATCTTACCGGCTAGCCGCTGGTTGTATGCCGCGCCCAGGCCACCGAGTCTGACATGTTCCAAGGGCCTCATGTGAA
AGCGATGTGCTGATCCAGCATGGCCTTCAGCCTCACAAGACTGAGAGTTGGTGTAGAAACCCGGTTTGTTGGTAGTATGA
TGCTTAAAAATCGGTTTAATTTAGCTTGGTTAGATTTGAACGTGAAGAGCCTATGTCCAAAGACCAACTAACCTACTCAG
ACTAAGATTTACCGATTTCTCGTTTTGACTGAGATACGTAGTTAGCTATACGGGGCGTCGCCATCCTCTACCCCTTGAAT
ATGATTCGTAAGTGATGCCATTCTCGTGCTCGTGCGCTATGAATAGCAATGTCACCACAATAGTGCCCGGTGGCCGACAA
TCCGAGACCATGTCCGTCCTCATTCATGCGTAGTTAGTAGTGTATCGACTCACCTGGAGATGACCTCTATGTCCTGCTGC
GGTACGGCACGGCAGGATCTTTCTAGAAACATCGGCGATGCCCCAGAGGTTCTCTAAACTGTGTCATCTTTTTCTGCGGT
GCGTGGCTGGTCAAACCATTCAAATATATGAGAGTCATGCGCTAGCTCCCGGTAAAACCCCACAATAACGGGATTTGGGG
TTGAAAATGCGAGGCAGCGCACATGGATCAGGCCATTTAGGGTGTGATTGGTATGTTCCGAAAAAGCTGATGAGAACTAT
GCCCCGTTTACTTTGCCAGTAGAAAGAGTAGGAAGTAGTGTAACGCGGGCACCGGAAAACGACCGACACGATACTTTCCC
TTGTGCTGGAATTGTTTGATATATTAGGACGAGAATAGTTCTCTCCATTCCGCCGGCGTATAGACCAGGCCATGCCGTTC
TATTTCCAAGTTCCTTTGAAGGATCGGTGTTATTTCGGATGTCGTCGAGTAGTCGTGGGGCGTTAGGGTCTAAGCGACCA
GTTGTAGCTAATCGGGTGTCCTCCACTAACTGCTATTAGCATAGTACACCATGCTGCTTCAACTCATTTGGGATTTGCAC
TACTCTAGGCCAGAGATACAGATGCAAGCCGACTTAGCTGCACTAGCCAAGATTGAGCCCTAGGGCGTAGCGACCCTGAG
AATCCTGGCTAGCCCCCCTAGCCCCGGAAACTGCCCGTGTTGCTCTCGAAGCAGTTAGTAATCATCGTAATTTTGTCAAG
ATGGCCGTAGAGTGCAACCTGCAAGGCTGCAGGGTGTGTTCGGCATATTAATGCGGACAGTGCAGTGGACAAAGAGTCTC
GGAAGCGACGTGTATACAGTAGTGAGTAAATTGTTAAGCTTACTATCGCAGGAACTTCTGATCTGCCCGACCGTCGAGAT
CGAGTCTAGTAACAAGATTGCAGTCCTCAAGACTAGAAGTACCTAACTCCCTCAAATGCGAGTCCCAACAACTCATCCTC
ACCTCTGGAATCTCACCTCTAAGCATGACAGGCTAGGACCATTTAACATAGTTACAGTGACCTGATACGTAACGCCTCGA
AAGGAAAGGCGCGCCTAGGCAGCAATTGCAACGAACTCGGACGTCAGTACTCAACCAACCGAGTTATTTCAGCGTCGCGA
ATCTAATGCATCTTCCTGCTTAAGACGTCGTACTCCGGTGTCACCCTTCGACTAACTTAAAATCGCGCATTGGCAAGCAT
ACCTCTAATACCGGATCGCTCCCCTCAGGTTTACTTGATGAGCGCTGAACCCAACCATAATTTCGGCAAGGTAATCGCTT
ATCGTTCCACCCAGGCGTGAATAATTCATGCCCAATGCAGGTGACCGTATCTCAGGGAGATATAAGTGAAAGTTATGTGG
AAGGTATTGTAGTACATTTCAGGGTTCATCTAAGCTTCAGGACCACAGATGCAGAAGACGGGTTAAAGGACATGTCGGCA
CGAAGACATATCTTGTGGATCCCTATTCAGTGCCCGTGACTCAACAGATAGACTCATTTTCTCCTTTGGCGTGTCCGCAG
GTAAGATAACTTCAGTGTCCGAGCGGCGGAGTGGGTGTTCACGTCGTCGAGTGTCCATTTCGATTGGGGATGTTGGCTCG
CATACCACACGACCCTTTAGTGGACGGTCTAGTTTGTATACTCCCCGATTGGACTTTACCCGCGAAGCTAGCACTGTAAA
TATTGCCCCGGAGTGGCGGGATACGTTGGATATGCCTATACTGGACCGCAGCCTATTACATCAGATCGGCTTACCGCCAA
TCTTAGTGCTCAGCTACCCAGAGCTGTATCCATGGACATAGGCGCTATGACAAGCAGTACTGTAAAGCAGGGACAGCTTT
ACTATGTCGACAGCCGGCGTACGTCGTTGGGCTACCGCCGGCCTAGTTCCTAGTCAGTTCCGAGGCATAATCTACAAGTC
TAAGTAGGGCGCCATGATGTCACGAGACTTATAGGAGTCCACCGGCAAGTGCGTTGACGTAAATACATATGTGTATGCAG
CACTTTTGACAGGCCCGTAGATTTTGCACAAACAGCCGGGTGTGGTTCAAAAAGGGTGTTGCGGTCAGCCGCAGCAGCGC
AATTGTAACCAATCTACCTCCCGTTTGCCGACCGCTCGCGCAGATTCATGTAGGAACCGCTAGTTCCCCGTCCGCTACAT
TTCAATGGGCTTTTGCATGTCCTATCGCGATACCATCTCTGGTGGCGATGATATTTTATCAGGTTGCATTGGCCCAGTCC
ATCTTCGCGGCCGATTGGGCACGCCATTAGTTGCTCTCTGCGCAATCGCAGCTATACTAATGGTATGGTTGGGGAAATCA
TTGGAGTGCATTATTCCGCCCTCTTAACCTGTTGTGAACGGAACCGCAGTACGGTAGATTGCAAAGTCCAAGGTTTCTGG
ATTGTCTCTGAAAATGAAAGTTCTGCTCTGTCAAGTGATAAACGGAACGTCCTAGGAGGCGCCTACCGCGTTTAGAGAAG
TAACCCACGATATCTCGAACAAAAAATCAAAAGCGGTGTAGGTACAAAACGTATCGCTTACATCCCGTGAAGCTTAGTAG
GACGCACCCTTCATCTCTCAGGGGGTCGCGTACACGATGTCCGTCTTTTATCGTGCCACGTGTACAAGTAGGGGTGCAGA
AATAACTGTACTAATGCAACGTTTCAGTTCTTCTGATGAACATCTCACACGGTTGTCCCCTCTAATATTAGGATTATCGG
CCTAGACGTGTTATGCACGCCGACCCCTTTCCCTAAGTGCTTCCGCAATGAAGTCGCGTATTTGTAACACACATTGGGTA
TGAATATAACTGTCCCTCCTGGAATAAGGAGTGCGCTTATCCTTTCGGACGTCAGAGCCCAGGGAGGAGACGCGTCAGAT
GCCATAGCCTATCGGGATCCGTCGTGCTAGTAGAACCAAAGTAACGGTGCTGTTAAATCGTTCCCTCAGCAGTCCGGGGT
TGACAGGTACAGTGTTTCCAACTACTAGAGGATCTCGCTGCTATAGAACGATACCACTGCGGGGGACACCGCCACTTTTC
GAGCTGTGGGGCCAGAAGTGCTGTCTGGATAATTCGGTAGGCATCAGCGGACAGCGGAGCTCGTAAGTGCCATGCACTTG
TAGCCCATTACGATGAGATCGGTTCCGCCAACTCCGTCAGCACAGGTATCCACGAGGAGTCACCGTCCGGCATGACGCAG
GACGGGGGCGGTGGAAATAATCGAGGCGGATACTCTTCATTTGTAGGCGCCAGCTACAGCGGTCCGGCGGGTCAATACCG
CATCCGCAGACATAATTGACCTCACTCAGCTCGCACGAACCTCCCTACTCTTTTCTACTCGAGGGTGCCTGATCAGAGAA
CGGAAGACGCTATAGGCAAGGCGGGCACGCCTGGAATACAGTCCCTTTCCGAATATTCACACCCGCTACCTATGTCGTAG
CGTTGCCGGGCCCGTTATCCAGACCGAACCCGTTGTCTGTTATAACATATTGAGTAATGGGCAGACAAACCCGTGTAGAG
TCCTCTCGGTTCCACATATCGAAGAGGGCGATCCGTACGA
ながい。
poo.fa として保存します。
wgsimを動かす 〜プーの全ゲノムシークエンス〜
さて、謎の生物プーは ヒツジ用の歯磨き粉から発見されました。ところでヒツジってハミガキするんでしょうかね?さあ、私は詳しいことはわかりません。ただ、プーは若いヒツジがこっそり使用していた歯磨き粉から発見されたと報告されてますので、それを信じることにしましょう。学名は、顕微鏡でみた様子がぷるぷるしていることから、プル・プルプルスと言うそうです。(今適当につけた)
さてプーを発見した人はその後人生に山あり谷ありプーに対する興味を失って、その後50年間プーは放置していました。しかし天と地の気のめぐり、赤道の気温の上昇、町内会長のおののき、ほのかな春のおとづれ、会長と社長の和解など、たまたまいろいろな偶然の成り行きがあって、プーが全ゲノムシークエンスされる時がやってきました。さて、あなたは神なのでゲノムシークエンスの結果をwgsimで求めます。
wgsimの実行に先立って、オプションをみてみます。
wgsim --help
Options: -e FLOAT base error rate [0.020]
-d INT outer distance between the two ends [500]
-s INT standard deviation [50]
-N INT number of read pairs [1000000]
-1 INT length of the first read [70]
-2 INT length of the second read [70]
-r FLOAT rate of mutations [0.0010]
-R FLOAT fraction of indels [0.15]
-X FLOAT probability an indel is extended [0.30]
-S INT seed for random generator [0, use the current time]
-A FLOAT discard if the fraction of ambiguous bases higher than FLOAT [0.05]
-h haplotype mode
ところどころ私にはよくわからないオプションがありますが(-s が何の標準偏差なのか、-A とか -h がどう動くのかとか)、それは後で勉強することにしましょう。(しない)
ここではリード数を1000本にします。
wgsim -N 1000 poo.fa poo_1.fq poo_2.fq
実行すると突然変異が表示されます。poo_1.fq と poo_2.fq が生成されたと思います。簡単ですね。
poo 977 A W +
poo 1210 T W +
poo 1655 G K +
poo 1874 T K +
poo 2043 C S +
poo 2378 G T -
生成されたfastaファイルの中味を見てみましょう。
head -n 12 poo_1.fq
@poo_418_960_2:0:0_1:0:0_0/1
TCGTCGAGTAGTCGTGGGGCGTTAGGGTCTAAGCGACCAGTTGTAGCTAATCGGGTGTCCTCCACTAACT
+
2222222222222222222222222222222222222222222222222222222222222222222222
@poo_624_1182_3:0:0_1:0:0_1/1
CTTAGGACATAGGCTCTTCACGTTCAAATCTAACCAAGCTAAATTAAACCGATTTTTAAGCATCATACTA
+
2222222222222222222222222222222222222222222222222222222222222222222222
@poo_2608_3166_3:0:0_0:0:0_2/1
GTCCAGTATAGGCATATCCAACGTATCCCGCCACTCCGGGGCAATATTTACAGTGCTAGCTTCGCGGGTA
+
2222222222222222222222222222222222222222222222222222222222222222222222
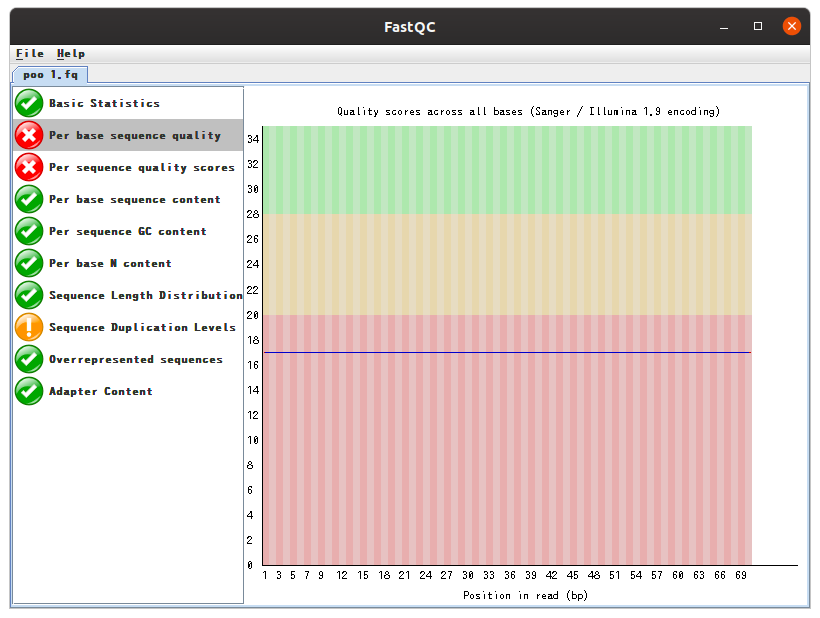
まあ、普通リードの後半の方がクオリティスコアが落ちてきたりすると思いますが、そうはなっていませんね。
Fastqcでsequence qualityを見て確認してみましょう。
クオリティが一定(しかも低い)ことがわかります。
一般的には下図のようにリードの後方にいくにつれて、クオリティが下がっていくはずです。(画像は Fastqcの公式ホームページから https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)

まあそこのへんは昔のソフトなので仕方がないということで。
BWAを使ってマッピング
さて、全ゲノムシークエンスなので、bwaでマッピングします。なになに。minimap だと? ここは10年前の世界観で、wgsimで遊ぶ記事なんだぞ、ロングリードなんて存在しないんだ!!おっと取り乱してしまいました。神は取り乱さないように気をつけなければなりません。
まずはともあれindexの作成します。
bwa index poo.fa
プーのゲノムサイズは小さいので一瞬で実行が完了すると思います。
それではbwaをかけてみましょう。(bwa も Heng Li氏の作ったツールです。まさに神といって良いでしょう。)
bwa mem poo.fa poo_1.fq poo_2.fq > poo_wgs.sam
さて、SAM → BAM、BAM → sorted BAM、インデックスまでつけてやりましょう。
samtools view -bS poo_wgs.sam > poo_wgs.bam
samtools sort poo_wgs.bam > poo_wgs.sorted.bam
samtools index poo_wgs.sorted.bam
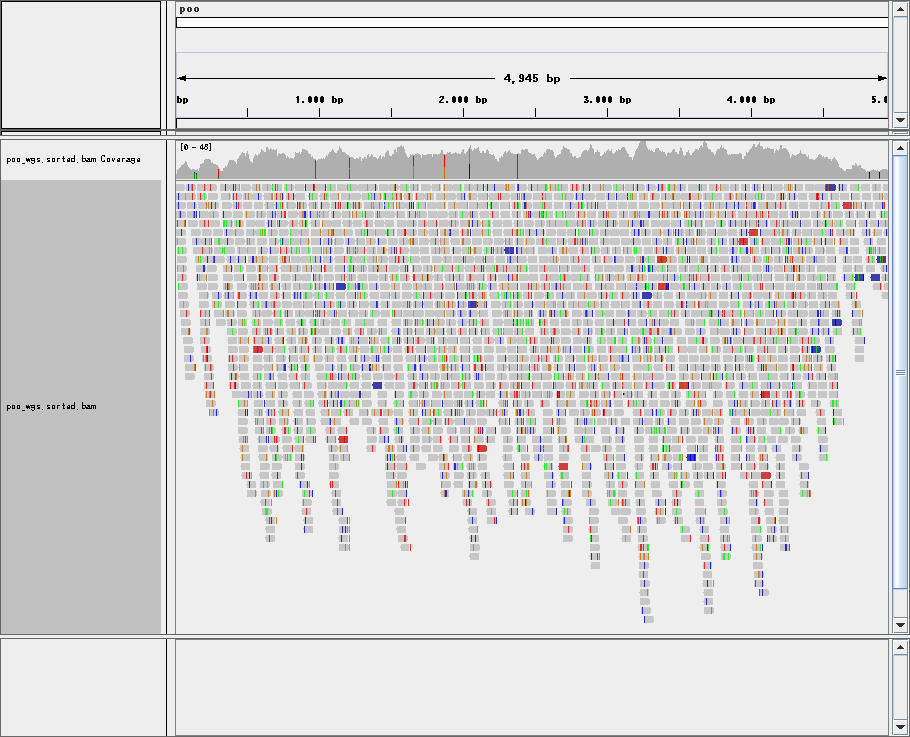
IGVでリードの様子を見てみよう
IGVで、poo.fa をリファレンスゲノムに設定し、poo_was.sorted.bam を開きましょう。
うおぉぉぉ!できたよ!できたよ兄さん!(うるさいな)
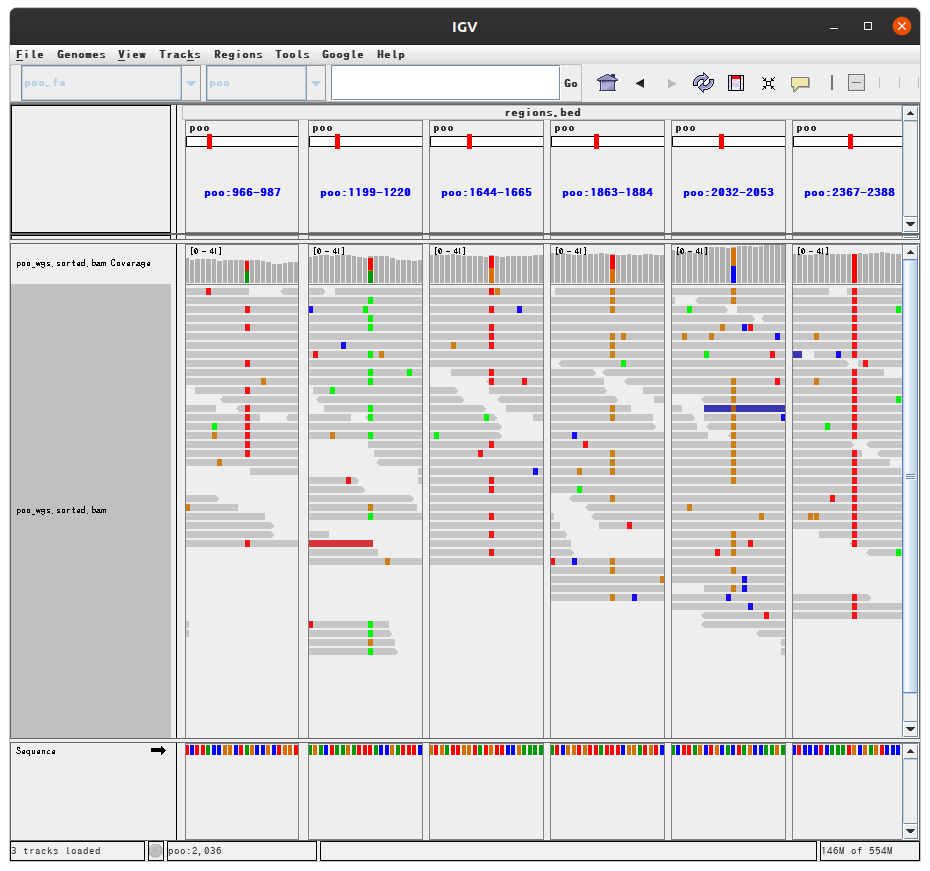
さてIGVには、実は便利なスプリットビューの機能がついています。これを利用して、突然変異の場所を観察してみましょう。
うおぉぉぉ!兄さん(略)
ちゃんと突然変異が検出されていますね。
バリアントの検出
人間の検体を使う場合はGATKとかで頑張るのかもしれないが、pooは適当な生物なので、bcftoolsとかでコールしておけばOK。
bcftools mpileup -f poo.fa poo_wgs.sorted.bam | bcftools call -mv -Ob -o poo.bcf
などとして、
bcftools view poo.bcf
とすればよし。
おわりに
さて、謎の生物 プー に関する記事を気まぐれで書いてみましたが、思いのほか書いていて楽しいのと、勉強にもなることが判明しました。プーシリーズ、また気が向いたら書くかも。