はじめに
掲題の通り。必要に迫られてやってみた。
やってみよう。
前置きはさて置きやってみよう。えいやっと。
from rdkit import Chem
from rdkit.Chem.Draw import rdMolDraw2D
from IPython.display import SVG
from io import BytesIO
from PIL import Image
from cairosvg import svg2png
import argparse
def generate_image(mol, highlight_atoms, highlight_bonds, atomColors, bondColors, radii, size, output, isNumber=False):
print(highlight_atoms)
print(highlight_bonds)
print(atomColors)
print(bondColors)
view = rdMolDraw2D.MolDraw2DSVG(size[0], size[1])
tm = rdMolDraw2D.PrepareMolForDrawing(mol)
option = view.drawOptions()
if isNumber:
for atom in mol.GetAtoms():
option.atomLabels[atom.GetIdx()] = atom.GetSymbol() + str(atom.GetIdx() + 1)
view.DrawMolecule(tm, highlightAtoms=highlight_atoms, highlightBonds=highlight_bonds,
highlightAtomColors=atomColors, highlightBondColors=bondColors, highlightAtomRadii=radii)
view.FinishDrawing()
svg = view.GetDrawingText()
SVG(svg.replace('svg:', ''))
svg2png(bytestring=svg, write_to=output)
return
def main():
parser = argparse.ArgumentParser()
parser.add_argument("-i", type=str, required=True)
parser.add_argument("-o", type=str, required=True)
parser.add_argument("-a", type=str, action="append")
parser.add_argument("-b", type=str, action="append")
args = parser.parse_args()
atom_hilights = {}
bond_hilights = {}
radii = {}
with open(args.i, "r") as f:
string_mol = f.read()
mol = Chem.MolFromMolBlock(string_mol)
if args.a:
for atom in args.a:
print(atom)
datas = atom.split(",")
atom_hilights[int(datas[0])] = (int(datas[1]), int(datas[2]), int(datas[3]))
radii[int(datas[0])] = 0.20
if args.b:
for bond in args.b:
datas = bond.split(",")
bond_hilights[int(datas[0])] = (int(datas[1]), int(datas[2]), int(datas[3]))
generate_image(mol,list(atom_hilights.keys()), list(bond_hilights.keys()), atom_hilights, bond_hilights, radii, (400, 200), args.o, False)
if __name__ == "__main__":
main()
使い方
$ python HilightChemExample.py -i ../sample/2-Acetylbenzothiophene.mol -o
usage: HilightChemExample.py [-h] -i I -o O [-a A] [-b B]
-i で入力MOLファイル
-o で出力画像
-a でハイライトさせたい原子を指定。molファイルの中での原子番号を0起点で指定し、その後続けてRGB色の指定をカンマ区切りで指定。
-b でハイライトさせたい結合を指定。molファイルの中での結合番号を0起点で指定し、その後続けてRGB色の指定をカンマ区切りで指定。
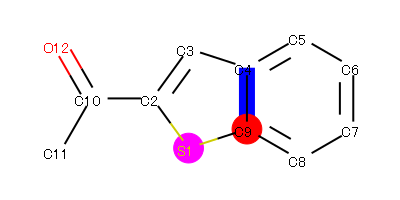
例
こんな指定を指定をすると...
python HilightChemExample.py -i ../sample/2-Acetylbenzothiophene.mol -o ./hoge.png -a 0,1,0,1 -a 8,1,0,0 -b 9,0,0,1
こんな感じででる。原子番号は結果を分かりやすくするために表示させているが、プログラムをいじれば表示されなくすることはできる。