概要
とある公共データベースから、データのスクレイピング方法を調べた時のメモ
※ スクレイピングする場合はサイトに負荷をかけないようにしましょう。
動機
- 論文に記載されているデータで機械学習を試したかった。
- WEB画面の検索機能を用いることで、必要なデータを取得できることが分かったが、件数がそこそこあったため、コピペミス等防止のため、自動的に取得したいと考えた。
- そのサイトではREST APIも用意されていたが、仕様やWEB画面と同じ結果が得られるかを調べる手間が惜しかった。
ゲットしたいデータ
蛋白質構造データベース PDB から、コロナウィルスのタンパク質を阻害する化合物(リガンド)を取得したい。
以下、データを手動で取得する場合の手順を示す。
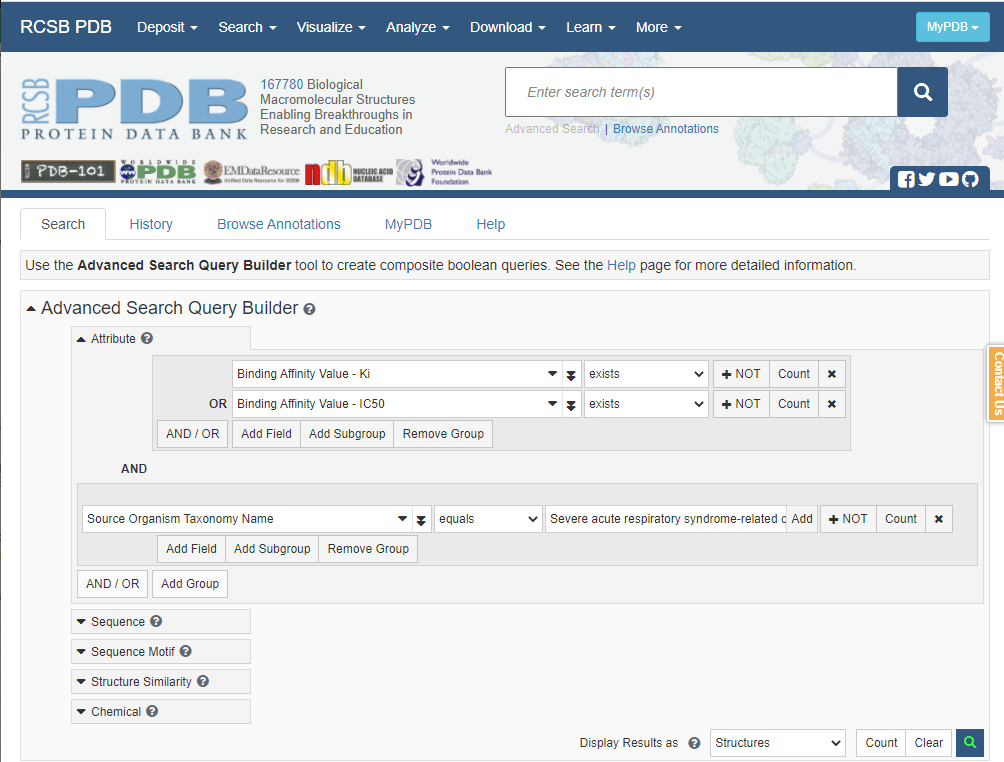
1. 検索画面
検索画面 (https://www.rcsb.org/search/advanced) で条件を入力し、検索ボタン(虫眼鏡アイコン)をクリックする。
※以下は、生物種が「コロナウィルス」であり、阻害データとしてKiまたはIC50が存在するデータを検索するという条件を入れている。
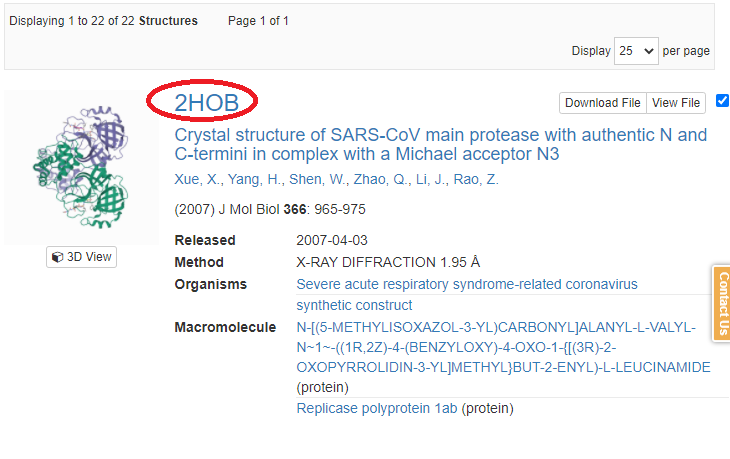
2. 検索結果一覧
検索結果としてタンパク質の構造一覧が表示されるので、取得したいIDをクリックする。
ちなみに、今回、全ID分について、以降の画面で得られるデータを収集したい。
3. 蛋白質構造詳細画面
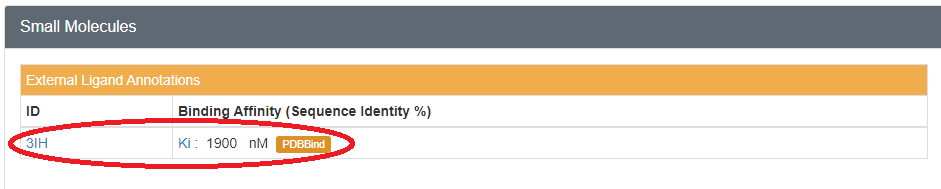
蛋白質構造詳細画面より、リガンドのIDとBinding Affinity(阻害定数、単位)を取得する。
その後IDをクリックしする。
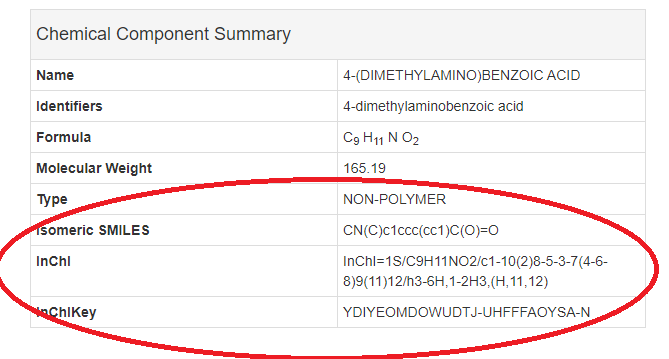
2. リガンド詳細画面
リガンド詳細画面より、SMILES、Inchi等の構造情報を得る
事前調査
スクレピングに必要となる画面の遷移の仕方、データ表現の仕方がHTMLレベルでどうなっているか確認した。
- 最初の検索画面における検索処理は、ブラウザの開発モードで通信を確認したところ、検索条件をJSONリスエストでPOSTし、検索結果がJSONレスポンスで受け取っていた。通信と画面の表示はJavaScriptで実施していると思われる。
- 蛋白質構造詳細画面では、"binding_row_N" (Nは数字)というidがついたtrタグ内のtdタグの中に、リガンドのURLや阻害データが格納されていた。
- リガンド詳細画面では、"chemicalIsomeric"等のidがついたtrタグ内のtdにてSMILES等の項目が出力されていた。inchi, inchiKeyも同様であった。
やってみる
事前調査が終わったのでやってみた。Scrapyにも興味があったため、Scrapyを使う場合とScrapyを使わない場合(生Python)の2パターンでやってみた。
方法1 生Python
コード
import requests
import json
import time
import lxml.html
import argparse
import csv
def get_ligand(ligand_id):
tmp_url = "https://www.rcsb.org" + ligand_id
response = requests.get(tmp_url)
if response.status_code != 200:
return response.status_code, []
html = response.text
root = lxml.html.fromstring(html)
print(tmp_url)
smiles = root.xpath("//tr[@id='chemicalIsomeric']/td[1]/text()")[0]
inchi = root.xpath("//tr[@id='chemicalInChI']/td[1]/text()")[0]
inchi_key = root.xpath("//tr[@id='chemicalInChIKey']/td[1]/text()")[0]
return response.status_code, [smiles, inchi, inchi_key]
def get_structure(structure_id):
structure_url = "https://www.rcsb.org/structure/"
tmp_url = structure_url + structure_id
print(tmp_url)
html = requests.get(tmp_url).text
root = lxml.html.fromstring(html)
binding_trs = root.xpath("//tr[contains(@id,'binding_row')]")
datas = []
ids = []
for tr in binding_trs:
print(tr.xpath("@id"))
d1 = tr.xpath("td[position()=1]/a/@href")
if d1[0] in ids:
continue
ids.append(d1[0])
status_code, values = get_ligand(d1[0])
ligand_id = d1[0][(d1[0].rfind("/") + 1):]
if status_code == 200:
smiles, inchi, inchi_key = values
item = tr.xpath("td[position()=2]/a/text()")[0]
item = item.strip()
value = tr.xpath("td[position()=2]/text()")[0]
value = value.replace(":", "")
value = value.replace(";", "")
value = value.replace(" ", "")
value = value.replace("\n", "")
print(value)
values = value.split(" ", 1)
print(values)
value = values[0].strip()
unit = values[1].strip()
datas.append([ligand_id, smiles, inchi, inchi_key, item, value, unit])
time.sleep(1)
return datas
def main():
parser = argparse.ArgumentParser()
parser.add_argument("-output", type=str, required=True)
args = parser.parse_args()
base_url = "https://www.rcsb.org/search/data"
payloads = {"query":{"type":"group","logical_operator":"and","nodes":[{"type":"group","logical_operator":"and","nodes":[{"type":"group","logical_operator":"or","nodes":[{"type":"group","logical_operator":"and","nodes":[{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.value","negation":False,"operator":"exists"},"node_id":0},{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.type","operator":"exact_match","value":"IC50"},"node_id":1}],"label":"nested-attribute"},{"type":"group","logical_operator":"and","nodes":[{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.value","negation":False,"operator":"exists"},"node_id":2},{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.type","operator":"exact_match","value":"Ki"},"node_id":3}],"label":"nested-attribute"}]},{"type":"group","logical_operator":"and","nodes":[{"type":"terminal","service":"text","parameters":{"operator":"exact_match","negation":False,"value":"Severe acute respiratory syndrome-related coronavirus","attribute":"rcsb_entity_source_organism.taxonomy_lineage.name"},"node_id":4}]}],"label":"text"}],"label":"query-builder"},"return_type":"entry","request_options":{"pager":{"start":0,"rows":100},"scoring_strategy":"combined","sort":[{"sort_by":"score","direction":"desc"}]},"request_info":{"src":"ui","query_id":"e757fdfd5f9fb0efa272769c5966e3f4"}}
print(json.dumps(payloads))
response = requests.post(
base_url,
json.dumps(payloads),
headers={'Content-Type': 'application/json'})
datas = []
for a in response.json()["result_set"]:
structure_id = a["identifier"]
datas.extend(get_structure(structure_id))
time.sleep(1)
with open(args.output, "w") as f:
writer = csv.writer(f, lineterminator="\n")
writer.writerow(["ligand_id", "canonical_smiles", "inchi", "inchi_key", "item", "value", "unit"])
for data in datas:
writer.writerow(data)
if __name__ == "__main__":
main()
解説
- mainメソッドでjsonリクエストにより検索を行っている。IDの配列が得られるので、それをループでまわし、get_structureメソッドを呼び出している。
- get_sctructureメソッドでは、BindingAffinityのtrタグを取得し、そこから阻害データを取得するとともに、get_ligandメソッドを呼び出している。
- get_ligandメソッドでは、idからSMILES、Inchi等を取得している。
- 最後にmainメソッドで取得した全データをcsvに出力している。
方法2 Scrapyを用いた方法
コード
class PdbGetItem(scrapy.Item):
ligand_id = scrapy.Field()
canonical_smiles = scrapy.Field()
inchi = scrapy.Field()
inchi_key = scrapy.Field()
item = scrapy.Field()
value = scrapy.Field()
unit = scrapy.Field()
import scrapy
from scrapy.http import Request, JsonRequest
import json
from ..items import PdbGetItem
class LigandsSpider(scrapy.Spider):
name = 'get_pdb'
allowed_domains = ['www.rcsb.org']
def start_requests(self):
base_url = "https://www.rcsb.org/search/data"
payloads = {"query":{"type":"group","logical_operator":"and","nodes":[{"type":"group","logical_operator":"and","nodes":[{"type":"group","logical_operator":"or","nodes":[{"type":"group","logical_operator":"and","nodes":[{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.value","negation":False,"operator":"exists"},"node_id":0},{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.type","operator":"exact_match","value":"IC50"},"node_id":1}],"label":"nested-attribute"},{"type":"group","logical_operator":"and","nodes":[{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.value","negation":False,"operator":"exists"},"node_id":2},{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.type","operator":"exact_match","value":"Ki"},"node_id":3}],"label":"nested-attribute"}]},{"type":"group","logical_operator":"and","nodes":[{"type":"terminal","service":"text","parameters":{"operator":"exact_match","negation":False,"value":"Severe acute respiratory syndrome-related coronavirus","attribute":"rcsb_entity_source_organism.taxonomy_lineage.name"},"node_id":4}]}],"label":"text"}],"label":"query-builder"},"return_type":"entry","request_options":{"pager":{"start":0,"rows":100},"scoring_strategy":"combined","sort":[{"sort_by":"score","direction":"desc"}]},"request_info":{"src":"ui","query_id":"e757fdfd5f9fb0efa272769c5966e3f4"}}
yield JsonRequest(url=base_url, data=payloads, callback=self.parse)
def parse(self, response):
jsonresponse = json.loads(response.text)
for a in jsonresponse["result_set"]:
ligand_id = a['identifier']
structure_url = "https://www.rcsb.org/structure/"
structure_url += ligand_id
yield Request(url=structure_url, callback=self.parse_structure)
def parse_structure(self, response):
ids = response.xpath("//tr[contains(@id,'binding_row')]/td[position()=1]/a/@href").getall()
items = response.xpath("//tr[contains(@id,'binding_row')]/td[position()=2]/a/text()").getall()
values = response.xpath("//tr[contains(@id,'binding_row')]/td[position()=2]/text()").getall()
for ligand_id, item, value in zip(ids, items, values):
ligand_url = "https://www.rcsb.org" + ligand_id
item = item.strip()
value = value.replace(":", "")
value = value.replace(";", "")
value = value.replace(" ", "")
value = value.replace("\n", "")
values = value.split(" ", 1)
value = values[0].strip()
unit = values[1].strip()
pdb_item = PdbGetItem()
ligand_id = ligand_id[(ligand_id.rfind("/") + 1):]
pdb_item["ligand_id"] = ligand_id
pdb_item["item"] = item
pdb_item["value"] = value
pdb_item["unit"] = unit
request = Request(url=ligand_url, callback=self.parse_ligand)
request.meta["item"] = pdb_item
yield request
def parse_ligand(self, response):
smiles = response.xpath("//tr[@id='chemicalIsomeric']/td[1]/text()").get()
inchi = response.xpath("//tr[@id='chemicalInChI']/td[1]/text()").get()
inchi_key = response.xpath("//tr[@id='chemicalInChIKey']/td[1]/text()").get()
pdb_item = response.meta['item']
pdb_item["canonical_smiles"] = smiles
pdb_item["inchi"] = inchi
pdb_item["inchi_key"] = inchi_key
yield pdb_item
ROBOTSTXT_OBEY = False
DOWNLOAD_DELAY = 3
ITEM_PIPELINES = {
'pdb_get.pipelines.PdbGetPipeline': 300,
}
from scrapy.exceptions import DropItem
import csv
class PdbGetPipeline:
def open_spider(self, spider):
self.f = open("output.csv", "w")
self.writer = csv.writer(self.f, lineterminator="\n")
self.writer.writerow(["ligand_id", "canonical_smiles", "inchi", "inchi_key", "item", "value", "unit"])
def close_spider(self, spider):
self.f.close()
def process_item(self, item, spider):
if item["canonical_smiles"]:
self.writer.writerow([item["ligand_id"], item["canonical_smiles"], item["inchi"],
item["inchi_key"], item["item"], item["value"], item["unit"]])
else:
raise DropItem(item["ligand_id"])
解説
- get_pdb.py がスクレイパー、items.py がデータを保持するitemクラス、settings.py は設定、piplelines.py はitemを処理するためのパイプラインクラスである。
- setting.pyで1リクエスト当たりのスリープの指定(3秒)、Robots.txtに従うかどうかの設定、pipeline.pyの有効化を行っている。
- get_pdb.py の LingadsSpider の start_requests メソッドで検索を行っている。
- get_pdb.py の LingadsSpider の parse メソッドで検索結果の処理、蛋白質構造詳細画面の呼び出しを行っている。
- get_pdb.py の LingadsSpider の parse_structure メソッドで蛋白質構造詳細画面から阻害データの取得、リガンド詳細画面への遷移を行っている。
- get_pdb.py の LingadsSpider の parse_ligand メソッドでリガンド詳細画面から構造情報の取得を行っている。
- parse_structure メソッドと parse_ligand メソッドの2つの処理で item に必要なデータを取得しているが、これらitemの受け渡しは、parse_structureメソッド内の
request.meta["item"] = pdb_itemと parse_ligand メソッド内のpdb_item = response.meta['item']で行っている。今回のプチ肝といえる部分だ。 - pipeline.pyでは、csvファイルへの結果出力のためにファイルのストリームの開閉、ストリームへの出力を行っている。
出力されたCSV
こんな感じ。構造と阻害データがあるので候補薬を予測するモデルを作成しちゃおう。
ligand_id,canonical_smiles,inchi,inchi_key,item,value,unit
CYV,CCOC(=O)CC[C@H](C[C@H]1CCNC1=O)NC(=O)[C@H](CC=C(C)C)CC(=O)[C@@H](NC(=O)[C@H](CO)NC(=O)OC(C)(C)C)C(C)C,"InChI=1S/C32H54N4O9/c1-9-44-26(39)13-12-23(16-22-14-15-33-28(22)40)34-29(41)21(11-10-19(2)3)17-25(38)27(20(4)5)36-30(42)24(18-37)35-31(43)45-32(6,7)8/h10,20-24,27,37H,9,11-18H2,1-8H3,(H,33,40)(H,34,41)(H,35,43)(H,36,42)/t21-,22-,23-,24+,27+/m1/s1",CSVHTXSZRNRRSB-AHPXAKOISA-N,IC50,80000,nM
GRM,C[C@@H](N1CC[C@@H](CC1)C(=O)NCc2ccc3OCOc3c2)c4cccc5ccccc45,"InChI=1S/C26H28N2O3/c1-18(22-8-4-6-20-5-2-3-7-23(20)22)28-13-11-21(12-14-28)26(29)27-16-19-9-10-24-25(15-19)31-17-30-24/h2-10,15,18,21H,11-14,16-17H2,1H3,(H,27,29)/t18-/m1/s1",IVXBCFLWMPMSAP-GOSISDBHSA-N,IC50,320,nM
XP1,CN(C)c1ccc(cc1)C(O)=O,"InChI=1S/C9H11NO2/c1-10(2)8-5-3-7(4-6-8)9(11)12/h3-6H,1-2H3,(H,11,12)",YDIYEOMDOWUDTJ-UHFFFAOYSA-N,IC50,5000,nM
S88,C[C@@H](N1CC[C@H](CC1)C(=O)NCc2cccc(F)c2)c3cccc4ccccc34,"InChI=1S/C25H27FN2O/c1-18(23-11-5-8-20-7-2-3-10-24(20)23)28-14-12-21(13-15-28)25(29)27-17-19-6-4-9-22(26)16-19/h2-11,16,18,21H,12-15,17H2,1H3,(H,27,29)/t18-/m1/s1",XJTWGMHOQKGBDO-GOSISDBHSA-N,IC50,150,nM
P85,C[C@@H](N1CC[C@@H](CC1)C(=O)NCc2ccc(F)cc2)c3cccc4ccccc34,"InChI=1S/C25H27FN2O/c1-18(23-8-4-6-20-5-2-3-7-24(20)23)28-15-13-21(14-16-28)25(29)27-17-19-9-11-22(26)12-10-19/h2-12,18,21H,13-17H2,1H3,(H,27,29)/t18-/m1/s1",VTAUBQMDNJOEAK-GOSISDBHSA-N,IC50,490,nM
3X5,O=C[C@H](Cc1[nH]cnc1)NC[C@H]2C[C@@H]3CCCC[C@H]3CN2C(=O)c4ccc(cc4)c5ccccc5,"InChI=1S/C29H34N4O2/c34-19-27(15-26-16-30-20-32-26)31-17-28-14-24-8-4-5-9-25(24)18-33(28)29(35)23-12-10-22(11-13-23)21-6-2-1-3-7-21/h1-3,6-7,10-13,16,19-20,24-25,27-28,31H,4-5,8-9,14-15,17-18H2,(H,30,32)/t24-,25-,27-,28+/m0/s1",VZCULZJNALRGNB-DNZWLJDLSA-N,IC50,240000,nM
3A7,Brc1ccc(cc1)C(=O)N2C[C@H]3CCCC[C@@H]3C[C@H]2CN[C@@H](Cc4[nH]cnc4)C=O,"InChI=1S/C23H29BrN4O2/c24-19-7-5-16(6-8-19)23(30)28-13-18-4-2-1-3-17(18)9-22(28)12-26-21(14-29)10-20-11-25-15-27-20/h5-8,11,14-15,17-18,21-22,26H,1-4,9-10,12-13H2,(H,25,27)/t17-,18-,21+,22+/m1/s1",SKLHMRHVVDDIOX-UBBRYJJRSA-N,IC50,63000,nM
SFG,N[C@@H](CC[C@H](N)C(O)=O)C[C@H]1O[C@H]([C@H](O)[C@@H]1O)n2cnc3c(N)ncnc23,"InChI=1S/C15H23N7O5/c16-6(1-2-7(17)15(25)26)3-8-10(23)11(24)14(27-8)22-5-21-9-12(18)19-4-20-13(9)22/h4-8,10-11,14,23-24H,1-3,16-17H2,(H,25,26)(H2,18,19,20)/t6-,7-,8+,10+,11+,14+/m0/s1",LMXOHSDXUQEUSF-YECHIGJVSA-N,IC50,740,nM
D3F,Cc1cc(c(Cl)cc1Cl)[S](=O)(=O)c2c(cc(cc2[N+]([O-])=O)C(F)(F)F)[N+]([O-])=O,"InChI=1S/C14H7Cl2F3N2O6S/c1-6-2-12(9(16)5-8(6)15)28(26,27)13-10(20(22)23)3-7(14(17,18)19)4-11(13)21(24)25/h2-5H,1H3",INAZPZCJNPPHGV-UHFFFAOYSA-N,IC50,300,nM
F3F,FC(F)(F)c1[nH]c(SC(=O)c2oc(cc2)C#Cc3ccccc3)nn1,"InChI=1S/C16H8F3N3O2S/c17-16(18,19)14-20-15(22-21-14)25-13(23)12-9-8-11(24-12)7-6-10-4-2-1-3-5-10/h1-5,8-9H,(H,20,21,22)",VNGWUVBXUIDQTK-UHFFFAOYSA-N,IC50,3000,nM
23H,CCC(C)(C)NC(=O)[C@H](N(C(=O)Cn1nnc2ccccc12)c3ccc(NC(C)=O)cc3)c4cccn4C,"InChI=1S/C28H33N7O3/c1-6-28(3,4)30-27(38)26(24-12-9-17-33(24)5)35(21-15-13-20(14-16-21)29-19(2)36)25(37)18-34-23-11-8-7-10-22(23)31-32-34/h7-17,26H,6,18H2,1-5H3,(H,29,36)(H,30,38)/t26-/m1/s1",BCIIGGMNYNWRQK-AREMUKBSSA-N,IC50,6200,nM
CY6,CCOC(=O)/C=C/[C@H](C[C@@H]1CCNC1=O)NC(=O)[C@H](CC=C(C)C)CC(=O)[C@@H](NC(=O)c2cc(C)on2)C(C)C,"InChI=1S/C29H42N4O7/c1-7-39-25(35)11-10-22(15-21-12-13-30-27(21)36)31-28(37)20(9-8-17(2)3)16-24(34)26(18(4)5)32-29(38)23-14-19(6)40-33-23/h8,10-11,14,18,20-22,26H,7,9,12-13,15-16H2,1-6H3,(H,30,36)(H,31,37)(H,32,38)/b11-10+/t20-,21+,22-,26+/m1/s1",CSNQHKJCKPMZCY-YFUAOJPXSA-N,IC50,70000,nM
0EN,CC(C)(C)NC(=O)[C@H](N(C(=O)c1occc1)c2ccc(cc2)C(C)(C)C)c3cccnc3,"InChI=1S/C26H31N3O3/c1-25(2,3)19-11-13-20(14-12-19)29(24(31)21-10-8-16-32-21)22(18-9-7-15-27-17-18)23(30)28-26(4,5)6/h7-17,22H,1-6H3,(H,28,30)/t22-/m1/s1",JXGIYKRRPGCLFV-JOCHJYFZSA-N,IC50,4800,nM
TTT,C[C@@H](NC(=O)c1cc(N)ccc1C)c2cccc3ccccc23,"InChI=1S/C20H20N2O/c1-13-10-11-16(21)12-19(13)20(23)22-14(2)17-9-5-7-15-6-3-4-8-18(15)17/h3-12,14H,21H2,1-2H3,(H,22,23)/t14-/m1/s1",UVERBUNNCOKGNZ-CQSZACIVSA-N,IC50,2640,nM
WR1,C[C@@H](NC(=O)[C@H](Cc1ccccc1)NC(=O)OCc2ccccc2)C(=O)CCO,"InChI=1S/C22H26N2O5/c1-16(20(26)12-13-25)23-21(27)19(14-17-8-4-2-5-9-17)24-22(28)29-15-18-10-6-3-7-11-18/h2-11,16,19,25H,12-15H2,1H3,(H,23,27)(H,24,28)/t16-,19+/m1/s1",UUOOAGBWJUGBMV-APWZRJJASA-N,Ki,2200,nM
S89,OC[C@H](Cc1ccccc1)NC(=O)[C@H](Cc2ccccc2)NC(=O)/C=C/c3ccccc3,"InChI=1S/C27H28N2O3/c30-20-24(18-22-12-6-2-7-13-22)28-27(32)25(19-23-14-8-3-9-15-23)29-26(31)17-16-21-10-4-1-5-11-21/h1-17,24-25,30H,18-20H2,(H,28,32)(H,29,31)/b17-16+/t24-,25-/m0/s1",GEVQDXBVGFGWFA-KQRRRSJSSA-N,Ki,2240,nM
ZU3,CC(C)C[C@H](NC(=O)[C@H](CNC(=O)C(C)(C)C)NC(=O)OCc1ccccc1)C(=O)N[C@H](CCC(C)=O)C[C@@H]2CCNC2=O,"InChI=1S/C32H49N5O7/c1-20(2)16-25(28(40)35-24(13-12-21(3)38)17-23-14-15-33-27(23)39)36-29(41)26(18-34-30(42)32(4,5)6)37-31(43)44-19-22-10-8-7-9-11-22/h7-11,20,23-26H,12-19H2,1-6H3,(H,33,39)(H,34,42)(H,35,40)(H,36,41)(H,37,43)/t23-,24+,25-,26-/m0/s1",IEQRDAZPCPYZAJ-QYOOZWMWSA-N,Ki,38,nM
ZU5,CC(C)C[C@H](NC(=O)[C@@H](NC(=O)OCc1ccccc1)[C@@H](C)OC(C)(C)C)C(=O)N[C@H](CCC(=O)C2CC2)C[C@@H]3CCNC3=O,"InChI=1S/C34H52N4O7/c1-21(2)18-27(31(41)36-26(14-15-28(39)24-12-13-24)19-25-16-17-35-30(25)40)37-32(42)29(22(3)45-34(4,5)6)38-33(43)44-20-23-10-8-7-9-11-23/h7-11,21-22,24-27,29H,12-20H2,1-6H3,(H,35,40)(H,36,41)(H,37,42)(H,38,43)/t22-,25+,26-,27+,29+/m1/s1",QIMPWBPEAHOISN-XSLDCGIXSA-N,Ki,99,nM
TLD,Cc1ccc(S)c(S)c1,"InChI=1S/C7H8S2/c1-5-2-3-6(8)7(9)4-5/h2-4,8-9H,1H3",NIAAGQAEVGMHPM-UHFFFAOYSA-N,Ki,1400,nM
PMA,OC(=O)c1cc(C(O)=O)c(cc1C(O)=O)C(O)=O,"InChI=1S/C10H6O8/c11-7(12)3-1-4(8(13)14)6(10(17)18)2-5(3)9(15)16/h1-2H,(H,11,12)(H,13,14)(H,15,16)(H,17,18)",CYIDZMCFTVVTJO-UHFFFAOYSA-N,Ki,700,nM
考察
Scrapyを使った場合と使わない場合について考察してみる。
Scrapyを用いる場合のメリット
- ログを見ると分かるが、同じURLへのアクセスを自動認識し、無駄なアクセスをしない。
- エラーになったときに、自動的にリトライしてくれる(デフォルトは3回っぽい)
- setting.pyの設定で、スリープ処理を一括で入れることができる。
- Roblots.txt のルールに従うかどうかなど、細かいスクレイピング流儀について調整することができる。
- ログ出力をうまいことやってくれる。
- ソースの構成が美しい。
Scrapyを用いる場合のデメリット
- エラーになった時に中断するなど細かい制御ができない (pipelineを利用すれば結局できちゃうわけだが。)
- 学習コストが高い。 (覚えてしまえば楽できるが。。。)
- 今回の検索処理に見られるように、JSON等の構造化データでrequest/responsするケースが増えてきており、その場合結局JSONの操作だけで済むので、あまりメリットがない?
Scrapyのデメリットが少ない。よし、今後はScrapyでガンガンデータを取ろう。
最後に今回の環境
今回は以下の環境で実施しました。
- python3.7
- scrapy 2.3
- requests 2.24
- lxml 4.5.2