はじめに
RDKit等で3次元構造を生成する際には、水素原子を付与することが推奨されている。しかしSDFから読み込んで生成したMolオブジェクトにChem#AddHsメソッドで付与しても正しく水素原子が付与されてない場合があるので、メモ
環境
- RDKit 2018/9/1
- Python 3.6
- Jupyter Notebook
うまくいかない方法
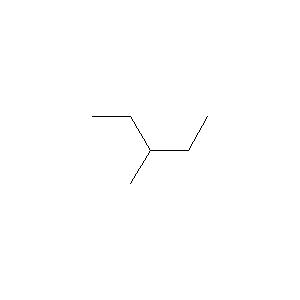
まずは読み込んで表示。
import rdkit.Chem
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem import AllChem
molBlock = '''3-methylpentane
RDKit 2D
6 5 0 0 0 0 0 0 0 0999 V2000
0.2606 0.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3000 0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6000 -1.2000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9000 0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9394 0.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0
2 3 1 0
3 4 1 0
3 5 1 0
5 6 1 0
M END'''
molFromSDF = Chem.MolFromMolBlock(molBlock)
Draw.MolToImage(molFromSDF)
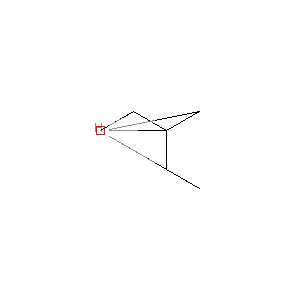
次にAddHsで水素を追加。
molFromSDFH = Chem.AddHs(molFromSDF)
Draw.MolToImage(molFromSDFH)
明らかに失敗。
正しい方法
MOLオブジェクトをいったんSMILES化し、SMILESからMOLオブジェクトを再作成する。
smiles = Chem.MolToSmiles(molFromSDF)
molFromSDFnorm = Chem.MolFromSmiles(smiles)
次にAddHsで水素追加
molFromSDFnormH = Chem.AddHs(molFromSDFnorm)
Draw.MolToImage(molFromSDFnormH)
おおう、正しく表示された。うまくいったようだ。
追記(重要) 2022/06/19
どうやら、AddHsを行う時に全ての水素の座標が(0,0)の位置に追加されるため、表示が崩れるらしい。addCoordsオプションをTrueに指定することで、水素の座標がいい感じで追加され表示が崩れないようである。
Chem.AddHs(ligand_mol, addCoords=True)