先日の#75 Tokyo.Rの初心者セッションでdata pipelineというトークをしました。データは「横」「縦」「畳み込み」という形式で整理して解析を進めましょうという大枠の流れと、それぞれのデータの特徴を紹介しました。

また、Qiitaに書いたR入門(裏口)でもnestについて触れました。が、データの畳み込みを解析の中でどうやって活かしていくのかという点には十分踏み込めていませんでした(初心者向けのコンテンツを超えてしまう気がしたのと、時間の都合です)。というわけで、別立てですが記事にしておきます。
途中で出てくるmapやmap2に関しては別記事にしました。
使用するライブラリ
library(tidyverse)
使用するデータ
みんな大好き iris 。
dat <- iris
> head(dat)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
> str(dat)
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
朴訥にやる。
Speciesごとに Sepal.Length ~ Petal.Width の回帰をしてみましょう。
# それぞれ取ってきて
dat_setosa <- filter(dat, Species == "setosa")
dat_versicolor <- filter(dat, Species == "versicolor")
dat_virginica <- filter(dat, Species == "virginica")
# それぞれ回帰
model_lm_setosa <- lm(Sepal.Length ~ Petal.Width, data = dat_setosa)
model_lm_versicolor <- lm(Sepal.Length ~ Petal.Width, data = dat_versicolor)
model_lm_virginica <- lm(Sepal.Length ~ Petal.Width, data = dat_virginica)
いやいやいや、せめてパイプ%>%を使いましょう。
model_lm_setosa <- dat %>%
filter(Species == "setosa") %>%
lm(Sepal.Length ~ Petal.Width, data = .)
model_lm_versicolor <- dat %>%
filter(Species == "versicolor") %>%
lm(Sepal.Length ~ Petal.Width, data = .)
model_lm_virginica <- dat %>%
filter(Species == "virginica") %>%
lm(Sepal.Length ~ Petal.Width, data = .)
っと、これでもコレはあんまりにもあんまりですよね。
独自関数を作って使う。
とりあえず、独自関数を定義して簡略化してみます。
filterd_lm <- function(dat, .tag){
dat %>%
filter(Species == .tag) %>%
lm(Sepal.Length ~ Petal.Width, data = .)
}
model_lm_setosa <- filterd_lm(dat, "setosa")
model_lm_versicolor <- filterd_lm(dat, "versicolor")
model_lm_virginica <- filterd_lm(dat, "virginica")
ん〜、どうせ関数化するならもうちょっと頑張りましょう。
filterd_lm <- function(dat, .tag,
.key = "Species", .x = "Sepal.Width", .y = "Petal.Length"){
dat %>%
select(.key, .x, .y) %>% # selectは文字列を引数に取れる。
rename_all(~c("key", "x", "y")) %>% # NSE問題を回避するために関数の中でカラム名を指定する。
filter(key == .tag) %>%
lm(y ~ x, data = .)
}
model_lm_setosa <- filterd_lm(dat, "setosa")
model_lm_versicolor <- filterd_lm(dat, "versicolor")
model_lm_virginica <- filterd_lm(dat, "virginica")
ちょっと汎用性が上がりました。
purrr::mapを使おう。
しかし、相変わらず3つの回帰を実行するのに3回コピペしなきゃいけない。
嫌ですね。
そんな時はpurrr::mapを使います1。
extract_levels <- function(dat, .key){
dat %>% select(.key) %>% unlist %>% factor %>% levels()
}
dat %>%
extract_levels("Species") %>%
map(~filterd_lm(dat, .))
結果はこうなります。
[[1]]
Call:
lm(formula = y ~ x, data = .)
Coefficients:
(Intercept) x
1.18292 0.08141
[[2]]
Call:
lm(formula = y ~ x, data = .)
Coefficients:
(Intercept) x
1.9349 0.8394
[[3]]
Call:
lm(formula = y ~ x, data = .)
Coefficients:
(Intercept) x
3.5109 0.6863
それぞれのlmの結果がlistの要素になって出力されています。
mapの導入に関しては、もうちょっと詳しく別の記事で解説しました。
nestしよう。
これはこれでいいんですけど、ここから先どうするのか考えると見通しが悪い。
元のデータと回帰モデルの対応が付いて、まとまって1つのオブジェクトになっていて欲しい。
そこでtidyr::nestを使います。
dat_lm <- dat %>%
group_by(Species) %>%
nest() %>%
mutate(model_lm = map(data, ~lm(Petal.Length ~ Sepal.Width, data = .)))
出力はこうなります2。
> dat_lm
# A tibble: 3 x 3
Species data model_lm
<fct> <list> <list>
1 setosa <tibble [50 x 4]> <S3: lm>
2 versicolor <tibble [50 x 4]> <S3: lm>
3 virginica <tibble [50 x 4]> <S3: lm>
levelsを抽出したりfilterをかける作業が全てgroup_byからのnestの畳み込み操作の中に含まれているので、独自関数を定義する必要がないのは大きなポイントです。
また、キーとなるSpecies・元データdata・線形回帰モデルmodel_lmが、行で対応づけられているのも大きなポイントで、モデルとデータを別々のオブジェクトに格納して別々に引っ張り出すよりも、その後の解析操作の見通し(風通し)を良くします。
例えば、$R^2$が欲しい場合、mapの一種map_dblを使って、下記の様に抽出します。
> dat_lm %>%
+ mutate(rsq = map_dbl(model_lm, ~summary(.) %>% .$r.squared))
# A tibble: 3 x 4
Species data model_lm rsq
<fct> <list> <list> <dbl>
1 setosa <tibble [50 x 4]> <S3: lm> 0.0316
2 versicolor <tibble [50 x 4]> <S3: lm> 0.314
3 virginica <tibble [50 x 4]> <S3: lm> 0.161
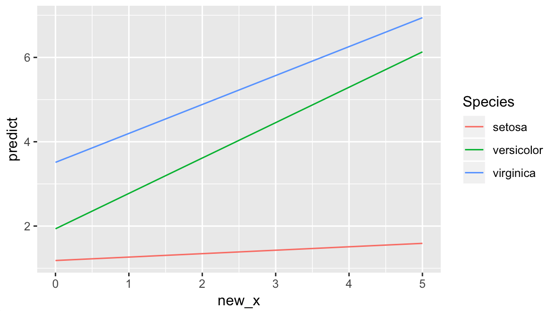
x = [0, 5]の範囲で予測値を出す事もこのままできます。
dat_predict <- dat_lm %>%
mutate(new_x = list(seq(0, 5, length = 5)),
predict = map2(new_x, model_lm,
~predict(.y, data.frame(.x) %>% set_names("Sepal.Width"))))
> dat_predict
# A tibble: 3 x 5
Species data model_lm new_x predict
<fct> <list> <list> <list> <list>
1 setosa <tibble [50 x 4]> <S3: lm> <dbl [5]> <dbl [5]>
2 versicolor <tibble [50 x 4]> <S3: lm> <dbl [5]> <dbl [5]>
3 virginica <tibble [50 x 4]> <S3: lm> <dbl [5]> <dbl [5]>
必要に応じてunnestします。
この様に見通しよくデータを整えてから、欲しい部分を抽出して、tidyr::unnestします。
例えば、線形モデルの予測値をグラフ化したい場合には、下記の様に抽出します。
dat_g <- dat_predict %>%
select(Species, new_x, predict) %>%
unnest()
# A tibble: 15 x 3
Species new_x predict
<fct> <dbl> <dbl>
1 setosa 0 1.18
2 setosa 1.25 1.28
3 setosa 2.5 1.39
4 setosa 3.75 1.49
5 setosa 5 1.59
6 versicolor 0 1.93
7 versicolor 1.25 2.98
8 versicolor 2.5 4.03
9 versicolor 3.75 5.08
10 versicolor 5 6.13
11 virginica 0 3.51
12 virginica 1.25 4.37
13 virginica 2.5 5.23
14 virginica 3.75 6.08
15 virginica 5 6.94
描画してみます。
g <- ggplot(dat_g, aes(new_x, predict, color = Species))+
geom_path()
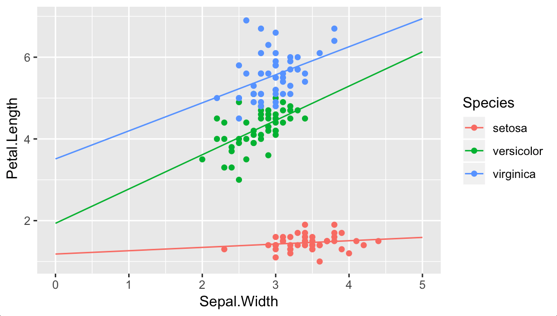
これに元のポイントを重ねてみるには、ちょっとギョーギが悪いですが、
g + geom_point(data = dat_predict %>% select(Species, data) %>% unnest,
aes(Sepal.Width, Petal.Length, color = Species))+
xlab("Sepal.Width")+
ylab("Petal.Length")
はい、終わり。
という訳にはならないんですよね。
可視化は1枚絵ができました、はいおしまいではなくて、何度もなんども何種類も絵を作ってみながらデータの特徴やモデルを考えていく探索的プロセスです。1個のモデルを当てはめて図示して、ふー、とため息をついて、えーと、と後ろを振り返って焼け野原では困るんです。
もう一度、元データとモデルに素早く戻れるという事が大切です。
> dat_lm
# A tibble: 3 x 3
Species data model_lm
<fct> <list> <list>
1 setosa <tibble [50 x 4]> <S3: lm>
2 versicolor <tibble [50 x 4]> <S3: lm>
3 virginica <tibble [50 x 4]> <S3: lm>
ここで、最初のdatに戻らないで済む、というのも大きいですね。
データを意図に合わせて整理した状態で保持しておくと、見通しが良くなります。
もちろん、元に戻りたければunnestで戻れます。
dat_lm %>%
select(Species, data) %>%
unnest()
pipeはいいぞ。
最後の利点は、解析の全般を通じて必要なモノ以外に名前をつけなくて良いという事です。

これはパイプ演算子の特徴ですが、途中経過にそれぞれオブジェクト名を振っていると、戻りたいポイントがどこだか分からなくなってしまうというデメリットがあります。そしてその度に適切なオブジェクト名を考え出すのに無駄なエネルギーを使うことになります。model_lm_setosaなんてオブジェクトを作るのは、やっぱりナンセンスだと思いませんか?
例えばこんな風に。
簡単な線形回帰をやりたいだけなら、もちろん、下記のようにできます。
dat %>%
ggplot(aes(Sepal.Width, Petal.Length, color = Species))+
geom_point()+
geom_smooth(method = "lm", se = F)

そうですね、水準ごとにPCAをかけて、それぞれの主成分スコアと因子負荷量をグラフ化したいとか。
df <- dat %>%
group_by(Species) %>% # Speciesでnest
nest %>%
mutate(pca = map(data,
~ prcomp(., scale = T)), # それぞれにpca
score = map(pca,
~ .$x %>% data.frame), # 主成分スコア抽出
fl = map2(data, pca, # 因子負荷量(factor loadings)抽出
~ cor(.x, .y$x) %>% data.frame %>% rownames_to_column("params")))
このdfが帰ってくるポイントですね。
> df
# A tibble: 3 x 5
Species data pca score fl
<fct> <list> <list> <list> <list>
1 setosa <tibble [50 x 4]> <S3: prcomp> <data.frame [50 x 4]> <data.frame [4 x 5]>
2 versicolor <tibble [50 x 4]> <S3: prcomp> <data.frame [50 x 4]> <data.frame [4 x 5]>
3 virginica <tibble [50 x 4]> <S3: prcomp> <data.frame [50 x 4]> <data.frame [4 x 5]>
必要部分をunnestしてggplotで描画。
# 主成分スコア描画
g1 <- df %>% select(Species, score) %>% unnest %>%
ggplot(aes(PC1, PC2, color = Species))+
geom_point()+
stat_ellipse(level = 0.68)+
facet_wrap(~Species)+
theme_bw()+
theme(legend.position = "none")+
scale_x_continuous(limits = c(-4, 4))+
scale_y_continuous(limits = c(-4, 4))+
coord_fixed(1/1)
# FL描画
g2 <- df %>% select(Species, fl) %>% unnest %>%
ggplot()+
geom_segment(aes(x = 0, y = 0, xend = PC1, yend = PC2),
arrow = arrow(type = "closed"), alpha = 0.5)+
geom_text(aes(label = params, x = PC1, y = PC2))+
facet_wrap(~Species)+
theme_bw()+
scale_x_continuous(limits = c(-1.2, 1.2))+
scale_y_continuous(limits = c(-1.2, 1.2))+
coord_fixed(1/1)
# 結合した絵を表示
library(patchwork)
g1 + g2 + plot_layout(ncol = 1)

-
purrrの取り扱いは、u_riboさんの解説そろそろ手を出すpurrrや、Heavy Watalさんの解説purrr — ループ処理やapply系関数の決定版が詳しいです。 ↩
-
あ!上の例とxとyが逆だった、と後から気づきましたが、まぁご愛嬌で。。 ↩