MCMC(*´Д`)ハァハァ
— MrUnadon (@MrUnadon) 2017年9月1日
ちょっと前ですが、TokyoRでベイズ特集回をやった事があります。
その際に、ベイズ統計の基礎という大げさなタイトルでトークをしました。
トークは基礎的(理論的)な話だけでしたので、これはRとstanを使った実践編という感じです。
環境設定
# libraryにattachしておく
library(tidyverse) # データ加工
library(rstan) # MCMCハァハァ
library(patchwork) # 図を並べる
library(lme4) # おまけ
# rstanのchainを並列計算させる呪文
rstan_options(auto_write=TRUE)
options(mc.cores=parallel::detectCores())
復習: 線形混合モデル
線形混合モデルの取り回しでは、種ごとのランダム効果を導入した線形モデルによる回帰をlme4::lmer関数を使って行いました。
例えば下記のようなモデル関数を使ってフィッティングします。
# 傾きと切片にそれぞれ独立のランダム効果を導入
model_lmm <- function(data)
lmer(Petal.Length ~ Sepal.Width + (1|Species) + (0 + Sepal.Width|Species), data = data)
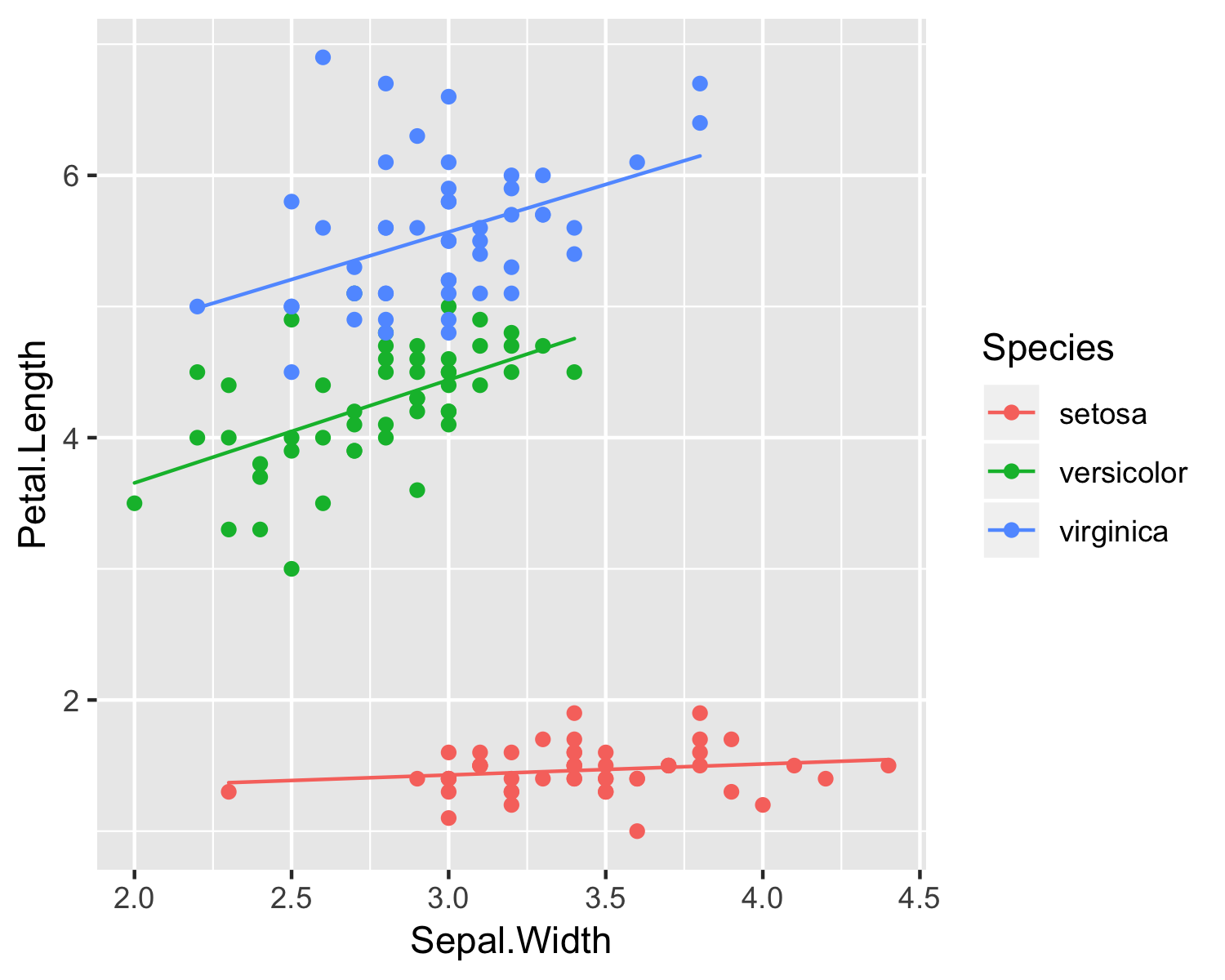
図を出力するコード
dat %>% model_lmm %>% predict %>% data.frame %>% set_names("predict") %>% cbind(dat, .) %>%
ggplot(aes(x = Sepal.Width, color = Species))+
geom_point(aes(y = Petal.Length))+
geom_path(aes(y = predict, group = Species))
ただの線形回帰
まずは簡単なモデルを組んでみる。
混合効果を考えない単なる回帰。
(ほとんどhoxo_mさんの二番目に簡単な rstan コードと同じです)
せっかくベイズ推定するので、切片bの最低値が0という縛りを入れました。
推定値Yも0以上ですね。(間接的にaの範囲を制限できる)
花弁の長さが0以上というのは自然な仮定です。
bの事前分布は明示的に指定していないのですが、この場合unif(0, Inf)になるようです。
stancode_iris_lm <- "
data { # 入力する既知データ
int N; # データ数
real x[N]; # 長さNのベクトル
real y[N];
}
parameters{ # 事後分布を推定されるパラメータ
real a; # 傾きa
real<lower=0> b; # 切片b
real<lower=0> sigma; # 正規誤差sigma
}
transformed parameters{
real<lower=0> Y[N]; # xに対応して推定される値Y
for(i in 1:N)
Y[i] = a * x[i] + b; # 回帰式
}
model{
for(i in 1:N)
y[i] ~ normal(Y[i], sigma); # y ~ Y + N(0, sigma)の変形
a ~ normal(0, 100); # 事前分布
sigma ~ inv_gamma(0.001, 0.001); # 事前分布
}
"
キックするRコードはこんな感じ。
dat <- iris
dat_stan <- list(
N = nrow(dat),
x = dat$Sepal.Width,
y = dat$Petal.Length
)
fit <- stan(model_code = stancode_iris_lm, data = dat_stan, seed = 71)
結果の確認。
Rhatは軒並み1で、収束は良さそう。
> fit
Inference for Stan model: 4bac703ee505e3edc67be58e74e56e8e.
4 chains, each with iter=2000; warmup=1000; thin=1;
post-warmup draws per chain=1000, total post-warmup draws=4000.
mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
a -1.73 0.01 0.29 -2.29 -1.92 -1.73 -1.53 -1.12 1026 1
b 9.04 0.03 0.91 7.14 8.44 9.05 9.63 10.80 1021 1
sigma 1.61 0.00 0.09 1.44 1.54 1.60 1.67 1.81 1682 1
線形回帰lmの結果と比較してみる。aとbの値は、ほぼ一緒。
> lm(Petal.Length ~ Sepal.Width, data = dat)
Call:
lm(formula = Petal.Length ~ Sepal.Width, data = iris)
Coefficients:
(Intercept) Sepal.Width
9.063 -1.735
念の為可視化。(code割愛)
まぁ、こうなりますよね〜、という結果。
ランダム効果を導入
transformed parametersのセクションが変わっています。
それに合わせてあちこち調整を入れています。
切片bの混合効果b_mを<lower=-b>としても良いです(結果はホボ変わらなかったですね)。
また、この入れ方では、speciesはintで入れないと怒られます。
stancode_iris_lmm <- "
data {
int N;
real x[N];
real y[N];
int N_s; # 種数
int species[N]; # データに対応する種のindexが入ったベクトル
}
parameters{
real a;
real<lower=0> b;
real<lower=0> sigma;
real a_m[N_s]; # 種ごとの傾き変動(ランダム効果)
real b_m[N_s]; # 種ごとの切片変動(ランダム効果)
real<lower=0> a_s; # 傾きランダム効果のばらつき
real<lower=0> b_s; # 切片ランダム効果のばらつき
}
transformed parameters{
real<lower=0> Y[N];
for(i in 1:N) # 種のindex番目のランダム効果が加わる回帰式
Y[i] = (a + a_m[species[i]]) * x[i] + b + b_m[species[i]];
}
model{
for(i in 1:N_s){
a_m[i] ~ normal(0, a_s); # ランダム効果は種ごとにサンプリング
b_m[i] ~ normal(0, b_s);
}
for(i in 1:N)
y[i] ~ normal(Y[i], sigma); # 回帰はデータごとにサンプリング
a ~ normal(0, 100); # 以下、事前分布
sigma ~ inv_gamma(0.001, 0.001);
a_s ~ inv_gamma(0.001, 0.001);
b_s ~ inv_gamma(0.001, 0.001);
}
"
キックするコードはほぼ一緒。
dat <- iris %>%
mutate(species = Species %>% factor %>% c)
dat_stan <- list(
N = nrow(dat),
x = dat$Sepal.Width,
y = dat$Petal.Length,
N_s = dat$species %>% max,
species = dat$species
)
fit <- stan(model_code = stancode_iris_lmm, data = dat_stan, seed = 71, iter = 5000, thin = 2)
結果。良さげ。ただしn_effが小さい。
> fit
Inference for Stan model: 8c68fd56debef05f291ad0da21a95901.
4 chains, each with iter=5000; warmup=2500; thin=2;
post-warmup draws per chain=1250, total post-warmup draws=5000.
mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
a 0.49 0.03 0.73 -0.58 0.32 0.52 0.72 1.59 456 1.01
b 2.35 0.03 1.06 0.44 1.74 2.26 2.80 4.86 1356 1.00
sigma 0.39 0.00 0.02 0.35 0.37 0.39 0.40 0.44 3671 1.00
a_m[1] -0.42 0.03 0.74 -1.59 -0.66 -0.41 -0.20 0.57 469 1.01
a_m[2] 0.28 0.03 0.74 -0.80 0.02 0.23 0.46 1.39 455 1.01
a_m[3] 0.25 0.03 0.74 -0.78 0.00 0.20 0.43 1.40 456 1.01
b_m[1] -1.14 0.03 1.19 -3.98 -1.71 -0.96 -0.33 0.67 1286 1.00
b_m[2] -0.21 0.03 1.08 -2.82 -0.67 -0.11 0.37 1.73 1425 1.00
b_m[3] 0.98 0.03 1.11 -1.42 0.36 0.96 1.61 3.14 1391 1.00
a_s 0.73 0.04 1.22 0.11 0.32 0.49 0.78 2.71 808 1.01
b_s 1.70 0.04 1.56 0.06 0.82 1.30 2.09 5.62 1665 1.00
lmmの結果と、似ているような似ていないような。だいたい似ているかな〜。
> dat %>% model_lmm %>% fixef
(Intercept) Sepal.Width
2.2182831 0.5314268
> dat %>% model_lmm %>% ranef
$Species
(Intercept) Sepal.Width
setosa -1.0415779 -0.4475732
versicolor -0.1347486 0.2544014
virginica 1.1763265 0.1931718
n_effも気になるし、traceplotを描いてみる。
traceplot(fit, pars = c("a", "b", "sigma", "a_m", "b_m"))
MCMC(*´Д`)ハァハァ
ん〜ちょっとaですっ飛んでいるところがありますね。
(thin=2にしたりiterを大きめにした理由はこのへんを考慮してみた結果)
事後分布を可視化してみましょう。ドンッ!
図を出力するコード
# 有効MCMCサンプルの抽出
ext_fit <- extract(fit)
# fixed effectを取り出して縦持ちにしてggplot
g_fixef <- data.frame(a = ext_fit$a,
b = ext_fit$b) %>%
gather(key, val) %>%
ggplot(aes(val, ..density.., color = key, fill = key))+
geom_density(alpha = 0.5)+
facet_wrap(~key)+
theme(axis.title.x = element_blank())+
ggtitle("Fixed effect")+
scale_x_continuous(limits = c(-10, 10))
# この関数は時々欲しい。今回はNSEで実装
extract_levels <- function(dat, .key){
dat %>% select(!!enquo(.key)) %>% unlist %>% factor %>% levels
}
# random effectを取り出して縦持ちにしてタグを整備してggplot
g_ranef <- cbind(ext_fit$a_m, ext_fit$b_m) %>%
data.frame() %>%
set_names(str_c("a_", extract_levels(iris, Species)),
str_c("b_", extract_levels(iris, Species))) %>%
gather(key, val) %>%
separate(key, into = c("param", "id"), remove = F) %>%
ggplot(aes(val, ..density.., color = id, fill = id))+
geom_density(alpha = 0.5)+
scale_x_continuous(limits = c(-5, 5))+
facet_wrap(~param)+
theme(axis.title.x = element_blank())+
ggtitle("Random effect")
# 回帰直線をプロット
g_predict <- ext_fit$Y %>%
data.frame() %>%
summarise_all(mean) %>%
t %>%
data.frame() %>%
set_names("predict") %>%
cbind(dat, .) %>%
ggplot(aes(x = Sepal.Width, color = Species))+
geom_point(aes(y = Petal.Length))+
geom_path(aes(y = predict, group = Species))
# 保存した絵を組み合わせたプロットをg_wに入れておいてggsave
g_w <- wrap_plots((g_fixef / g_ranef) | g_predict)
ggsave("iris_lmm.png", g_w, width = 12, height = 6)
無茶な推定ではなさそうです。
ただ、線形混合モデルの取り回しでみた通り、このモデルはそもそもあまり「良い」とは言えないんですよね。
「MCMCが収束する=良いモデル」というのは大変危険で、それをいうなら上に挙げた「単なる線形回帰」も収束しているわけです。やはりモデル選択の考え方が必要になってくるでしょう。それはまた後日。
(ちなみに単なる線形回帰モデルのAICは、LMMのAICより明らかにデカいので「良くない」と判断されます)
> dat %>% lm(Petal.Length ~ Sepal.Width, data = .) %>% AIC
[1] 570.7537
> dat %>% model_lmm %>% AIC
[1] 171.819


