はじめに
Microsoft様のご好意により、Githubに無料公開されているデータサイエンス初心者向け講座をベースにCovid-19の科学論文を分析していきます。
対象読者
Pythonとデータサイエンスに興味があって、英語が苦手な人(英語が得意な人は、参考文献を直接解いてください。
Day2
この記事では、Day2のWork With Data, Work With PythonのAnalyzing COVID-19 Papersチャレンジを翻訳しながらやってみます。
Day2ゴール
本記事では2.非構造データである論文を扱います。pandasを使った分析はこちらの記事でやって見ました。
- COVID-19国別感染者の集計(構造化されたデータ)
- COVID-19論文の解析(非構造データ)
利用するライブラリの概要
- pandas
pandasライブラリでは、リレーショナルテーブルに類似した、いわゆるDataframeを操作することができます。名前付きカラムを持つことができ、行、カラム、データフレームに対して一般的に異なる操作を行うことができます。
- numpy
numpyライブラリは、テンソル、すなわち多次元配列を扱うためのライブラリである。配列は同じ型の値を持ち、dataframeより単純ですが、より多くの数学的操作を提供し、オーバーヘッドを少なくします。
- matplotlib
matplotlibはデータ可視化・グラフ描画に利用するライブラリです。少ないコードで綺麗なグラフが描けます。
CORD Papersデータの取得
CORD papersのデータをkaggleから取得し、DataFrameに格納する。
366MBほどあるのでネットワーク環境にもよるが取得に時間かかるので、
httpsアクセスで直接取得しても良し。
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
df = pd.read_csv("https://datascience4beginners.blob.core.windows.net/cord/metadata.csv.zip",compression='zip')
# df = pd.read_csv("metadata.csv")
df.head()
データ変換
publish_timeをpandasのdatetime型に変換する。
df['publish_time'] = pd.to_datetime(df['publish_time'])
df['publish_time'].hist()
plt.show()
治療薬と診断リストのキーワード頻度集計
Abstractからどのような情報を簡単に抽出できるか見ていく。ひとつは、どのような治療戦略が存在し、それが時間とともにどのように進化してきたかを確認する。 まず、COVIDの治療に使用される可能性のある薬剤のリストと、診断のリストを手作業でまとめる。そして、それらを調べ、論文のabstractの中から対応する用語を検索する。
medications = [
'hydroxychloroquine', 'chloroquine', 'tocilizumab', 'remdesivir', 'azithromycin',
'lopinavir', 'ritonavir', 'dexamethasone', 'heparin', 'favipiravir', 'methylprednisolone']
diagnosis = [
'covid','sars','pneumonia','infection','diabetes','coronavirus','death'
]
for m in medications:
print(f" + Processing medication: {m}")
df[m] = df['abstract'].apply(lambda x: str(x).lower().count(' '+m))
for m in diagnosis:
print(f" + Processing diagnosis: {m}")
df[m] = df['abstract'].apply(lambda x: str(x).lower().count(' '+m))
dfm = df[medications]
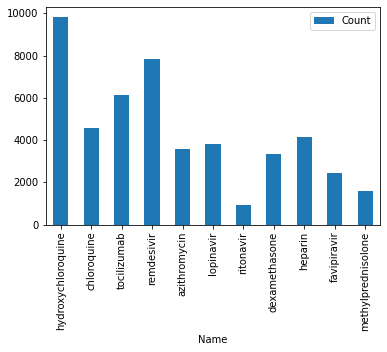
dfm = dfm.sum().reset_index().rename(columns={ 'index' : 'Name', 0 : 'Count'})
dfm.sort_values('Count',ascending=False)
- Processing medication: hydroxychloroquine
- Processing medication: chloroquine
- Processing medication: tocilizumab
- Processing medication: remdesivir
- Processing medication: azithromycin
- Processing medication: lopinavir
- Processing medication: ritonavir
- Processing medication: dexamethasone
- Processing medication: heparin
- Processing medication: favipiravir
- Processing medication: methylprednisolone
- Processing diagnosis: covid
- Processing diagnosis: sars
- Processing diagnosis: pneumonia
- Processing diagnosis: infection
- Processing diagnosis: diabetes
- Processing diagnosis: coronavirus
- Processing diagnosis: death
| Idx | Name | Count |
|---|---|---|
| 0 | hydroxychloroquine | 9806 |
| 3 | remdesivir | 7861 |
| 2 | tocilizumab | 6118 |
| 1 | chloroquine | 4578 |
| 8 | heparin | 4161 |
| 5 | lopinavir | 3811 |
| 4 | azithromycin | 3585 |
| 7 | dexamethasone | 3340 |
| 9 | favipiravir | 2439 |
| 10 | methylprednisolone | 1600 |
| 6 | ritonavir | 948 |
治療薬と診断リストのキーワード頻度集計結果をmatplotlibでグラフ表示
dfm.set_index('Name').plot(kind='bar')
plt.show()
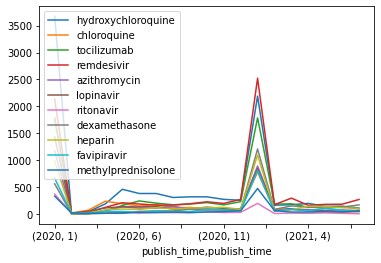
治療薬のトレンドを集計
dfm = df[['publish_time']+medications].set_index('publish_time')
dfm = dfm[(dfm.index>="2020-01-01") & (dfm.index<="2021-07-31")]
dfmt = dfm.groupby([dfm.index.year,dfm.index.month]).sum()
治療薬のトレンドを可視化
dfmt.plot()
plt.show()
2カ所で大きなスパイクが発生していることは興味深い。2020年1月と2021年1月である。これは、発表時期が明確に指定されていない論文があり、それぞれの年の1月と指定されていることが原因である。
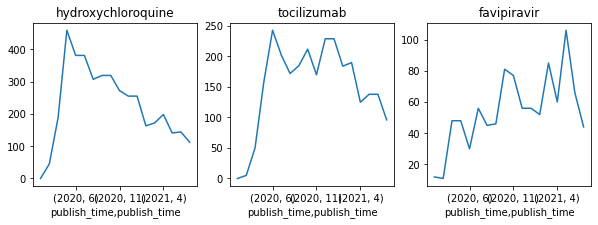
データをより理解するために、ほんの数種類の薬を可視化してみよう。また、よりきれいにプロットするために、1月のデータを「消去」して、中程度の値で埋めることにする。
meds = ['hydroxychloroquine','tocilizumab','favipiravir']
dfmt.loc[(2020,1)] = np.nan
dfmt.loc[(2021,1)] = np.nan
dfmt.fillna(method='pad',inplace=True)
fig, ax = plt.subplots(1,len(meds),figsize=(10,3))
for i,m in enumerate(meds):
dfmt[m].plot(ax=ax[i])
ax[i].set_title(m)
plt.show()
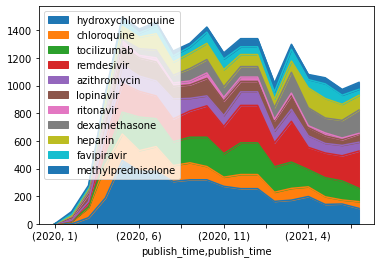
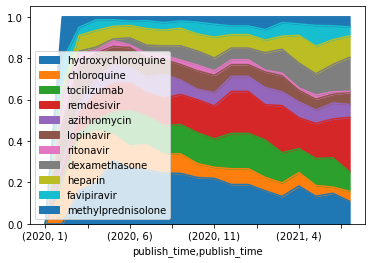
ヒドロキシクロロキンの人気は最初の数ヶ月は上昇し、その後低下し始め、一方ファビピラビルの言及数は安定した上昇を示している。相対的なトレンドを可視化するためには、積み重ねプロット(またはPandasの用語でエリアプロット)が有効である。
dfmt.plot.area()
plt.show()
dfmtp = dfmt.iloc[:,:].apply(lambda x: x/x.sum(), axis=1)
dfmtp.plot.area()
plt.show()
パーセンテージでやるともっとわかりやすい。
診断と薬の共起頻度マップ
最も興味深い関係の1つは、異なる診断が異なる薬でどのように扱われるかということだ。これを可視化するためには、共起頻度マップを計算する必要がある。これは、2つの用語が同じ論文で何回言及されているかを示すものである。 このようなマップは本質的に2次元行列であり、numpyの配列で表現するのが最適である。このマップを計算するには、すべての抄録を走査し、そこに現れるエンティティに印をつける。
m = np.zeros((len(medications),len(diagnosis)))
for a in df['abstract']:
x = str(a).lower()
for i,d in enumerate(diagnosis):
if ' '+d in x:
for j,me in enumerate(medications):
if ' '+me in x:
m[j,i] += 1
plt.imshow(m,interpolation='nearest',cmap='hot')
ax = plt.gca()
ax.set_yticks(range(len(medications)))
ax.set_yticklabels(medications)
ax.set_xticks(range(len(diagnosis)))
ax.set_xticklabels(diagnosis,rotation=90)
plt.show()
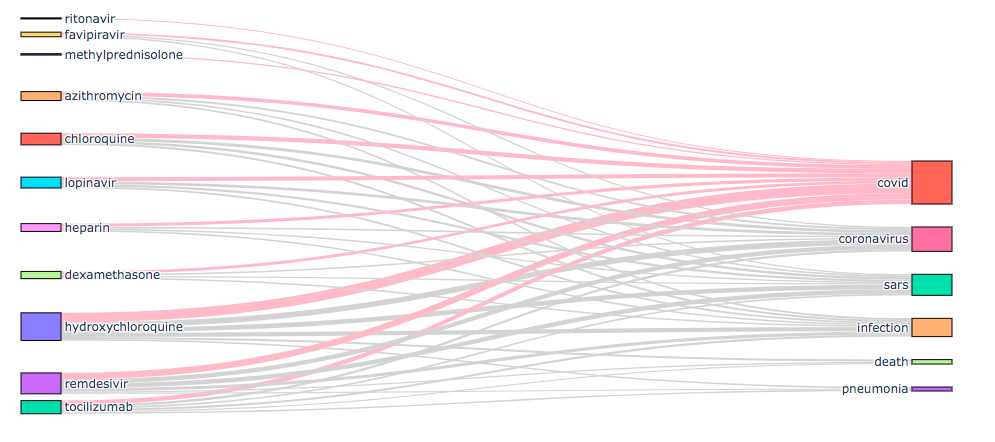
Plottyを使うとこの関係性をサンキーチャートという可視化技法で
さらにわかりやすく表示できる。
import plotly.graph_objects as go
def sankey(cat1, cat2, m, treshold=0, h1=[], h2=[]):
all_nodes = cat1 + cat2
source_indices = list(range(len(cat1)))
target_indices = list(range(len(cat1),len(cat1)+len(cat2)))
s, t, v, c = [], [], [], []
for i in range(len(cat1)):
for j in range(len(cat2)):
if m[i,j]>treshold:
s.append(i)
t.append(len(cat1)+j)
v.append(m[i,j])
c.append('pink' if i in h1 or j in h2 else 'lightgray')
fig = go.Figure(data=[go.Sankey(
# Define nodes
node = dict(
pad = 40,
thickness = 40,
line = dict(color = "black", width = 1.0),
label = all_nodes),
# Add links
link = dict(
source = s,
target = t,
value = v,
color = c
))])
fig.show()
sankey(medications,diagnosis,m,500,h2=[0])
簡単な頻度集計や共起頻度の集計だけでもいろんなことが見えてくる。
データサイエンスって面白い。
著者のTWITTERアカウント
ホームページ(pytorch/python/nlp)
参考文献