SPSS Modelerの平均値ノードをPythonのscipyとstatsmodelsで書き換えてみます。
以下の記事でModelerでの使い方は解説していますので、この内容をscipyとstatsmodelsで行ってみます。
SPSS Modeler ノードリファレンス 6-9 平均値 - Qiita
1m.2つのフィールドの平均を比較 Modeler
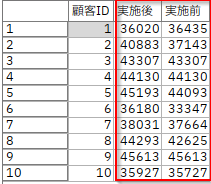
同じ顧客の施策前と施策後の平均を比較します。これは対応ある2標本のt検定になります。
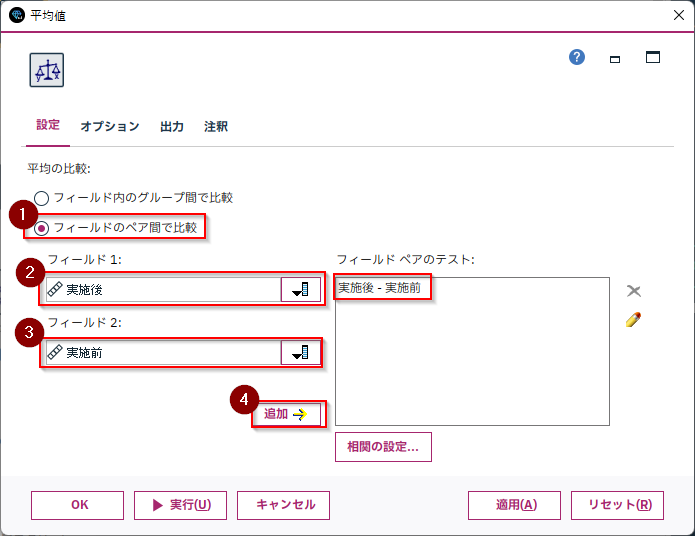
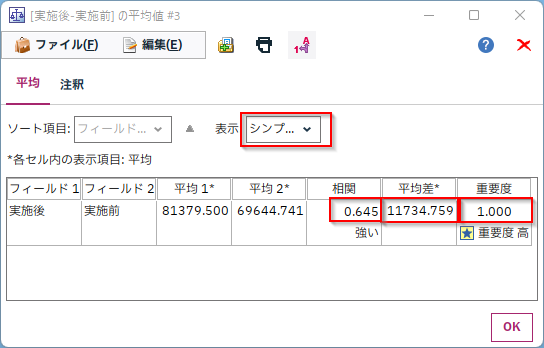
平均値ノードの「フィールドのペア間で比較」を選び「実施後」と「実施前」の列のペアを追加します。
「実施後」と「実施前」の相関係数と平均値差、そして「重要度」はt検定の「1-p値」をあらわしています。
デフォルトでは、相関係数もt検定も「1-p値」が0.95以上だと「強い」または「重要」と判定されます。つまり相関または平均値差が統計的に優位であるかどうかを示しています。これで施策の効果に優位な差があったのかを判断することができます。
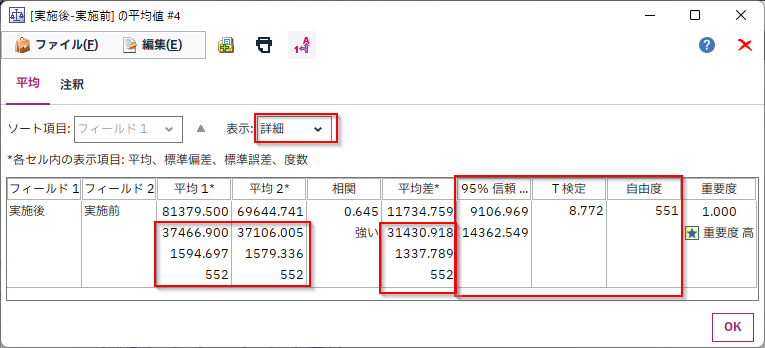
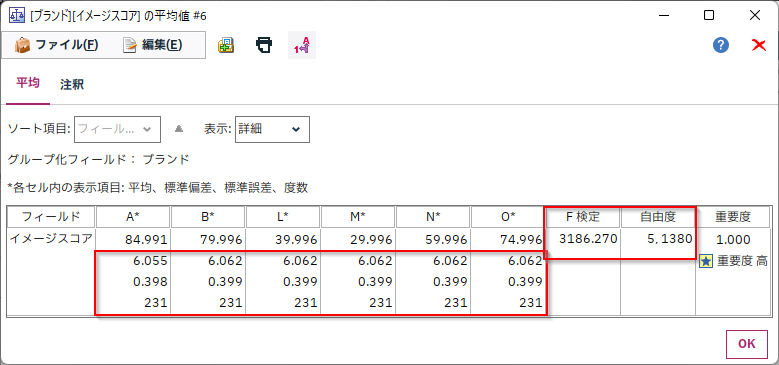
なお、表示を「詳細」にすると各列と平均値差の標準偏差、標準誤差、度数、また平均値差の95%信頼区間、T値、自由度が表示されます。
- 参考 24-5. 対応のある2標本t検定 | 統計学の時間 | 統計WEB
1p.2つのフィールドの平均を比較 scipy
まず列の基本指標を、pandasのメソッドなどで計算します。
mean:平均
std:標準偏差
sem:標準誤差
len:度数
columns=['実施後','実施前']
-----中略------
#列の基本指標
for i,c in enumerate(columns):
sttest['平均'+str(i+1)+'m']=df[c].mean()
if details==True:
sttest['平均'+str(i+1)+'s']=df[c].std()
sttest['平均'+str(i+1)+'se']=df[c].sem()
sttest['平均'+str(i+1)+'n']=len(df[c])
次に相関係数とp値をscipyのstats.pearsonrで計算します。
from scipy import stats
r,p=stats.pearsonr(df[columns[0]],df[columns[1]])
sttest['相関係数']=r
sttest['相関係数検定1-p']=1-p
次に2列の平均値差を絶対値absで求めます。
m = abs(df[columns[0]].mean()-df[columns[1]].mean())
s = (df[columns[0]]-df[columns[1]]).std()
そして、stats.ttest_relをつかって、対応ある2標本のt検定を行います。
stats.t.intervalをつかって95%信頼区間を求めます。
t,p=stats.ttest_rel(df[columns[0]],df[columns[1]])
m = abs(df[columns[0]].mean()-df[columns[1]].mean())
s = (df[columns[0]]-df[columns[1]]).std()
se = m/t
n = len(df[columns[0]])
d_f = len(df[columns[0]])-1
ci = stats.t.interval( alpha=0.95, loc=m, scale=se, df=d_f )
Modelerの「シンプル」表示と同等の項目だと以下の結果が得られます。

Modelerの「詳細」表示と同じ項目だと以下の結果が得られます。
pandasでは複数項目を1セルに入れられないので、横に長くなってしまっています。

- 参考
- Python で無相関検定をしてみる - GIS奮闘記
- 【統計】Pythonでt検定をやってみる | naoの学習&学習
- Pythonでt検定 2クラスの試験成績の比較 - Qiita
2m.複数カテゴリの平均を比較 Modeler
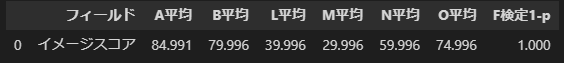
今度は6ブランドのイメージスコアにブランド毎の平均に差があるかを検定します。3つ以上のカテゴリ比較が可能になるように、一元配置分散分析で検定しています。
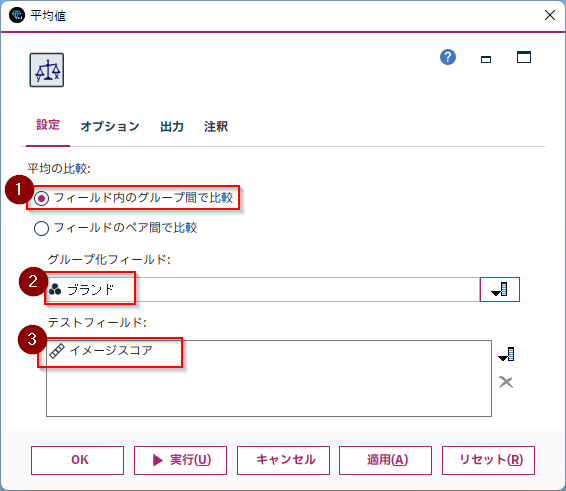
「フィールド内のグループ間で比較」を選び「グループ化フィールド」に比較したいカテゴリ列を指定します。この例では「ブランド」です。
「テストフィールド」に比較したい値を設定します。この例では「イメージスコア」です。
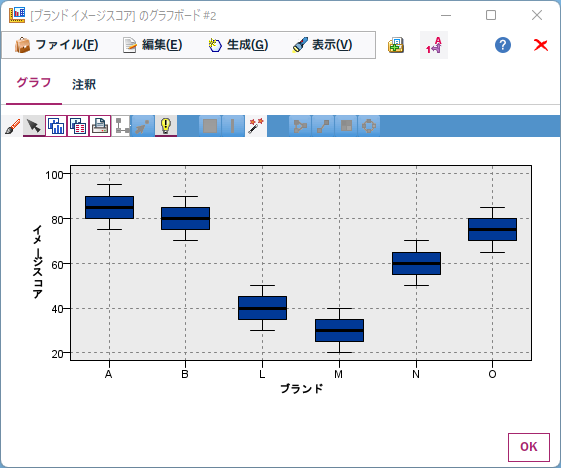
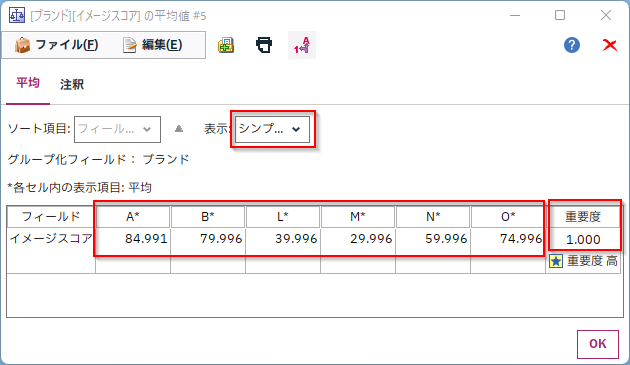
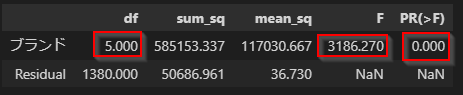
各ブランド列内にあったA,B,L,M,N,Oというブランド毎に平均が計算され、この平均の差が統計的に優位かどうかを検定しています。「重要度」はF検定の「1-p値」をあらわしています。
デフォルトでは、F検定も「1-p値」が0.95以上だと「重要」と判定されます。つまり平均値差が統計的に優位であるかどうかを示しています。これでブランド毎のイメージスコアに優位な差があったのかを判断することができます。
表示を「詳細」にすると各列の標準偏差、標準誤差、度数、またF値、自由度が表示されます。
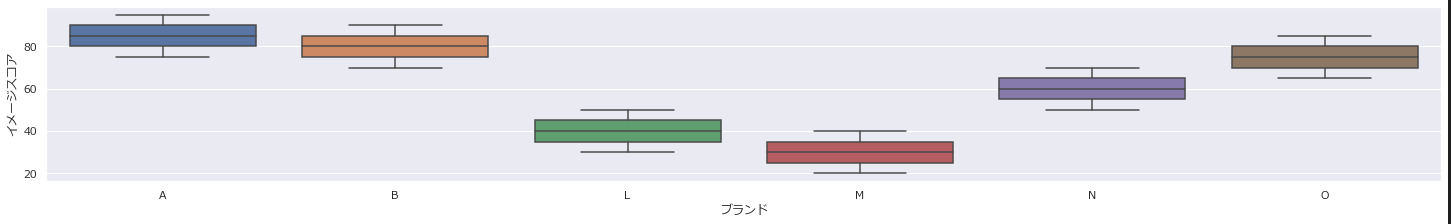
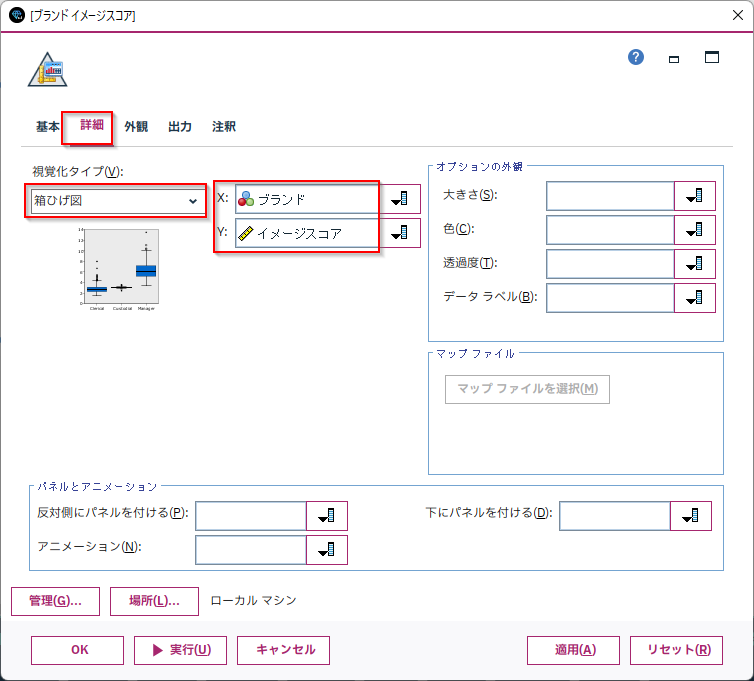
「グラフボード」ノードで箱ひげ図をつくると視覚的にも確認ができます。
2p.複数カテゴリの平均を比較 scipyとstatsmodels
最初に「グループ化フィールド」に比較したいカテゴリ列を指定します。この例では「ブランド」です。
「テストフィールド」に比較したい値を設定します。この例では「イメージスコア」です。
「グループ化フィールド」になる列のデータ型はastype("category")でカテゴリ型に変更しておきます。
grpcolumn='ブランド'
testcolumns=['イメージスコア']
df1[grpcolumn]=df1[grpcolumn].astype("category")
各カテゴリ(cat)の集計対象フィールド(col)の平均値meanなどを集計します。
query(grpcolumn+'==@cat')で、各カテゴリごとに集計しています。
具体的には
df1.query('ブランド=="A"')['イメージスコア'].mean()
のような計算が行われます。
for cat in df1[grpcolumn].cat.categories:
sanova[cat+'平均']=df1.query(grpcolumn+'==@cat')[col].mean()
if details==True:
sanova[cat+'s']=df1.query(grpcolumn+'==@cat')[col].std()
sanova[cat+'se']=df1.query(grpcolumn+'==@cat')[col].sem()
sanova[cat+'n']=len(df1.query(grpcolumn+'==@cat')[col])
次にstatsmodelsで一元配置分散分析を行います。
最小二乗法で回帰モデルをつくって、そこから分散分析結果を取り出す手順になります。
回帰モデルは以下で作ります。
ols(col+' ~ ' + grpcolumn, data=df1).fit()
具体的には
ols('イメージスコア ~ ブランド', data=df1).fit()
のような計算が行われます。
回帰モデルから分散分析の結果を抽出します。
anov = sm.stats.anova_lm(model)
分散分析の結果は以下のようなpandasのDataFrameで取得できますのでここからloc[grpcolumn,'F']などで必要な指標を取り出します。
from statsmodels.formula.api import ols
model = ols(col+' ~ ' + grpcolumn, data=df1).fit()
import statsmodels.api as sm
anov = sm.stats.anova_lm(model)
if details==True:
sanova['F検定']=anov.loc[grpcolumn,'F']
sanova['自由度']=anov.loc[grpcolumn,'df']
sanova['F検定1-p']=1-anov.loc[grpcolumn,'PR(>F)']
Modelerの「シンプル」表示と同等の項目だと以下の結果が得られます。
Modelerの「詳細」表示と同じ項目だと以下の結果が得られます。
pandasでは複数項目を1セルに入れられないので、横に長くなってしまっています。
箱ひげ図はseabornで書きました。
具体的には
sns.boxplot(x='ブランド', y='イメージスコア',
data=df, showfliers=False, ax=ax)
のように書いています。
sns.set(font='Meiryo')
for col in testcolumns:
fig = plt.figure(figsize=(20, 3), constrained_layout=True)
ax = fig.add_subplot(1, 1, 1)
sns.boxplot(x=grpcolumn, y=col,
data=df, showfliers=False, ax=ax)
plt.show()
-
参考
-
pythonで統計学基礎:03 検定・分散分析 | 化学の新しいカタチ
- Pythonを用いた分散分析(ANOVA)あれこれ(1) - 工場裏のアーカイブス
- statsmodel
3m.複数カテゴリの複数フィールドの平均を比較 Modeler
ノードリファレンスにはなかったのですが、Modelerの「平均値」ノードを使うと複数カテゴリで複数フィールドの平均値を比較して、特徴量を重要度順に並べ替えることができます。これはとても強力なので例をご紹介します。
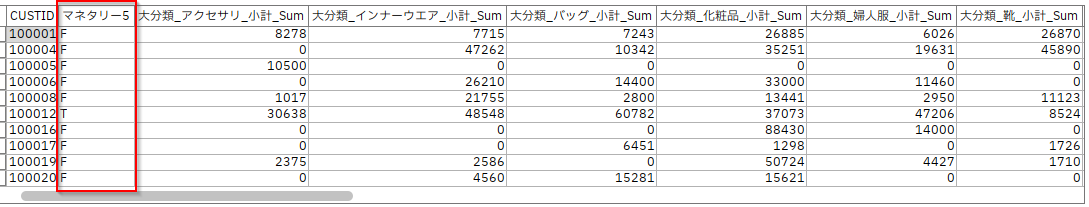
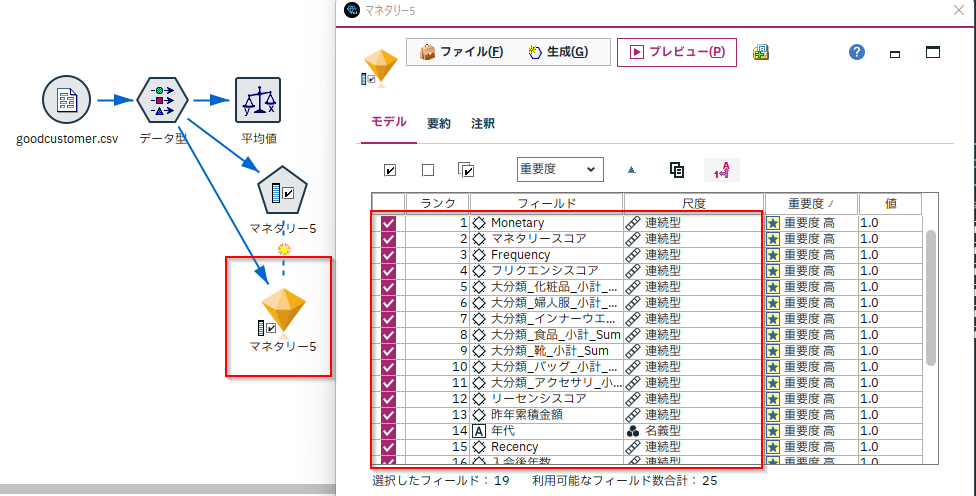
以下のようなデータを用意しました。
各顧客に対して、アクセサリの購入金額合計などのさまざまな集計がなされています。
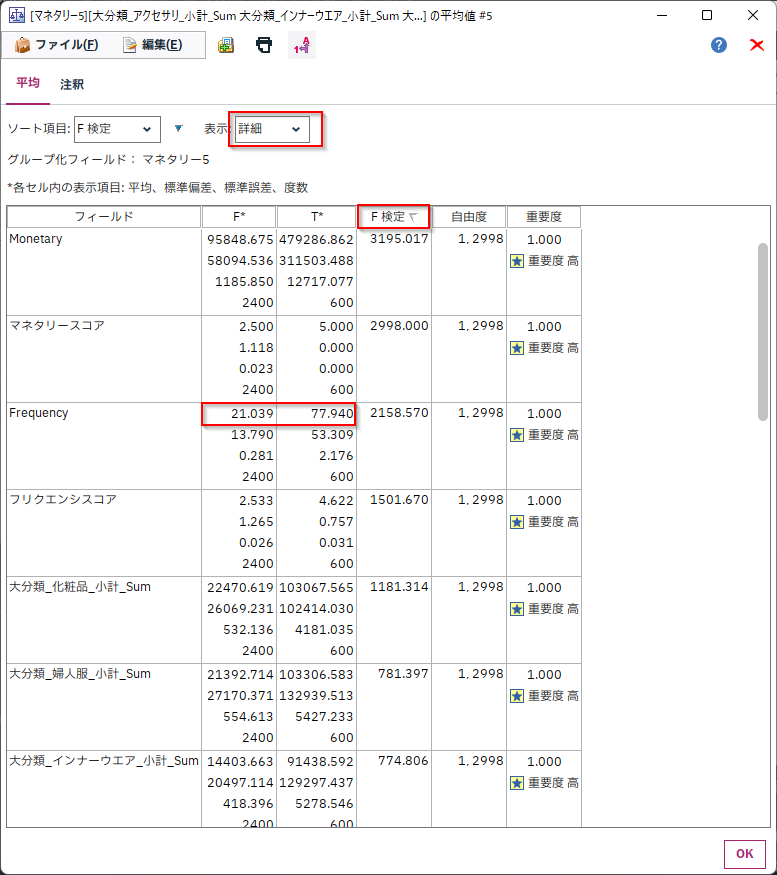
「マネタリー5」というフラグ列があり、これは購入金額が全体の上位20%の優良顧客であるということを示しています。
この優良顧客とそうでない顧客でどんな差があるかどうかを「平均値」ノードを使って調べてみます。
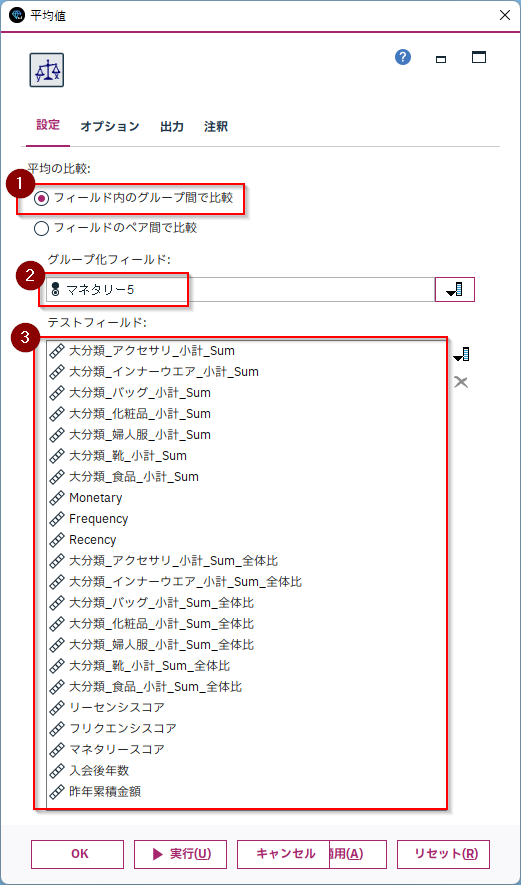
「フィールド内のグループ間で比較」を選び「グループ化フィールド」に比較したいカテゴリ列を指定します。この例では「マネタリー5」です。
「テストフィールド」に比較したい値を設定します。ここではすべての数値列を選びました。この中から特に平均値の差を注目すべき特徴量を探します。
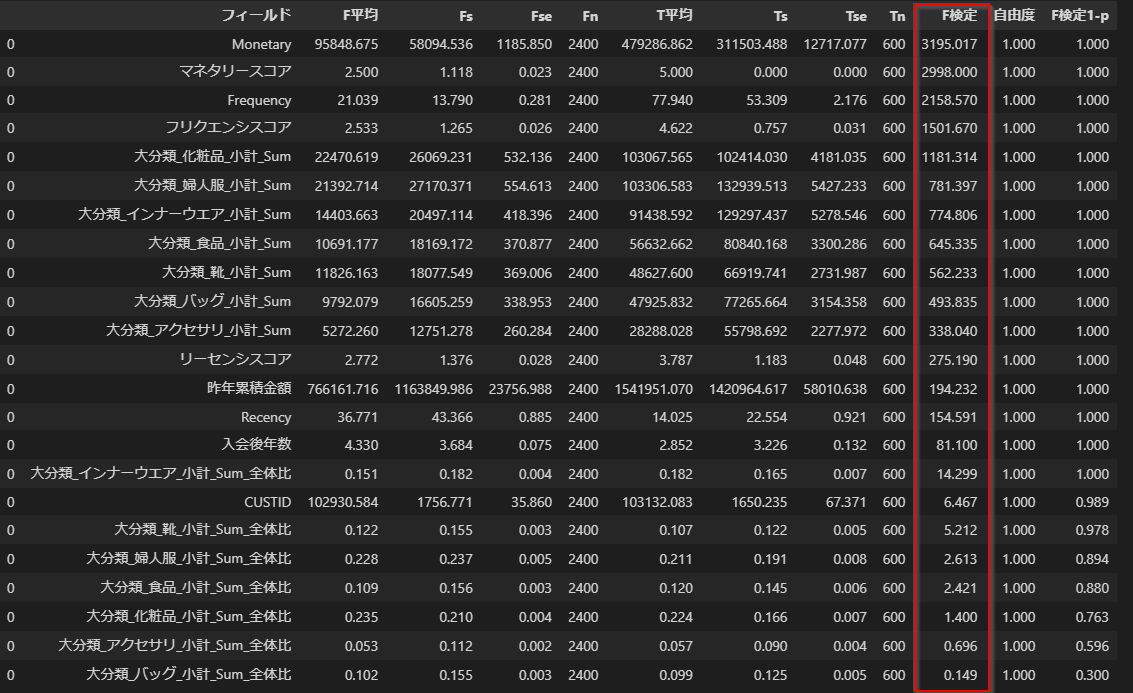
表示を「詳細」にして、「F検定」の降順で並べ替えます。
そうするとF値が大きい順に並びますので、優良顧客とそれ以外で平均値の違いが顕著な順に特徴量が並べ替えられます。(「重要度」で並べ替えても本来は同じ順序になるべきなのですが、p値がとても小さいと丸められてしまって順位がつかなくなるのでF値で並べ替えるのがコツです)
中身をみてみると、「Monetary」や「マネタリースコア」が最上位に来ています。これは「購入金額が全体の上位20%の優良顧客」が「マネタリー5」=Trueなのでチートになり、当たり前の結果です。
次点の「Frequency」は優良顧客は77.9回、そうでない顧客は21回なので大きな差があることがわかります。
このようにチートも含まれますが、たくさんの特徴量候補があるときに、とりあえず、すべての特徴量から重要そうな特徴量を知りたいときにとても便利です。
ちなみに「特徴量選択」ノードでも、目的変数がカテゴリ型で説明変数が数値型の場合は、同様のロジックを使っています。ただ「平均値」ノードの場合は各カテゴリの平均値も表示されるので、具体的な値で各カテゴリでどちらがどれだけ大きいのか小さいのかも比べることができるのがメリットです。
3p.複数カテゴリの複数フィールドの平均を比較 scipyとstatsmodels
Pythonの場合は各列の中から
re.sub(r'[0-9]+', '', df2[col].dtype.name) in ['int', 'float']:
で数値列を探して分散分析を行います。
そして、sort_values('F検定',ascending=False)でF値順に並べ替えています。
import re
grpcolumn='マネタリー5'
testcolumns=[]
for col in df2:
if re.sub(r'[0-9]+', '', df2[col].dtype.name) in ['int', 'float']:
testcolumns.append(col)
df2[grpcolumn]=df2[grpcolumn].astype("category")
df_anova=anova(df2, testcolumns,grpcolumn,details=True).sort_values('F検定',ascending=False)
以下のようにF値の大きな順で特徴量を並べ替えることができました。
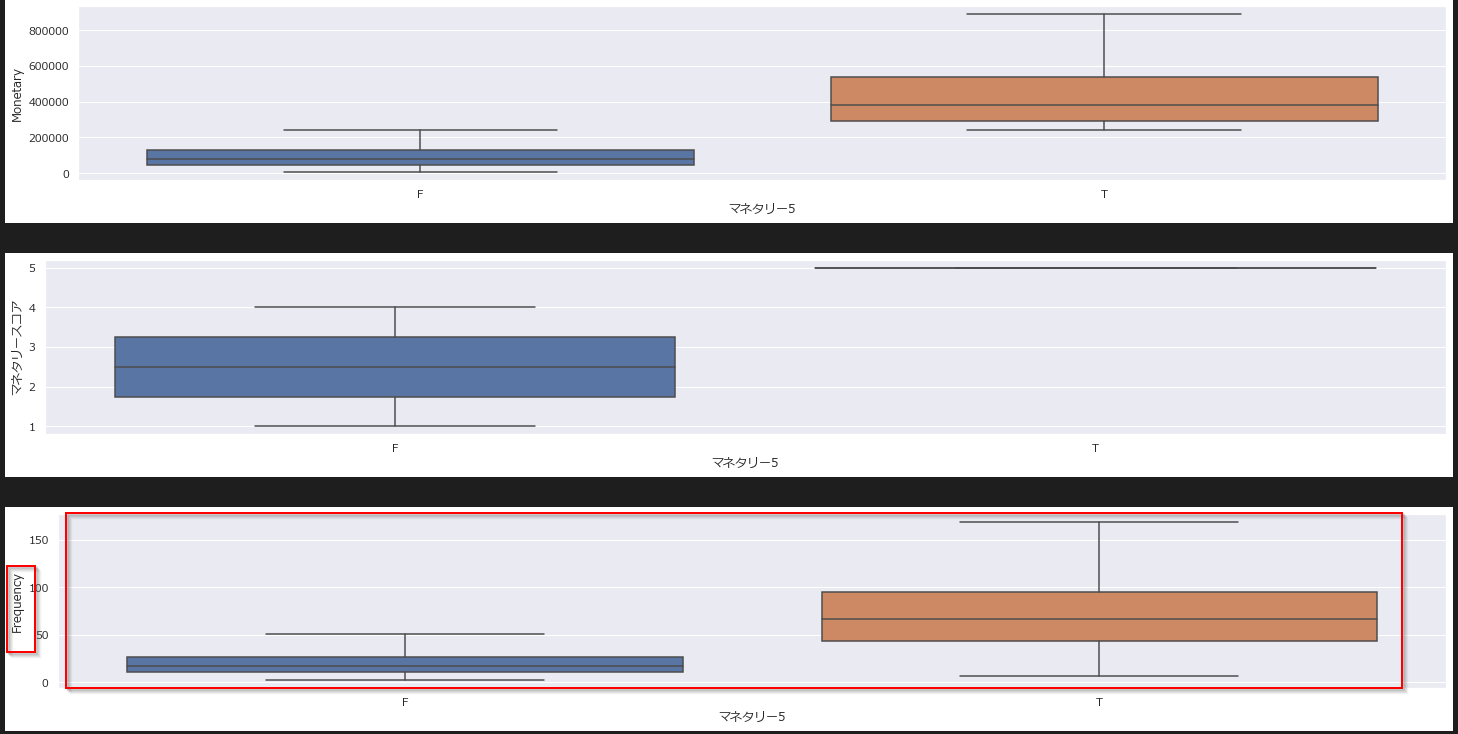
さらにこの並べ替えた特徴量順に箱ひげ図を表示します。
boxplot(df2,df_anova['フィールド'],grpcolumn)
確かにFrequencyで優良顧客かどうかで大きな差があることがよくわかります。
サンプル
サンプルは以下に置きました。
サンプルストリーム
notebook
利用データ
■テスト環境
Modeler 18.4
Windows 10 64bit
Python 3.8.10
pandas 1.4.1
scipy 1.8.0
statsmodels 0.13.2